杭州西溪湿地沉积物中不同培养基分离的细菌种群多样性
2016-12-16褚文珂谢蔚鹏
黄 媛,褚文珂,谢蔚鹏,陈 敏
(杭州师范大学生命与环境科学学院,浙江 杭州310036)
杭州西溪湿地沉积物中不同培养基分离的细菌种群多样性
黄 媛,褚文珂,谢蔚鹏,陈 敏
(杭州师范大学生命与环境科学学院,浙江 杭州310036)
以西溪湿地沉积物的细菌群落为对象,对4种不同培养基(M1、R2A、NB和DNB)分离细菌的能力及分离物的种群多样性进行了比较研究.共分离得到191株菌株,经16S rRNA基因测序和系统发育分析,可归于13个不同的属,其中R2A培养基分离的细菌种类最多,共有11个不同的属,其次是NB培养基,分离得到8个属,M1和DNB培养基均分离得到7个属.优势种属为根瘤菌属(Rhizobium),占总数的39.27%.研究结果表明,R2A培养基分离的培养物种群多样性更为丰富.
西溪湿地;沉积物;培养基;细菌多样性
湿地生态系统中,沉积物是一个由各种微生物参与的、物质发生频繁交换的、具有高度生物活性的特殊生境,其中微生物的多样性对整个水体系统有重要影响.与水体中的悬浮微生物比较,沉积物微生物往往在单位数量和功能多样性上更胜一筹,因此,对水体生态系统平衡的重要性也更大.目前,沉积物微生物正受到越来越多研究者的关注.
杭州西溪国家湿地公园坐落于杭州市区的西部,是在自然湿地基础上经一千多年农渔耕作用形成的罕见的城中次生湿地,其中河港、池塘、沼泽等水域约占湿地面积的70%.近20多年来,随着经济和社会的发展,西溪湿地的面积由60 km2萎缩至不足12 km2,并且人类活动的干扰日趋频繁,对湿地的生态系统造成了影响[1-2].本研究以西溪湿地不同生境下的表层沉积物为研究对象,通过设计不同的培养基,用纯培养的方法研究沉积物中细菌的群落特征和多样性,以期为丰富西溪湿地生态系统内涵,开发西溪湿地的微生物资源,进而为西溪湿地的保护和修复提供理论和实践依据.
1 材料与方法
1.1 采 样
选择了湿地内6种典型的水域生境:1)挺水植物区(蒲苇,白茅等);2)浮水植物区(睡莲,凤眼莲等);3)沉水植物区(金鱼藻,狐尾藻等);4)湿生植物区(美人蕉,黄菖蒲等);5)开阔的河道(岸边有杂草树木);6)野生养殖塘(水草、杂草丛生).采集上述6类生境的表层(0~10 cm)沉积物,每个样品由采样点中心及周围共5个点的土样混合而成.样品编为1~6号,采集完成后迅速放入无菌保鲜袋,4 ℃保存,带回实验室立即用于菌株分离.
1.2 培养基
M1培养基:蛋白胨0.1 g,可溶性淀粉0.25 g,酵母浸粉0.05 g,甘油磷酸钠0.01 g,琼脂15 g,蒸馏水1 L.
R2A培养基:胰蛋白胨0.25 g,酸水解酪蛋白0.5 g,酵母浸粉0.5 g,可溶性淀粉0.5 g,K2HPO30.3 g,MgSO40.1 g,葡萄糖0.5 g,丙酮酸钠0.3 g,蛋白胨0.25 g,琼脂15 g,蒸馏水1 L.
牛肉膏蛋白胨培养基(以下简称NB):牛肉膏3 g,蛋白胨10 g,NaCl 5 g,琼脂15 g,蒸馏水1 L.
稀释100倍牛肉膏蛋白胨培养基(以下简称DNB):牛肉膏0.03 g,蛋白胨0.1 g,NaCl 0.05 g,琼脂15 g,蒸馏水1 L.
LB培养基:胰蛋白胨10 g,酵母提取物5 g,NaCl 10 g,琼脂15 g,蒸馏水1 L.
1.3 菌株的分离纯化
称取10 g沉积物样品放入盛有90 mL灭菌生理盐水的锥形瓶(含玻璃珠)中振荡20 min,静置后取液体进行梯度稀释,取10-4、10-5、10-6稀释液0.1 mL,分别涂布于M1、R2A、NB及DNB培养基平板上,用保鲜膜包好倒置于28 ℃培养箱,培养5~7 d,待平板上长出单菌落后,挑取单菌落反复划线分离至获得纯化菌株,试管斜面4 ℃保藏.
1.4 细菌DNA的提取
采用上海生工生物工程公司生产的细菌基因组DNA快速抽提试剂盒进行菌体DNA的抽提,步骤如下:1.5 mL离心管中加入1 mL过夜培养的菌液,室温8 000 r/min离心1 min,移去上清,收集菌体.取400 μL裂解液加入菌体沉淀中,摇匀后置于泡沫板上在65 ℃水浴锅中水浴1 h,水浴过程每隔10 min观察一次直至裂解完全.裂解完成后,加入PB溶液200 μL,将离心管反复颠倒使液体混匀,离心管置于-20 ℃冰箱.5 min后取出离心管,室温10 000 r/min离心5 min,将尽可能多的上清液取出置于干净离心管.加一倍体积的异丙醇后上下颠倒离心管10次,混匀后室温放置3 min,室温10 000 r/min离心5 min,弃上清.加入75%乙醇1 mL,漂洗3 min,漂洗过程中要让沉淀漂浮起来,10 000 r/min离心2 min,弃上清.重复漂洗2次,室温晾干至乙醇完全挥发.TE Buffer溶解所得DNA,低温保存.
1.5 16S rRNA基因扩增
采用细菌通用引物进行PCR扩增,引物序列如下:27F,5′-AGAGTTTGATCCTGGCTCAG-3′;1492R,5′-TACGGTTACCTTGTTACGACTT-3′.PCR扩增体系为25 μL:ddH2O 15 μL;10×buffer 2.5 μL;dNTP(25 mmol/L)4.0 μL;27F(25 pmol/μL)0.5 μL;1492R(25 pmol/μL) 0.5 μL;Taq DNA polymerase 0.5 μL;DNA template 2.0 μL.PCR扩增条件为:95 ℃ 90 s;95 ℃ 30 s, 60 ℃ 30 s, 72 ℃ 90 s, 5个循环;95 ℃ 30 s, 55 ℃ 30 s, 72 ℃ 90 s, 5个循环;95 ℃ 30 s, 50 ℃ 30 s, 72 ℃ 90 s, 25个循环;72 ℃ 10 min, 60 ℃ 10 min.扩增产物经1%琼脂糖凝胶电泳检测,稳定电压130 V,20 min后根据电泳条带结果,将合适条带的扩增产物送往上海英俊生物公司测序.
1.6 系统发育及多样性分析
通过PCR扩增及测序后,将得到的基因序列提交到RDP(http://rdp.cme.msu.edu/),利用Classifier选项进行序列比对,得到各个菌株的分类学地位.利用Sequence 5.4.1软件,对获得的16S rRNA基因序列进行分类(序列完全相同的暂视为同一株),取代表菌株在GenBank中进行Blastn比对,查找相似度较高的模式菌株.
2 结果与分析
2.1 细菌的分离和纯化
实验共设计了4种不同类型培养基对6个沉积物样品进行平板分离培养,根据菌落的颜色、大小、形态等特征,从各个平板上挑取不同的单菌落,共得到191株细菌菌株(表1).从4种不同培养基的分离效果来看,NB培养基分离得到的菌株数量最多,共61株,其次是R2A培养基53株,M1培养基分离得到的细菌数量最少,为31株.从6个不同样品来看,1号样品分离得到细菌数量最多,为41株,其次是2号样品33株,5号样品分离得到的细菌数量最少,只有26株.
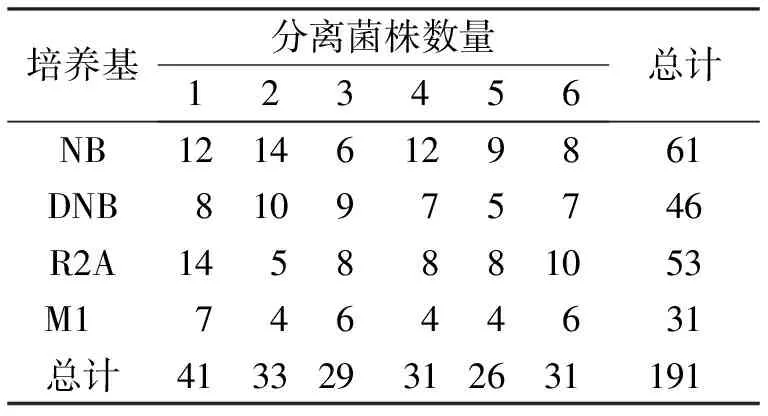
表1 细菌分离结果
2.2 分离物的多样性分析
本次分离得到的191株细菌分属于13个属(表2),根据每个种属所含有的细菌数量来看,优势菌群为根瘤菌属(Rhizobium),占总菌数的39.27%;其次是假单胞杆菌属(Pseudomonas),占总菌数26.70%;微球菌属(Micrococcus)、不动杆菌属(Acinetobacter)、贪嗜菌属(Variovorax)、罗氏菌属(Rothia)及赖氨酸芽孢杆菌属(Lysinibacillus)均只分离得到1株细菌.此外,4种不同培养基分离得到的细菌种类以R2A培养基最多,有11个不同的属,其次是NB培养基8个属,M1培养基和DNB培养基都是7个属.
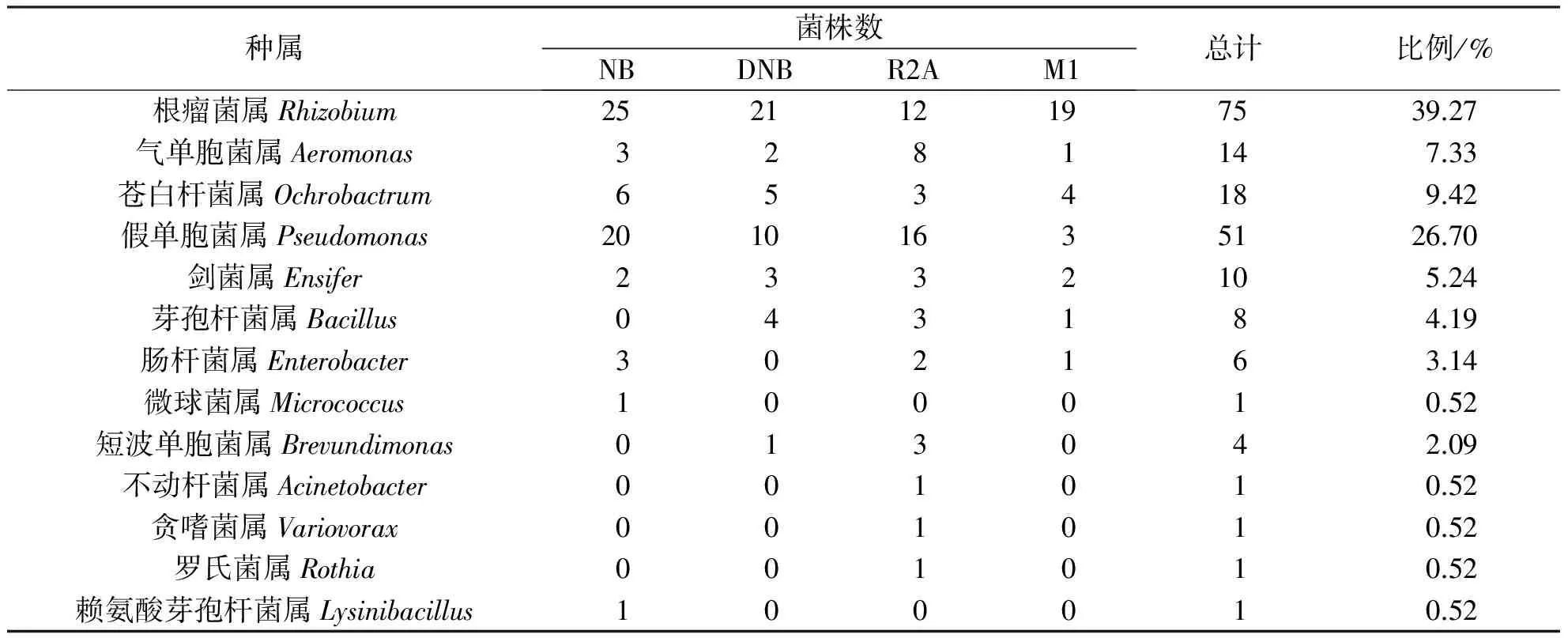
表2 细菌的种属分布
2.3 分离物的系统发育分析
分离物的16S rRNA基因序列相似性及系统发育分析结果如表3所示.结果显示,根瘤菌属(Rhizobium)共有Rhizobiumpusense和Rhizobiumrosettiformans2个不同的种.其中与R.pusense相似的菌株共71株,同源性为99.4%~99.8%;与R.rosettiformans相似的菌株较少,仅4株,同源性为100%.
假单胞菌属(Pseudomonas)的细菌种类最丰富,分别属于Pseudomonasstutzeri、Pseudomonasplecoglossicida、Pseudomonashunanensis、Pseudomonastoyotomiensis、Pseudomonaschengduensis和Pseudomonasalcaligenes6个不同的种.其中与P.stutzeri相似的有20株,同源性为99.8%~100%;与P.plecoglossicida相似的有19株,同源性为99.7%~99.8%;与P.hunanensis相似的有9株,同源性为99.2%~99.9%.与P.toyotomiensis、P.chengduensis及P.alcaligenes相似的各有一株,同源性分别为99.4%、99.5%及99.8%.
芽孢杆菌属(Bacillus)的细菌属于4个不同的种,分别是Bacillussiamensis4株,同源性99.8%~99.9%;Bacillussubtilis和Bacillusaryabhattai各1株,同源性分别为99.9%和100%;Bacillusmethylotrophicus有2株,同源性99.8%~100%.
短波单胞菌属(Brevundimonas)的细菌分属3个不同的种:Brevundimonasdiminuta2株,同源性99.4%~99.7%;Brevundimonasintermedia和Brevundimonasvesicularis各1株,同源性分别为99.8%及99.3%.
苍白杆菌属(Ochrobactrum)的细菌共有18株,均属于Ochrobactrumanthropi,同源性为99.9%~100%.气单胞菌属(Aeromonas)的细菌有14株,均属于Aeromonashydrophila,同源性为99.9%~100%.剑菌属(Ensifer)的细菌有10株,均属于Ensiferadhaerens,同源性为99.5%~99.8%.肠杆菌属(Enterobacter)的细菌有6株,均属于Enterobacteraerogenes,同源性均为99.0%.
不动杆菌属(Acinetobacter)、贪嗜菌属(Variovorax)、罗氏菌属(Rothia)、赖氨酸芽孢杆菌属(Lysinibacillus)及微球菌属(Micrococcus)的细菌各有1株,分别属于Acinetobacterjohnsonii、Variovoraxginsengisoli、Rothiaterrae、Lysinibacillusfusiformis及Micrococcusendophyticus,同源性分别为99.1%、98.6%、97.6%、99.7%及99.8%.
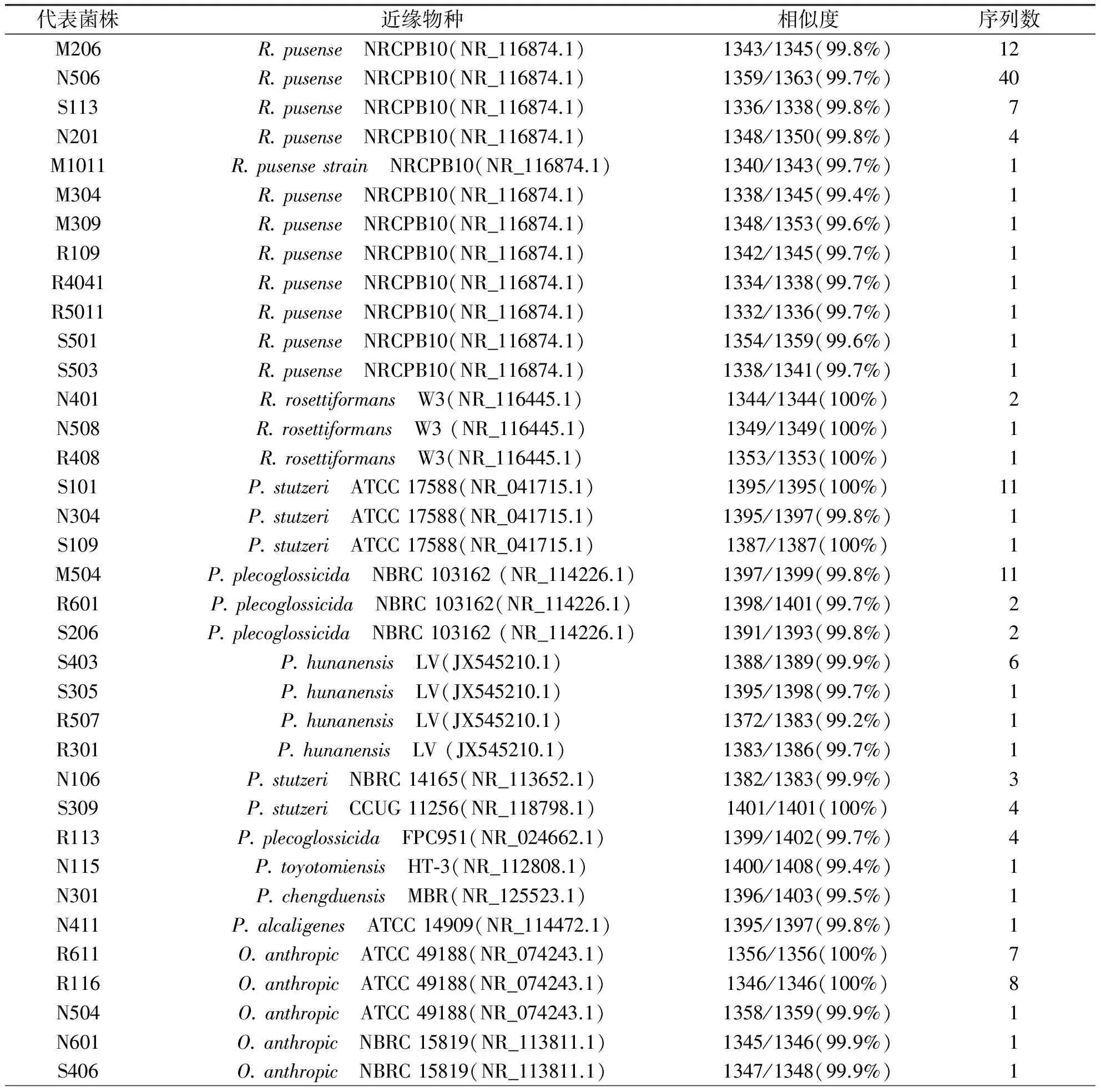
表3 分离物的测序比对结果
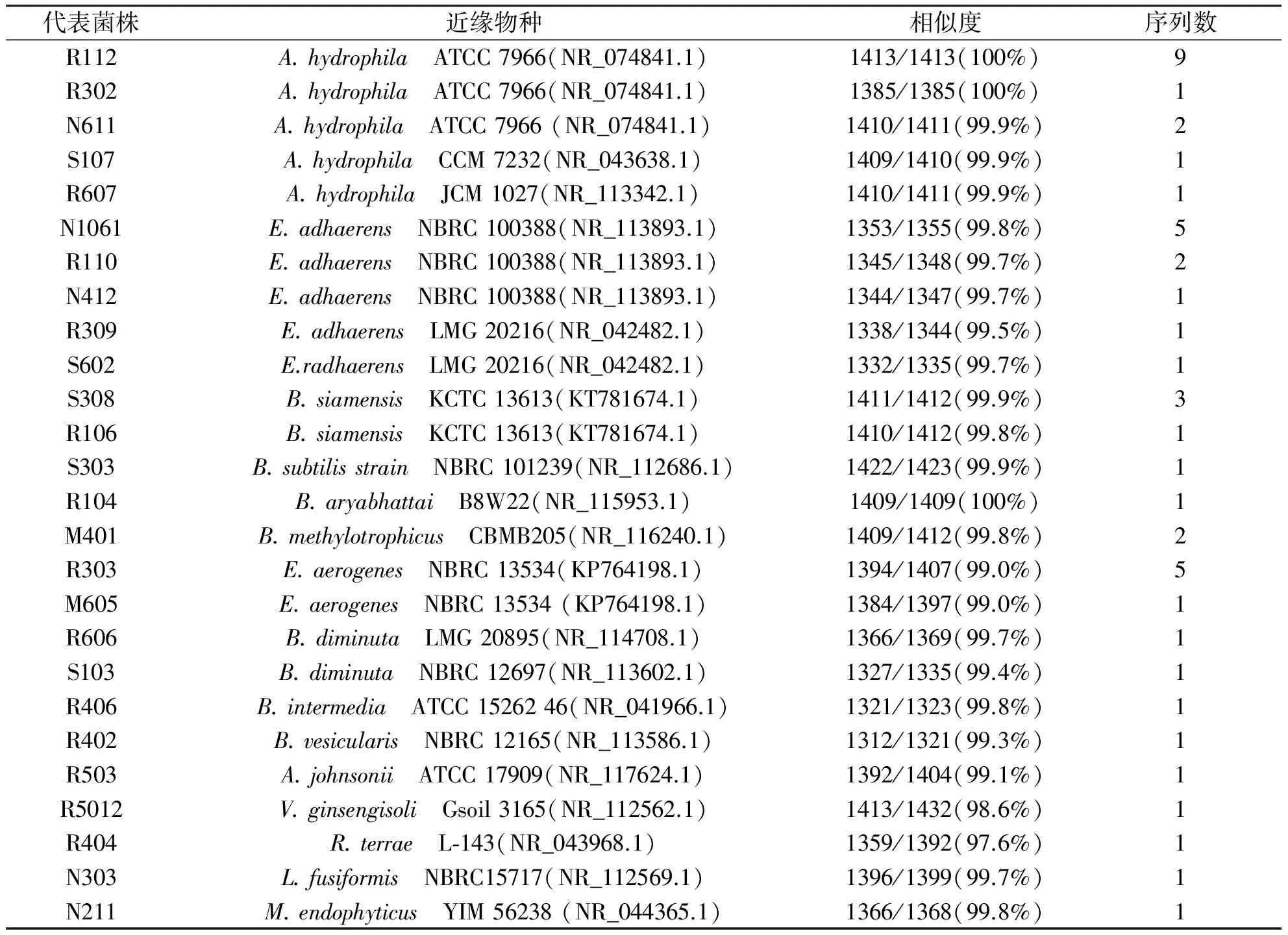
续表3
3 讨 论
微生物纯培养技术通过采用成分各异的培养基可以对不同的微生物进行分离培养[3-4].这种方法最大的优势在于可以获得微生物的纯培养物,是研究、开发和利用微生物资源的前提,也不失为研究微生物种群多样性的一类方法[5].刘玉娟等[6]通过纯培养对中国南海海域20个沉积物样品进行了细菌多样性分析,获得了200株细菌,分属于47个属,99个种;戴欣等[7]通过改良基础培养基,对太湖沉积物可培养细菌多样性进行了研究,发现稀释培养基上细菌的生长数量比普通培养基上高3~5倍.田晓燕等[8]通过纯培养方法从人工湿地中得到9种菌株,分别为严格好氧菌和兼性厌氧菌,都具有很强的有机物降解能力.
由于微生物生存环境复杂,人工培养环境与原生境的偏差,环境样品中可培养微生物的数量仍然非常有限[9].近年来,在改进和开发新型培养技术等方面,国内外学者展开了较广泛的研究工作.据报道,提高微生物的可培养性可以从改变培养基成分和培养条件[10-12]、添加相关的信号物质[13]、降低培养基营养成分浓度[14-15]、延长培养时间以及采用多种培养基组合[16]等方面进行改进.本研究采用4种不同培养基分离湿地沉积物样品中的细菌,结果表明,NB培养基分离得到的细菌数量最高,且细菌种类也较多,仍是分离培养细菌的最常规培养基之一.R2A培养基上分离的细菌种类是最多的,甚至分离到了NB培养基上不能生长的3个属:不动杆菌属、贪嗜菌属和罗氏菌属,可作为沉积物样品细菌分离的较适用的一种培养基.而DNB及M1培养基无论是分离的细菌数量还是种类均较低.这些差异的出现主要与培养基中所含不同的营养物质有关,例如,与经典的NB培养基相比较,R2A培养基中添加了酸水解酪蛋白及丙酮酸钠.据报道,丙酮酸钠经三羧酸循环后产生的NADH和H+可与超氧化物结合,从而减少超氧化物对细胞的损害[17];酸水解酪蛋白是天然牛奶蛋白经酸水解等过程处理后的产物,含有18种游离氨基酸,可被微生物直接利用[18].此外,R2A是一类低营养浓度的培养基,有利于把那些在菌群之间处于劣势的微生物以及只能形成微小菌落或生长缓慢的类群分离出来[19].总之,应根据研究目的和样品的不同来源等选择合适的培养基,并采取多种培养基结合使用的方法来提高分离物的多样性,获得较全面的数据.
[1] 金云峰,杨玉鹏,蒋祎.国外湿地公园保护与管理研究综述[J].中国城市林业,2015,13(6):1-5.
[2] 陈久和.城市边缘湿地生态环境脆弱性研究:以杭州西溪湿地为例[J].科技通报,2003,19(5):395-398.
[3] 孙风芹,汪保江,李光玉,等.南海南沙海域沉积物中可培养微生物及其多样性分析[J].微生物学报,2008,48(12):1578-1587.
[4] 王宪斌.若尔盖高原湿地沼泽土嗜冷菌多样性及系统发育研究[D].雅安:四川农业大学,2012.
[5] 王永霞.云南高原程海湖沉积物中的细菌多样性研究[D].昆明:云南大学,2014.
[6] 刘玉娟,田新朋,黄小芳,等.中国南海沉积环境可培养细菌多样性研究[J].微生物学通报,2014,41(4):661-673.
[7] 戴欣,王保军,黄燕,等.普通和稀释培养基研究太湖沉积物可培养细菌的多样性[J].微生物学报,2005,45(2):161-165.
[8] 田晓燕,李丽萍.人工湿地污泥处理系统微生物组成的分离与鉴定[J].吉林建筑工程学院学报,2008,25(4):14-17.
[9] AMANN R I, LUDWIG W, SCHLEIFER K H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J]. Microbiol Rev,1995,59(1):143-169.
[10] CHIN K J, HAHN D, HENGSTMANN U, et al. Characterization and identification of numerically abundant culturable bacteria from the anoxic bulk soil of rice paddy microcosms[J]. Appl Environ Microbiol,1999,65(11):5042-5049.
[11] JANSSEN P H, YATES P S, GRINTON B E, et al. Improved culturability of soil bacteria and isolation in pure culture of novel members of the divisions Acidobacteria, Actinobacteria, Proteobacteria, and Verrucomicrobia[J]. Appl Environ Microbiol,2002,68(5):2391-2396.
[12] JOSEPH S J, HUGENHOLTZ P, SANGWAN P, et al. Laboratory cultivation of widespread and previously uncultured soil bacteria[J]. Appl Environ Microbiol,2003,69(12):7210-7215.
[13] BRUNS A, NÜBEL U, CYPIONKA H, et al. Effect of signal compounds and incubation conditions on the culturability of freshwater bacterioplankton[J]. Appl Environ Microbiol,2003,69(4):1980-1989.
[14] AAGOT N, NYBROE O, NIELSEN P, et al. An altered Pseudomonas diversity is recovered from soil by using nutrient-poor Pseudomonas-selective soil extract media[J]. Appl Environ Microbiol,2001,67(11):5233-5239.
[15] STEVENSON B S, EICHORST S A, WERTZ J T, et al. New strategies for cultivation and detection of previously uncultured microbes[J]. Appl Environ Microbiol,2004,70(8):4748-4755.
[16] 袁志辉,王健,杨文蛟,等.土壤微生物分离新技术的研究进展[J].土壤学报,2014,51(6):1183-1191.
[17] 岳秀娟,余利岩,李秋萍,等.自然界中难分离培养微生物的分离和应用[J].微生物学通报,2006,33(3):77-81.
[18] 杨震,王爱霞,姬元忠,等.盐酸快速水解酪蛋白工艺研究[J].食品研究与开发,2011,32(7):44-47.
[19] 陈敏,赵立平.焦化废水处理系统中不同培养基分离的细菌种群多样性[J].微生物学报,2003,43(3):366-371.
Biodiversity of Bacterial Isolates on Four Different Culture Medias from Xixi Wetland
HUANG Yuan, CHU Wenke, XIE Weipeng, CHEN Min
(College of Life and Environmental Sciences, Hangzhou Normal University, Hangzhou 310036, China)
A total of 191 bacteria strains were isolated from the sediment samples collected from Xixi wetland by culture-dependent method based on four different medias (M1、R2A、NB and DNB). The phylogenetic analyses data of 16S rRNA gene sequencing revealed 13 genera, of which there were 11 genera related bacteria were isolated from R2A media, 8 genera from NB media, 7 genera from M1 and DNB media respectively. The dominant group was genusRhizobium,which accounted for 39.27% of total sequences. So R2A may be recommended to be used for a non-selective media for isolating more diversified bacteria from sediment samples.
Xixi wetland; sediment; culture media; bacterial diversity
2016-05-20
浙江省自然科学基金项目(Y307452).
陈 敏(1963—),女,教授,主要从事环境微生物研究.E-mail:mchen63@163.com
10.3969/j.issn.1674-232X.2016.06.008
Q938
A
1674-232X(2016)06-0600-06
