适于蚁蚕的基因组DNA提取方法
2016-12-12张俊辉
张 洋 金 英 薛 强 沈 群 张俊辉
(吉林省蚕业科学研究院,吉林吉林 132112)
适于蚁蚕的基因组DNA提取方法
张 洋 金 英 薛 强 沈 群 张俊辉
(吉林省蚕业科学研究院,吉林吉林 132112)
昆虫基因组DNA提取是对昆虫在分子水平上进行研究的关键,提取的DNA的纯度、浓度及完整性是进行基因工程各项研究所必须的条件。本研究主要对应用4种方法,以柞蚕蚁蚕时期组织材料为样品提取DNA后进行比较分析。结果发现,利用BIOMIGA试剂盒,置于36℃条件,经50 μL蛋白酶K 70℃消化6 h后,5 μLRNase A酶37℃消化10 min,提取的DNA质量较好。且样品易于保存,保存时间长,提取时间短的优点,对提高柞蚕研究效率具有实用价值。
蚁蚕 基因组DNA 提取
分子生物学技术已成为生命科学强劲的推动力量,为传统的及新兴的生物学科注入了新的能量,因此21世纪被称为生命科学的时代。利用分子生物学技术从核酸水平对动物进行物种鉴定以及探讨物种的起源和进化,是现在生物系统学和生物进化学的研究热点之一,这类研究的第一步也是最关键步骤是获得物种的脱氧核糖核酸(DNA)[1]。
DNA又称去氧核糖核酸,是生物体的重要组成部分,是生命科学研究的物质基础。高质量的基因组DNA是进行分子标记、基因克隆及基因表达研究等下游技术的必要前提[2-3]。因此DNA的分离纯化成为分子生物学研究中的一个重要环节。为探讨一种方便、省时、省力、效果好、简便易行的提取方法,本研究对蚁蚕为样品提取DNA的不同方法和条件进行了对比分析,期望探索出一种快速、便捷的柞蚕基因组DNA提取方法,以达到快速高效的目的。
1 材料与方法
1.1 材料
供试蚁蚕样品来源于吉林省蚕业科学研究院。
1.2 主要试剂与仪器
试剂:Tris(三羟甲基氨基乙烷)、EDTA(乙二胺四乙酸二钠)、SDS(十二烷基磺酸钠)、DTT(二硫苏糖醇)、CTAB(溴化十六烷三甲基铵)、氯化钠、β-巯基乙醇、琼脂糖购自于上海生工。三氯甲烷、异丙醇购自于北京化工厂。
两种昆虫DNA提取试剂盒购自于Omega和Biomiga。蛋白酶K、RNase A酶购于北京天根生物技术有限公司。
仪器:低温离心机、紫外成像仪、微量紫外分光光度计、电泳仪、水浴锅、温箱。
1.3 样品处理
单蛾交配后置于产卵袋,置于25℃恒温育卵,出蚕后(未添食)置于1.5 mL离心管中,加入无水乙醇于-20℃保存。实验时,每组取10头蚁蚕,用蒸馏水冲洗,晾干后放入1.5 mL离心管中,加入液氮充分研磨至粉末状,备用。
1.4 提取方法
1)方法A(SDS)与方法B(CTAB),参考文献[4-9]:
①将液氮小心的倒入预冷的含有样品的1.5 mL离心管中,用研磨杵将样品充分研磨粉碎。②A加入650 μL预热的SDS斑迹抽提液(10 mMol/L Tris-HCl,pH 8.0; 10 mMol/L EDTA, pH 8.0;100 mMol/L NaCl;2% SDS; 0.039 Mol/L DTT);B加入650 μL预热的CTAB抽提液(100mMol/L Tris-HCl, pH 8.3; 20 mMol/L EDTA, pH 8.3;1.4 Mol/L NaCl; 2% CTAB)漩涡振荡混匀后加入蛋白酶K,置于56℃条件下水浴。③加入2 μL(10 mg/mL)RNase A酶液,置于36℃条件下水浴。④样品消化液于4℃,12000 rpm离心10 min。⑤上清移入新的1.5 mL离心管中,加入等体积的苯酚,混匀后静置片刻,然后于4℃,12000 rpm离心5 min。⑥上清移入新的1.5 mL离心管中,再加入等体积的三氯甲烷∶异戊醇(24∶1),上下颠倒混匀后静置片刻,然后于4℃,12000 rpm离心5 min。⑦再将上清移入新的1.5 mL离心管中,加入2.5倍体积预冷的无水乙醇,轻轻混匀,于-20℃静置30 min。⑧于4℃,10000 rpm离心10 min,去上清,留沉淀。⑨沉淀用500 μL 70%的乙醇洗涤,于4℃,4000 rpm离心5 min,彻底弃上清。⑩将离心管倒扣在滤纸上,室温自然干燥后加入75 μL TE缓冲液溶解干燥的DNA。加入2 μL(10 mg/mL)RNase A酶液,于37℃水浴锅中保温30 min,然后用氯仿抽提一次。
2)方法C(Omega)、方法D(Biomiga)试剂盒提取方法按照使用说明书步骤进行提取,相关步骤进行了调整。
根据提取过程中的蛋白酶K、RNase A酶消化时间及使用量,设置蛋白酶K消化2 h、4 h、6 h、8 h,加入量为12.5 μL、25 μL、37.5 μL、50 μL,RNase A酶消化时间10 min、15 min、20 min、25 min,加入量为2.5 μL、5 μL、7.5 μL、10 μL,设计四因素四水平的正交试验,如表1。
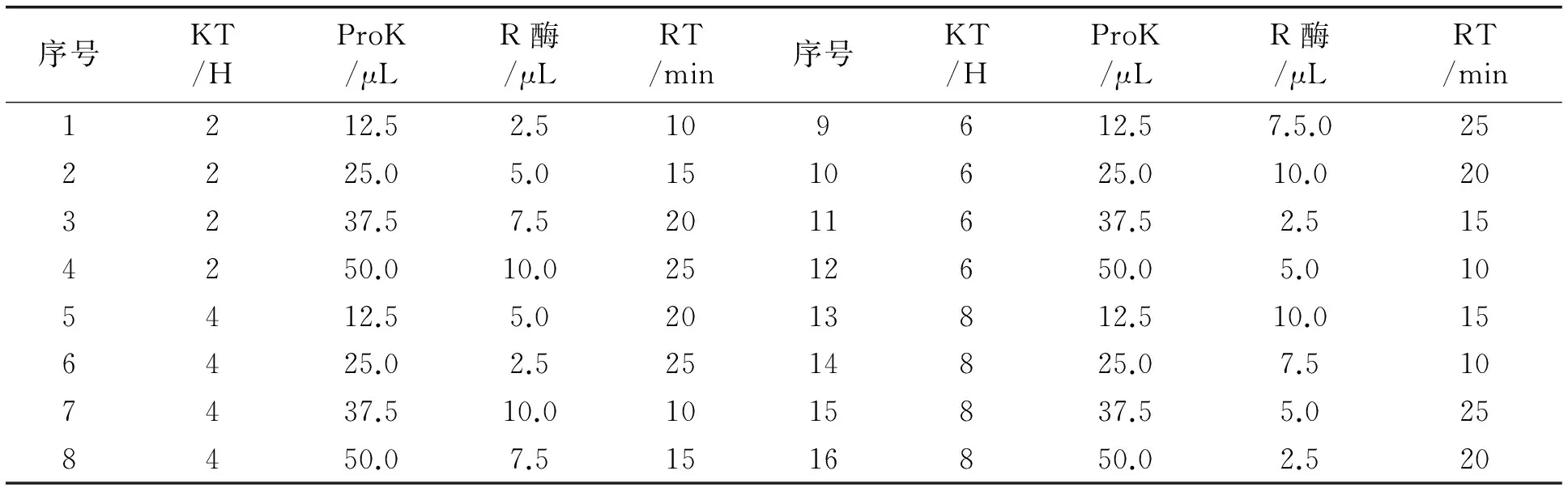
表1 正交试验设计表
注:KT-蛋白酶K消化时间;ProK-蛋白酶K;R酶-RNase A酶;RT-RNase A酶消化时间
1.5 基因组总DNA琼脂糖电泳及紫外分光光度计检测
取4 μL DNA溶液与适量上样缓冲液(6ⅹLoading buffer)混合,用1.2%琼脂糖经GelRedTM染色后,电泳后经紫外成像检测DNA片段大小及质量。用微量紫外分光光度计检测样品DNA的OD260/230和OD260/280值及浓度,判断DNA的纯度[10]。
2 结果与分析
液氮研磨后的蚁蚕为样品,分别经过蛋白酶K消化2 h、4 h、6 h、8 h,加入量分别为12.5 μL、25 μL、37.5 μL、50 μL,RNase A酶消化时间10 min、15 min、20 min、25 min,加入量分别为2.5 μL、5 μL、7.5 μL、10 μL,消化后提取柞蚕基因组,经1.2%琼脂糖凝胶电泳展示如图1-4,结果可以看出不同消化时间及方法均能成功提取的基因组,DNA的电泳条带位置在1.5 kb分子量标准带的上方,且处于同一水平排列,目的条带清晰、完整。
从图1-4中可以看出,在不同消化时间及酶处理条件下SDS和CTAB法提取的柞蚕基因组条带均出现弥散现象,存在一定程度的降解情况。在250 bp条带下方有明显的弥散条带,表明存在大量RNA,且随着蛋白酶K、RNase A酶的消化时间的延长及酶添加量的加大而未出现明显的减弱现象。
应用Omega和Biomiga试剂盒在相同的条件下提取基因组,当消化时间延长至6 h时,DNA条带拖尾的现象明显减弱,当酶处理达到4 h时在电泳图片中未检测到RNA及出现DNA的降解情况。
不同方法提取后的DNA样品经微量紫外分光光度计检测DNA的OD260/230和OD260/280值及浓度,结果见表2。结果可见,应用Omega和Biomiga试剂盒在相同的条件下提取基因组,OD260/230均大于2.0,Omega试剂盒提取的基因组OD260/280均大于2.0,Biomiga试剂盒提的基因组绝大多数在1.8≤OD260/280≤2.0范围内,两种试剂盒提取的DNA浓度随着蛋白酶K的消化时间的延长而降低,但不具有规律性。
应用SDS、CATB两种提取方法在相同的条件下提取基因组,OD260/230值绝大多数均大于2.0,OD260/230值在1.28≤OD260/280≤1.68范围内,绝大部分在相同条件下较Omega和Biomiga试剂盒比值小,说明SDS、CATB两种提取方法在提取过程中存在蛋白质或者氨基酸的浓度污染问题。
观察表2,可以看出在相同的处理条件下SDS、CATB两种提取方法的浓度是两种试剂盒的提取浓度的8~10倍。
比较不同提取方法的浓度含量可以看出,SDS、CTAB方法核酸提取量明显大于应用Omega和Biomiga试剂盒提取的结果。结合图1-4与表2,可以分析出可能是由于大量的DNA降解以及RNA残留造成浓度结果偏高。
3 讨论
重复3次试验,结合以上电泳图片和DNA样品OD值和浓度表。可以看出,利用Biomiga试剂盒提取柞蚕基因组DNA OD260/280值绝大多数在1.8≤OD260/280≤2.0范围内,OD260/230均大于2.0,在相同的处理条件下为提取效果最佳。无需其他纯化可直接用于下游的分子生物学试验。其他不同组方法织均出现DNA裂解、拖尾、点样孔出现蛋白核酸复合体等现象。或是由于样品材料含有脂肪、蛋白质、糖类杂志程度不同对提取结果造成干扰。
多糖(其他杂质等)在OD230具有最大的吸收值,双链DNA在OD260下具有最大的吸收值,蛋白质或者氨基酸的浓度在OD280下具有最大的吸收值。样品中如果含有蛋白质及苯酚,OD260/280值会明显下降。样品中若含有碳水化合物(糖类)、盐类或有机溶剂污染,OD260/230值小于2.0。一般情况下DNA的OD值约为1.8≤OD260/280≤2.0,OD260/230≥2.0。
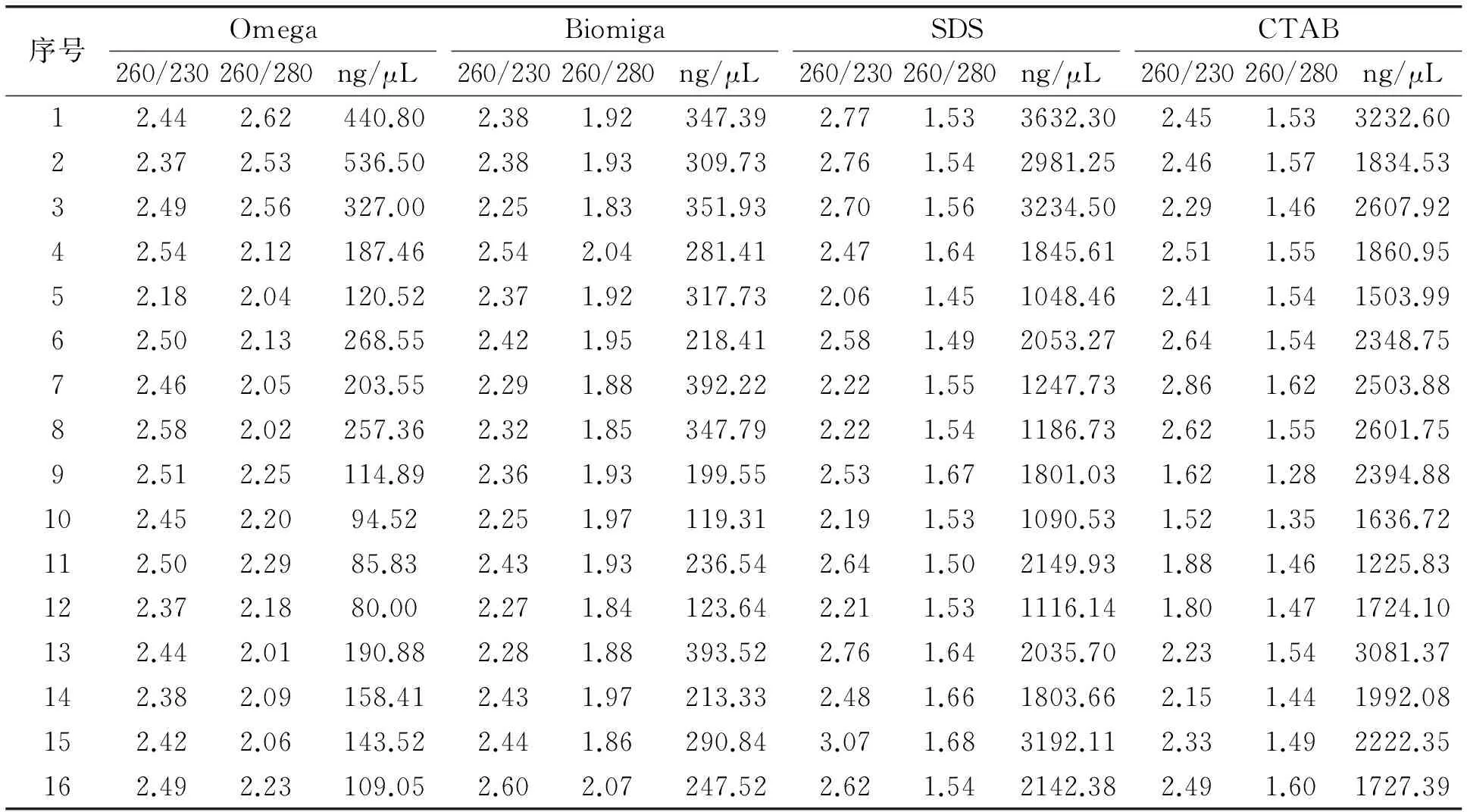
表2 DNA OD值及浓度
注:处理方法与表1序号相同
比较表2中OD值及浓度可以看出,Omega试剂盒OD260/280≥2.0,存在RNA污染,这一点也可以在图1-4中看出。SDS、CTAB两种提取方法OD260/280≤1.8,说明在提取过程中存在蛋白质、多酚等物质的污染。
SDS、CTAB两种提取方法在抽提过程中,需要经过两次的转移上清溶液,分析原因应该是由于此过程在吸取上清过程中中间层、有机层溶液吸取,造成对提取结果的影响。
现有的报道大多从中肠、丝腺[9-11]、蚕蛹[12-15]、五龄蚕足部血液[16]中提取。这些组织在进行液氮研磨时较蚁蚕耗时耗力,或提取较多数量样本时容易出现样品间污染的情况,或取样时间特定、样品制备时间长,或在保存方面也存在一定程度的困难。相对于蚁蚕在保存时期较上述样品的保存较容易,可长时间保存于-20℃无水乙醇中。
另一方面,且上述样品中含有大量的脂肪和蛋白质类物质等杂质,这些物质易与DNA结合形成粘稠的胶状物,影响基因组DNA的提取。又易抑制Tag酶活性,从而影响PCR反应,进而影响实验结果。而蚁蚕可能由于体内组织、杂质相对较少、简单等原因,从而适合提取DNA。
综上所述,Biomiga试剂盒提取的DNA,质量较好,能够满足实验需求。可以用于对DNA量不是要求很高的分子生物学实验,如PCR实验等。
[1] Chapco W, Ashton N W, Martel R K B, et al . A feasibility study of the use of random amplified polymorphic DNA in the population genetics and systematics of grasshopper[J]. Genome, 1992, 35:569-574.
[2] 程道军,鲁成,周泽扬,等.几种绢丝昆虫遗传多样性的RAPD研究[J].蚕业科学, 2002, 28(4):277-282.
[3] 游源浅,陈艳,易建平,等.松突圆蚧基因组DNA提取方法比较[J].华东昆虫学报. 2007, 16(1):26-29.
[4] 陈冬妹,林英,王艳霞,等.一种简便分离高质量家蚕基因组DNA的方法. 蚕学通讯, 2007, 27: 5-9.
[5] 候成香,李木旺,苗霄霞,等.ISSR方法在蓖麻蚕品种遗传多样性研究中的应用[J].蚕业科学, 2005, 31: 358-341.
[6] Yasukochi Y.A dense geneticmap of the silkworm, Bombyxmori, coving all chromosomes based on 1018 molecular markers[J]. Genetics,1998,150:1513-1525.
[7] [美]萨姆布鲁克D W.拉塞尔分子克隆实验指南(第三版)(上册)[M].黄培堂,译.北京:科学出版社,2002:461-487.
[8] 杨建雄.生物化学与分子生物学实验技术教程[M].北京:科学出版社,2002:57-59.
[9] 夏庆友,周泽扬,鲁成,等.家蚕 RAPD的扩增条件、重复性及遗传模型研究[J].蚕业科学,1996,22 (1):20-25.
[10] 卢圣栋.现代分子生物学实验技术[M].北京:中国协和医科大学出版社,1999.
[11] 林青松,刘朝良.家蚕与柞蚕的基因组RAPD分析[J].安徽农业科学,2007,35(9):2579-2580.
[12] 赵巧玲,张志芳,何家禄.家蚕蛹体基因组DNA的快速制备方法[J].蚕业科学,2000,26(3):63-64.
[13] 刘春宇,张春玲,陈元霖.家蚕和蓖麻蚕的基因组RAPD检测[J].蚕业科学,1997,23(4):215-220.
[14] 陈冬妹,林英,王艳霞,等.一种简便分离高质量家蚕基因组DNA的方法[J].蚕学通讯,2007,27(2):5-9.
[15] 姜振,蔡明文,徐世清,等.基于RAPD和SSR分子标记的家蚕部分实用品种多态性及其亲缘关系分析[J].蚕业科学,2010,36(1):157-164.
[16] 张洋,万军,郝大东,等.柞蚕幼虫与蛹期DNA提取分析[J].北方蚕业,2015, 36(1): 1-5.
The Extraction Method of Genomic DNA for Newly Hatched Antheraeapernyi
ZHANG Yang,JIN Ying,XUE Qiang,SHEN Qun,ZHANG Junhui
(Jinlin Sericultural Research Institute,132112,Jilin,Jinlin,China)
Insect genomic DNA extraction is the key to the study of insects at molecular level. The purity, density and completeness of extracted DNA are necessary criteria for genetic engineering researches. This study did a comparative analysis on extracted DNA from samples of newly-hatched silkworms with four methods. The results show the extracted DNA quality is better by using BIOMIGA kit which was placed under 36 degrees Celsius and for 6 hours after 50μl pro K of 70 ℃, digested for 10min by 5μl RNase at 37℃. And the sample was easy to be stored for a long time. These merits plus short extraction time make the study having practical value.
newly-hatchedAntheraeapernyi;genomic DNA;extraction
吉林省财政行业与基本课题(吉科发财[2016]40号)
张洋(1983—),男,硕士,研究方向为柞蚕病虫害防治。
