土壤宏蛋白质组学之土壤蛋白质提取技术的发展①
2016-11-21林欣萌
熊 艺,林欣萌,兰 平
(1 土壤与农业可持续发展国家重点实验室(中国科学院南京土壤研究所),南京 210008;2 南京农业大学资源与环境科学学院,南京 210095)
土壤宏蛋白质组学之土壤蛋白质提取技术的发展①
熊 艺1,2,林欣萌1,2,兰 平1*
(1 土壤与农业可持续发展国家重点实验室(中国科学院南京土壤研究所),南京 210008;2 南京农业大学资源与环境科学学院,南京 210095)
随着土壤宏基因组学的日益成熟和发展,作为后基因组时代重要技术平台的土壤宏蛋白质组学越来越受到关注。土壤宏蛋白质组学的研究是解析土壤宏基因功能的重要手段之一,对于碳氮磷的生物地球化学循环以及土壤有机质积累的研究具有重大价值,可将蛋白信息与相关的生态系统过程联系起来。但是,土壤蛋白含量少,样品复杂程度高,极大限制了土壤蛋白的分离提取和进一步分析。因此,通过改进和优化土壤蛋白的提取技术,得到高浓度的蛋白是土壤宏蛋白质组学研究的前提条件。本文总结了近几年来土壤蛋白提取方法,并对其适用的土壤性质以及适用的不同种类和功能的蛋白进行了分析。此外,本文也对现有的土壤蛋白提取技术以及土壤蛋白鉴定技术的方法改进进行了探讨。
土壤蛋白质;宏蛋白质组学;蛋白质提取方法
众所周知,蛋白是生理功能的最终执行者。土壤蛋白质在碳氮磷的生物地球化学循环以及有机物的积累方面发挥重要作用,与土壤质量与可持续发展息息相关[1]。宏蛋白质组学是研究整个生态系统内所有微生物来源的蛋白质种类和丰度变化的技术手段[2]。虽然,总体而言宏蛋白组学的研究较少,特别是明确鉴定在生态系统中发挥重要功能的蛋白的研究目前几乎是微生物生态学研究中的一个空白[3-7]。但是,在过去的10年中,宏蛋白质组学极大地加深了我们对于微生物群落以及它们所处的地球化学环境的理解[8-10]。蛋白质组学的研究提供了不同微生物类群在凋落物分解过程中所起到的作用,对于胞外酶的研究可以揭示微生物量的限制因素、酶与凋落物分解的关系、酶活性与相关蛋白丰度的关系、酶的微生物起源以及落叶层生物与生物地球化学作用之间的关系[11]。微生物蛋白与其生境的时间分布可以为微生物的多样性与其在生物地球化学作用过程中所扮演的角色的研究提供帮助[12]。宏蛋白质组学将从一个新的水平上提供微生物演替和特定种群分布活动的信息,并且这些信息都能从表达的蛋白上得到解析。例如,预测未来微生物群落的演替规律,哪种微生物在分解作用中的贡献最大,以及哪个因素影响了整个分解过程[12]。土壤微生物生物量巨大、种类繁多、不易分离培养[13],宏基因组学与蛋白质组学结合为解决这一难题提供了契机,特别是宏蛋白质组学可以深入地研究种群的多样性,可以直接观察蛋白质在环境样本中的变化,能够提供细胞的功能信息,可以细致地分析群落的结构与功能[14]。宏蛋白质组学可以通过分析微生物酶的来源功能和活动来解决不同种类蛋白的混合表达问题[11],从而克服寻找酶信息与相关生态系统过程联系的困难[15]。但是,土壤蛋白含量少,样品复杂程度高,极大限制了土壤蛋白的分离提取和进一步分析。因此,通过改进和优化土壤蛋白的提取技术,得到高浓度的蛋白是土壤宏蛋白质组学研究的前提条件[16]。
1 高效分离提取土壤蛋白的限制因素
1.1 土壤样品的复杂性
土壤具有高度的微生物多样性,土壤生态系统中,微生物数量巨大,种类丰富,微生物与环境之间的相互作用多样;并且,土壤环境具有高度多样的生物化学途径,不同种类的微生物在不同生长时期代谢利用的底物与最终产物不同,这也就造成了土壤环境中海量的蛋白种类[17]。此外土壤中的蛋白质样本分布并不均匀,这是因为土壤养分分布是由结构性因素和随机性因素共同作用的结果。结构性因素,如气候、母质、地形、土壤类型自然因素等可以导致土壤养分强的空间相关性;而随机性因素,如施肥、耕作措施、种植制度等各种人为活动使得土壤养分的空间相关性减弱,朝均一化方向发展[18]。不同区块的土壤蛋白含量可能也有类似的变化情况。
1.2 土壤蛋白质的丰度低
土壤含有大量干扰物质,尤其是天然有机物质。然而在大部分土壤当中,蛋白质含量低,现有的土壤蛋白的提取方法无法获得适用于蛋白组分析的高质量土壤蛋白样品[19]。
1.3 高度的生物多样性与微生物群落的高度动态性变化
叶片来源和微生物来源的蛋白比例在凋落物分解的过程中在不断变化,然而这个比例却很难被统计[20]。在凋落物形成早期,植物来源的蛋白质占大多数,在1 ~ 2年的凋落物中,微生物来源的蛋白质则占90%[21-22]
1.4 蛋白质的吸附作用
虽然,很多暴露在土壤中的蛋白质会被土壤微生物很快地分解成肽段碎片,但小部分蛋白被认为通过与土壤黏土矿物和有机质结合,从而有了抵抗微生物降解的能力[23]。
胞外酶经常被土壤矿物吸收或者黏附,也常被土壤腐殖质胶体捕获,蛋白质的吸附作用使酶稳定,使酶免受蛋白质水解酶的影响。但是蛋白质的吸附作用会造成酶在被黏粒吸附之后催化活性的降低,这将导致稳定之后的酶与具体的微生物活动并无关联[24]。这也将会对酶的提取造成影响,在分离过程中,酶的形态已经发生改变[25]。Keiblinger等[6]用已经完全测序的微生物蛋白与土壤颗粒混合进行稀释实验,然而胞外酶的肽段覆盖率比胞内酶更低,且在与土壤颗粒作用后,仅有40% 的肽段光谱能够被数据库检测到。
由于某些蛋白与有机质之间的联接,与其他大量分泌到胞外的酶(比如磷酸酶、葡萄糖苷酶、脲酶)相比,利用质谱仪对他们的鉴定依然具有不确定性。蛋白质与腐殖质之间的强联系(酶-腐殖质复合体)已被多位专家证实[26-27]。
1.5 提取缓冲区选择范围小
从土壤中提取蛋白质具有一定难度,一方面由于提取缓冲区的选择范围小,另一方面需要从提取出的有机土壤组分中对蛋白质进行激烈地纯化,而这会导致蛋白质的损失和降解[28]。
2 土壤蛋白提取方法的发展
2.1 提取方法的改变
目前,土壤蛋白的提取还没有统一的方法,各种方法都有其局限性[29]。
最初,土壤酶的活性能够通过间接的方法被检测到,比如通过土壤溶液或者土壤浸出液测定酶的活性,但是却没有胞外酶分子或者胞外蛋白分子被检测到的报告。在最初的实验方法中,通过六号滤纸并不能完全除去细菌细胞,胞内蛋白也能被SDS-PAGE检测到[30]。
从土壤基质的无机和有机成分中分离蛋白质时,Benndorf等人[31]采用了NaOH处理与酚提取相结合的方式。其中,0.1 mol/L的NaOH的处理使得腐殖酸和蛋白质从土壤矿物中释放出来,同时破坏了微生物。而随后的酚提取步骤将蛋白质从腐殖质中分离出来,并用于蛋白质组学的研究。此种方法可分别提取出胞内和胞外蛋白,且利于蛋白组学的功能分析。但是蛋白质的提取率很低,要通过预培养和接种才可获得足够分析的蛋白质含量[29]。
柠檬酸盐提取法同时提取出的杂质较多,但是提取物的颜色较深,因此可以促进随后的SDS提取,而单纯的SDS提取方法,提取物颜色浅黄,含有杂质较少。两个方法连续使用,能够提取出更多的蛋白并且能够显示更多的条带。仅用NaOH或者柠檬酸盐是不够的,因为在接下来的SDS缓冲液提取步骤中仍然能够从土壤基质中洗脱出大量蛋白[19]。
土壤蛋白提取的困难在于蛋白提取方法的缺失。蛋白质功能的鉴定受到提取方法低效率的严重影响,也许大部分蛋白在纯化步骤中未被高效地提取,或者在过程中被丢失[28]。
采用原位或浸出法直接提取出总蛋白,再由过膜、沉淀和离心等分离手段分离出细胞蛋白的蛋白直接提取法具有一些缺陷,例如提取出的蛋白质的浓度通过比色法或免疫学技术来测定,而这种测定方法会因为含有的酚类物质而发生偏差。除此之外,蛋白质的性质通过没有任何氨基酸序列标识的电泳来展现。土壤蛋白直接提取方案的最大缺点是提取率和对土壤微生物区系的影响一直未被研究[28]。2010年Chourey等[32]提出了一个新的土壤微生物蛋白的直接提取方法,即SDS-TCA法。此法采用热变性和SDS相结合的方法处理土壤样品,接着通过TCA沉淀及丙酮洗涤等步骤,提取出蛋白样品。这种方法在不同的土壤样品中均能鉴定出500个以上的蛋白,并且与纯培养的相比,在蛋白大小和功能分类等方面均无明显偏差。与间接提取法相比,此法具有明显的优势,被认为是目前为止最有效的土壤微生物蛋白组学的提取方法[29]。
如表1所示,一开始的土壤蛋白质的提取方法只包括简单的过滤、离心与渗析,随后为了通过电泳获得更多的蛋白条带与斑点,大家开始采用柠檬酸盐、SDS、酚类物质相结合的方法。由于需要蛋白质条带或斑点的可视化体现,此阶段的蛋白质提取手段需要考虑到浓缩产物的颜色。从2010年左右开始,随着质谱分析技术的进步,为了适应定量化的质谱分析的需求,大家开始使用超声波破碎或者加入液氮研磨的手段使细胞更容易破碎,加入NaOH浸提、乙酸铵沉淀、TCA沉淀、洗涤剂(DTT)与尿素清洗、丙酮清洗等步骤使得提取产物种类更加全面和纯净。然而SDS与苯酚始终是去除有机质与土壤矿物黏粒的有力武器。
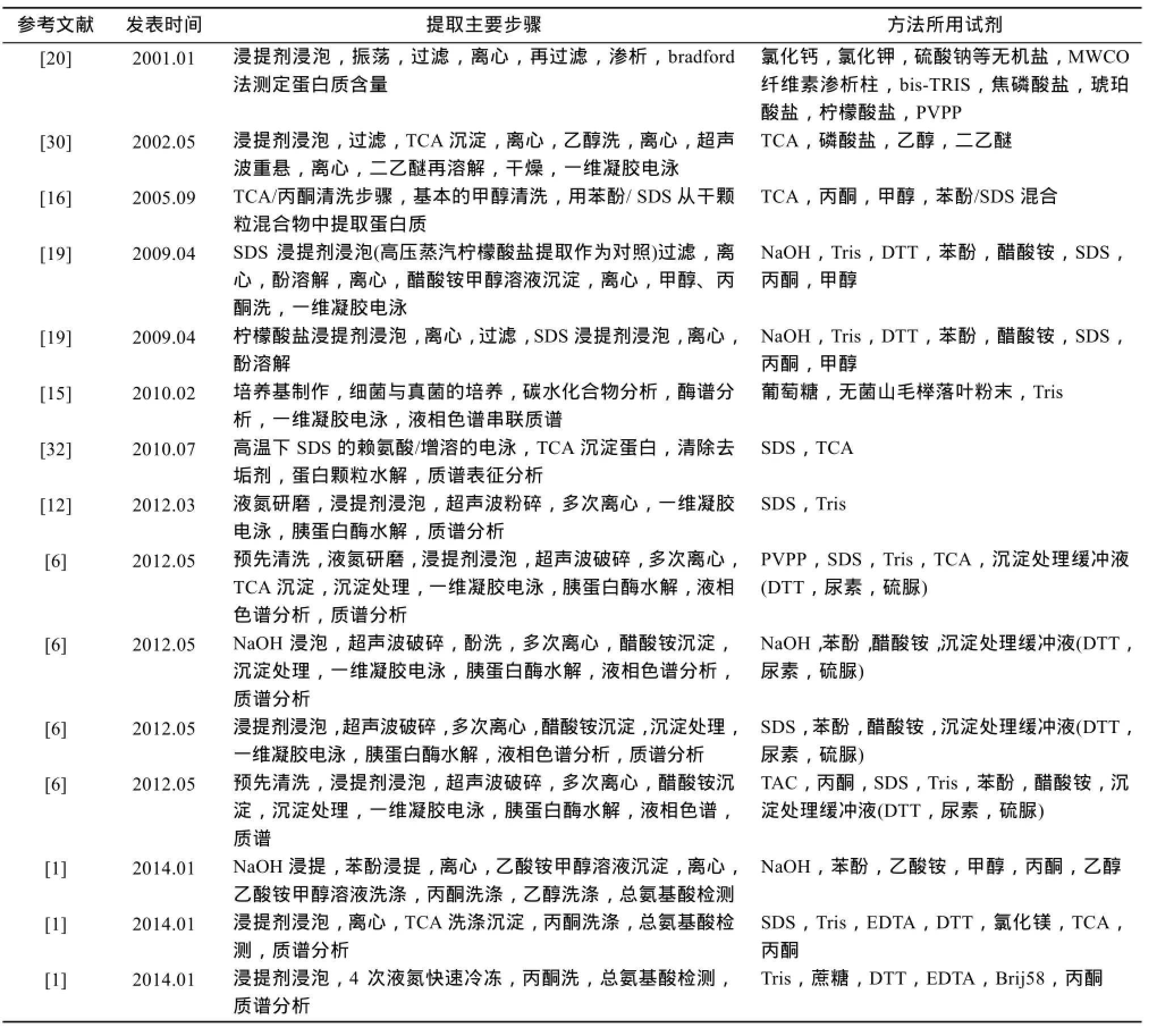
表1 近年来土壤蛋白提取方法的主要步骤以及所用试剂Table1 Main steps and reagents used for the soil protein extraction in recent years
2.2 蛋白鉴定方法的进步
在对土壤蛋白质进行一维凝胶电泳的过程中,在pH = 6.0时,得到了最清晰的电泳图谱,然而在电泳过程中,蛋白质条带只能在拥有足够的分子质量相同的蛋白质的情况下才会显露出来,而土壤蛋白仅有很少的量并且拥有大量不明确的相对分子质量[30]。
在2002年以前的蛋白质浓缩物的检测方法主要包括紫外光吸收法、色谱分析法、茚三酮法、通过比色的蛋白质定量分析(比如双缩脲法、lowry法、bradford法)以及二喹啉甲酸法[20]。
然而,不恰当的蛋白质计量方法也会影响蛋白质的的鉴定效果。某些土壤蛋白一般会和腐殖质形成复合物从而被共同提取,然而这会对标准比色法对于蛋白质含量的测定产生影响,比如bradford法[33],一些已经商用的发色基团以及荧光素都是无效的,不能提供正确的量比指标[34]。因此我们需要不受干扰物质影响的蛋白质含量的测定方法,比如氨基酸计量法[1]。
如表1所示,最初,在土壤蛋白的定量分析上,主要使用bradford法等比色法。而后出现了一维凝胶电泳和二维凝胶电泳。随着质谱分析技术和色谱分析技术的发展又出现了液相色谱串联质谱,或者单独的色谱分析和质谱分析。而如今在常用的土壤蛋白提取物分析技术中,电泳则作为在质谱分析或者色谱分析所必需的胰蛋白酶水解步骤之前的预处理手段。如表2所示,当然更加先进的检测手段(比如利用液相色谱仪和质谱仪检测蛋白质的种类和数量)和与之相适应的提取手段必然会检测到更多的蛋白种类与更加精确的蛋白含量。

表2 近年来不同土壤蛋白提取方法的提取效果Table2 Performance of different methods for the soil protein extraction in recent years
3 不同土壤蛋白提取方法适用的情景
3.1 不同性质土壤适合的提取方法
表3中罗列了近年来适宜不同性质土壤类型的土壤蛋白提取方法,采用不同的方法提取同一种性质的土壤中的蛋白,结果差异很大。
由于在提取过程中,溶液中的蛋白质的水解作用以及部分蛋白在TCA中的溶液化,TCA预处理可能导致高分子量的蛋白质的丢失[35]。SDS和酚共同使用提取效果更佳。酚类对于干扰物(比如腐殖质)的去除最有效果,避免了蛋白质和干扰物的共同提取[34]。但是,由于腐殖质主要位于水相,蛋白质主要位于酚相[31],这也可能造成蛋白质与腐殖质复合体在移去水相的过程中的丢失,从而导致相关蛋白的缺失[25]。使用NaOH的提取方法也可能造成类似的影响[31]。此外酚类物质和SDS共同使用还能提高蛋白提取的效果[19]。但是单一的提取方法可能会偏向于某些群落结果的信息。Keiblinger等[6]用了4种方法测定提取土壤蛋白,然而4种方法共同检测到的光谱量很少,也许特定的方法对于特定的种类的蛋白有特殊的偏好。

表3 不同理化性质的土壤或凋落物适合的土壤蛋白提取方法Table3 Soil protein extraction methods suitable for the soil or leaf litter with different physical and chemical properties
柠檬酸盐和SDS缓冲液连续使用的提取方法在不同利用类型的土地的土壤中都表现得很不错[19]。
为了检测到所有的蛋白,Benndorf等[31]采用紧接着有机溶剂反复清洗的酚类物质提取方法,这个方法对于去除土壤或者地下水中的有机质干扰物非常有效。这种方法似乎也减少了黄铁矿和蛋白质之间的联系,因为蛋白质在酚中呈现出了比在水中更高的溶解度。虽然说这种方法对低分子量蛋白质提取具有显著效果,但同时也丢失了一些高分子量的蛋白[36]。
3.2 不同种类、功能蛋白适合的提取方法
如表4所示,不同的土壤蛋白提取方法对不同种类、功能的蛋白质存在不同的偏好。
不同的蛋白质会被蛋白酶水解到不同的程度,其他修饰酶也会将不同的蛋白质修饰成不同的性状,而在加入酚类物质的提取过程中,蛋白质又不可避免地会在水相中溶解,每种蛋白又会有不同的沉淀和溶解的能力,在TCA沉淀的过程中,蛋白质又会被不同程度地灭活,故每种蛋白质提取方法可能造成专一性蛋白质的丢失[35]。
根据前人的经验,可以在进行光谱分析之前,将不同提取方法得到的蛋白混合起来,这样可以使能够检测到的蛋白质种类更加全面[6]。
将已经完全测序的真菌与细菌作为模式系统可以解决数据库相关序列不全面的问题。但这样将会受到生物多样性单一以及加入的培养基和凋落物使得生境过于单一[15]。
可以制备土壤悬浊液,通过差速离心法得到富含土壤微生物细胞的层次,从而实现土壤蛋白的富集[37]。也可以利用NaOH和苯酚去除土壤中的腐殖质直接从土体中提取土壤蛋白[31]。

表4 不同种类、功能的土壤蛋白质适合的提取方法Table4 Extracting methods for the soil protein with different kinds and functions
4 总结和展望
4.1 现有土壤蛋白质提取技术的局限性
由于土壤样品成分复杂,土壤样品中蛋白质含量低,土壤蛋白的种类和数量受土壤生物群落动态性变化影响很大,土壤物质对于蛋白质有吸附作用以及提取土壤蛋白的浸提液缓冲区可选择的范围狭窄等原因,目前尚无一种可以从不同性质的土壤样品中无差别地提取所有蛋白质的方法。
适用于土壤蛋白质组学的蛋白质提取方法发展到现在,主要有先将土壤中的微生物细胞分离再提取胞内蛋白,或者提取细胞分泌到土壤中的蛋白;以及直接将土壤中的微生物细胞溶解破碎,再提取土壤中总蛋白的3种思路。
然而提取过程中使用试剂的不同,缓冲体系选择的pH不同,离心、过滤、沉淀等的操作顺序、时间、强度的不同又会使得不同的提取方法对某些种类的蛋白产生偏好[6]。从目前所能达到的最高技术水平来看,通过土壤蛋白质组学的研究得到的数据,由于其提取完整性较低、变异性太大以及肽段序列数据库匮乏的原因,往往需要结合利用土壤宏基因组学、转录组学得到的数据才能对研究对象做出较为客观的结论[38]。
4.2 解决思路
4.2.1 通过特定的提取方法从特定类型的土壤中提取特定类型的蛋白 如表3、4所示,不同的蛋白质提取方法可以从不同类型的土壤中提取出不同种类和功能的蛋白质。洗涤剂的使用以及高温会更加高效地促进细胞裂解以及矿物质与蛋白质的分离[32],高浓度的DTT也许能通过减弱蛋白质与矿物和腐殖质之间的二硫键来提取更多的土壤蛋白[1]。
Chourey等[32]和Singleton等[39]的方法在检测主要的细胞活动和新陈代谢过程中蛋白质比例的信息的能力是相同的,但Singleton的方法在森林土壤蛋白功能的检索能力方面还是有所欠缺的,而Chourey的方法能够在研究C与N固定的过程以及有机物水解的方面提供更多的信息。而且两种方法在分类上也有偏见,前者偏向于提取真菌蛋白,而后者倾向于提取细菌蛋白。使用DTT的Chourey的方法提取出的蛋白多样性更高[1]。
4.2.2 通过蛋白质肽段数据库的完善鉴定出更多的蛋白 本文所涉及的土壤蛋白质组学的研究大多涉及了蛋白质肽段数据库的匮乏导致的土壤蛋白鉴定的不全面和偏差。Baldrian和López-Mondéjar[40]在实验中发现,分泌蛋白的数据库组要由氧化酶类和水解酶类组成,能检测到真菌的蛋白信息而没有对应的基因序列主要是由于将检测到的蛋白去对应已知基因序列的能力不足造成的。
宏转录学虽然能直接地鉴定代谢途径,但是也缺乏mRNA和蛋白质之间的对应关系。蛋白质组学的质谱鉴定也是依赖于相关基因图谱的丰富程度,蛋白质组学也能从更加丰富的基因序列中受益[2]。
一些根际微生物生态的研究使用了稳定同位素探测技术,利用C13与N15标记核酸,从而探测和鉴定在土壤微生物群落中参与了特定的新陈代谢过程或者其他生化途径的微生物种群[33]。此外,稳定同位素同时也可以标记蛋白质,若能解决土壤蛋白质的分离纯化上的困难以及蛋白质数据库缺失的问题,SIP-Protein techniques(稳定同位素标记蛋白技术)也能用来准确鉴定具体的酶和特定种类的微生物在土壤群落生物化学过程中起到的作用。
4.2.3 建立广谱高效的蛋白质提取方法 配合更高精度的质谱分析更全面地提取并鉴定蛋白的质谱分析与色谱分析是鉴定土壤蛋白的关键[1]。一种较为可行的提高土壤蛋白提取的广谱性和公平性的方法是将不同提取方法提取出的土壤蛋白浓缩液混合在一起然后再进行电泳或者液相色谱以及质谱仪检测,这样能够使提取出来的土壤蛋白种类更加全面[6]。
[1] Bastida F, Hernández T, García C. Metaproteomics of soils from semiarid environment: Functional and phylogenetic information obtained with different protein extraction methods[J]. Journal of Proteomics, 2014, 101: 31-42
[2] Siggins A, Gunnigle E, Abram F. Exploring mixed microbial community functioning: Recent advances inmetaproteomics[J]. FEMS Microbiol. Ecol., 2012, 80:265-280
[3] VerBerkmoes N C, Denef V J, Hettich R L, et al. Functional analysis of natural microbial consortia using community proteomics[J]. Nat. Rev. Microbiol., 2009, 7:196-205
[4] Morris R M, Nunn B L, Frazar C, et al. Comparative metaproteomics reveals ocean-scale shifts in microbial nutrient utilization and energy transduction[J]. ISME J.,2010, 4: 673-685
[5] Sowell S M, Abraham P E, Shah M, et al. Environmental proteomics of microbial plankton in a highly productive coastal upwelling system[J]. ISME J., 2011, 5: 856-865
[6] Keiblinger K M, Wilhartitz I C, Schneider T, et al. Soil metaproteomics — comparative evaluation of protein extraction protocols[J]. Soil Biol. Biochem., 2012, 54:14-24
[7] Hettich R L, Pan C, Chourey K, et al. Metaproteomics:Harnessing the power of high performance mass spectrometry to identify the suite of proteins that control metabolic activities in microbial communities[J]. Anal. Chem., 2013, 85: 4 203-4 214
[8] Newman D K, Banfield J F. Geomicrobiology: How molecular-scale interactions underpin biogeochemical systems[J]. Science, 2002, 296: 1 071-1 077
[9] Schneider T, Riedel K. Environmental proteomics: Analysis of structure and function of microbial communities[J]. Proteomics, 2010, 10: 785-798
[10] Wilmes P, Bond P L. Microbial community proteomics:Elucidating the catalysts and metabolic mechanisms that drive the Earth’s biogeochemical cycles[J]. Curr. Opin. Microbiol., 2009, 12: 310-317
[11] Sinsabaugh R L, Lauber C L, Weintraub M N, et al. Stoichiometry of soil enzyme activity at global scale[J]. Ecol. Lett., 2008, 11: 1 252-1 264
[12] Thomas S, Katharina M K, Emanuel S, et al. Who is who in litter decomposition: Metaproteomics reveals major microbial players and their biogeochemical functions[J]. The ISME Journal, 2012, 6: 1 749-1 762
[13] Mocali S, Benedetti A. Exploring research frontiers in microbiology: The challenge of metagenomics in soil microbiology[J]. Research in Microbiology, 2010, 161:497-505
[14] Bastida F, Algora C, Hernandez T, et al. Feasibility of a cell separation-proteomic based method for soils with different edaphic properties and microbial biomass[J]. Soil Biology & Biochemistry, 2012, 45: 136-138
[15] Thomas S, Bertran G, Regula G, et al. Proteome analysis of fungal and bacterial involvement in leaf litter decomposition[J]. Proteomics, 2010, 10: 1 819-1 830
[16] Wang W, Vignani R, Scali M, et al. A universal and rapid protocol for protein extraction from recalcitrant plant tissues for proteomic analysis[J]. Electrophoresis, 2006, 27:2 782-2 786
[17] Becher D, Bernhardt J, Fuchs S, et al. Metaproteomics to unravel major microbial key players in leaf litter and soil environments: Challenges and perspectives. Proteomics,2013, 13: 2 895-2 909
[18] 郭旭东, 傅伯杰, 马克明, 等. 基于 GIS和地统计学的土壤养分空间变异特征研究——以河北省遵化市为例[J].应用生态学报, 2000, 11(4): 557-563
[19] Shaoning C, Matthias C R, Wei W. Improving soil protein extraction for metaproteome analysis and glomalin-related soil protein detection[J]. Proteomics, 2009, 9: 4 970-4 973
[20] Criquet S, Farnet A M, Ferre E. Protein measurement in forest litter[J]. Biol. Fertil. Soils, 2002, 35: 307-313
[21] Miltner A, Zech W. Microbial degradation and resynthesis of proteins during incubation of beech leaf litter in the presence of mineral phases[J]. Biol. Fertil. Soils, 1999, 30:48-51
[22] Ziegler F, Zech W. Veränderungen in der stofflichen-Zusammen-setz.ung von buchenstren und gerstenstrohbeim abbauunter laborbedingungen[J]. Z. Pflanzenernaehr Bodenkd, 1991, 154: 377-385
[23] Boyd S A, Mortland M M. Enzyme interaction with clays and clay-organic matter complexes[J]. Soil Biochemistry,1990, 6: 1-28
[24] Nannipieri P. Role of stabilised enzymes in microbial ecology and enzyme extraction from soil with potential applications in soil proteomics // Nannipieri P, Smalla K. Nucleic acids and proteins in soil[M]. Berlin, Heidelberg:Springer Berlin Heidelberg; 2006: 75-94.
[25] Giagnoni L, Magherini F, Landi L, et al. Soil solid phases effects on the proteomic analysis of Cupriavidus metallidurans CH34[J]. Biology and Fertility of Soils, 2012, 48(4):425-433
[26] Nannipieri P, Sequi P, Fusi P. Humus and enzyme activity // Piccolo A. Humic substances in terrestrial ecosystems[M]. Amsterdam: Elsevier, 1996: 293-328
[27] Masciandaro G, Macci C, Doni S, et al. Comparison of extraction methods for recovery of extracellular βglucosidase[J]. Soil Biol. Biochem., 2008, 40: 2 156-2 161
[28] Giagnoni L, Magherini F, Landi L, et al. Extraction of microbial proteome from soil: Potential and limitations assessed through a model study[J]. European Journal of Soil Science, 2011, 62: 74-81
[29] 王小丽, Pablo G P, 叶俊, 等. 土壤宏蛋白质组学蛋白质提取方法及其应用[J]. 应用与环境生物学报, 2012, 18(4):691-696
[30] Akifumi M, Masaki Y, Risa U, et al. Isolation of extracellular protein from greenhouse soil[J]. Soil Biology & Biochemistry, 2003, 35: 733-736
[31] Benndorf D, Gerd U B, Hauke H, et al. Functional metaproteome analysis of protein extracts from contaminated soil and groundwate[J]. The ISME Journal,2007, 1: 224-234
[32] Chourey K, Janet J, Nathan V, et al. Direct cellular lysis/protein extraction protocol for soil metaproteomics[J]. J. Proteome Res., 2010, 9(12): 6 615-6 622
[33] Bastida F, Moreno J L, Nicolas C, et al. Soil metaproteomics: A review of an emerging environmental science. Significance, methodology and perspectives[J]. Eur. J. Soil Sci., 2009, 60: 845-859
[34] Roberts P, Jones D L. Critical evaluation of methods for determining total protein in soil solution[J]. Soil Biol. Biochem., 2008, 40: 1 485-1 495
[35] Carpentier S C, Witters E, Laukens K, et al. Preparation of protein extracts from recalcitrant plant tissues: An evaluation of different methods for two- dimensional gel electrophoresis analysis[J]. Proteomics, 2005, 5: 2 497-2 507
[36] Benndorf D, Carsten V, Nico J, et al. Improving protein extraction and separation methods for investigating the metaproteome of anaerobic benzene communities within sediments[J]. Biodegradation, 2009, 20: 737-750
[37] Williams M A, Taylor E B, Mula H P. Metaproteomic characterization of a soil microbial community following carbon amendment[J]. Soil Biol. Biochem., 2010, 42:1 148-1 156
[38] Jenni H, Mark P, Rache M, et al. Multi-omics of permafrost,active layer and thermokarst bog soil microbiomes[J]. Nature, 2015, 521(7551): 208-212
[39] Singleton I, Merrington G, Colvan S, et al. The potential of soil protein-based methods to indicate metal contamination[J]. Appl. Soil Ecol., 2003, 23: 25-32
[40] Baldrian P, López-Mondéjar R. Microbial genomics,transcriptomics and proteomics: New discoveries in decomposition research using complementary methods[J]. Appl. Microbiol. Biotechnol., 2014, 98: 1 531-1 537
The Development of Soil Protein Extraction Methods in Soil Metaproteomics
XIONG Yi1,2, LIN Xinmeng1,2, LAN Ping1*
(1 State Key Laboratory of Soil and Sustainable Agriculture (Institute of Soil Science, Chinese Academy of Sciences), Nanjing 210008, China; 2 College of Resources and Environmental Sciences, Nanjing Agricultural University, Nanjing 210095, China)
There has been increasing concern about the soil metaproteomics in recent years. Proteomics platform in soil is helpful for understanding the biogeochemical cycles of carbon, nitrogen and phosphorus and the oxidation of soil organic matter. By analyzing the sources, functions and activities of soil microbial protein, soil metaproteomics can address the issue of different kinds of protein expression and then link protein information with the associated ecosystem processes. However, although the species of protein in soils are very rich, the contents of proteins in soils are usually very low, which dramatically hinders the application of proteomics techniques. A critical step thus is to get a high concentration of protein solution by pretreatment of soil samples. This review summarized some typical methods for extracting proteins from soils and leaf litter in recent years. In addition, the potential directions of improvements for current methods were discussed.
Soil protein; Proteomics; Protein extraction methods
S154.3
10.13758/j.cnki.tr.2016.05.001
国家重点基础研究发展计划(973计划)项目(No. 2015CB150501)和国家重点研发计划项目(2016YFD0200308)资助。
*通讯作者(plan@issas.ac.cn)
熊艺(1995—),男,四川泸州人,主要从事土壤蛋白组学研究。E-mail: 13613229@njau.edu.cn
