饲粮精粗比对泌乳水牛瘤胃细菌和甲烷菌区系的影响
2016-11-15李丽莉韦升菊邹彩霞
林 波 梁 辛 李丽莉 韦升菊 李 萍 邹彩霞,*
(1.广西大学动物科技学院,南宁530004;2.中国农业科学院广西水牛研究所,农业部水牛遗传繁育重点实验室,广西水牛遗传繁育重点实验室,南宁530001)
饲粮精粗比对泌乳水牛瘤胃细菌和甲烷菌区系的影响
林波1梁辛2李丽莉2韦升菊2李萍1邹彩霞1,2*
(1.广西大学动物科技学院,南宁530004;2.中国农业科学院广西水牛研究所,农业部水牛遗传繁育重点实验室,广西水牛遗传繁育重点实验室,南宁530001)
本试验旨在研究饲粮精粗比对泌乳水牛瘤胃细菌和甲烷菌区系的影响。选取15头健康杂交后备泌乳水牛,按体重、采食量等相近原则随机分为3组,每组5头,分别饲喂精粗比为0∶100(全粗料组)、35∶65(低精料组)、50∶50(中等精料组)的混合饲粮。试验为期40 d,其中前10 d为预试期。在试验结束后第1天,经口腔抽取瘤胃液抽提微生物DNA,采用Illumina Miseq PE250平台研究瘤胃细菌和甲烷菌区系组成。结果表明:1)在门水平上,水牛瘤胃内拟杆菌门(45%~65%)、厚壁菌门(13%~27%)和变形菌门(13%~18%)为主要细菌类别,与低精料组和中等精料组相比,全粗料组提高了拟杆菌门、黄杆菌门和SR1细菌的比例,降低了厚壁菌门、疣微菌门、放线菌门和绿弯菌门细菌的比例;在科水平上,普雷沃氏科(15%~32%)和黄杆菌科(8%~21%)为主要细菌类别,与中等精料组相比,全粗料组、低精料组提高了普雷沃氏科细菌的比例,降低了黄杆菌科、毛螺菌科、瘤胃球菌科、红蝽杆菌科和双歧杆菌科细菌的比例;中等精料组瘤胃细菌相比全粗料组有更高的多样性。2)水牛瘤胃内,90%以上的甲烷菌为甲烷短杆菌属,其次为热原体属,饲粮精粗比的改变并未影响甲烷短杆菌属在瘤胃内的优势地位。综合得出,全粗料饲粮有提高水牛瘤胃内纤维降解相关细菌比例的趋势,却降低了瘤胃细菌的多样性;饲粮精粗比的差异对水牛瘤胃甲烷菌在属水平上的组成并无显著影响。
水牛;精粗比;瘤胃;细菌区系;甲烷菌区系
水牛是典型的以粗料饲喂的反刍动物,其瘤胃发酵特点及微生物区系组成有何特点尚无定论。饲粮精粗比是决定瘤胃发酵的主要因素,而改变饲粮精粗比以研究瘤胃微生物区系的反应也是阐明反刍动物瘤胃微生物区系特点的重要手段[1-2]。Mao等[3]研究表明,在饲喂高精料饲粮的奶牛瘤胃内,厚壁菌门(Firmicutes)和放线菌门(Actinobacteria)细菌的比例则较高,而变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes)细菌的比例较低。Pitta等[4]对水牛的研究发现,全粗料饲粮饲喂后瘤胃内瘤胃球菌属(Ruminococcus)和黄杆菌门(Fibrobacter)细菌的比例较高;而精粗比为50∶50的饲粮饲喂后瘤胃内普雷沃菌属(Prevotella)细菌的比例较高,表明瘤胃球菌属和黄杆菌门细菌是瘤胃内纤维消化的重要细菌类别。本课题组前期的研究也表明,提高饲粮精粗比降低了瘤胃内产琥珀酸丝状杆菌和丁酸弧菌数量[5]。高粗饲粮条件下,纤维降解菌如丁酸弧菌、栖瘤胃普雷沃氏菌等是瘤胃中的优势菌种;高精料条件下,嗜淀粉类细菌如反刍兽新月单胞菌、消化链球菌、乳酸杆菌以及嗜淀粉瘤胃杆菌为优势菌种[6-7]。然而,在不同精粗比饲粮下水牛瘤胃微生物区系的变化情况研究报道还较少,不足以阐明水牛瘤胃微生物区系的特点。因此,本研究采用高通量测序技术对饲喂不同精粗比饲粮的水牛瘤胃细菌和甲烷菌区系的变化进行了研究,以期阐明饲粮精粗比构成对水牛瘤胃细菌和甲烷菌区系的影响,为进一步了解水牛瘤胃提供理论依据。
1 材料与方法
1.1试验动物
本试验在广西水牛研究所水牛养殖示范基地进行。选取15头健康杂交[广西本地水牛×(尼里-拉菲水牛)×摩拉水牛]后备泌乳水牛,按体重、采食量等相近原则随机分为3组,每组5头水牛。试验牛进行单槽专人饲喂,采取先粗后精的饲喂顺序。饲喂后,试验牛放于运动场自由运动和饮水。
1.2试验设计及饲养管理
采用单因素设计原则,3组水牛分别饲喂精粗比为0∶100(全粗料组)、35∶65(低精料组)和50∶50的混合饲粮(中等精料组),试验期共40 d,其中预试期10 d,正试期30 d。预试期内逐步过渡饲粮精粗比,到正试期前达到试验所设定的精粗比。精料组成、精料和粗料饲喂量及采食营养水平见表1。
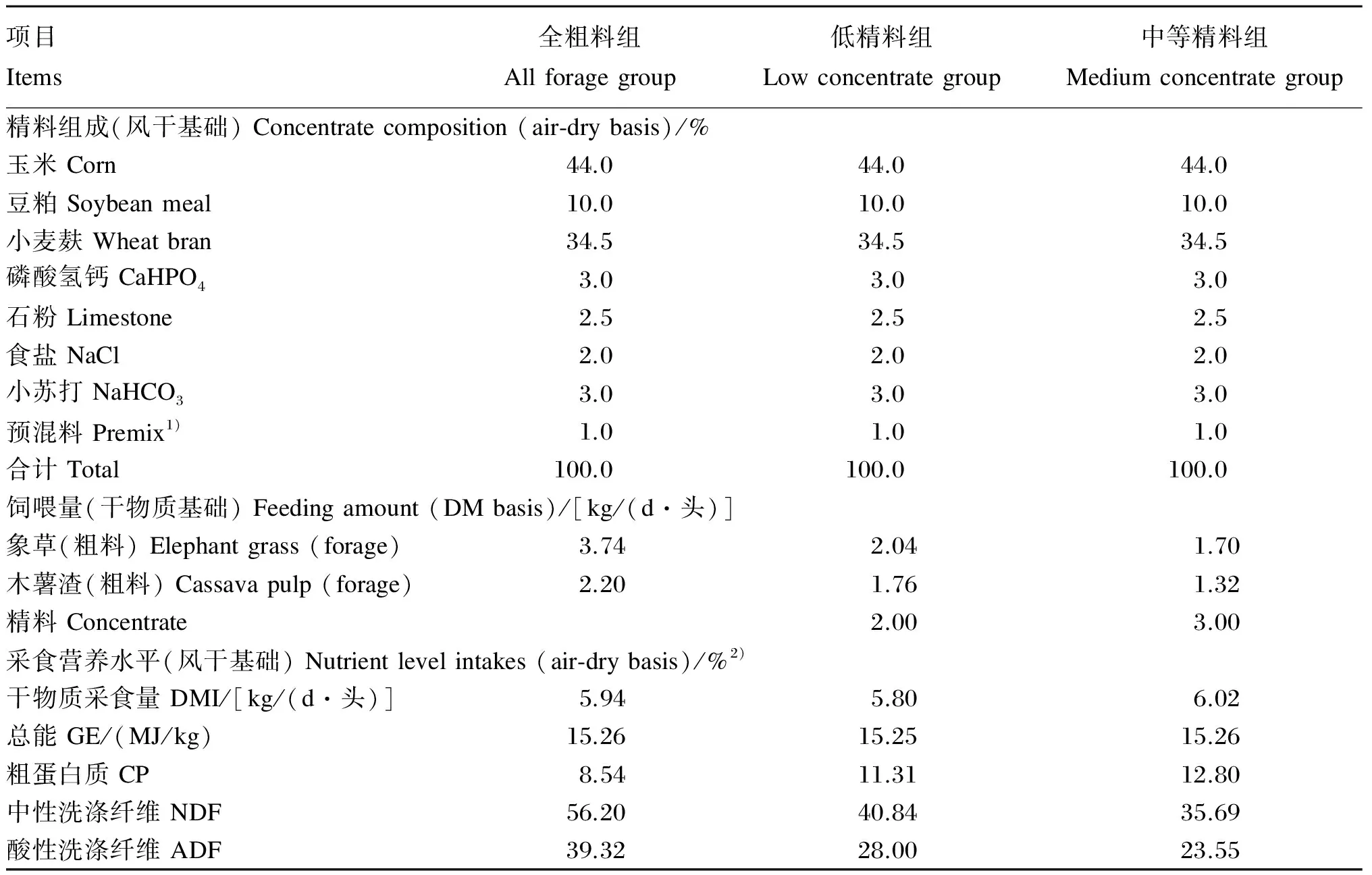
表1 精料组成、精料和粗料饲喂量及采食营养水平
1)每千克预混料含有 One kg of premix contained the following:VE 3 000 IU,VD 150 000 IU,VA 500 000 IU,Cu 1.3 g,Fe 4.0 g,Mn 3.0 g,I 80 mg,Zn 6.0 g,Co 80 mg,Se 50 mg。
2)实测值 Measured values。
1.3瘤胃液采集
正试期结束后第1天,每头水牛采集瘤胃液100 mL,以组为单位混合,通过4层纱布过滤,取
50 mL滤液冷冻干燥(Christ Alpha 1-4 LD plus, Christ Alpha, Germany),用于DNA提取测定微生物区系。
1.4瘤胃微生物区系测定
1.4.1瘤胃液DNA提取
DNA提取方法按照Rius等[8]的报道进行。具体方法为:称取25 mg冷冻干燥瘤胃液置于装有0.7 g锆珠(0.1 mm)、200 μL的20%十二烷基苯磺酸钠(SDS)、282 μL的QIAquick Buffer A和268 μL的QIAquick Buffer PB(Qiagen,德国)以及550 μL的饱和酚/氯仿/异戊醇溶液(25∶24∶1)的离心管中。封闭离心管后于小型珠磨器(Biospec Mini-Beadbeater-16,美国)上破碎2 min,然后于16 000×g(4 ℃)下离心20 min,取500 μL上清液与650 μL的Buffer PB混合后,用QIAquick PCR purification kit(Qiagen,德国)纯化混合液获得DNA样品。
1.4.2水牛瘤胃微生物区系组成的测序分析
瘤胃细菌和甲烷菌的16S rRNA基因片段扩增与处理均按照Kittelmann等[9]提供的方法进行。PCR引物由Illumina Miseq PE250测序接头、1个2碱基的连接(linker)和样品标签(barcode)组成,细菌PCR引物为515f/806r,甲烷菌PCR引物为519f/915r(表2)。PCR扩增采用Promega PCR mix进行,体系为50 μL,每个样品的2种微生物分别扩增2管。PCR扩增程序为:94 ℃预变性3 min;然后进入94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸45 s的循环,共进行30个循环。PCR扩增后,将同一样品的同一种微生物的2管PCR产物混合,电泳后切胶回收,采用胶回收试剂盒(天根生化科技有限公司)纯化;采用ND2000超微量核酸蛋白测定仪(Thermo,德国)准确测定纯化产物的DNA浓度(精确到0.1 ng/μL)后,每个样品的PCR纯化产物按照细菌∶甲烷菌=5∶1的浓度比例混合。最后,将各样品全部PCR产物混合后,送广州海基生物技术有限公司应用Miseq PE250平台进行测序分析。
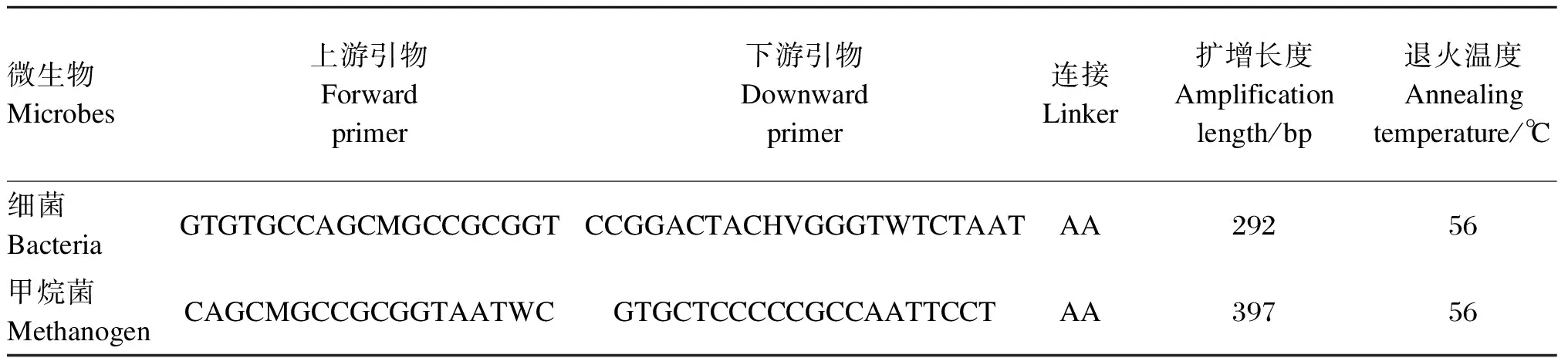
表2 瘤胃微生物区系测序分析采用的引物信息
1.4.3测序序列的聚类分析和注释
测序数据分析采用QIIME 1.5进行,经过去除接头污染、低复杂度以及低质量reads等质量控制后,对获得的clean reads进行拼接并去除引物,用于后续分析[10]。序列分类操作单元(OTU)注释和聚类分析按照与细菌和古菌97%相似度以上的原则进行。OTU聚类分析和注释通过与Ribosomal Database Project(RDP)数据库中的细菌和古菌序列比对进行,细菌聚类分析到门和科水平,甲烷菌分类到属水平。
1.5统计与分析
所有数据均采用SAS 8.02软件的GLM程序进行单因素方差分析,多重比较采用Duncan氏法,P<0.05为差异显著性判定标准。
2 结果与分析
2.1高通量测序数据质量控制与多样性分析
高通量测序数据经质量控制后,各组细菌和甲烷菌测序clean reads数、OTU数以及样品物种多样性指数如表3。水牛瘤胃内细菌测序后发现,在种水平上得到的clean reads数平均值为26 613,OTU数平均值为969,测序覆盖率为0.89~0.90;细菌香农多样性指数平均值为7.58,Chao1多样性指数平均值为1 233。除中等精料组香农指数显著高于全粗料组(P<0.05)外,组间细菌各指标均无显著差异(P>0.05)。水牛瘤胃内甲烷菌测序后发现,在种水平上得到的clean reads数平均值为8 607,OTU数平均值为136,测序覆盖率为0.74~0.93;甲烷菌香农多样性指数平均值为1.25,Chao1多样性指数平均值为237。组间甲烷菌各指标均无显著差异(P>0.05)。
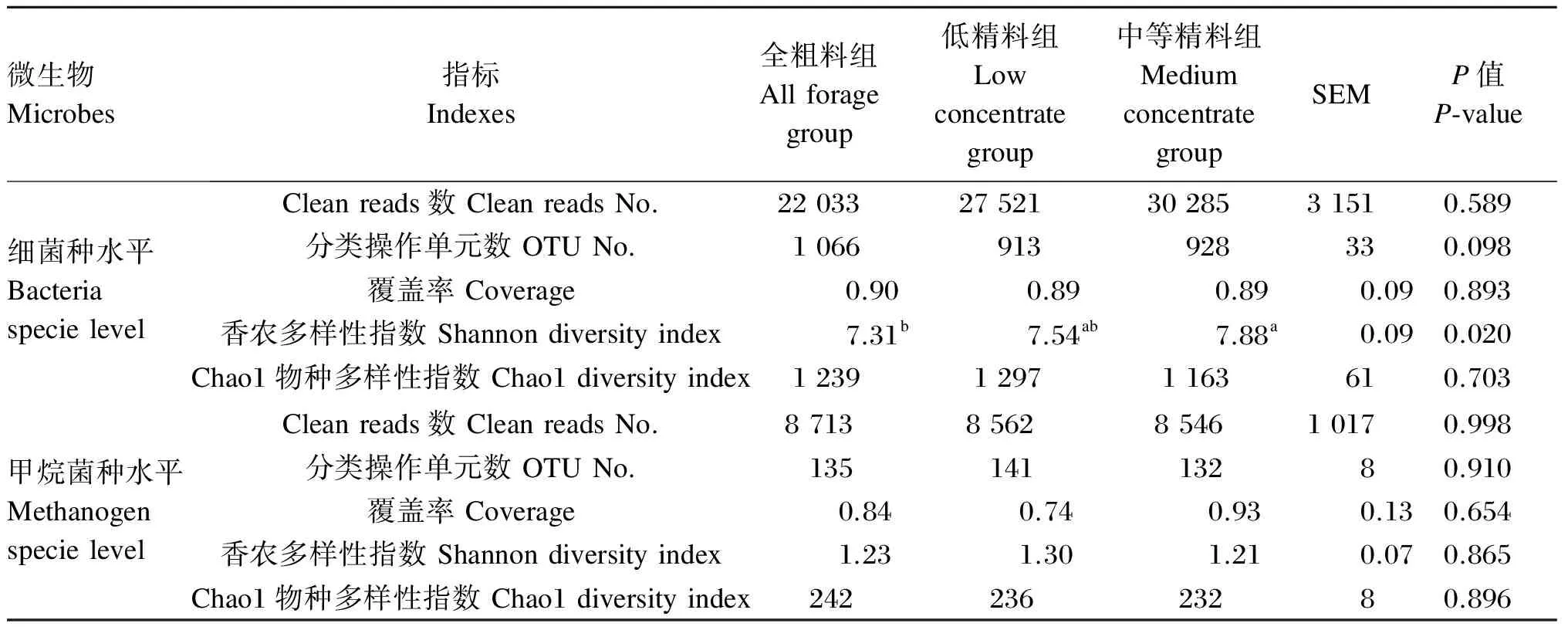
表3 瘤胃细菌和甲烷菌高通量测序质量控制与样品多样性指数
2.2饲粮精粗比对瘤胃细菌区系的影响
不同精粗比饲粮下,瘤胃细菌组成在门水平和科水平的组成见表4。水牛瘤胃细菌在门水平上主要的细菌门是拟杆菌门、厚壁菌门和变形菌门。全粗料组拟杆菌门细菌的比例显著高于中等精料组(P<0.05),黄杆菌门和SR1细菌的比例显著高于低精料组和中等精料组(P<0.05),但全粗料组厚壁菌门、疣微菌门细菌的比例显著低于中等精料组(P<0.05),放线菌门和绿弯菌门细菌的比例显著低于低精料组和中等精料组(P<0.05)。
在科水平上,瘤胃细菌以普雷沃氏科(Prevotellaceae)和黄杆菌科(Fibrobacteraceae)为主。全粗料组、低精料组普雷沃氏科细菌的比例显著高于中等精料组(P<0.05),而全粗料组和低精料组黄杆菌科、毛螺菌科、红蝽杆菌科和双歧杆菌科细菌的比例均显著低于中等精料组(P<0.05),全粗料组瘤胃球菌科细菌的比例显著低于中等精料组(P<0.05)。
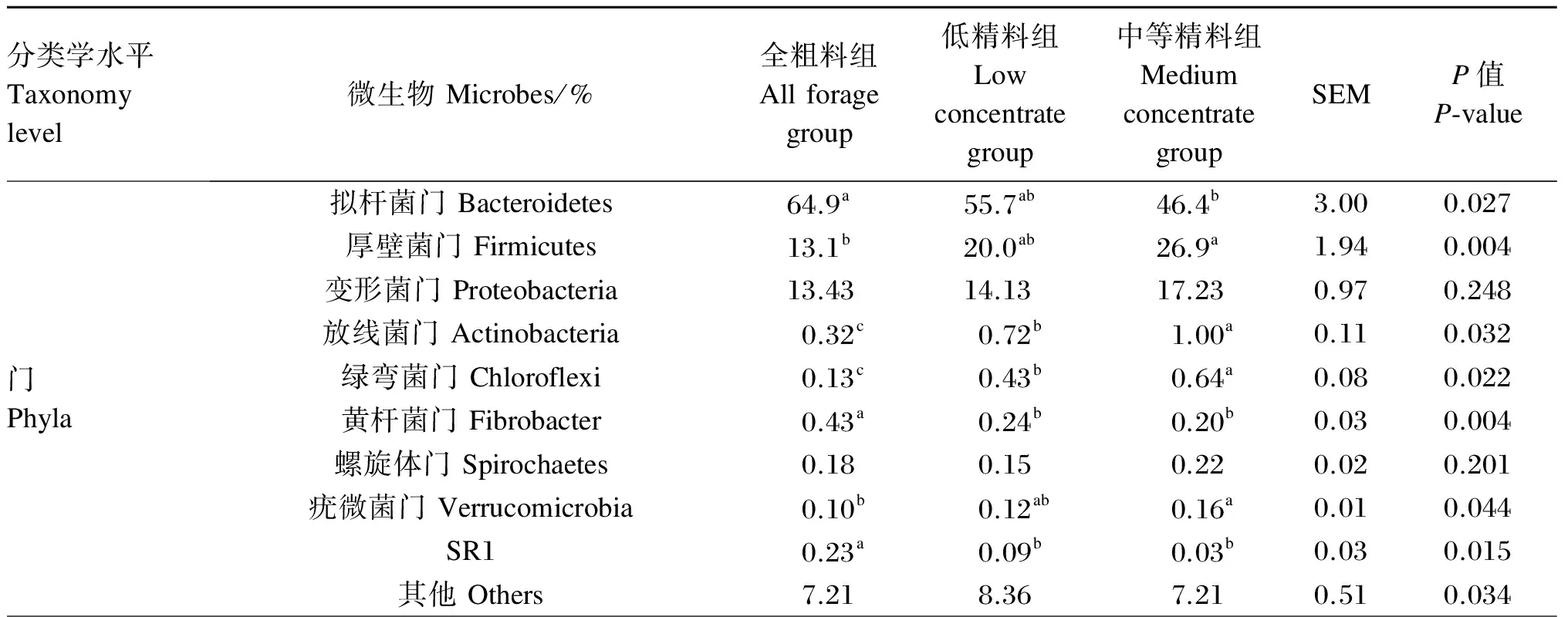
表4 瘤胃细菌门和科水平上的细菌比例(占总菌比例大于1%)

续表4分类学水平Taxonomylevel微生物Microbes/%全粗料组Allforagegroup低精料组Lowconcentrategroup中等精料组MediumconcentrategroupSEMP值P⁃value科Family普雷沃氏科Prevotellaceae31.6a25.7a15.1b2.09<0.001黄杆菌科Fibrobacteraceae8.3b11.3b20.5a5.75<0.001肠杆菌科Enterobacteriaceae7.617.699.580.570.348莫拉氏菌科Moraxellaceae4.254.335.370.420.505毛螺菌科Lachnospiraceae2.28b2.74b4.73a0.34<0.001瘤胃球菌科Ruminococcaceae2.55b3.18ab4.62a0.24<0.001韦荣球菌科Veillonellaceae0.760.860.670.050.339紫单胞菌科Porphyromonadaceae0.640.630.760.040.059丛毛单胞菌科Comamonadaceae0.600.410.260.060.005红蝽杆菌科Coriobacteriaceae0.25b0.31b0.49a0.04<0.001未分类菌科XⅢIncertaesedisXⅢ0.170.250.580.230.078双歧杆菌科Bifidobacteriaceae0.10b0.17b0.65a0.310.001
不同精粗比饲粮饲喂的各组水牛瘤胃细菌组成的柱状分布图和热图分布分别如图1和图2所示。虽然组间各样品瘤胃细菌组成相近,但不同组间瘤胃细菌组成比例仍存在差异,特别是全粗料组的普雷沃氏科、链球菌科(Streptococcaceae)和气单胞菌科(Aeromonadaceae)细菌的比例较高,而中等精料组中黄杆菌科、瘤胃球菌科、毛螺菌科、双歧杆菌科和厌氧绳菌科(Anaerolineaceae)细菌的比例较高。
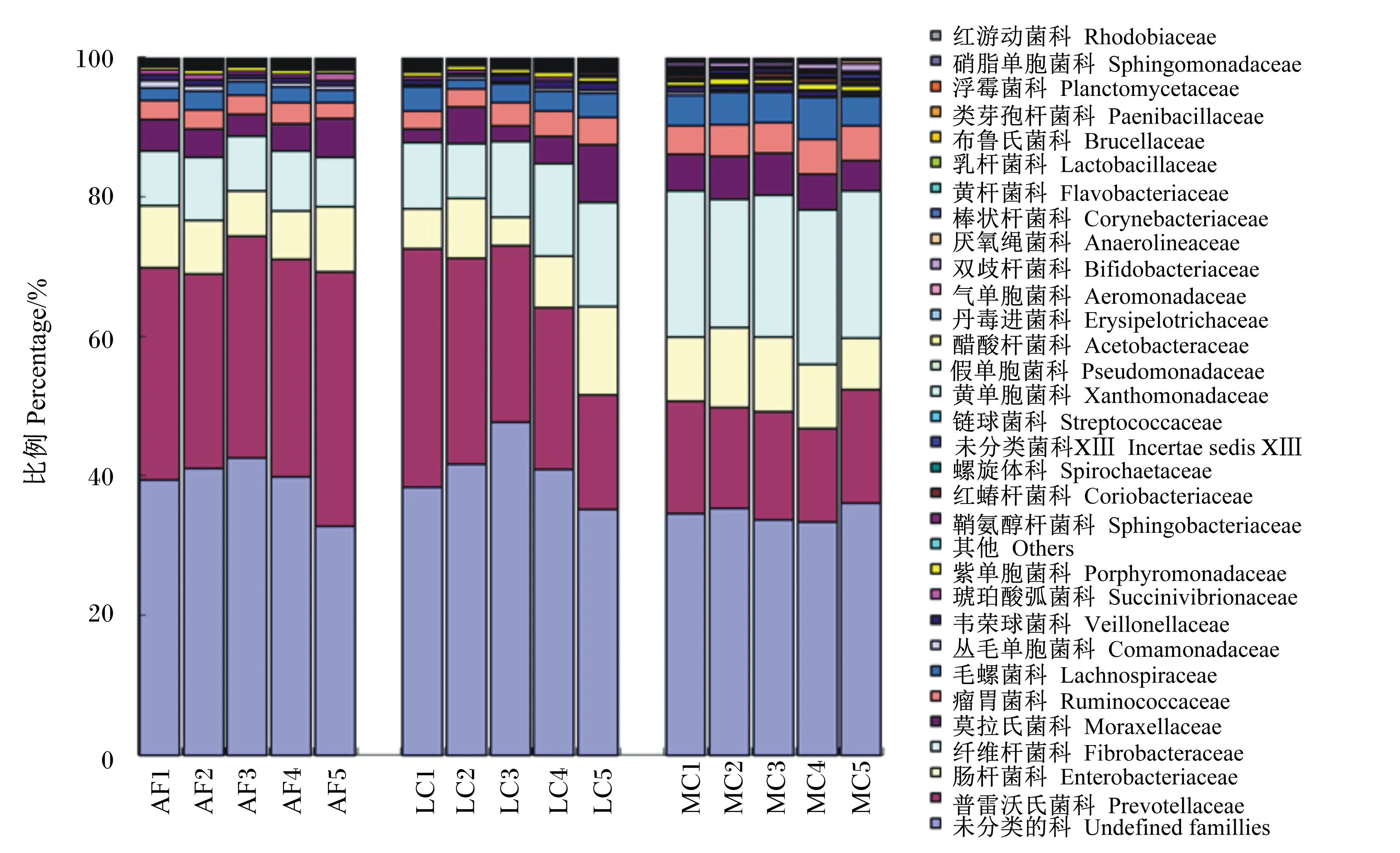
AF:全精料组 all forage group;LC:低精料组 low concentrate group;MC:中等精料组 medium concentrate group。下图同 The same as below。
图1饲粮精粗比对瘤胃细菌在科水平上组成的影响
Fig.1Effects of forage to concentrate ratio on ruminal bacteria composition at family level

图2 饲粮精粗比对瘤胃细菌在科水平上热图分布的影响
2.3饲粮精粗比对泌乳水牛瘤胃甲烷菌区系的影响
饲粮精粗比对瘤胃甲烷菌区系在属水平上组成的影响如图3所示。可鉴定到属水平的水牛瘤胃甲烷菌共6个,其中主要的甲烷菌是甲烷短杆菌属(Methanobrevibacter)、热原体属(Thermoplasma)和甲烷球菌属(Methanosphaera),这3类菌约占甲烷菌总数的95%,其中各组甲烷短杆菌属细菌的比例均在90%以上。饲粮精粗比均未影响各组甲烷菌的组成比例。

图3 饲粮精粗比对瘤胃甲烷菌在属水平上组成的影响
3 讨 论
3.1饲粮精粗比对瘤胃细菌区系的影响
饲粮精粗比是决定瘤胃发酵的主要原因之一,我们前期的研究结果表明饲粮精粗比影响了水牛瘤胃的发酵指标[5],而其调控的途径主要通过影响瘤胃微生物区系或功能。研究表明,随着饲粮精料比例的提高,纤维降解菌和真菌数量逐渐减少,当精料增加到一定比例时,瘤胃内低pH最终会抑制纤维降解菌的生长[7,11]。Kocherginskaya等[12]发现高精料饲粮瘤胃内细菌数量和种类较多,变形菌门是门水平上的主要细菌;对高粗饲粮而言,低G+C含量的革兰氏阳性菌为主要的细菌类别。Wetzels等[13]报道,与30%精料水平组相比,0和60%精料水平组瘤胃壁上的厚壁菌门和SR1含量较高。Mao等[3]研究表明,在饲喂高精料饲粮引起亚急性酸中毒的奶牛瘤胃内,变形菌门和拟杆菌门细菌的比例显著降低,而厚壁菌门和放线菌门细菌的比例则提高;在属水平上,普雷沃菌属、密螺旋体属(Treponema)、厌氧支原体属(Anaeroplasma)等属细菌的比例在高精料饲粮组中较低,但瘤胃球菌属、奇异菌属(Atopobium)和梭菌目(Clostridiales)等细菌的比例较高。本研究结果表明,精粗比为50∶50组水牛瘤胃细菌在门水平上具有较高比例的厚壁菌门、变菌门和放线菌门细菌,而全粗料组则具有较高比例的拟杆菌门、黄杆菌门和SR1细菌,该结果与其他一些关于奶牛和羊的结果相似[3,13],均表明高精料饲粮可提高瘤胃内厚壁菌门和变形菌门,降低拟杆菌门细菌的比例。在科水平上,本研究发现全粗料组普雷沃氏科细菌比例(31.6%)为最高,而中等精饲粮组黄杆菌科细菌的比例(20.5%)为最高;普雷沃氏科是拟杆菌门的主要细菌,含有较多高活性的半纤维素降解菌,广泛存在于瘤胃和动物后肠道中负责蛋白质和碳水化合物的分解[14],其在高粗饲粮中含量较高表明该科的细菌与纤维消化密切相关。然而,Pitta等[4]对水牛饲喂不同精粗比饲粮研究结果表明,全粗料饲喂组水牛瘤胃内瘤胃球菌属和黄杆菌门细菌数量较高,而在50∶50精粗比饲粮组,普雷沃氏属是细菌数量最多的属,与本研究结果并不一致,其原因可能本试验与该研究的动物试验设计差异有关,本试验3组水牛为同时饲喂,而该研究则为同组水牛顺序饲喂,即先喂全粗料饲粮再喂低精料饲粮,最后喂中等精料饲粮;此外,也有可能与引物的差异有关,Pitta等[4]的研究采用不同的引物得到了不同的细菌区系组成,表明引物的合理选择对区系的影响较大。本研究发现精粗比为50∶50组瘤胃内细菌的香农多样性指数显著高于全粗料组,表明饲喂适宜的精料有提高瘤胃内细菌多样性的趋势,该结果与孙云章[15]的研究表明饲粮由全粗料转为精粗比为30∶70时,山羊瘤胃细菌区系多样性增加一致。此外,Kocherginskaya等[12]也研究表明,高精料饲粮饲喂下山羊瘤胃内细菌数量和种类较多。本研究中,精粗比为50∶50组瘤胃内细菌多样性高于全粗料饲粮组的原因可能与精粗比为50∶50组瘤胃内分解淀粉和纤维的细菌共存,而全粗料饲粮组则以高效分解纤维的细菌为主,种类较少有关,因此给水牛饲喂精粗平衡的饲粮有利于维持瘤胃细菌的多样性和稳定性。
3.2饲粮精粗比对瘤胃甲烷菌区系的影响
饲粮精粗比会显著影响反刍动物瘤胃甲烷产量,高粗饲粮甲烷产量和甲烷菌数量均显著高于高精料饲粮,但饲粮精粗比对甲烷菌区系的研究报道还较少。现有研究表明,反刍动物瘤胃甲烷菌以甲烷短杆菌属为主,其次是甲烷球菌属和热原体属[16];较多印度学者的研究表明,水牛瘤胃内甲烷菌以甲烷微菌属(Methanomicrobium)细菌为主,表现出不同于其他反刍动物不同的区系[17-19]。本研究表明,水牛瘤胃内甲烷菌共7个属,其中甲烷短杆菌属在各组中的占比均在90%以上,与Franzolin等[20]报道水牛瘤胃甲烷菌以甲烷短杆菌属为主一致,且与反刍动物的瘤胃甲烷菌以甲烷杆菌属为主一致。本研究结果不同于其他印度学者研究结果的原因可能与水牛所处的地理位置或品种有关[21],另一重要原因可能与引物的差异有关,研究表明不同甲烷菌引物会产生不同的菌群组成[22]。本研究发现,水牛饲粮精粗比并未影响甲烷短杆菌属在水牛瘤胃内的主导地位,因此饲粮精粗比对瘤胃甲烷产量的影响可能主要归因于甲烷菌数量的变化,但饲粮精粗比的改变是否影响了不同种的甲烷短杆菌组成尚有待进一步研究。有研究表明,反刍动物瘤胃内甲烷菌组成有其个体特异性[23],并不容易随外界环境和饲粮组成的改变的而变化。
4 结 论
全粗料饲粮有提高瘤胃内纤维降解相关细菌比例的趋势,却降低了瘤胃细菌的多样性;饲粮精粗比的差异对瘤胃甲烷菌在属水平上的组成并无显著影响。
[1]娜仁花,董红敏,陈永杏,等.日粮精粗比对瘤胃发酵特性的影响[J].中国畜牧杂志,2011,47(9):49-54.
[2]PETRI R M,FORSTER R J,YANG W,et al.Characterization of rumen bacterial diversity and fermentation parameters in concentrate fed cattle with and without forage[J].Journal of Applied Microbiology,2012,112(6):1152-1162.
[3]MAO S Y,ZHANG R Y,WANG D S,et al.Impact of subacute ruminal acidosis (SARA) adaptation on rumen microbiota in dairy cattle using pyrosequencing[J].Anaerobe,2013,24:12-19.
[4]PITTA D W,KUMAR S,VEICCHARELLI B,et al.Bacterial diversity associated with feeding dry forage at different dietary concentrations in the rumen contents of Mehshana buffalo (Bubalusbubalis) using 16S pyrotags[J].Anaerobe,2014,25:31-41.
[5]李萍,梁辛,韦升菊,等.饲粮精粗比对后备母水牛瘤胃发酵参数和纤维降解菌数的影响[J].畜牧与兽医,2015,47(7):36-40.
[6]易学武,张石蕊.奶牛日粮精粗比应用研究进展[J].中国草食动物,2006,26(3):51-55.
[7]孙云章,毛胜勇,姚文,等.不同精粗比底物下瘤胃真菌和纤维降解细菌共培养发酵特性及菌群变化[J].微生物学报,2006,46(3):422-426.
[8]RIUS A G,KITTELMANN S,MACDONALD K A,et al.Nitrogen metabolism and rumen microbial enumeration in lactating cows with divergent residual feed intake fed high-digestibility pasture[J].Journal of Dairy Science,2012,95(9):5024-5034.
[9]KITTELMANN S,SEEDORF H,WALTERS W A,et al.Simultaneous amplicon sequencing to explore co-occurrence patterns of bacterial,archaeal and eukaryotic microorganisms in rumen microbial communities[J].PLoS One,2013,8(2):e47879.
[10]CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods,2010,7(5):335-336.
[11]王海荣.不同日粮精粗比及氮源对绵羊瘤胃纤维降解菌群和纤维物质降解的影响[D].博士学位论文.呼和浩特:内蒙古农业大学,2006:2-3.
[12]KOCHERGINSKAYA S A, AMINOV R I,WHITE B A.Analysis of the rumen bacterial diversity under two different diet conditions using denaturing gradient gel electrophoresis,random sequencing,and statistical ecology approaches[J].Anaerobe,2001,7(3):119-134.
[13]WETZELS S U,MANN E,METZLER-ZEBELI B U,et al.Pyrosequencing reveals shifts in the bacterial epimural community relative to dietary concentrate amount in goats[J].Journal of Dairy Science,2015,98(8):5572-5587.
[14]MATSUI H,OGATA K,TAJIMA K,et a1.Phenotypic characterization of polysaccharidases produced by fourPrevotellatype strains[J].Current Microbiology,2000,41(1):45-49.
[15]孙云章.不同底物下瘤胃微生物的发酵特性及细菌菌群变化的分子描述[D].博士学位论文.南京:南京农业大学,2005:6-9.
[16]ST-PIERRE B,WRIGHT A D G.Diversity of gut methanogens in herbivorous animals[J].Animal,2013,7(Suppl.1):49-56.
[17]CHAUDHARY P P,SIROHI S K.Dominance ofMethanomicrobiumphylotype in methanogen population present in Murrah buffaloes (Bubalusbubalis)[J].Letters in Applied Microbiology,2009,49(2):274-277.
[18]SINGH K M,PANDYA P R,PARNERKAR S,et al.Molecular identification of methanogenic archaea from Surti buffaloes (Bubalusbubalis),reveals more hydrogenotrophic methanogens phylotypes[J].Brazilian Journal of Microbiology,2011,42(1):132-139.
[19]SINGH K M,AHIR V B,TRIPATHI A K,et al.Metagenomic analysis of Surti buffalo (Bubalusbubalis) rumen:a preliminary study[J].Molecular Biology Reports,2012,39(4):4841-4848.
[20]FRANZOLIN R,ST-PIERRE B,NORTHWOOD K,et al.Analysis of rumen methanogen diversity in water buffaloes (Bubalusbubalis) under three different diets[J].Microbial Ecology,2012,64(1):131-139.
[21]WRIGHT A D G,AUCKLAND C H,LYNN D H.Molecular diversity of methanogens in feedlot cattle from Ontario and Prince Edward Island,Canada[J].Applied and Environmental Microbiology,2007,73(13):4206-42107.
[22]CHENG Y F,MAO S Y,LIU J X,et al.Molecular diversity analysis of rumen methanogenicArchaeafrom goat in eastern China by DGGE methods using different primer pairs[J].Letters in Applied Microbiology,2009,48(5):585-592.
[23]ZHOU M,HÜNERBERG M,BEAUCHEMIN K A,et al.Individuality of ruminal methanogen/protozoa populations in beef cattle fed diets containing dried distillers’ grain with solubles[J].Acta Agriculturae Scandinavica,Section A:Animal Science,2012,62(4):273-288.
(责任编辑王智航)
Dietary Forage to Concentrate Ratio Affects Ruminal Bacterial and Methanogen Community Composition of Water Buffaloes
LIN Bo1LIANG Xin2LI Lili2WEI Shengju2LI Ping1ZOU Caixia1,2*
(1. College of Animal Science, Guangxi University, Nanning 530004, China; 2. Key Laboratory of Buffalo Genetics, Breeding and Reproduction of Ministry of Agriculture, Key Laboratory of Buffalo Genetics, Breeding and Reproduction of Guangxi, Buffalo Research Institute, Chinese Academy of Agricultural Sciences, Nanning 530001, China)
This study was conducted to study the effects of dietary forage to concentrate ratio on ruminal bacterial and methanogen community composition of water buffaloes. Fifteen healthy similar hybrid water buffaloes were selected and divided into three groups with 5 heads per group according to body weight and feed intake. The three groups were fed diets with concentrate to forage ratio at 0∶100 (all forage group), 35∶65 (low concentrate group) and 50∶50 (medium concentrate group), respectively. The total experimental period was 40 days, including 10 days of advanced experiment feeding period. On day 1 after the experiment, rumen fluid sample was taken from rumen through mouth, and ruminal fluid DNA was extracted to research ruminal bacterial and methanogen community by Illumina Miseq 250PE. The results showed as follows: 1) at phyla level, ruminal bacterial community of buffaloes was dominated by Bacteroidetes (45% to 65%) and Firmicutes (13% to 27%) and Proteinobacter (13% to 18%); compared with low concentrate group and medium concentrate group, all forage group increased the proportions of Bacteroidetes, Fibrobacter and SR1, but decreased the proportions of Firmicutes, Verrucomicrobia, Actinobacteria and Chloroflexi; at the family level, the dominant bacteria were Prevotellaceae (15% to 32%) and Fibrobacteraceae (8% to 21%); compared with medium concentrate group, all forage group and low concentrate group increased the proportions of Prevotellaceae, while decreased the proportions of Fibrobacteraceae, Lachnospiraceae, Ruminococcaceae, Coriobacteriaceae and Bifidobacteriaceae; additionally, the bacterial community in medium concentrate group was more diverse than that in all forage group. 2) Ruminal methanogen community of water buffaloes was dominated byMethanobrevibacter(over 90%), followed byThermoplasma; dietary concentrate to forage ratio had no influence on ruminal methanogen community of water buffaloes. In conclusion, bacteria related with fiber digestion tends to be increased by all forge diet, but ruminal bacterial diversity is decreased by all forage diet; there is no influence of concentrate to forage ratio on ruminal methanogen composition of water buffaloes at genus level.[ChineseJournalofAnimalNutrition, 2016, 28(10):3101-3109]
water buffalo; concentrate to forage ratio; rumen; bacteria community; methanogen community
, professor, E-mail: caixiazou2002@hotmail.com
10.3969/j.issn.1006-267x.2016.10.012
2016-03-28
国家自然科学基金(31160470);广西自然科学基金(2013GXNSFBA019114)
林波(1983—),男,四川雅安人,副研究员,博士,主要从事反刍动物营养研究。E-mail: linbobri@foxmail.com
邹彩霞,研究员,硕士生导师,E-mail: caixiazou2002@hotmail.com
S823
A
1006-267X(2016)10-3101-09
