益生菌生物被膜的研究进展
2016-11-14李平兰
刘 蕾,刘 义,李平兰*
(中国农业大学食品科学与营养工程学院,北京食品营养与人类健康高精尖创新中心,北京 100083)
益生菌生物被膜的研究进展
刘 蕾,刘 义,李平兰*
(中国农业大学食品科学与营养工程学院,北京食品营养与人类健康高精尖创新中心,北京 100083)
益生菌是指当以足够数量存在时可对机体健康产生有益作用的活的微生物。近年来,由于其对机体具有保健和治疗作用,因而受到越来越多的关注。生物被膜是细菌分泌的多糖,纤维蛋白和脂蛋白等物质将细菌自身包裹其中,吸附在生物材料或机体腔道等表面而形成的膜样复合物,是自然状态下许多细菌所选择的生存方式。但是关于益生菌生物被膜的研究还比较少,且尚处于起步阶段。本综述围绕益生菌生物被膜的形成、阶段特征、影响因素、优势及调控机制等展开分析,并指出益生菌生物被膜的相关研究将会是益生菌研究领域的一个重要发展方向。
益生菌;生物被膜;影响因素;调控机制
根据世界卫生组织(World Health Organization,WHO)对益生菌的定义,益生菌是指当以足够数量存在时可对机体健康产生有益作用的活的微生物,包括细菌和真菌。长久以来,传统观点一直认为益生菌是一种食品补充剂。但是近年来的研究结果表明益生菌还具有许多治疗作用,如用于治疗皮肤炎、哮喘、节段性回肠炎、肝源性脑病、鼻炎、结肠直肠癌以及胃肠道感染等[1-4]。在英国,大约有70%的医生利用益生菌作为处方药物对胃肠道疾病的患者进行治疗[5]。目前,益生菌不再被简单的认为仅仅是一种食品补充剂,其营养保健及治疗作用也得到越来越多的关注。因此,如何更好地发挥益生菌的益生作用引起了研究人员的广泛关注。
生物被膜是细菌利用分泌的多糖,纤维蛋白和脂蛋白等物质将其自身包裹其中,并吸附在生物材料或机体腔道等表面而形成的膜样复合物[6-7],是大多数细菌在自然状态下的生长方式。由于生物被膜的形成与细菌的毒力、耐药性及群体性行为密切相关,所以关于有害微生物生物被膜的研究非常多,也取得了很大的进展[8-11]。但与此同时,关于有益微生物生物被膜的研究却相当匮乏,尚处于起步阶段。有研究报道称生物被膜状态下的益生菌比浮游状态下的益生菌具有更显著的免疫调节作用[12]。也有相关报道称与常规益生菌微制剂相比,高密度生物被膜状态下益生菌制备的微制剂具有更好的抗冷冻干燥能力、耐热性和耐酸性[13]。因此,对益生菌生物被膜展开相关的研究,不仅对阐明益生菌益生机理具有重要意义,而且对研发、生产高效的益生菌微制剂具有指导作用。本文概括总结了益生菌生物被膜的形成过程、阶段特征和影响因素,益生菌生物被膜状态时的优势,以及益生菌生物被膜形成的调控机制。
1 益生菌生物被膜的形成过程、阶段特征及影响因素
近数十年来,研究人员发现生物被膜不是细菌个体的简单堆积,而是细菌菌体及胞外分泌物如多糖、纤维蛋白、脂蛋白等相互作用而形成的具有复杂结构的膜样物质[14-16]。生物被膜形成过程中的细菌会启动一系列基因的表达,并且与浮游状态时的基因表达方式显著不同[17]。在自然环境中大多数细菌都以生物被膜的状态生存,以传统的培养方式培养益生菌并不能模拟其真实的生长状态,故在此基础上对其益生功能展开研究也具有一定的缺陷。因此,阐明益生菌生物被膜的形成过程、特征及影响因素等有利于进一步揭示益生菌的益生功效。
与大多数细菌一样,益生菌的生物被膜形成及发展过程主要分为以下5 个阶段:第一阶段是细菌细胞对表面基质的初始附着;第二阶段是附着的细菌分泌胞外多聚物从而促进细菌对表面基质进行不可逆地附着;第三阶段是生物被膜结构形态的初步形成和发展;第四阶段是生物被膜结构形态的成熟;第五阶段是生物被膜中个体细胞的脱落。脱落的个体细胞可以再次附着,并形成一个新的生物被膜。通过这5 个阶段,细菌生物被膜完成了附着、形成、成熟、老化脱落、再重新附着的循环过程(图1)[18]。在生物被膜形成的各个阶段,细菌不同的基因转录翻译成不同的蛋白质,参与生物被膜的形成。Sauer等[17]发现在Pseudomonas aeruginosa生物被膜形成的各阶段中有35%(大约有525 种蛋白)的蛋白表现出了差异。而且,浮游状态下的菌体细胞与生物被膜脱落期的细胞更为相似,但是与生物被膜成熟时期的菌体细胞相比,二者有超过800 个蛋白(占所有蛋白的50%都表现出了6 倍以上的差异)[17]。此外,研究表明益生菌可以抑制或干扰致病菌生物被膜的形成,如益生菌Lactobacillus rhamnosus GR-1和Lactobacillus reueri RC-14可以完全抑制Candida glabrata形成生物被膜[19]。Vuotto等[20]报道可以利用益生菌来对抗生物被膜相关的感染性疾病,如蛀齿、牙周病、口臭、泌尿生殖道及伤口感染。然而对于益生菌自身而言,在其生物被膜形成的各阶段,哪些基因和蛋白质发挥了关键作用还尚未阐明。
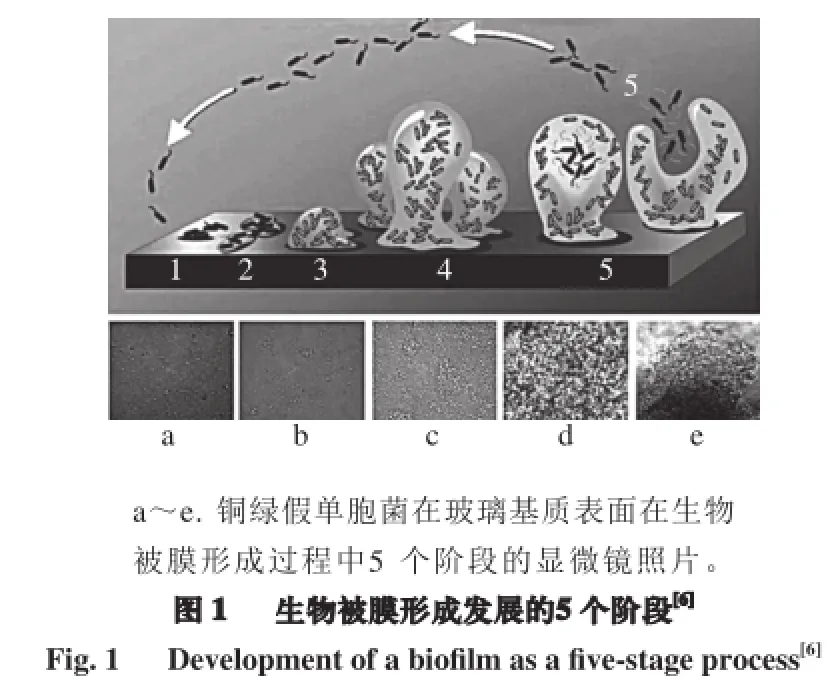
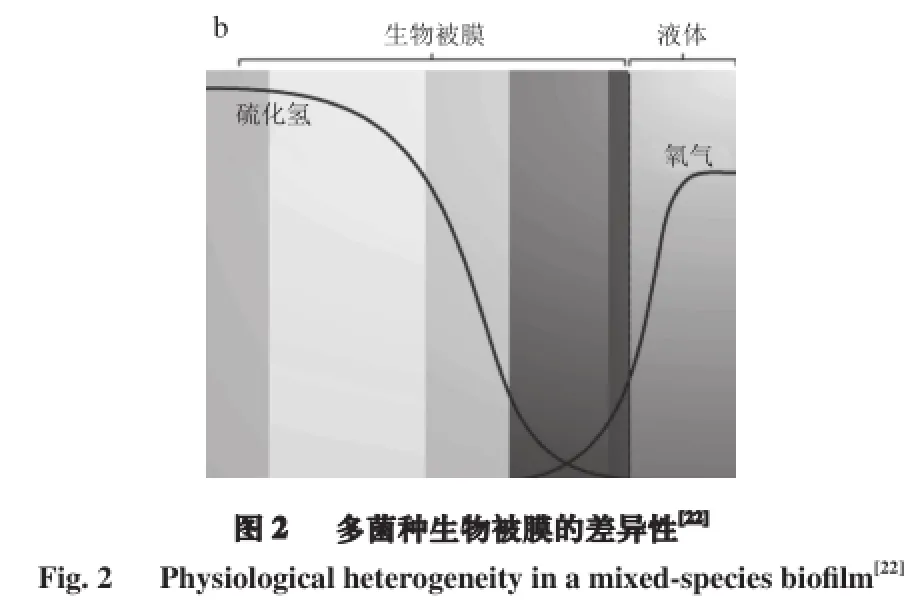
细菌生物被膜是高度结构化的,在其形成发展的过程中发生了一系列形态学的变化。在最初的阶段,附着在表面基质的细菌个体细胞被其分泌的胞外多聚物包裹,开启了生物被膜的形成,其中许多细菌还可以通过菌毛的运动来自由地移动。这些初始附着的细菌还没有开始进行生物被膜形成过程中的分化阶段,因此很多细菌还可以离开基质表面重新回到之前的浮游状态。之后细菌开始启动一系列基因的表达,分泌胞外多糖等物质,使细菌不可逆地黏附到基质表面。有研究报道称Pseudomonas aeruginosa在初始附着的15 min内,藻酸盐基因表达迅速上调,产生大量藻酸盐,促进生物被膜的形成。在生物被膜的成熟阶段,细菌生物被膜的复杂结构及水通道结构逐渐形成,位于底层的细菌也开始进行重新分布。接下来,一些细菌小菌落可能从基质表面脱落,重新回到浮游状态,同时留下一些空洞形成水通道。除此之外,小菌落可能在没有明显的外界干扰时整体从生物被膜中脱落。在整个生物被膜中,这些现象并不是同步发生的,因此在同一时间生物被膜中的小区域常常处在不同的发展阶段(图2)[21]。益生菌中包含多数兼性厌氧菌和严格厌氧菌,其生物被膜的结构是怎样的以及在其形成过程中哪些基因发挥重要作用还要需要进一步研究。

影响益生菌生物被膜形成的因素众多,除去自身基因调控的因素外,外界环境的诸多因素都会影响其生物被膜的形成,例如菌体附着表面的材质、水流的冲刷作用、pH值、过高的渗透压、胆盐的存在、黏液素的存在以及非消化性多糖的存在等,都会明显干扰益生菌生物被膜的形成的发展[23]。有研究报道称培养基中营养物质的缺乏及限制碳源营养物质供给都会促进Lactobacillus rhamnosus GG生物被膜的形成,黏液素等物质可能促进Lactobacillus rhamnosus GG附着到基质表面,与此同时生物被膜状态下的Lactobacillus rhamnosus GG比悬液培养时对低pH值的环境更为敏感,而且研究结果表明胞外多糖编码基因wzb,脂磷壁酸编码基因dltD,中心代谢参与基因luxS与Lactobacillus rhamnosus GG生物被膜的形成有关[23]。此外,Brink等[24]发现当表面液体流速降低时,葡萄糖代谢产生的3-羟基-丁酮减少,NADH的消耗量减少,乳酸菌可以产生的更多的胞外聚合物,这有利于生物被膜的形成。Slizova等[25]研究了分离自猪肠道内的罗伊氏乳杆菌Lactobacillus reuteri L2/6生物被膜的形成条件和影响因素,发现培养基中吐温-80及糖的含量和种类在生物被膜形成过程中发挥重要作用。因此,益生菌生物被膜的形成受到多种因素的影响,通过对外界因素的控制,可以提高益生菌的生物形成量,对实际生产具有指导意义。
2 益生菌生物被膜生长状态的优势
自然环境中大多数的细菌都选择以生物被膜的状态生存,这是因为无论是单菌种形成的生物被膜,还是多菌种形成的更为复杂的生物被膜,对细菌本身而言都具有浮游状态所不可比拟的优势。概括地说,像大多数细菌一样,益生菌生物被膜也具有以下几个优势。首先,形成生物被膜的细菌在生理功能上像一个整体般发挥作用。在牛的瘤胃中,各种环境和细菌形成一个独特的小生态系统,多种细菌共生,它们常常附着在纤维素等表面,形成复杂的多菌种生物被膜,共同协调作用对营养物质进行分解代谢[26]。其次,生物被膜中的细菌在行为表现上像一个整体起到相互协调的作用。研究发现黏细菌在营养物质充足的条件下常常滑动至营养物质浓度高的地区,而在干燥或营养物质匮乏的条件下个体细胞分化形成包囊,整个生物被膜形成更为复杂的结构[27]。当然,生物被膜高度的结构化也为其中的细菌个体提供了有利的生存环境。位于生物被膜不同位置的细菌个体接触不同的微环境,其生长状态不同,但是高度的结构化使得不同位置的细菌都能够接触到营养物质,并且采取与所处微环境相适宜的方式生存,从整体上保障整个细菌群体的生存[21]。此外,研究发现非致病性细菌的生物被膜形成对于其在体内生态系统中稳定存在发挥重要作用,而且可以抑制致病菌的生长和黏附[28-29]。
目前有研究报道发现,生物被膜的形成有助于乳杆菌发挥免疫调节作用,Rieu等[12]将Lactobacillus casei ATCC334生物被膜和浮游状态下的培养上清处理人巨噬细胞分化的单核细胞THP-1细胞,发现只有生物被膜状态下的上清可以抑制肿瘤坏死因子-α(tumor necrosis factor-α,TNF-α)的产生和核转录因子-κB(nuclear factor-κB,NF-κB)通路的激活。此外,Cheow等[30]将形成高密度生物被膜的Lactobacills rhamnosus制备成微胶囊,其抗冷冻干燥的能力提升了40 倍,而且其耐热性能也显著提高。因此,生物被膜中的益生菌通过其生理和行为上的共同调节,表现出其浮游状态下所不具备的优势(图3)。由于目前对于益生菌生物被膜的研究还处于起步阶段,因此相关研究报道都还很少。生物被膜状态下益生菌所具备的优势还有待进一步阐明。

3 益生菌生物被膜形成的调控
益生菌生物被膜形成的调控分为两个方面,一是益生菌菌体初始对表面基质的感知机制,二是形成生物被膜后的调控机制。由于不同的培养材料材质可以直接影响细菌生物被膜形成的多少和有无,所以细菌如何识别表面基质并作出生理和行为上的调控是非常重要的。对于细菌而言,识别表面基质并快速地从浮游生存状态转变到生物被膜生存状态是一个巨大的挑战。其中,Guttenplan等[31]发现在Bacillus subtilis的生物被膜形成过程中,EpsE蛋白在菌体鞭毛上形成簇状结构,从而阻碍菌体的运动。该EpsE蛋白序列与糖基转移酶家族酶类序列相似,而且其保守活性基团负责胞外多糖(exopolysaccharides,EPS)的合成[31]。本实验室通过对动物双歧杆菌RH、植物乳杆菌L31-1和类植物乳杆菌L-ZS9全基因组进行测序,发现三者都具有胞外多糖EPS合成基因,其中动物双歧杆菌RH的EPS基因簇包括42 个基因,其中包括10 个糖基转移酶编码基因[32]。然而,EPS合成相关基因在益生菌生物被膜形成过程中的作用还有待进一步研究。同时有研究发现这种对基质表面的应答反应有可能是通过cyclic-di-GMP信号转导途径来实现的,而且该信号转导途径也参与调控胞外多糖的产生。胞外多糖不仅仅是生物被膜结构的填充物质,同时也具有正反馈调节机制,产生的胞外多糖可以作为信号刺激菌体产生更多的胞外多糖,从而形成适宜厚度的生物被膜[33]。然而,Kolddkin-Gal等[34]报道称细菌的生物被膜也具有一定的生命周期,当营养物质匮乏和有害代谢产物累积时,细菌自身产生的D型氨基酸通过引起生物被膜基质中蛋白质组分的流失从而调控了生物被膜的脱落。此外,研究人员通过动态光散射和扫描电子显微镜实验发现去甲基咪可以直接并特异性地与胞外多糖互作,因此D型氨基酸和去甲基咪共同作用可以破坏成熟的生物被膜[34]。对于益生菌的生物被膜,D型氨基酸和去甲基咪是否发挥同样的作用还未阐明。
在生物被膜的逐渐形成和发展过程中,群体感应系统和双组分调控系统发挥着重要的调控作用[35]。益生菌中的乳杆菌双歧杆菌等都属于革兰氏阳性菌,其种内交流的主要信号分子是自诱导肽(autoinducing peptides,AIPs)。在金黄色葡萄球菌中,适宜水平的AIP与ArgC结合,导致非编码的小RNA(small RNA,sRNA)即RNA-Ⅲ的转录表达,从而下调一系列基因的表达,包括对生物被膜形成所必须的黏附素基因[36]。对于益生菌,本实验室通过对乳杆菌和双歧杆菌等全基因组序列的分析发现,它们的基因组都存在产生种内群体感应系统的相关基因,如类植物乳杆菌L-ZS9的基因组序列显示L-ZS9具有3 个组分群体感应调节系统基因,包括两个应答调节子和一个组氨酸激酶编码基因。而且,编码细菌素的ATP结合盒式蛋白(ATP-bindingcassette transporter,ABC)、细菌素免疫蛋白以及细菌素前体肽PlnE、PlnJ和PlnK等的基因都与群体感应调节基因相邻,这提示我们细菌素的产生可能受到群体感应系统的调控。在自然状态或活体肠道中,不同的细菌总是以生物被膜的形态共存,而且益生菌常常通过分泌细菌素来抑制有害病原体的生长,因此群体感应系统、细菌素的产生及生物被膜形成之间的关系值得研究人员进一步研究。此外,本实验室曾鉴定出维氏气单胞菌的1型信号分子的种类。为了进一步明确益生菌群体感应信号分子的种类和作用,需要研究人员更深入的分析。而且AIP如何调控基因表达从而调节生物被膜的形成还没有阐明,需要进一步的研究。
与此同时,在革兰氏阳性菌和革兰氏阴性菌中都存在种间群体感应系统,其产生的信号分子是2型自体诱导物AI-2,其前体物质是4,5-羟基-2,3-戊二酮(4,5-dihydroxy-2,3-pentanedion,DPD),DPD是S-腺苷甲硫氨酸(S-adenosylmethionine,SAM)代谢的副产物。其中SAM是主要的细胞甲基化供体,广泛存在于各种细菌、真菌及多细胞生物中,不同的物种具有不同的SAM代谢途径。SAM将甲基基团转移给多种不同的底物,生成了具有毒性的产物S-腺苷同型半胱氨酸(S-adenosylhomocysteine,SAH),通过甲硫腺苷核苷酶MTAN/Pfs和LuxS 2 个蛋白依次转化为S-核糖同型半胱氨酸(S-ribosylhomocysteine,SRH)和同型半胱氨酸,同时生成AI-2的前体物质DPD。DPD是一种高活性分子,它经过重排及一系列的附加反应生成不同的DPD衍生物,可以作为信号分子被不同的细菌识别,即AI-2。不同的细菌产生的AI-2结构类似,因此可以实现不同细菌之间信号的交流[37-39]。由于在自然环境中,细菌常常以多菌种共存的状态生存,故自然状态下也常常是多菌种共同形成的复杂的生物被膜,因此种间群体感应系统可能发挥着重要的作用[40-41]。本实验室通过对多种乳杆菌和双歧杆菌的全基因组序列分析,发现它们的基因组中都存在合成AI-2的关键基因luxS和pfs/mtan,其中乳杆菌的luxS基因全长477 bp,pfs/mtan基因全长693 bp,双歧杆菌luxS基因全长501 bp,pfs/mtan基因全长693 bp,乳杆菌和双歧杆菌种内luxS基因和pfs基因同源性较高,但是二者之间的差异性较大。Sun Zhongke等[42]研究发现已有的双歧杆菌基因组序列显示所有的双歧杆菌都具有luxS基因,而且其LuxS蛋白的氨基酸序列与哈维氏弧菌的LuxS蛋白相似度很高。但是,研究人员指出传统培养方法培养与MRS肉汤培养基的双歧杆菌的上清中并未检测到AI-2的活性,高浓度的葡萄糖、较低的pH值都会抑制AI-2的活性。同时,过表达luxS基因的双歧杆菌重组菌株及外源添加AI-2都使其生物被膜形成能力提高。除了群体感应系统,细菌常常通过双组份系统对外界信号和刺激进行感知和应答[43]。益生菌对肠黏膜的黏附能力是其在人体胃肠道内定植的先决条件。Uchida等[44]发现嗜酸乳杆菌利用BIACORE生物传感器可识别肠黏膜上的A型血抗原从而与其结合。短乳杆菌ATCC8287的SrtA表面蛋白可参与调节菌株对人肠道上皮细胞系和纤连蛋白的黏附[45]。对于植物乳杆菌而言,其表面有多种蛋白成分,单独任何一种蛋白都对菌株体外黏附上皮细胞的能力有促进作用[46]。张莉[47]曾分析过植物乳杆菌C88的黏附机理,发现其甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)在黏附过程中发挥重要作用,GAPDH重组蛋白能够实现黏附在Caco-2单层细胞的表面、形成占位,从而阻碍了植物乳杆菌C88、大肠杆菌、金黄色葡萄球菌、鼠伤寒沙门氏菌和福氏志贺氏菌对Caco-2细胞的黏附。但是对于大多数益生菌而言,黏附与生物被膜之间的关系及生物被膜的调控机制都还未清楚阐明,本实验室在该领域进行相关研究,力图揭示益生菌生物被膜调控的相关机制。
4 结 语
国内外对生物被膜的研究已经进行了数十年,并取得了长足的发展。但是,关于生物被膜的研究主要集中在致病菌上,这主要是由于目前抗生素耐药性的问题已迫在眉睫,临床上生物被膜导致的慢性感染危害人类健康并带来重大的经济损失等。然而,对于益生菌生物被膜的相关研究还处于起步阶段。以生物被膜形式生存是大多数细菌在自然环境下的生存状态,传统的实验室培养方法并不能模拟益生菌的自然生存状态,因此研究益生菌的生物被膜形成、发展、调控以及生物被膜状态下益生菌的生理特性,对于阐明益生菌的益生特性和开发更好的益生菌产品具有非常重大的意义。由于许多乳酸菌和双歧杆菌都具有产胞外多糖的能力,因此通过控制外界因素可以改变其产糖能力,从而控制益生菌生物被膜的形成能力。此外,利用分子手段改造益生菌,构建相关基因缺失菌株和过表达菌株,可以进一步阐明益生菌生物被膜的形成机制。目前的研究表明益生菌具有群体感应系统,因此通过调节群体感应信号分子受体蛋白及其调控通路,阐明益生菌群体感应系统与生物被膜形成的关系,是揭示益生菌生物被膜形成及调控机制的突破口之一。
[1] SANDERS M E, GUARNER F, GUERRANT R, et al. An update on the use and investigation of probiotics in health and disease[J]. Gut,2013, 62(5): 787-796. DOI:10.1136/gutjnl-2012-302504.
[2] KAILASAPATHY K, CHIN J. Survival and therapeutic potential of probiotic organisms with reference to Lactobacillus acidophilus and Bifidobacterium spp.[J]. Immunology and Cell Biology, 2000, 78(1):80-88.
[3] DELLEY M, BRUTTIN A, RICHARD M, et al. In vitro activity of commercial probiotic Lactobacillus strains against uropathogenic Escherichia coli[J]. FEMS Microbiology Letters, 2015, 362(13):fnv096. DOI:10.1093/femsle/fnv096.
[4] CAMPANIELLO D, BEVILACQUA A, SINIGAGLIA M, et al. Screening of Propionibacterium spp. for potential probiotic properties[J]. Anaerobe, 2015, 34: 169-173. DOI:10.1016/ j.anaerobe.2015.06.003.
[5] CORDINA C, SHAIKH I, SHRESTHA S, et al. Probiotics in the management of gastrointestinal disease: analysis of the attitudes and prescribing practices of gastroenterologists and surgeons[J]. Journal of Digestive Diseases, 2011, 12(6): 489-496. DOI:10.1111/j.1751-2980.2011.00534.x.
[6] STOODLEY P, SAUER K, DAVIES D G, et al. Biofilms as complex differentiated communities[J]. Annual Review of Microbiology, 2002,56(1): 187-209.
[7] CHAN C, HARDIN T C, SMART J I. A review of telavancin activity in in vitro biofilms and animal models of biofilm-associated infections[J]. Future Microbiology, 2015, 10(8): 1-14. DOI:10.2217/ fmb.15.53.
[8] O'LOUGHLIN C T, MILLER L C, SIRYAPORN A, et al. A quorum-sensing inhibitor blocks Pseudomonas aeruginosa virulence and biofilm formation[J]. Proceedings of the National Academy of Sciences, 2013, 110(44): 17981-17986. DOI:10.1073/ pnas.1316981110.
[9] YONEZAWA H, OSAKI T, KAMIYA S. Biofilm formation by Helicobacter pylori and its involvement for antibiotic resistance[J]. Biomed Research International, 2015, 2015: 914791. DOI:10.1155/2015/914791.
[10] FRANK K L, VERGIDIS P, BRINKMAN C L, et al. Evaluation of the Enterococcus faecalis biofilm-associated virulence factors AhrC and Eep in rat foreign body osteomyelitis and in vitro biofilm-associated antimicrobial resistance[J]. PLoS ONE, 2015, 10(6): e130187. DOI:10.1371/journal.pone.0130187.
[11] MACHADO A, CERCA N. The influence of biofilm formation by Gardnerella vaginalis and other anaerobes on bacterial vaginosis[J]. Journal of Infectious Diseases, 2015, 212(12): 1856-1861. DOI:10.1093/infdis/jiv338.
[12] RIEU A, AOUDIA N, JEGO G, et al. The biofilm mode of life boosts the anti-inflammatory properties of Lactobacillus[J]. Cellular Microbiology, 2014, 16(12): 1836-1853. DOI:10.1111/cmi.12331.
[13] CHEOW W S, KIEW T Y, HADINOTO K. Controlled release of Lactobacillus rhamnosus biofilm probiotics from alginate-locust bean gum microcapsules[J]. Carbohydrate Polymers, 2014, 103(4): 587-595. DOI:10.1016/j.carbpol.2014.01.036.
[14] van HOEK M L. Biofilms: an advancement in our understanding of Francisella species[J]. Virulence, 2014, 4(8): 833-846. DOI:10.4161/ viru.27023.
[15] HOBLEY L, HARKINS C, MACPHEE C E, et al. Giving structure to the biofilm matrix: an overview of individual strategies and emerging common themes[J]. FEMS Microbiology Reviews, 2015, 39(5): 649-669. DOI:10.1093/femsre/fuv015.
[16] CUTLER N A, CHAPUT D L, OLIVER A E, et al. The spatial organization and microbial community structure of an epilithic biofilm[J]. FEMS Microbiology Ecology, 2015, 91(3): 1-9. DOI:10.1093/femsec/fiu027.
[17] SAUER K, CAMPER A K, EHRLICH G D, et al. Pseudomonas aeruginosa displays multiple phenotypes during development as a biofilm[J]. Journal of Bacteriology, 2002, 184(4): 1140-1154.
[18] BLACKLEDGE M S, WORTHINGTON R J, MELANDER C. Biologically inspired strategies for combating bacterial biofilms[J]. Current Opinion in Pharmacology, 2013, 13(5): 699-706. DOI:10.1016/j.coph.2013.07.004.
[19] CHEW S Y, CHEAH Y K, SEOW H F, et al. In vitro modulation of probiotic bacteria on the biofilm of Candida glabrata[J]. Anaerobe,2015, 34: 132-138. DOI:10.1016/j.anaerobe.2015.05.009.
[20] VUOTTO C, LONGO F, DONELLI G. Probiotics to counteract biofilm-associated infections: promising and conflicting data[J]. International Joutnal of Oral Science, 2014, 6(4): 189-194. DOI:10.1038/ijos.2014.52.
[21] GU H, HOU S, YONGYAT C, et al. Patterned biofilm formation reveals a mechanism for structural heterogeneity in bacterial biofilms[J]. Langmuir, 2013, 29(35): 11145-11153. DOI:10.1021/ la402608z.
[22] BELAS R. When the swimming gets tough, the tough form a biofilm[J]. Molecular Microbiology, 2013, 90(1): 1-5. DOI:10.1111/ mmi.12354.
[23] LEBEER S, VERHOEVEN T L A, PEREA VELEZ M, et al. Impact of environmental and genetic factors on biofilm formation by the probiotic strain Lactobacillus rhamnosus GG[J]. Applied and Environmental Microbiology, 2007, 73(21): 6768-6775.
[24] BRINK H G, NICOL W. The influence of shear on the metabolite yield of Lactobacillus rhamnosus biofilms[J]. New Biotechnology,2014, 31(5): 460-467. DOI:10.1016/j.nbt.2014.06.003.
[25] SLIZOVA M, NEMCOVA R, MAD'AR M, et al. Analysis of biofilm formation by intestinal lactobacilli[J]. Canadian Journal of Microbiology, 2015, 61(6): 437-446. DOI:10.1139/cjm-2015-0007.
[26] CHENG K J, FAY J P, HOWARTH R E, et al. Sequence of events in the digestion of fresh legume leaves by rumen bacteria[J]. Applied and Environmental Microbiology, 1980, 40(3): 613-625.
[27] SUN H, ZUSMAN D R, SHI W. Type IV pilus of Myxococcus xanthus is a motility apparatus controlled by the frz chemosensory system[J]. Current Biology, 2000, 10(18): 1143-1146.
[28] VENTOLINI G, MITCHELL E, SALAZAR M. Biofilm formation by vaginal Lactobacillus in vivo[J]. Medical Hypotheses, 2015, 84(5):417-420. DOI:10.1016/j.mehy.2014.12.020.
[29] LEPARGNEUR J P, ROUSSEAU V. Protective role of the doderlein flora[J]. Journal de Gynecologie, Obstetrique et Biologie de la Reprodroduction (Paris), 2002, 31(5): 485-494.
[30] CHEOW W S, HADINOTO K. Biofilm-like Lactobacillus rhamnosus probiotics encapsulated in alginate and carrageenan microcapsules exhibiting enhanced thermotolerance and freeze-drying resistance[J]. Biomacromolecules, 2013, 14(9): 3214-3222. DOI:10.1021/ bm400853d.
[31] GUTTENPLAN S B, KEARNS D B. Regulation of flagellar motility during biofilm formation[J]. FEMS Microbiology Reviews, 2013,37(6): 849-871. DOI:10.1111/1574-6976.12018.
[32] LIU L, QIN Y, WANG Y, et al. Complete genome sequence of Bifidobacterium animalis RH, a probiotic bacterium producing exopolysaccharides[J]. Journal of Biotechnology, 2014, 189: 86-87. DOI:10.1016/j.jbiotec.2014.08.041.
[33] JENAL U M J. Mechanisms of cyclic-di-GMP signaling in bacteria[J]. Annual Review of Genetics, 2006, 40(1): 385-407.
[34] KOLODKIN-GAL I, CAO S, CHAI L, et al. A self-produced trigger for biofilm disassembly that targets exopolysaccharide[J]. Cell, 2012,149(3): 684-692. DOI:10.1016/j.cell.2012.02.055.
[35] HOLM A, VIKSTRÃ M E. Quorum sensing communication between bacteria and human cells: signals, targets, and functions[J]. Frontiers in Plant Science, 2014, 5: 309. DOI:10.3389/fpls.2014.00309.
[36] BORDI C, de BENTZMANN S. Hacking into bacterial biofilms: a new therapeutic challenge[J]. Annals of Intensive Care, 2011, 1: 19. DOI:10.1186/2110-5820-1-19.
[37] GUO L, HE X, SHI W. Intercellular communications in multispecies oral microbial communities[J]. Frontiers in Microbiology, 2014, 5:328. DOI:10.3389/fmicb.2014.00328.
[38] di FRANCESCO S, NEGLIA G, VECCHIO D, et al. Influence of season on corpus luteum structure and function and AI outcome in the Italian Mediterranean buffalo (Bubalus bubalis)[J]. Theriogenology,2012, 78(8): 1839-1845. DOI:10.1016/j.theriogenology.2012.07.022.
[39] ZHU P, PENG H, NI N, et al. Novel AI-2 quorum sensing inhibitors in Vibrio harveyi identified through structure-based virtual screening[J]. Bioorganic and Medicinal Chemistry Letters, 2012, 22(20): 6413-6417. DOI:10.1016/j.bmcl.2012.08.062.
[40] JAKUBOVICS N S. Talk of the town: interspecies communication in oral biofilms[J]. Molecular Oral Microbiology, 2010, 25(1): 4-14. DOI:10.1111/j.2041-1014.2009.00563.x.
[41] ADLER L, ALTER T, SHARBATI S, et al. Phenotypes of Campylobacter jejuni luxS mutants are depending on strain background, kind of mutation and experimental conditions[J]. PLoS ONE, 2014, 9(8): e104399. DOI:10.1371/journal.pone.0104399.
[42] SUN Z K, HE X, BRANCACCIO V F, et al. Bifidobacteria exhibit luxS-dependent autoinducer 2 activity and biofilm formation[J]. PLoS ONE, 2014, 9(2): e88260. DOI:10.1371/journal.pone.0088260.
[43] TOMARAS A P, FLAGLER M J, DORSEY C W, et al. Characterization of a two-component regulatory system from Acinetobacter baumannii that controls biofilm formation and cellular morphology[J]. Microbiology, 2008, 154(11): 3398-3409. DOI:10.1099/mic.0.2008/019471-0.
[44] UCHIDA H, FUJITAN K, KAWAI Y, et al. A new assay using surface plasmon resonance (SPR) to determine binding of the Lactobacillus acidophilus group to hunman colonic mucin[J]. Bioscience,Biotechnology, and Biochemistry, 2004, 68(5): 1004-1010.
[45] HYNONEN U, WESTERLUND-WIKSTROM B, PALVA A, et al. Identification by flagellum display of an epithelial cell- and fibronectinbinding function in the SlpA surface protein of Lactobacillus brevis[J]. Journal of Bacteriology, 2002, 184(12): 3360-3367.
[46] ADLERBERTH I, AHRNE S, JOHANSSON M L, et al. A mannose-specific adherence mechanism in Lactobacillus plantarum conferring binding to the human colonic cell line HT-29[J]. Applied and Environmental Microbiology, 1996, 62(7): 2244-2251.
[47] 张莉. 植物乳杆菌的黏附特性研究及其在益生菌干酪中的应用[D].长春: 吉林大学, 2013: 61-82.
Progress in Research on Biofilm Formation by Probiotics
LIU Lei, LIU Yi, LI Pinglan*
(Beijing Advanced Innovation Center for Food Nutrition and Human Health, College of Food Science and Nutritional Engineering,China Agricultural University, Beijing 100083, China)
Probiotics are defined as ‘live microorganisms which when administered in adequate amounts confer health benefits to the host'. In recent years, probiotics have gained increasing attention due to their therapeutic effects. Biofilms are formed by multiple bacterial cells attached to a surface that arranges themselves into a complex tertiary structure encased in an extracellular matrix comprised of carbohydrates, proteins, and other macromolecules, which constitute the majority of bacteria in most natural ecosystems. However, little research has been done concerning probiotic biofilms. Therefore, the formation, characteristics, influencing factors, advantages and regulatory pathways of probiotic biofilms are discussed in this paper. In the future, probiotic biofilms will be an important research area.
probiotics; biofilms; influencing factors; regulatory pathway
10.7506/spkx1002-6630-201609040
Q939.99
A
1002-6630(2016)09-0214-06
刘蕾, 刘义, 李平兰. 益生菌生物被膜的研究进展[J]. 食品科学, 2016, 37(9): 214-219. DOI:10.7506/spkx1002-6630-201609040. http://www.spkx.net.cn
LIU Lei, LIU Yi, LI Pinglan. Progress in research on biofilm formation by probiotics[J]. Food Science, 2016, 37(9): 214-219.(in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201609040. http://www.spkx.net.cn
2015-06-20
国家自然科学基金面上项目(31271827;31471707);国家高技术研究发展计划(863计划)项目(2011AA100805-0-1)
刘蕾(1988—),女,博士研究生,研究方向为食品微生物。E-mail:liulei0606@gmail.com
*通信作者:李平兰(1964—),女,教授,博士,研究方向为食品微生物。E-mail:lipinglan@cau.edu.cn
