4 株E. coli O157:H7毒力基因检测及其冷应激损伤
2016-11-11水新云王虎虎南京师范大学金陵女子学院食品科学系江苏南京0097南京农业大学国家肉品质量安全控制工程技术研究中心江苏南京0095
水新云,王虎虎,高 峰,江 芸,(.南京师范大学金陵女子学院食品科学系,江苏 南京 0097;.南京农业大学 国家肉品质量安全控制工程技术研究中心,江苏 南京 0095)
4 株E. coli O157:H7毒力基因检测及其冷应激损伤
水新云1,王虎虎2,高 峰2,江 芸1,*
(1.南京师范大学金陵女子学院食品科学系,江苏 南京 210097;2.南京农业大学 国家肉品质量安全控制工程技术研究中心,江苏 南京 210095)
采用多重聚合酶链式反应对4 株大肠埃希氏菌O157:H7(Escherichia coli O157:H7)进行毒力特性评价,研究3 种常用选择性生长基质对E.coli O157:H7的精确定量对比,筛选出的适宜选择性培养基用于冷应激时菌体损伤的研究。结果显示,菌株CICC 21530的stx1、stx2、eae基因均为阳性,NCTC 12900和牛肉分离菌1的eae呈现阳性,牛肉分离菌2三种毒力基因均为阴性,表明4 株菌的致病性不同;4 株测试菌在改良山梨醇麦康凯琼脂(cefiximetellurite sorbitol macconkey agar,CT-SMAC)上计数均显著低于胰蛋白胨大豆琼脂(tryptose soya agar,TSA)上计数(P<0.05),而SMAC和改良伊红美蓝琼脂(modified eosin methylene blue agar,mEMB)上的计数与TSA相比无显著性差异,表明改良SMAC对正常菌体既有较强抑制作用,不适合用于E. coli O157:H7的精确计数,可以选用SMAC或mEMB;进一步以SMAC和mEMB作为选择性培养基研究菌体在4 ℃冷应激时的损伤情况,结果表明冷藏过程中SMAC、mEMB及TSA上的菌数均逐渐下降,第10天时4 株菌均发生了一定程度的损伤或死亡。本方法可为食品安全中E. coli O157:H7的定量评估和风险控制提供科学依据。
大肠埃希氏菌O157:H7;毒力基因;半致死损伤;冷应激;选择性培养基
大肠埃希氏菌O157:H7(Escherichia coli O157:H7)是一种重要的食源性人畜共患病原菌,已成为世界性的重要公共卫生问题之一[1-2]。E. coli O157:H7是肠出血性大肠埃希氏菌(enterohemorrhagic E. coli,EHEC)的一个主要血清型,可引起腹泻、出血性肠炎,极易继发溶血性尿毒综合症和血栓性血小板减少性紫癜两种严重的并发症[3]。该菌株最主要的毒力因子是志贺毒素(Stx),包括Stx1和Stx2。由eae基因编码的分子质量在94~97 kD的紧密素对E. coli O157:H7在肠道定居起重要作用,其黏附作用导致特征性黏附-抹去损伤(attaching and effacing lesion,A/E)病变的发生。
食品在加工贮藏过程中,食品中的微生物可受到不利于其生长的多种胁迫环境,如加热、冷冻、酸渍、盐渍、防腐剂等。经这些胁迫条件处理后微生物存在3 种状态:死亡细胞(致死的或不可逆的损伤)、未损伤细胞(正常细胞)和损伤细胞(应激的、亚致死的或可逆的损伤)[4]。研究表明,常见的食品加工处理工艺均会导致E. coli O157:H7等微生物产生亚致死损伤[5-7]。微生物的亚致死损伤状态被认为是介于微生物死亡和正常生长的中间状态。研究表明,4 ℃冷藏条件也会对细菌造成冷应激,从而可能导致菌体发生应激损伤[8]。损伤菌失去了一些内在特性,如致病特性、在选择性培养基上的生长能力,进而在食品安全的常规检测中被低估或忽视。然而,已有研究表明,处于亚致死损伤状态的食源性致病菌在一定条件下可以修复,最终恢复正常生理特性,包括致病性[9]。因而,这部分亚致死损伤微生物的存在给食品的安全性以及货架期造成极大隐患。
亚致死损伤菌在非选择性培养基上可形成菌落而在选择性培养基上不能形成菌落,因此非选择性培养基上菌落数与选择性培养基上菌落数的差值即可反映亚致死损伤菌的数量[10]。对食源性致病菌进行准确计数是食品安全性评估的重要前提。关于E. coli O157:H7亚致死损伤菌计数的选择性培养基,目前报道并不一致。有的应用山梨醇麦康凯琼脂(sorbitol macconkey agar,SMAC)[11-13],或应用改良山梨醇麦康凯琼脂,即山梨醇麦康凯琼脂添加头孢克肟-亚碲酸钾(cefiximetellurite SMAC,CT-SMAC)[8,14-15],也有的应用改良伊红美蓝琼脂(modified eosin methylene blue agar,mEMB)[16]。此外,还有研究应用大肠菌群显色培养基、乳糖胆盐琼脂,或胰蛋白胨大豆琼脂(tr yptose soya agar,TSA)中添加一定浓度NaCl作为选择性培养基来检测E. coli O157:H7损伤情况[17-18]。
因此本实验首先应用多重聚合酶链式反应(polymerase chain reaction,PCR)方法对实验用4 株E. coli O157:H7进行stx1、stx2、eae毒力基因的检测,以了解其致病性;进一步选用常见的3 种选择性培养基SMAC、CT-SMAC、mEMB对其新鲜菌液进行培养计数,以非选择性培养基TSA为对照,拟筛选出合适的选择性培养基;在此基础上研究冷应激条件下E. coli O157:H7的损伤情况。本实验将为E. coli O157:H7的精确计数提供合理手段,为食品安全危险性评估提供科学依据。
1 材料与方法
1.1 菌种与试剂
本实验所用E. coli O157:H7共4 株,1 株(编号CICC 21530)由南京农业大学国家肉品质量安全控制工程技术研究中心惠赠,菌种原购自中国工业微生物菌种保藏管理中心;1 株(编号NCTC12900) 广东环凯微生物科技有限公司;2 株由南京农业大学江苏省动物源食品生产与安全保障重点实验室惠赠,均分离自牛肉。
胰蛋白胨大豆肉汤(tryptic soy broth,TSB)、TSA、SMAC、亚碲酸钾、P-09噻孢霉素 北京路桥技术有限公司;mEMB 山东青岛捷世康生物科技有限公司;TaqTM(with Mg2+free buffer)、100 bp DNA Marker 大连宝生物(TaKaRa)工程有限公司;琼脂糖 北京天根生化科技有限公司;4S Red染料 上海生工生物科技有限公司;所有引物均由上海英俊生物技术有限公司合成。
1.2 仪器与设备
Mastercycler ep personal PCR仪 德国Enperndorf公司;GelDoc 2000 system凝胶成像仪 美国BioRad公司;TCL-16G高速离心机 上海安亭仪器厂;DYY-2C琼脂糖凝胶电泳仪 上海天能科技有限公司;SZX超净工作台 上海浦东跃欣仪器厂;DHP-9052电热恒温培养箱 上海一恒科技公司。
1.3 方法
1.3.1 菌株的培养
将4 株E. coli O157:H7分别经TSA划线培养,37 ℃培养24 h,挑取单菌落分别接种于3 mL TSB中,37 ℃培养18 h,再转接至100 mL TSB中,37 ℃培养18 h。得到菌液置于4 ℃,不同时间取样进行传统培养计数。
1.3.2 细菌基因组DNA提取
将4 株实验用E. coli O157:H7、E. coli Sakai(阳性对照菌)和DH5α(阴性对照菌)共6株菌分别用TSA划线培养24 h。挑一环单菌落于150 μL双蒸水,放于灭菌1.5 mL离心管中,彻底涡旋,使其充分混匀。99 ℃水浴处理10 min,冰浴2 min,12 000×g离心2 min。吸取上清液至另一离心管中即为细菌基因组DNA,-20 ℃冻存备用。
1.3.3 多重PCR
采用多重PCR方法对4 株实验用E. coli O157:H7进行stx1、stx2、eae毒力基因检测[19-20]。对应stx1、stx2、eae毒力基因的3 对引物分别是:stx1F(5’-ACA CTG GAT GAT CTC AGT GG-3’),stx1R(5’-CTG AAT CCC CCT CCA TTA TG-3’);stx2F(5’-GGC ACT GTC TGA AAC TGC TCC-3’),stx2R(5’-TCG CCA GTT ATC TGA CAT TCT G-3’);eaeF(5’-GTG GCG AAT GGC ACT GAG ACT-3’),eaeR(5’-CCC CAT TCT TTT TCA CCG TCG-3’)。PCR反应体系(20 μL):10×PCR buffer 2 μL,dNTP 1.4 μL,MgCl21.2 μL,6条引物(10 μmol/L)各0.2 μL,Taq polymerase 0.1 μL,DNA模板1 μL。PCR反应程序:94 ℃预变性4 min;然后90 ℃变性45 s,55 ℃退火30 s,72 ℃退火50 s,进行30 个循环;最后72 ℃延伸8 min。PCR产物经1.4%琼脂糖凝胶电泳检测。
1.3.4 传统培养计数
取新鲜TSB菌液及4 ℃放置一定时间的菌液,经过系列10 倍梯度稀释,选择3 个合适稀释梯度,取100 μL菌液至非选择性培养基TSA和选择性培养基CTSMAC、SMAC、mEMB上,采用平板涂布法,37 ℃培养24 h,对E. coli O157:H7进行菌落计数,并进一步计算损伤率:
损伤率/%=[(非选择性培养基上的平均菌落数-选择性培养基上的平均菌落数)/非选择性培养基上的平均菌落数]×100
式中:非选择培养基TSA平板上的菌落数为未损伤、损伤和已繁殖的菌数,选择性培养基上的菌落数为未损伤、已修复、已繁殖的菌落数。
1.4 统计分析
数据采用SPSS V 17.0进行数据分析处理,采用方差分析进行单因素方差分析中的Duncan’s多重比较检验(P<0.05),数据均以
2 结果与分析
2.1 菌株毒力基因的检测
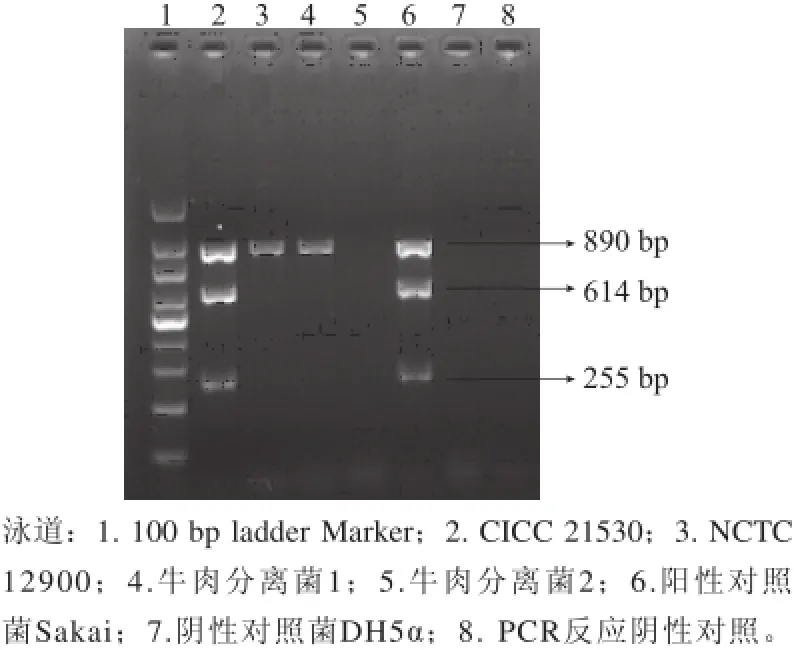
图1 E. coli三种毒力基因多重PCR电泳图Fig.1 Electrophoresis profile for three virulence genes of E. coli amplified by multiplex PCR
如图1所示,阳性对照菌Sakai成功扩增出3 条阳性条带,分别是eae基因条带(890 bp)、stx1基因条带(614 bp)、stx2基因条带(255 bp)。本实验所用菌株结果显示,CICC 21530三种毒力基因均阳性,NCTC 12900只有eae基因阳性,牛肉分离菌1 eae基因阳性,牛肉分离菌2三种毒力基因均为阴性。结果表明,4 株E. coli O157:H7所带毒力基因不尽相同,表明它们的致病性是不同的。
2.2 选择性培养基的筛选
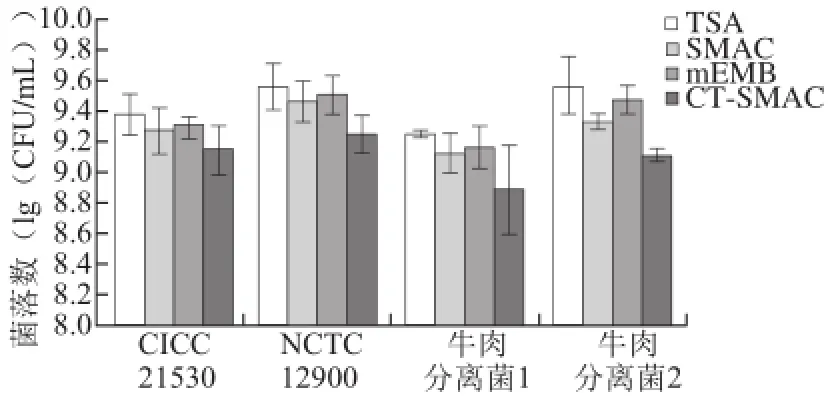
图2 2 E. coli coli O157:H7新鲜菌液在不同培养基上的菌落数Fig.2 Counts of fresh E. coli culture on different medium plates
如图2所示,本实验应用非选择性培养基TSA和选择性培养基CT-SMAC、SMAC、mEMB对新鲜菌液进行菌落计数,与TSA计数相比,4 株菌在选择性培养基CT-SMAC上的菌落数CICC 21530、NCTC 12900、牛肉分离菌1、牛肉分离菌2分别下降了0.26、0.32、0.36、0.45 (lg(CFU/mL)),进一步统计分析表明4 株菌在CT-SMAC上的计数均显著低于TSA上的计数(P<0.05),表明CT-SMAC对正常菌可产生显著的抑制作用,会导致计数结果偏差;4 株菌在选择性培养基SMAC上的计数与TSA计数相比下降了约0.1~0.2(lg(CFU/mL)),而在mEMB上的计数与TSA计数相比下降数<0.1(lg(CFU/mL)),进一步统计分析表明4 株菌在TSA、SMAC、mEMB上的菌落数无显著性差异(P>0.05),表明这两种选择性培养基对正常菌抑制较小。本实验结果表明,可以选用SMAC或mEMB用于后续实验中E. coli O157:H7损伤菌的选择性培养计数,而CT-SMAC并不合适。
2.3 冷应激条件下E. coli O157:H7损伤的研究
本实验进一步将菌液放置4 ℃不同时间,应用上述筛选的适宜选择性培养基SMAC和mEMB进行培养计数来观察冷应激条件下E. coli O157:H7的损伤情况,结果见图3。冷藏过程中4株菌在SMAC、mEMB及TSA上的计数整体呈下降趋势,在放置12 h时即低于初始菌数。放置10 d时与初始菌数相比,TSA上菌数降低了0.2~0.5(lg(CFU/mL))。统计分析发现,菌株CICC 21530、NCTC 12900以及牛肉分离菌2在0 d和10 d在TSA上的计数结果有显著性差异(P<0.05),表明4 ℃放置10 d时有细菌发生了一定程度的死亡。

图3 3 E. coli coli O157:H7菌液放置4 ℃条件下在不同培养基上的菌落数Fig.3 Counts of E. coli during storage in 4 ℃ cultured on different medium plates
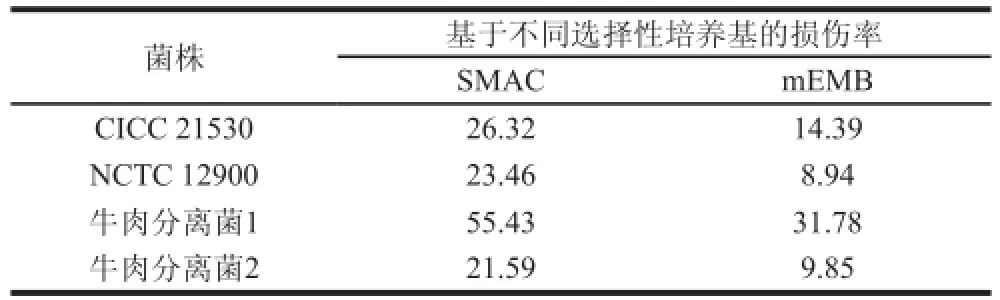
表1 4 株E. coli O157:H7 TSB培养液4 ℃放置10 d时的损伤率Table 1 Injury rates of four E. coli strains cultured in TSB stored in 4 ℃ for 10 days %
进一步计算10 d时的损伤率,结果如表1所示,基于SMAC或mEMB上的计数,各菌损伤率不尽相同,其中牛肉分离菌1在SMAC、mEMB上的损伤率均较高。实验结果表明,新鲜菌液置于4 ℃冰箱放置一段时间后,各菌发生了不同程度的损伤,甚至死亡。
3 讨 论
E. coli O157:H7的毒力基因主要有stx1、stx2、rfbE、flic、hly、ehxA等[21-22],E. coli O157:H7的致病机理并不仅仅因为单一的毒力因子,更是多种毒力因子共同作用的结果。其最主要的毒力因子是Stx,包括Stx1和Stx2,此外还有由eae基因编码的紧密素。多重PCR是在常规PCR基础上发展起来的一种PCR扩增技术,是在同一反应体系中加入多对引物同时扩增多条目的DNA片段的方法,利用扩增片段在琼脂糖凝胶电泳上的位置差异来区分和鉴定,该方法节省模板DNA、节省时间和费用[23]。本实验首先应用多重PCR方法对4 株实验用E. coli O157:H7进行了stx1、stx2、eae毒力基因的检测,结果表明,4 株E. coli O157:H7所带毒力基因不尽相同,表明它们的致病性是不同的。
已有较多研究应用选择性培养基与非选择性培养基进行传统培养来计数某特定食源性致病菌的半致死损伤菌,如单增李斯特菌、沙门氏菌、空肠弯曲菌、金黄色葡萄球菌以及E. coli O157:H7等。关于损伤性E. coli O157:H7培养计数方面,常见的报道有应用CTSMAC[8,14-15]、SMAC[11-13]、mEMB[16]等作为选择性培养基。这些选择性培养基中含有一些抑菌成分,使得E. coli O157:H7可以生长而抑制其他细菌的生长。我国食品卫生微生物学检验国家标准采用CT-SMAC来分离培养E. coli O157:H7[24]。但本实验对E. coli O157:H7新鲜菌液进行菌落计数时发现,CT-SMAC对正常菌体产生了显著的抑制作用,导致计数偏低,实验结果表明,CT-SMAC可用于E. coli O157:H7的分离培养,但并不适合用于其精确计数,因而不适合用于后续实验E. coli O157:H7损伤菌的计数。Jasson等[8]研究也发现CT-SMAC对E. coli O157:H7正常菌会产生一定的抑制作用。而选择性培养基SMAC、mEMB上E. coli O157:H7计数结果亦低于TSA上的计数,但无显著差异,表明这两种选择性培养基对正常菌抑制较小,可用于其精确计数。因此本实验进一步应用SMAC和mEMB作为选择性培养基研究了冷应激时E. coli O157:H7的损伤情况,结果发现4 ℃冷藏10 d时4 株菌均发生了一定程度的损伤。Jasson等[8]研究表明冷应激一段时间后E. coli O157:H7损伤率显著增加,而空肠弯曲菌和单增李斯特菌的损伤率则无显著性变化。本实验还发现,4 株菌损伤程度不尽相同,其中牛肉分离菌1较为敏感,表明冷应激损伤与菌株有关。
4 结 论
本实验采用多重P C R方法,确定4 株实验用E. coli O157:H7携带毒力基因stx1、stx2、eae不尽相同,表明它们的致病性是不同的。E. coli O157:H7的选择性培养基,CT-SMAC对正常菌有明显的抑制作用,不适合用于E. coli O157:H7的精确计数,而SMAC和mEMB抑制小,可以使用。冷应激损伤实验发现,4 ℃冷藏10 d时4 株菌均发生了不同程度的损伤,甚至死亡。本实验确定了4 株不同毒力基因的E. coli O157精确计数的选择性培养基,可为食品安全中E. coli O157的定量评估和风险控制提供科学依据。
[1] ATKINSON R, JOHNSON G, ROOT T, et al. Importance of culture confirmation of Shiga toxin-producing Escherichia coli infection as illustrated by outbreaks of gastroenteritis: New York and North Carolina, 2005[J]. Morbidity and Mortality Weekly Report, 2006, 55(38): 1042-1045. DOI:10.1037/e550322006-003.
[2] MAGWEDERE K, DANG H A, MILLS E W, et al. Incidence of Shiga toxin-producing Escherichia coli strains in beef, pork, chicken, deer, boar, bison, and rabbit retail meat[J]. Journal of Veterinary Diagnostic Investigation, 2013, 25(2): 254-258. DOI:10.1177/1040638713477407.
[3] PAGE A V, LILES W C. Enterohemorrhagic Escherichia coli infections and the hemolytic-uremic syndrome[J]. The Medical Clinics of North America, 2013, 97(4): 681-695. DOI:10.1016/ j.mcna.2013.04.001.
[4] NORIEGA E, VELLIOU E, van DERLINDEN E, et al. Effect of cell immobilization on heat-induced sublethal injury of Escherichia coli, Salmonella typhimurium and Listeria innocua[J]. Food Microbiology, 2013, 36(2): 355-364. DOI:10.1016/j.fm.2013.06.015.
[5] PILAVTEPE-ÇELIK M, BUZRUL S, ALPAS H, et al. Multi-pulsed high hydrostatic pressure treatment for inactivation and injury of Escherichia coli[J]. Journal of Consumer Protection and Food Safety, 2011, 6(3): 343-348. DOI:10.1007/s00003-010-0647-2.
[6] GUYOT S, POTTIER L, FERRET E, et al. Physiological responses of Escherichia coli exposed to different heat-stress kinetics[J]. Archives of Microbiology, 2010, 192(8): 651-661. DOI:10.1007/s00203-010-0597-1.
[7] BACCUS-TAYLOR G S, FALLOON O C, HENRY N. Pressure resistance of cold shocked Escherichia coli O157:H7 in ground beef, beef gravy and peptone water[J]. Journal of Applied Microbiology, 2015, 118(6): 1521-1529. DOI:10.1111/jam.12794.
[8] JASSON V, UYTTENDAELE M, RAJKOVIC A, et al. Establishment of procedures provoking sub-lethal injury of Listeria monocytogenes, Campylobacter jejuni and Escherichia coli O157 to serve method performance testing[J]. International Journal of Food Microbiology, 2007, 118(3): 241-249. DOI:10.1016/j.ijfoodmicro.2007.07.016.
[9] RITZ M, PILET M F, JUGIAU F, et al. Inactivation of Salmonella typhimurium and Listeria monocytogenes using high-pressure treatments: destruction or sublethal stress[J]. Letters in Applied Microbiology, 2006, 42(4): 357-362. DOI:10.1111/j.1472-765X.2006.01868.x.
[10] BESSE N G. Influence of various environmental parameters and of detection procedures on the recovery of stressed L. monocytogenes: a review[J]. Food Microbiology, 2002, 19(2/3): 221-234. DOI:10.1006/ fmic.2001.0448.
[11] SMITH A R, ELLISON A L, ROBINSON A L, et al. Enumeration of sublethally injured Escherichia coli O157:H7 ATCC 43895 and Escherichia coli strain B-41560 using selective agar overlays versus commercial methods[J]. Journal of Food Protection, 2013, 76(4): 674-679. DOI:10.4315/0362-028X.JFP-12-363.
[12] PATEL J, MACARISIN D, SANGLAY G, et al. Inactivation and injury of pathogens on intact beef treated with hydrodynamic pressure[J]. Innovative Food Science and Emerging Technologies, 2012, 14(1): 38-45. DOI:10.1016/j.ifset.2011.08.002.
[13] HONG S, MENDONÇA A F, DARABA A, et al. Radiation resistance and injury in starved Escherichia coli O157:H7 treated with electronbeam irradiation in 0.85% saline and in apple juice[J]. Foodborne Pathogens and Disease, 2014, 11(11): 900-906. DOI:10.1089/ fpd.2014.1782.
[14] RODRIGUEZ E, ARQUES J L, NUÑEZ M, et al. Combined effect of high-pressure treatments and bacteriocin-producing lactic acid bacteria on inactivation of Escherichia coli O157:H7 in raw-milk cheese[J]. Applied and Environmental Microbiology, 2005, 71(7): 3399-3404. DOI:10.1128/AEM.71.7.3399-3404.2005.
[15] YOSHITOMI K J, ZAPATA R, JINNEMAN K C, et al. Recovery of E. coli O157 strains after exposure to acidification at pH 2[J]. Letters in Applied Microbiology, 2012, 54(6): 499-503. DOI:10.1111/j.1472-765X.2012.03250.x.
[16] YUSTE J, CAPELLAS M, DANIEL Y C, et al. Inactivation and sublethal injury of foodborne pathogens by high pressure processing: evaluation with conventional media and thin agar layer method[J]. Food Research International, 2004, 37(9): 861-866. DOI:10.1016/ j.foodres.2004.05.002.
[17] GABRIEL A A, NAKANO H. Influences of simultaneous physicochemical stress exposures on injury and subsequent responses of E. coli O157:H7 to resuscitative and inactivative challenges[J]. International Journal of Food Microbiology, 2010, 139(3): 182-192. DOI:10.1016/j.ijfoodmicro.2010.02.028.
[18] YANG Baowei, SHI Ying, XIA Xiaodong, et al. Inactivation of foodborne pathogens in raw milk using high hydrostatic pressure[J]. Food Control, 2012, 28(2): 273-278. DOI:10.1016/ j.foodcont.2012.04.030.
[19] PATON A W, PATON J C. Direct detection and characterization of Shiga toxigenic Escherichia coli by multiplex PCR assays for stx1, stx2, eae, ehxA, and saa[J]. Journal of Clinical Microbiology, 2002, 40(1): 271-274. DOI:10.1128/JCM.40.1.271-274.2002.
[20] SVOBODA A L, DUDLEY E G, DEBROY C, et al. Presence of Shiga toxin-producing Escherichia coli O-group in small and verysmall beef-processing plant and resulting ground beef detected by a multiplex polymerase chain reaction assay[J]. Foodborne Pathogens and Disease, 2013, 10(9): 789-795. DOI:10.1089/fpd.2012.1445.
[21] SON I, BINET R, LIN A, et al. Identification of five Shiga toxinproducing Escherichia coli genes by luminex microbead-based suspension array[J]. Journal of Microbiological Methods, 2015, 111: 108-110. DOI:10.1016/j.mimet.2015.02.008.
[22] BUGAREL M, BEUTIN L, MARTIN A, et al. Micro-array for the identification of Shiga toxin-producing Escherichia coli (STEC) seropathotypes associated with hemorrhagic colitis and hemolytic uremic syndrome in humans[J]. International Journal of Food Microbiology, 2010, 142(3): 318-329. DOI:10.1016/ j.ijfoodmicro.2010.07.010.
[23] S˘NCHEZ S, LLORENTE M T, ECHEITA M A, et al. Development of three multiplex PCR assays targeting the 21 most clinically relevant serogroups associated with Shiga toxin-producing E. coli infection in humans[J]. PLoS One, 2015, 10(1): e0117660. DOI:10.1371/journal. pone.0117660.
[24] GB/T 4789.36—2008 食品卫生微生物学检验: 大肠埃希氏菌O157:H7/NM检验[S]. 北京: 中国标准出版社, 2008.
Detection of Virulence Genes of Four Escherichia coli O157:H7 Strains and Cold Stress Injury in the Bacteria
SHUI Xinyun1, WANG Huhu2, GAO Feng2, JIANG Yun1,*
(1. Department of Food Science, Ginling College, Nanjing Normal University, Nanjing 210097, China; 2. National Center of Meat Quality and Safety Control, Nanjing Agricultural University, Nanjing 210095, China)
In this study, the virulence genes of four Escherichia coli O157:H7 strains were detected by multiplex polymerase chain reaction (PCR). Then three selective media were compared for enumeration of E. coli O157:H7 and the injury of E. coli O157:H7 under cold stress was also investigated with the appropriate selective medium. The results showed that CICC 21530 strain was positive for all the three genes stx1, stx2 and eae, while NCTC 12900 and one beef isolate were eae-positive, and two other isolates were negative for all the three virulence genes, which indicated that the pathogenicity of four strains was different. The counts of all four strains on three selective media were lower than those on tryptose soya agar (TSA) (P < 0.05) but there was no significant difference between them except for cefixime-tellurite sorbitol macconkey agar (CT-SMAC), which indicated that CT-SMAC was not suitable for counting E. coli O157:H7 due to the greater inhibition on normal bacteria, while SMAC or mEMB could be used. Then the injury of E. coli O157:H7 stored at 4 ℃ (cold stress) was investigated with each of these two selective media. The results showed that the counts on SMAC, mEMB, or TSA were decreased during the whole storage and some bacteria were injured or killed on the 10thday. In the current study, the selective media for accurate enumeration of four E. coli O157:H7 strains with different virulence genes were finalized, which would provide the scientific basis for quantitative assessment and risk control of E. coli O157:H7 in food safety evaluation.
Escherichia coli O157:H7; virulence genes; sub-lethal injury; cold stress; selective media
10.7506/spkx1002-6630-201604031
TS201.3
A
1002-6630(2016)04-0176-05
水新云, 王虎虎, 高峰, 等. 4 株E. coli O157:H7毒力基因检测及其冷应激损伤[J]. 食品科学, 2016, 37(4): 176-180.
DOI:10.7506/spkx1002-6630-201604031. http://www.spkx.net.cn
SHUI Xinyun, WANG Huhu, GAO Feng, et al. Detection of virulence genes of four Escherichia coli O157:H7 strains and cold stress injury in the bacteria[J]. Food Science, 2016, 37(4): 176-180. (in Chinese with English abstract) DOI:10.7506/ spkx1002-6630-201604031. http://www.spkx.net.cn
2015-04-05
国家自然科学基金面上项目(31371861)
水新云(1989—),女,硕士研究生,研究方向为肉品安全与质量控制。E-mail:951619906@qq.com
*通信作者:江芸(1971—),女,教授,博士,研究方向为肉品安全与质量控制。E-mail:jiangyun@njnu.edu.cn
