实时荧光定量PCR技术监测腌制麻竹笋中乳酸乳球菌动态变化
2016-11-11夏雪娟叶秀娟吴金松阚建全南大学食品科学学院重庆40075重庆市特色食品工程技术研究中心重庆40075
夏雪娟,郑 炯,2,,叶秀娟,吴金松,阚建全,2(.西 南大学食品科学学院,重庆 40075;2.重庆市特色食品工程技术研究中心,重庆 40075)
实时荧光定量PCR技术监测腌制麻竹笋中乳酸乳球菌动态变化
夏雪娟1,郑 炯1,2,*,叶秀娟1,吴金松1,阚建全1,2
(1.西 南大学食品科学学院,重庆 400715;2.重庆市特色食品工程技术研究中心,重庆 400715)
采用实时荧光定量聚合酶链式反应(quantitative real-time polymerase chain reaction,qRT-PCR)技术定量监测6 g/100 mL盐质量浓度腌制麻竹笋中乳酸乳球菌(Lactococcus lactis)的动态变化。经乳酸乳球菌标准菌株基因组DNA提取、标准阳性质粒制备、标准曲线绘制、各时期竹笋腌制发酵液中细菌基因组DNA提取和乳酸乳球菌qRTPCR特异性扩增,对腌制液中乳酸乳球菌进行定量检测。结果表明,在腌制过程中(0~63 d),随着腌制时间的延长,乳酸乳球菌含量逐渐升高,在腌制14 d时达到最大值(4.63×108copies/øL),与0 d(2.41×102copies/øL)相比增加了6 个数量级,而后浓度缓慢降低,在腌制63 d时浓度为5.02×106copies/øL。qRT-PCR技术为定量监测腌制麻竹笋中微生物的动态变化提供了一条可靠、快速的有效途径。
大叶麻竹笋;腌制;实时荧光定量PCR;乳酸乳球菌;动态变化
大叶麻竹笋(Dendrocalamus latiflorus)是著名的高产型竹笋,广泛分布于我国南亚热带和热带地区[1]。腌制大叶麻竹笋加工简易,成本低廉,易于保存,且具有较高的营养价值、独特的风味,深受消费者的喜爱[2]。在腌制发酵过程中,麻竹笋会形成多种风味化合物,使其具有特殊的风味和营养价值[3]。而在风味形成过程中,除了其本身的风味外,其他的风味形成途径均与微生物的发酵作用有关[4],因此,了解麻竹笋腌制过程中优势微生物的动态变化对于研究腌制麻竹笋的风味品质变化具有重要作用。
乳酸菌具有良好的感官特性和防腐特性,在蔬菜发酵过程中发挥着重要作用[5]。其中,乳酸乳球菌(Lactococcus lactis)可形成柠檬酸,参与乙酰风味化合物的合成,并参与发酵初始风味的形成[6]。近年来,国内外诸多学者从腌制发酵蔬菜中分离鉴定出乳酸乳球菌,并对其益生作用进行研究。如付琳琳等[7]采用聚合酶链式反应-变性梯度凝胶电泳技术 从不同家庭制作的成熟期的泡菜液中鉴定出乳酸乳球菌。尹军霞等[8]研究了分离自酸菜汁的乳酸乳球菌的体外去除胆固醇特性。连元元等[9]从泡菜中提取并保存了一株泡菜乳酸乳球菌,并发现其具有较高的耐镉能力。Seema等[10]从腌制山药中分离出一株乳酸乳球菌并对其益生特性进行初步研究。Aso等[11]从日本传统“津田芜菁”泡菜中分离出一株乳酸乳 球菌,并对其产Nisin Z的能力进行探讨。本课题组前期的研究也表明乳酸乳球菌是腌制麻竹笋中的优势菌群,但其 含量及在腌制加工过程中的动态变化还有待进一步研究[4]。
实时荧光定量聚合酶链式反应(quantitative real-time polymerase chain reaction,qRT-PCR)技术可定量研究微生物菌落结构组成及数量变化,为全面、快速、准确地分析鉴定食品[12-15]中的各种微生物提供了一种崭新的技术工具和平台。近年来,也有部分学者采用qRT-PCR技术对食品中的乳酸乳球菌进行定量研究,如Grattepanche[6]和Achilleos[16]等分别采用qRT-PCR技术对混合发酵乳和芝士中的乳酸乳球菌进行鉴定。而有关麻竹笋腌制过程中乳酸乳球菌的qRT-PCR研究尚未见报道。因此,本实验采用qRT-PCR技术对腌制麻竹笋中的乳酸乳球菌变化进行定量监测,以期为腌制麻竹笋中风味物质的形成及变化机理研究提供实验依据。
1 材料与方法
1.1 材料与试剂
腌制麻竹笋:大叶麻竹笋原料采于重庆市北碚区施家梁镇大叶麻竹笋种植基地,将麻竹笋洗净、切分、漂烫、沥干、冷却后,按传统加工方法加盐(6 g/100 mL)在室温条件下腌制。从腌制当天开始取样,第3天取一次样,之后每隔一周取一次样,共取11 次样(0、3、7、14、21、28、35、42、49、56 d和63 d),取样完成时产品已基本成熟。样品-20 ℃冰箱保存,至最后一次取样完成后统一进行DNA提取。
乳酸乳球菌乳亚种(Lactococcus lactis subsp. lactis)CGMCC1.1936 中国普通微生物菌种保藏管理中心。
MRS培养基用试剂及配制方法参照文献[17];基因组DNA提取和克隆用试剂及配制方法参考文献[18];细菌基因组DNA提取试剂盒(DP302)、溶菌酶、琼脂糖、GeneGreen核酸染料(RT210)、琼脂糖凝胶回收试剂盒(DP209)、pGM-T克隆试剂盒(VT202)、DH5α感受态细胞(CB101-03)、质粒小提试剂盒(DP103) 天根生化科技(北京)有限公司;TaKaRa Taq扩增体系套装(R001A)、SYBR Premix Ex TaqTMⅡ(DRR820A) 宝生物工程(大连)有限公司;毛细管 上海大菱制塑有限公司;Lambda DNA/Hind ⅢMarker(NMW009)、100 bp ladder Ⅱ(NMW004)北京鼎国昌盛生物技术有限责任公司;实验所用引物由生工生物工程(上海)股份有限公司合成;化学试剂均为分析纯。
1.2 仪器与设备
S1000伯乐1000系列高性能PCR仪、164-5050基础电源电泳仪 美国Bio-Rad公司;Syngene GeneGenius凝胶成像系统 基因科技(上海)有限公司;LightScanner 32荧光定量PCR仪 美国Idaho公司;NanoDrop 2000微量紫外分光光度计 美国Thermo公司;SIM-F140AY65型制冰机 三洋电机国际贸易有限公司;SW-CJ-1F型单人双面净化工作台 苏州净化设备有限公司;1-15PK小型台式离心机 德国Sigma公司;ES-315高压灭菌锅日本Tomy公司;HWS-26电热恒温水浴锅、DHP-9272电热恒温培养箱 上海齐欣科学仪器有限公司。
1.3 方法
1.3.1 标准品的制备及标准曲线的建立
1.3.1.1 标准菌株模板DNA的制备
将乳酸乳球菌按照标准菌株培养条件接种至MRS液体培养基中,30 ℃恒温振荡培养过夜,经2 次活化后采用天根细菌基因组DNA提取试剂盒提取细菌基因组DNA。用溶菌酶对菌体进行破壁处理,提取结束时用80 μL TE buffer将DNA溶出。经0.8%琼脂糖凝聚电泳,GeneGreen染色检测后,-20 ℃保存备用,目标片段应在23 kb左右[19]。
1.3.1.2 目的片段的扩增纯化
采用特异性引物对标准菌株基因组DNA进行常规PCR扩增,用以制备标准阳性质粒[20]。正向引物L. lactis-F:5’-TGA AGA ATT GAT GGA ACT CG-3’,反向引物L. lactis-R:5’-CAT TGT GGT TCA CCG TTC-3’,目标片段约为126 bp[16]。扩增体系为:5 μL DNA模板,正向和反向引物各0.25 μL,10×PCR buffer(Mg2+plus)10 μL,dNTP Mixture 3 μL,Taq DNA聚合酶0.5 μL,灭菌超纯水补足至50 μL。反应程序为:94 ℃预变性5 min,94 ℃变性40 s,56 ℃退火40 s,72 ℃延伸1 min,35 个循环,72 ℃延伸7 min,4 ℃保存。扩增产物经3%琼脂糖凝胶电泳检测后,用无菌手术刀在紫外灯下切取目标片段,并采用琼脂糖凝胶回收试剂盒对其进行纯化回收。回收产物经琼脂糖凝胶电泳检测纯度后-20 ℃保存备用。
1.3.1.3 标准阳性质粒的制备[21]
使用天根pGM-T克隆试剂盒将已纯化标准菌株目的片段与质粒进行连接,并将4 øL连接反应物加入至50 øL DH5α感受态细胞中进行转化。利用蓝白斑筛选法挑选白色阳性克隆菌落后采用质粒小提试剂盒提取质粒,具体操作详见试剂盒说明书。
为避免质粒假阳性,采用通用引物T7(5’-TAA TAC GAC TCA CTA TAG GG-3’)和SP6(5’-ATT TAG GTG ACA CTA TAG-3’)对提取质粒进行扩增,扩增产物用做荧光定量PCR反应的标准品以绘制标准曲线。扩增体系同1.3.1.2节,扩增程序为:94 ℃预变性5 min,94 ℃变性40 s,55 ℃退火40 s,72 ℃延伸1 min,35 个循环,72 ℃延伸7 min,4 ℃保存。扩增产物用3%的琼脂糖凝胶电泳和微量紫外分光光度计检测其浓度与纯度后,送至生工生物工程(上海)股份有限公司测序。
1.3.1.4 qRT-PCR条件优化
以L. lactis-F和L. lactis-R为引物,以得到最小的Ct值(荧光信号达到设定阈值时所经历的循环数)、最大的荧光值和熔解曲线不产生非特异性峰为指标,对乳酸乳球菌qRT-PCR的退火温度、退火时间、引物浓度以及荧光数据收集温度等条件进行优化,确定扩增的最佳条件[22]。
1.3.1.5 标准曲线的绘制[12,23]
由微量紫外分光光度计测定的标准阳性质粒PCR产物浓度,结合产物测序得到的片段大小,根据公式(1)计算出PCR产物中所含的初始拷贝数。

稀释质粒扩增产物,观察不同浓度模板对扩增效率及荧光吸收强度的影响,从而确定合适的模板浓度范围。在合适的浓度范围内以无菌超纯水连续10 倍梯度稀释,将其作为阳性模板,按照优化后的条件进行荧光定量PCR反应,得到不同浓度梯度模板的PCR扩增曲线图。再以不同浓度阳性模板的拷贝数对数为横坐标,Ct值为纵坐标绘制出标准曲线,得到标准菌株的线性回归方程及相关系数(R2)。
1.3.2 各时期竹笋腌制发酵液中乳酸乳球菌含量的qRT-PCR检测
取3 mL各时期竹笋腌制发酵液,10 000 r/min离心1 min,弃上清液,加入180 øL 20 mg/mL的溶菌酶,37 ℃处理2 h后采用天根细菌基因组DNA提取试剂盒提取细菌基因组DNA。DNA用80 μL TE buffer溶出后置于-20 ℃冰箱保存备用。采用优化好的qRT-PCR条件和体系对各时期竹笋腌制发酵液细菌基因组DNA中乳酸乳球菌含量进行检测。
1.4 数据处理
实验数据采用Origin(Version 8.6)和Excel(Version 2013)软件进行处理与分析。
2 结果与分析
2.1 实时荧光定量PCR方法的建立
2.1.1 标准菌株模板DNA提取结果
乳酸乳球菌标准菌株基因组DNA电泳结果见图1,菌株基因组DNA在目标位置处有亮带,且无拖尾,说明基因组质量较好。
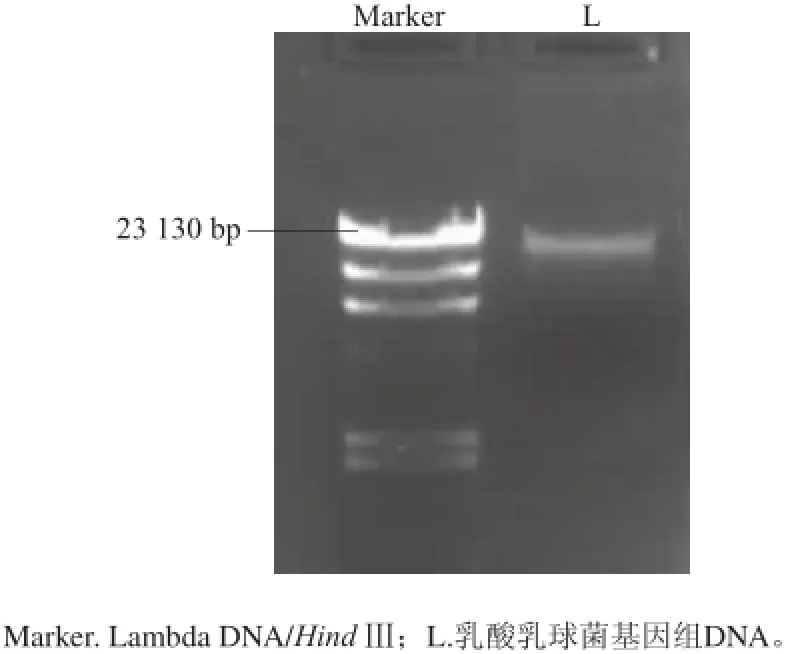
图1 乳酸乳球菌DNA凝胶电泳图Fig.1 Agarose gel electrophoresis of L. lactis genomic DNA
2.1.2 标准阳性质粒制备结果

图2 乳酸乳球菌常规PCR及阳性质粒PCR产物电泳图Fig.2 Agarose gel electrophoresis of PCR amplified products of L. lactis and its positive plasmids
以提取的乳酸乳球菌基因组DNA为模板,经特异性引物常规PCR扩增后,对目的片段进行连接、转化、克隆,再采用通用引物T7和SP6对质粒进行扩增。常规PCR产物和质粒PCR产物的琼脂糖凝胶电泳检测结果如图2所示。
由图2可知,常规PCR和质粒PCR扩增产物条带均清晰,无杂带、无拖尾,且常规PCR产物片段大小在126 bp左右,与预期结果一致。由于质粒扩增得到的片段是目的片段与pGM-T载体的部分序列(约145 bp)的连接产物,因此菌株的阳性质粒扩增产物大小约为271 bp,与图中一致。
经紫外分光光度计检测,菌株阳性质粒扩增产物的OD260nm为5.266±0.220,OD280nm为2.846±0.072,O D260nm/O D280nm为1.8 5±0.0 3,质量浓度为(263.3±11.0) ng/μL,质量较好。经序列分析,除去两端质粒片段,得到127 bp的目的片段,与预期结果相一致。且经BLAST比对(http://blast.ncbi.nlm.nih.gov/Blast. cgi)所得序列与乳酸乳球菌乳亚种(Lactococcus lactis subsp. lactis)的核苷酸序列同源性为100%,说明所构建的重组质粒含有目的片段,可作为标准品用于qRT-PCR中。2.1.3 qRT-PCR反应条件优化结果经优化,扩增采用25 øL的反应体系,反应体系见表1。

表1 qRT-PCR反应体系Table 1 Reaction system for qRT-PCR amplification
利用SYBR Green Ⅰ染料法进行实时监测,选择两步法PCR扩增程序进行扩增。反应程序为:第一步:95 ℃预变性30 s,20 ℃/s,1 个循环,不采集信号;第二步:95 ℃变性5 s,20 ℃/s,60 ℃退火20 s,20 ℃/s,每个循环采集荧光信号,50 个循环。熔解程序为:95 ℃、0 s,20 ℃/s;65 ℃、15 s,20 ℃/s;95 ℃、0 s,0.1 ℃/s,持续采集信号;50 ℃冷却30 s,20 ℃/s。
2.1.4 标准曲线的建立
对阳性标准品进行系列稀释(10-2、10-4、10-6、10-8、10-10),采用优化的条件进行qRT-PCR,不同浓度梯度阳性模板的PCR扩增荧光曲线图如图3A所示,其熔解曲线见图3B。由图3可知,各浓度扩增产物均在(78.7±0.4) ℃出现荧光峰值,无杂峰,说明产物均一性良好,无非特异扩增[24]。以相对拷贝数的对数为横坐标,以Ct值为纵坐标,得出菌株标准曲线。回归方程为:y=-3.868x+45.142,R2=0.998 5,线性关系良好。

图3 乳酸乳球菌荧光定量PCR荧光曲线(A)与熔解曲线(B)Fig.3 Quantitative PCR fluorescence and melting curves for Lactococcus lactis
2.2 各时期竹笋腌制发酵液中乳酸乳球菌含量检测结果
由于3 mL腌制发酵液中细菌较少,细菌基因组DNA条带较暗,故未提供其电泳结果。采用优化好的qRTPCR条件和体系对各时期竹笋腌制发酵液中乳酸乳球菌含量进行检测,根据熔解曲线分析PCR产物的特异性,根据样品的扩增曲线可计算得出Ct值,再带入标准曲线方程中,可得各样品中乳酸乳球菌的拷贝数对数值(lgC)见图4。使用Power函数对纵坐标进行换算,得出各时期竹笋腌制发酵液中乳酸乳球菌的拷贝数,在腌制0~3 d时,乳酸乳球菌含量略有上升,但浓度较低(分别为2.41×102copies/øL和1.78×103copies/øL)。随着腌制时间的延长,乳酸乳球菌浓度迅速升高,在腌制7 d时浓度为3.47×105copies/øL,而在腌制14 d时达到最大值,含量为4.63×108copies/øL,与腌制0 d相比增加了6 个数量级。之后浓度缓慢下降,在腌制21~63 d期间,乳酸乳球菌浓度从1.40×108copies/øL波动下降至5.02×106copies/øL。
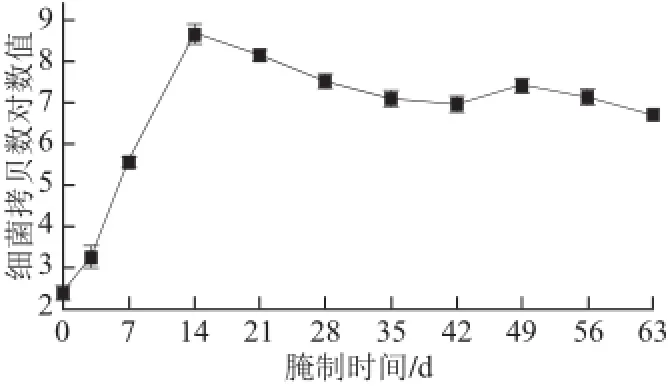
图4 麻竹笋腌制液中乳酸乳球菌的动态变化Fig.4 Changes in of Lactococcus lactis concentration in pickled ma bamboo shoots during pickling
3 讨论与结论
本研究采用qRT-PCR技术对6 g/100 mL盐质量浓度腌制麻竹笋发酵液中乳酸乳球菌含量的动态变化进行监控。经标准菌株基因组DNA提取、扩增、克隆、制备出标准阳性质粒,再经扩增获得阳性模板,再用优化后的qRT-PCR技术制备出标准曲线。提取各时期竹笋腌制发酵液中细菌基因组DNA,对其中乳酸乳球菌的含量进行qRT-PCR定量检测。结果表明,随着腌制时间的延长,乳酸乳球菌的含量逐渐升高,在腌制14 d时达到最大值(4.63×108copies/øL),而后浓度缓慢降低,在腌制63 d时浓度为5.02×106copies/øL。
乳酸乳球菌(L. l a c t i s)归属于硬壁菌门(F i r m i c u t e s)杆菌纲(B a c i l l i)乳杆菌目(Lactobacillales)链球菌科(Streptococcaceae)乳球菌属(Lactococcus)[25]。Aso[11]和Harri[26]等研究表明乳酸乳球菌能在发酵初期产生细菌素Nisin,抑制存在于盐水中的一些食源性致病菌和一些革兰氏阴性菌生长,且乳酸乳球菌为同型发酵,通过糖酵解途径产生乳酸。Xiong Tao等[27]研究表明,乳酸乳球菌在酸菜腌制过程中生长速度快,且产酸能力强,但耐酸能力差。本课题组之前的研究也表明,随着发酵时间的延长,麻竹笋发酵液pH值逐渐降低[2]。因此,可推测本研究中乳酸乳球菌浓度先升高再降低的原因是由于发酵开始时乳酸乳球菌产酸、产细菌素,抑制了其他细菌的生长,使得它的浓度迅速升高,而随着发酵时间的延长,发酵液pH值降低,乳酸乳球菌活力下降。Xiong Tao等[28]研究表明,在泡菜发酵过程中,乳酸乳球菌在发酵初期慢慢出现,在后期死亡,与本研究相一致。
综上所述,qRT-PCR技术为腌制麻竹笋中的微生物动态变化的定量监测提供了一条可靠、快速的有效途径。本实验仅对腌制大叶麻竹笋中乳酸乳球菌的定量变化进行了检测,进一步的研究可对腌制麻竹笋中不同微生物进行定量检测,以探讨腌制发酵液中微生物的交互作用对其风味物质的形成作用,为麻竹笋的风味形成机理和腌制工艺的改进提供一定的参考依据。
[1] SANTOSH S, LALIT M B, POONAM S, et al. Bamboo shoot processing: food quality and safety aspect[J]. Trends in Food Science and Technology, 2010, 21(4): 181-189. DOI:10.1016/ j.tifs.2009.11.002.
[2] 陈光静, 汪莉莎, 郑炯, 等. 食盐质量浓度对大叶麻竹笋腌制过程中品质特性的影响[J]. 食品科学, 2013, 34(15): 48-52. DOI:10.7506/ spkx1002-6630-201315010.
[3] 郑炯, 宋家芯, 陈光静, 等. 顶空-固相微萃取-气质联用法分析腌制麻竹笋挥发性成分[J]. 食品科学, 2013, 34(18): 193-196. DOI:10.7506/spkx1002-6630-201318039.
[4] 郑炯, 夏雪娟, 叶秀娟, 等. PCR-DGGE技术分析腌制麻竹笋中微生物多样性[J]. 食品科学, 2014, 35(21): 170-174. DOI:10.7506/ spkx1002-6630-201421033.
[5] HOLZAPFEL W. Use of starter cultures in fermentation on a household scale[J]. Food Control, 1997, 8(5): 241-258. DOI:10.1016/ S0956-7135(97)00017-0.
[6] GRATTEPANCHE F, LACROIX C, AUDET P, et al. Quantification by real-time PCR of Lactococcus lactis subsp cremoris in milk fermented by a mixed culture[J]. Applied Microbiology and Biotechnology, 2005, 66(4): 414-421. DOI:10.1007/s00253-004-1822-0.
[7] 付琳琳, 曹郁生, 李海星, 等. 应用PCR-DGGE技术分析泡菜中乳酸菌的多样性[J]. 食品与发酵工业, 2005, 31(12): 103-105. DOI:10.3321/j.issn:0253-990X.2005.12.027.
[8] 尹军霞, 沈国娟, 谢亚芳. 分离自酸菜汁的乳酸乳球菌体外去除胆固醇特性[J]. 食品与生物技术学报, 2009, 28(6): 850-853. DOI:10.3321/j.issn:1673-1689.2009.06.025.
[9] 连元元, 王颖, 李晨, 等. 镉胁迫下泡菜乳酸乳球菌的形态变化[J]. 食品科学, 2015, 36(1): 124-127. DOI:10.7506/spkx1002-6630-201501024.
[10] SEEMA B, ARASHDEEP S, ABHIJIT G. Probiotic characterization of potential hydrolases producing Lactococcus lactis subsp. lactis isolated from pickled yam[J]. International Journal of Food Sciences and Nutrition, 2014, 65(1): 53-61. DOI:10.3109/09637486.2013.832175.
[11] ASO Y, TAKEDA A, SATO M, et al. Characterization of lactic acid bacteria coexisting with a nisin Z producer in Tsuda-turnip pickles[J]. Current Microbiology, 2008, 57(1): 89-94. DOI:10.1007/s00284-008-9161-5.
[12] XU W, HUANG Z Y, ZHANG X J, et al. Monitoring the microbial community during solid-state acetic acid fermentation of Zhenjiang aromatic vinegar[J]. Food Microbiology, 2011, 28(6): 1175-1181. DOI:10.1016/j.fm.2011.03.011.
[13] ZHANG W J, CAI Q, GUAN X, et al. Detection of peanut (Arachis hypogaea) allergen by real-time PCR method with internal amplification control[J]. Food Chemistry, 2015, 174: 547-552. DOI:10.1016/j.foodchem.2014.11.091.
[14] ZHAO Y W, WU Z F, SHEN X Q, et al. Bacteria community analysis by quantitative real-time PCR of fermenting wax gourd and its changes of organic acids[J]. Journal of Food Processing and Preservation, 2014, 38(4): 1653-1659. DOI:10.1111/jfpp.12127.
[15] ROSANNA T, MARIA S, GIORGIA P, et al. Development and application of a real-time PCR-based assay to enumerate total yeasts and Pichia anomala, Pichia guillermondii and Pichia kluyveri in fermented table olives[J]. Food Control, 2012, 23(2): 356-362. DOI:10.1016/j.foodcont.2011.07.032.
[16] ACHILLEOS C, BERTHIER F. Quantitative PCR for the specific quantification of Lactococcus lactis and Lactobacillus paracasei and its interest for Lactococcus lactis in cheese samples[J]. Food Microbiology, 2013, 36(2): 286-295. DOI:10.1016/j.fm.2013.06.024.
[17] 李平兰, 贺稚非. 食品微生物学实验原理与技术[M]. 北京: 中国农业出版社, 2005: 264.
[18] 萨姆布鲁克 J, 拉塞尔 D W. 分子克隆实验指南[M]. 2版. 黄培堂,译. 北京: 科学出版社, 2002: 1564-1594.
[19] 夏雪娟, 陈芝兰, 陈宗道, 等. 16S rDNA序列分析法快速鉴定西藏地区传统乳制品中的乳酸菌[J]. 食品科学, 2013, 34(14): 245-249. DOI:10.7506/spkx1002-6630-201314050.
[20] XU W T, LI L Y, LU J, et al. Analysis of caecal microbiota in rats fed with genetically modified rice by real-time quantitative PCR[J]. Journal of Food Science, 2011, 76(1): M88-M93. DOI:10.1111/1750-3841.12604.
[21] LIU J X, FENG Y M, CHEN Q S. The distribution of somatostatin precursor 1 and preproenkephalin mRNA in chicken intestinal nerve of Remak[J]. Acta Zoologica Sinica, 2007, 53(2): 294-302. DOI:10.3969/ j.issn.1674-5507.2007.02.014.
[22] 赵雪涛. 荧光定量PCR测定食品中腊样芽孢杆菌[D]. 上海: 复旦大学, 2006.
[23] KAMILA O S, OLGA K R, ADLEY C C. Development of realtime pcr assays for detection and quantification of Bacillus cereus group species: differentiation of B. weihenstephanensis and rhizoid B. pseudomycoides isolates from milk[J]. Applied and Environmental Microbiology, 2011, 77(1): 80-88. DOI:10.1128/AEM.01581-10.
[24] 骞宇. 抗消化淀粉对实验鼠肠道生理环境和胃肠功能性作用的影响[D]. 重庆: 西南大学, 2013.
[25] 张家超, 王芳, 徐海燕, 等. 6 种区分乳酸乳球菌乳酸亚种和乳酸乳球菌乳脂亚种的分子生物学方法比较[J]. 微生物学报, 2010, 50(12): 1670-1676. DOI:10.13343/j.cnki.wsxb.2010.12.007.
[26] HARRI L J, FLEMING H P, KLAENHAMMER T R. Characterization of 2 nisin-producing Lactococcus-lactis subsp. lactis strains isolated from a commercial sauerkraut fermentation[J]. Applied and Environmental Microbiology, 1992, 58(5): 1477-1483.
[27] XIONG T, LI X, GUAN Q Q, et al. Starter culture fermentation of Chinese sauerkraut: growth, acidification and metabolic analyses[J]. Food Control, 2014, 41: 122-127. DOI:10.1016/ j.foodcont.2013.12.033.
[28] XIONG T, GUAN Q Q, SONG S H, et al. Dynamic changes of lactic acid bacteria flora during Chinese sauerkraut fermentation[J]. Food Control, 2012, 26(1): 178-181. DOI:10.1016/j.foodcont.2012.01.027.
Detection of Lactococcus lactis in Pickled Ma Bamboo Shoots by Quantitative Real-time PCR
XIA Xuejuan1, ZHENG Jiong1,2,*, YE Xiujuan1, WU Jinsong1, KAN Jianquan1,2
(1. College of Food Science, Southwest University, Chongqing 400715, China; 2. Chongqing Engineering Research Center of Regional Food, Chongqing 400715, China)
The dynamic changes of Lactococcus lactis in pickled Ma bamboo shoots with 6 g/100 mL salt concentration were studied by quanti tative real-time polymerase chain reaction (qRT-PCR). After the DNA extraction from standard Lactococcus lactis, preparation of positive plasmids, protraction of standard curve, DNA extraction from samples at different fermentation stages and qRT-PCR, and quantitative detection were performed. The results showed that during fermentation (0–63 days), the concentration of Lactococcus lactis increased gradually from 2.41 × 102copies/øL (day 0) to the maximum value of 4.63 × 108copies/øL (day 14), an increase by six orders of magnitude, followed by a slow decrease to 5.02 × 106copies/øL at day 63. In conclusion, qRT-PCR technology provides a reliable, fast and effective way for the quantitative analysis of bacteria in pickled Ma bamboo shoots.
Ma bamboo shoots (Dendrocalamus latiflorus); pickling; quantitative real-time polymerase chain reaction (qRT-PCR); Lactococcus lactis; dynamic changes
10.7506/spkx1002-6630-201604016
TS201.3
A
1002-6630(2016)04-0088-05
夏雪娟, 郑炯, 叶秀娟, 等. 实时荧光定量PCR技术监测腌制麻竹笋中乳酸乳球菌动态变化[J]. 食品科学, 2016, 37(4): 88-92. DOI:10.7506/spkx1002-6630-201604016. http://www.spkx.net.cn
XIA Xuejuan, ZHENG Jiong, YE Xiujuan, et al. Detection of Lactococcus lactis in pickled ma bamboo shoots by quantitative real-time PCR[J]. Food Science, 2016, 37(4): 88-92. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201604016. http://www.spkx.net.cn
2015-03-29
中央高校基本科研业务费专项(XDJK2013C131);重庆市特色食品工程技术研究中心能力提升项目(cstc2014pt-gc8001)
夏雪娟(1988—),女,博士研究生,研究方向为食品化学与营养学。E-mail:xiaxuej1989@163.com
*通信作者:郑炯(1982—),男,讲师,博士,研究方向为果蔬加工与质量控制。E-mail:zhengjiong_swu@126.com
