不同植物中推定蓖麻烯合酶基因的生物信息学分析
2016-11-10刘洪伟杨艳芳熊王丹吴平治吴国江邱德有
刘洪伟 杨艳芳 熊王丹 吴平治 吴国江 邱德有*
(1.林木遗传育种国家重点实验室,中国林业科学研究院林业研究所,北京 100091; 2.中国科学院华南植物园植物资源保护与可持续利用重点实验室,广州 510650)
不同植物中推定蓖麻烯合酶基因的生物信息学分析
刘洪伟1杨艳芳1熊王丹2吴平治2吴国江2邱德有1*
(1.林木遗传育种国家重点实验室,中国林业科学研究院林业研究所,北京 100091;2.中国科学院华南植物园植物资源保护与可持续利用重点实验室,广州 510650)
利用GenBank中已登录的完整的麻风树、乳浆大戟、蓖麻和乌桕中的13个蓖麻烯合酶(Casbene synthase,CS;EC 4.6.1.7)基因序列,通过生物信息学方法对其核酸及氨基酸序列、组成成分、导肽、信号肽、跨膜结构域、疏水性/亲水性、蛋白质的二级结构、三级结构及功能域等进行了分析预测。结果表明,13个CS基因的ORF长度均在1 647~1 845 bp,蛋白分子量均在63.0~70.8 kD,终止密码子为TGA或TAA,理论等电点均小于7.0,表明CS蛋白呈酸性。氨基酸含量最高的均为亮氨酸。核苷酸同源性比较分析表明,CS基因主要分为两类。导肽预测发现其中6个CS具有导肽,均为叶绿体导肽。信号肽和扩模结构域预测发现这些CS不存在信号肽和跨膜结构域,肽链整体呈现为亲水性。这些CS的主要二级结构元件为α-螺旋,并且都包含两个萜类合酶功能域。以上研究为进一步探索CS基因的功能提供一定理论依据。
巴豆烷;蓖麻烯合酶;生物信息学
二萜类化合物通常都是由牻牛儿基牻牛儿基焦磷酸(Geranylgeranyl pyrophosphate,GGPP)环化得到。巴豆烷是一类非常重要的二萜化合物的母核,这类化合物统称为佛波酯类化合物,它是麻风树(JatrophacurcasL.)主要毒性因素之一,并因为它的癌症诱导作用而受到广泛关注[1]。近年热门化合物prostratin也是一种佛波酯类化合物,它是在1992年由美国国立癌症研究所(NCS)分离得到,并在2001年证实其具有抵抗艾滋病病毒的作用[2]。因此,研究佛波酯类化合物的母核的生物合成途径很有必要,很多科学家也对这类化合物的母核生物合成途径产生了浓厚的兴趣。
巴豆烷类化合物的生物合成途径涉及多个酶促反应,而且路径尚不明晰,但是人们基本确定巴豆烷型二萜生物合成过程中的关键酶是蓖麻烯合酶(Casbene synthase,CS;EC 4.6.1.7),它首先催化GGPP环化生成反应中间体西松烷(Cembrane),接着西松烷通过其他途径变成蓖麻烯(Casbene),蓖麻烯再通过一系列电子转移形成巴豆烷(Tigliane)[3]。目前科学家已从蓖麻(Ricinuscommunis)、乌桕(Triadicasebifera)、乳浆大戟(Euphorbiaesula)、白角麒麟(Euphorbiaresinifera)和Mamala树(Homalanthusnutans)等多种植物中克隆得到CS基因[4]。由于麻风树具有作为生物能源材料的潜质,Sato等在2010年完成了其基因组测序,发现了9个CS类似基因(经分析认为其中一个为假基因),并公布了其中6个基因的氨基酸序列[5]。
生物信息学是当代生命科学与信息科学、计算机科学、统计学、数学、物理学、化学等多种学科彼此渗透而高度交织形成的一门新兴的前沿学科[6]。它可以通过DNA或cDNA序列的信息分析作为出发点,研究基因的功能,预测研究基因的产物即蛋白质,预测蛋白质的定位及行使功能的区域,模拟蛋白质的空间结构,分析蛋白质的性质等[7]。本研究通过生物信息学的方法,以麻风树的CS基因为重点,对从GenBank数据库中得到的13个完整的麻风树、乳浆大戟、蓖麻和乌桕蓖麻烯合酶CS基因的核苷酸及氨基酸序列的组成、生化特性、结构特点等进行推测和分析,为今后深入研究该类酶的功能和结构特征提供依据。
1 材料和方法
1.1 试验材料
利用GenBank数据库进行搜索,找到13个完整CS基因,其中乳浆大戟中1个(GenBank No.:ADB90273),麻风树中6个(BAJ53213、BAJ53216、BAJ53218、BAJ53219.1、BAJ53220、BAJ53221),蓖麻中5个(XP_002513369、XP_002513343、XP_002513340、XP_002513334、XP_002519897),乌桕中1个(ADB90272)。分别重新命名为:乳浆大戟EeCS,麻风树JcCS1-6,蓖麻RcCS1-5和乌桕TsCS。
1.2 试验方法
依据NCBI、CBS、ExPASy、SWISS-MODEL等网站提供的各类生物信息学软件进行在线分析。其中CS的查找在NCBI的GenBank(http://www.ncbi.nlm.nih.gov/genbank)进行;CS开放阅读框(open reading frame,ORF)的确定使用NCBI-ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)完成;核酸和氨基酸序列的组成成分、理化性质分析则利用ProtParam(http://www.expasy.ch/tools/protparam.html)在线工具进行;氨基酸序列的同源性比对及进化树的构建利用BioEdit和MEGA4.0完成;蛋白质导肽预测通过TargetP1.1(http://www.cbs.dtu.dk/services/TargetP/)以及ChloroP 1.1(http://www.cbs.dtu.dk/services/ChloroP/)完成;蛋白的信号肽使用SignalP3.0(http://www.cbs.dtu.dk/services/SignalP/)进行预测;预测蛋白的跨膜结构域以及亲水性/疏水性时使用在线工具TMHMM2.0(http://www.cbs.dtu.dk/services/TMHMM/)和ProtScale(http://www.expasy.ch/tools/protscale.html)进行;利用SMART(http://smart.embl-heidelberg.de/smart/set_mode.cgi?GENOMIC=1)完成蛋白功能域的预测;蛋白质二级结构预测利用软件SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page/NPSA/npsa_sopma.html)进行;蛋白质的三级结构预测利用ESyPred3D(http://www.fundp.ac.be/sciences/biologie/urbm/bioinfo/esypred/)在线工具完成;蛋白质三级结构模型稳定性分析利用Swiss-Model的ProCheck(http://swissmodel.expasy.org/workspace/index.php?func=tools_structureassessment1)。
2 结果与分析
2.1 核苷酸及氨基酸序列的组成及生化特性分析
用NCBI-ORF Finder、DNAMAN及ProtParam[8]在线工具对上述CS基因的核苷酸序列和氨基酸序列进行分析,结果显示(表1)13个CS基因编码的完整ORF长度在1 647~1 845 bp,预测蛋白分子量在63.0~70.8 kD,终止密码子为TGA或TAA,在麻风树中使用TGA较多。预测蛋白理论等电点均小于7.0,说明CS蛋白呈酸性。麻风树的JcCS 3蛋白中酸性氨基酸含量最高为15.3%,JcCS1蛋白中碱性氨基酸含量最高为12.1%。在这些植物CS蛋白中含量最高的氨基酸均为Leu,但其他氨基酸含量变化较大。JcCS 1和RcCS 1为稳定蛋白,其余CS蛋白均为不稳定蛋白。
表1不同植物CS基因及对应氨基酸序列的组成成分及理化性质分析
Table1Compositionandphysicochemicalcharactercomparisonsofthe13CSgenesandthe13deducedproteins

基因Gene开放阅读框长度Openreadingframe(bp)推导氨基酸残基Deducedaminoacidsresidues(aa)分子量Molecularweight(kD)理论等电点值Theoreticalisoelectricpoint(PI)含量最丰富的氨基酸Themostabundantaminoacidsresidues酸性氨基酸比例Acidicaminoacidsratio(%)碱性氨基酸比例Basicaminoacidsratio(%)不稳定系数Instabilityindex(%)终止密码子TerminatorcodonEeCS179759869.27915.69LeuSerLysGluAsp13.911.743.49不稳定TAAJcCS1180960269.44555.95LeuGluLysSerVal13.512.137.25稳定TGAJcCS2165655163.9465.29LeuGluAlaLysSer14.511.341.82不稳定TAAJcCS3167155664.30235.06LeuGluSerLysVal15.311.244.20不稳定TGAJcCS4167155664.01695.25LeuGluSerLysAla14.711.345.14不稳定TGAJcCS5164754863.01765.23LeuGluSerAlaLys14.610.945.94不稳定TGAJcCS6166855564.20945.39LeuGluLysSerAla14.411.743.55不稳定TGARcCS1166855564.35915.32LeuGluSerAlaIle14.811.737.29稳定TAARcCS2180059968.77535.44LeuSerGluAlaVal14.010.942.01不稳定TGARcCS3180660168.96555.35LeuSerGluAlaLys14.010.641.00不稳定TGARcCS4184561470.81476.10LeuSerGluAlaLys12.911.643.28不稳定TAARcCS5165655163.81965.29LeuGluAlaLysSer15.111.644.41不稳定TAATsCS179459768.91455.40LeuGluSerValLys13.910.746.56不稳定TGA
2.2氨基酸序列的多比对分析及系统进化树的构建
根据氨基酸序列的相似性,利用BioEdit和MEGA4.0[9]软件对13个CS氨基酸序列进行多序列比对,并对其构建系统进化树,采用默认参数,自检1 000次。结果如图1所示,13个CS基因被分成了两大类,其中JcCS2-6、RcCS1和RcCS5八个CS基因被聚成了一大类,其中JcCS3-6聚成了一个亚类,RcCS5、JcCS2和RcCS1聚成了一个亚类;JcCS1、EeCS、TsCS及RcCS2-4为一大类,其中JcCS1单独聚成了一个亚类,EeCS、TsCS和RcCS2-4聚成了一个亚类。这说明麻风树和蓖麻中的CS基因都有两个进化起源,而且相互之间也出现了进化分歧。
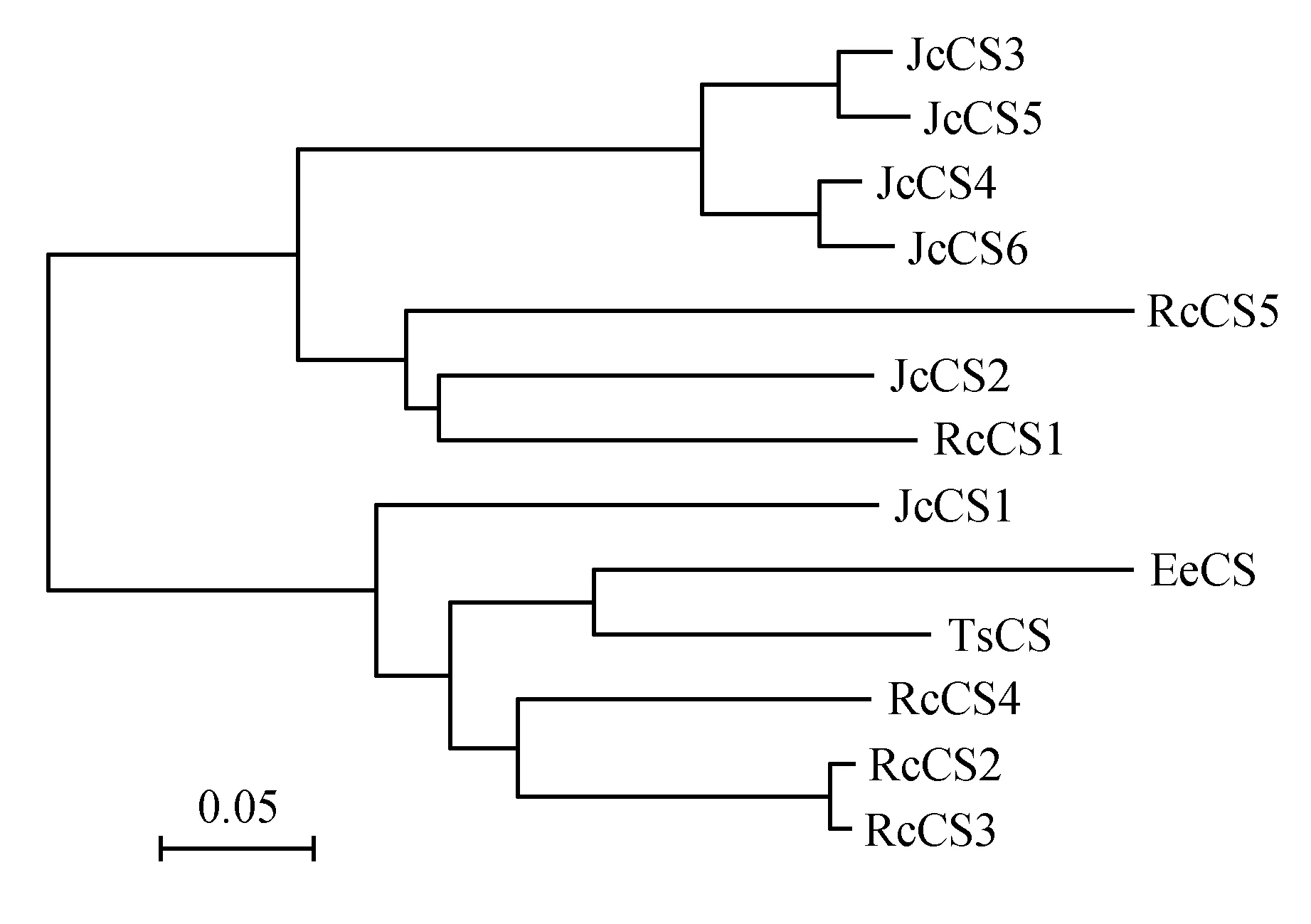
图1 不同植物CS氨基酸序列的系统进化树Fig.1 Phylogenetic tree of 13 CS amino acid sequences
2.3 导肽的预测和分析
有些蛋白能够在细胞中行使其特定功能,需要在合成多肽后先被定位运输到特定的细胞部位,如叶绿体、线粒体等,再装配形成具有一定结构的蛋白质。进行新合成多肽定位的氨基酸序列就是导肽(leader peptite)[10]。导肽通常含有丰富的带正电荷的碱性氨基酸(特别是精氨酸和赖氨酸),如果这些带正电荷的氨基酸被不带电荷的氨基酸取代,这段氨基酸序列就不起引导作用,说明这些带正电荷的氨基酸对于蛋白质的定位具有非常重要的意义[11]。本实验中,利用在线工具TargetP1.1Server[12],选择plant version及默认参数,对13个CS氨基酸系列进行预测,结果如表2所示。通过预测得到EeCS、JcCS1、RcCS2、RcCS3、RcCS4及TcCS可能具有叶绿体导肽,其中EeCS、JcCS1、RcCS3、RcCS4可靠级别在Ⅲ级以内,RcCS2和TcCS可靠级别为Ⅴ级,预测导肽长度在51~72个氨基酸残基。JcCS2、JcCS3、JcCS4、JcCS5、JcCS6、RcCS1及RcCS5没有氨基酸残基分裂位点,因此可能不存在导肽酶切位点,不具导肽。再通过ChloroP 1.1[13]验证发现,EeCS、JcCS1、JcCS2、RcCS3、RcCS4及TcCS具有叶绿体导肽,两者数据并不完全统一。

表2 不同CS氨基酸序列的导肽预测
注:Len.长度;cTP.叶绿体导肽;mTP.线粒体导肽;SP.分泌途径;Loc.定位;C.叶绿体导肽;RC.可靠级别,越小越高;TPlen.预测导肽长度;*. ChloroP 1.1预测结果
Note:Len. Sequence length; cTP. Chloroplast transit peptide; mTP. Mitochondrial targeting peptide; SP. Secretory pathway; Loc. Prediction of localization; C. Chloroplast; RC. Reliability class; TPlen. Predicted presequence length; *. The analysis results of ChloroP 1.1
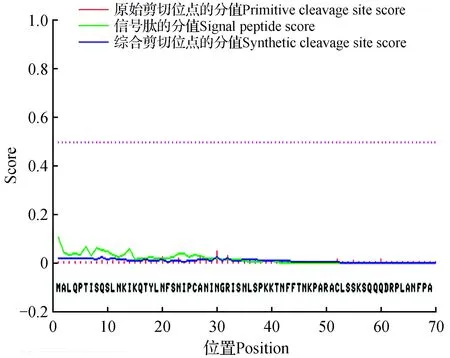
图2 JcCS1氨基酸序列信号肽的预测分析Fig.2 Predicted signal peptide of JcCS1 amino acid sequence
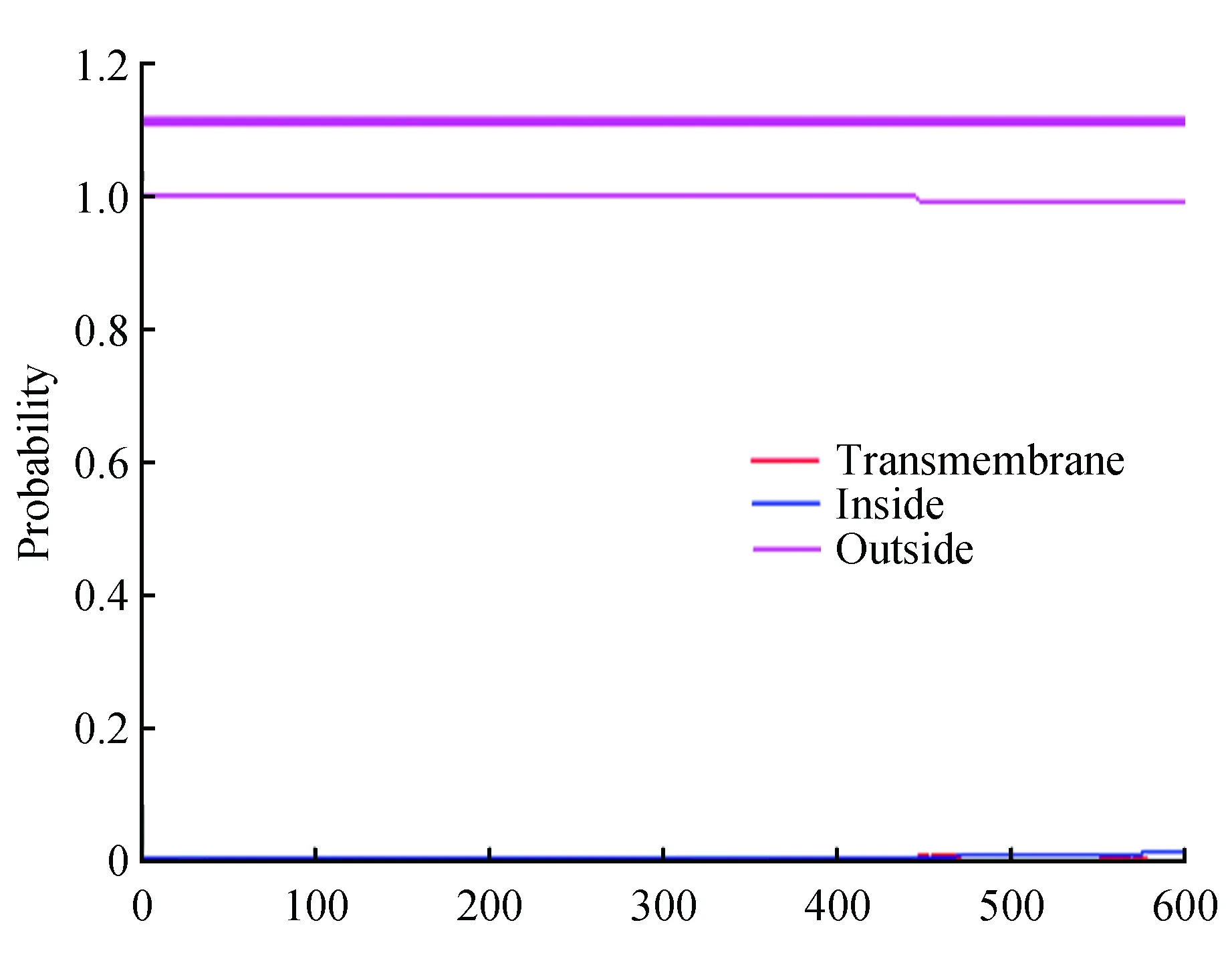
图3 JcCS1氨基酸序列跨膜结构域的预测分析Fig.3 Predicted transmembrane domain of JcCS 1 amino acid sequence
2.4 信号肽的预测与分析
信号肽指导分泌性蛋白到内质网膜上合成,它通常位于蛋白质的N端,在蛋白质合成结束之前就会被切除,它一般有16~26个氨基酸残基。利用在线工具SignalP3.0Server[14]预测JcCS1氨基酸序列的信号肽存在位置及序列(图2),结果表明JcCS1的氨基酸序列不存在信号肽。结合导肽预测的结果可以推测JcCS1在游离核糖体上合成以后,直接运输到叶绿体中发挥作用。运用同样的方法对另外12个CS的氨基酸序列进行分析,均不存在信号肽。
2.5 跨膜结构域的预测与分析
跨膜结构域一般由20个左右疏水性氨基酸残基组成,通常是跨膜蛋白的功能区域,主要形式为α-螺旋。利用TMHMM2.0 Server[15]在线预测JcCS1氨基酸序列的跨膜结构域(图3),结果表明JcCS1整条肽链都在膜的一侧,不具有跨膜结构。运用相同的方法对其他CS氨基酸序列进行分析,均不存在跨膜结构域。
2.6 疏水性/亲水性的预测和分析
蛋白质折叠时形成疏水内核和亲水表面,据此可以测定跨膜螺旋等二级结构和蛋白质表面氨基酸分布[16]。利用在线工具ProtScal[17],选定默认参数,预测JcCS1氨基酸序列的疏水性/亲水性(图4)。预测结果显示,多肽链在第507位(Q)具有最小值-2.889,亲水性最强;在第345位(V)和第349位(I)具有最大值2.611,疏水性最强。从整体上来看,预测结果为亲水性。运用相同的方法对其余CS氨基酸序列进行预测,结果和JcCS1相似,均为亲水性蛋白。

图4 JcCS1氨基酸序列疏水性/亲水性的预测分析Fig.4 Predicted hydrophobicity or hydrophilicity of JcCS1 amino acid sequence
2.7 二级结构的预测与分析
蛋白质的二级结构通常有α-螺旋(α-helix)、β折叠(β-sheet)、转角(turn)、无规则卷曲(coil)以及基序(motif)等[16]。用SOPMA[18]对JcCS 1氨基酸序列的二级结构进行预测(图5),结果表明JcCS 1蛋白的主要结构原件是α-螺旋,其次是无规则卷曲,β-转角和延伸链的含量都很少。对其他CS蛋白进行预测,结果如表3所示,主要结构原件都是α-螺旋和无规则卷曲。
表313个CS基因蛋白质二级结构主要构成组件比例
Tabel3Constitutedthemaincomponentsofproteinsecondarystructureratioofthe13CSgenes

组件名称Componentnameα⁃螺旋Alphahelix(%)β⁃转角Betaturn(%)延伸链Extendedstrand(%)无规则卷曲Randomcoil(%)EeCS67.062.684.1826.09JcCS164.122.825.4827.57JcCS271.693.093.4521.78JcCS369.963.422.7023.92JcCS469.062.523.6024.82JcCS570.073.284.2022.45JcCS669.913.602.7023.78RcCS171.352.883.6022.16RcCS266.113.344.3426.21RcCS365.393.164.4926.96RcCS461.892.775.0530.29RcCS572.052.723.6321.60TsCS66.832.683.6926.80

图5 JcCS1氨基酸序列二级结构的预测Fig.5 Predicted second structure of JcCS1 amino acid sequence
2.8 功能域的预测和分析
功能域(functional domain)是能独立存在于蛋白质分子中的功能单位,功能域可以是一个或多个结构域[19]。结构域是一种介于二级与三级结构之间的独立的结构和功能单位,具有一定的生物学功能[20]。利用在线工具SMART[21]分析JcCS1氨基酸序列功能结构域的结果表明,它具有两个结构域Terpene-synth和Terpene-synth-C。Terpene-synth为N-末端结构域从第72位氨基酸开始到247位氨基酸结束,它行使转运肽功能,将蛋白转运到对应的质体中发挥作用;Terpene-synth-C为金属结合位点区域从第277位氨基酸开始到546位氨基酸结束,可以与Mn2+结合从而行使催化功能[22]。利用相同的方法对其他CS氨基酸序列进行预测,结果与JcCS1相同,均包含这两个功能域,只是起始位置略有偏差。
2.9 三级结构的预测与分析
利用ESyPred3D Web Server 1.0[23]同源建模的方法,选用神经网络和新展示技术预测CS蛋白的三级结构,如图7所示为麻风树(JcCS 1)、乳浆大戟、蓖麻(RcCS 1)和乌桕CS蛋白质的三级结构。由三级结构可以看出蓖麻烯合酶属Ⅰ类萜类合酶(α区,蓝色区)[24],其包含金属离子结合区DDXXD和(N/D)DXX(S/T)XXXE(分别为红色和橙色区域)[25];同时连着一个小的退化区(β区,绿色区),这类区主要在Ⅱ类萜类合酶中发挥作用[26];紫色区域为第一个α螺旋,在I类萜类合酶中起给活性中心加盖的作用[25]。
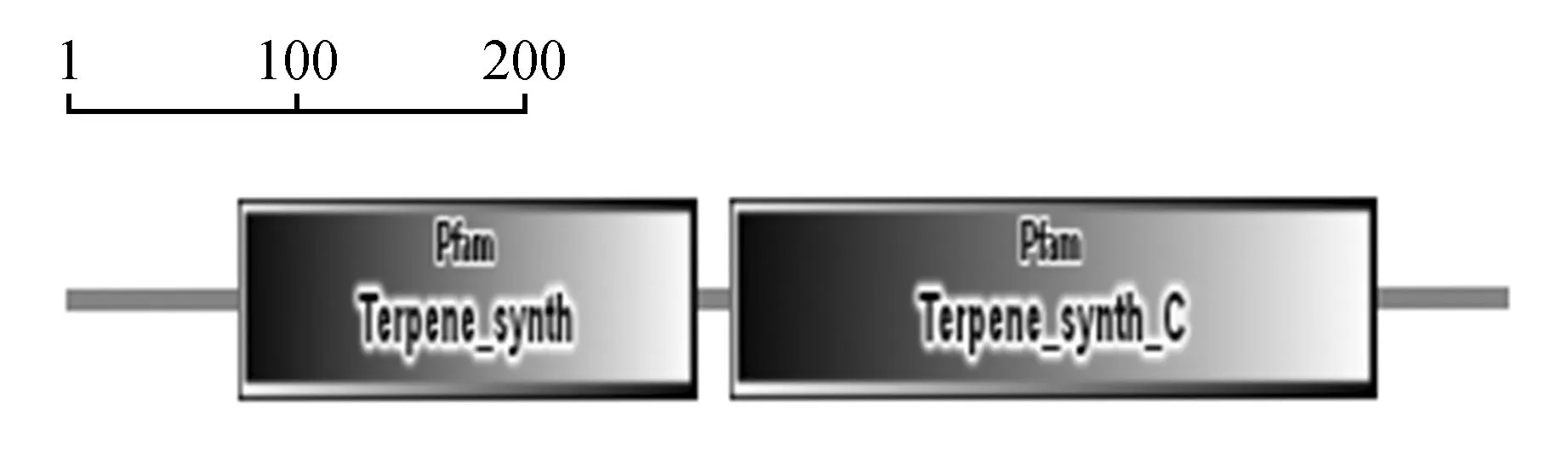
图6 JcCS1蛋白结构域位点预测Fig.6 Predicted protein domain sites of JcCS1 amino acid sequence
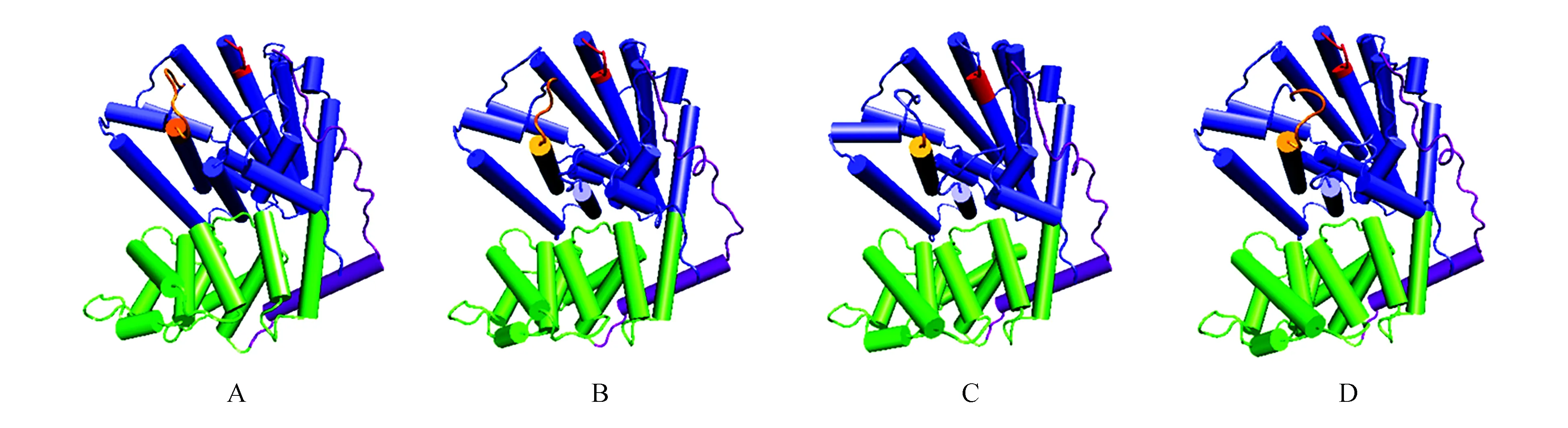
图7 麻风树JcCS1(A)、乳浆大戟EeCS(B)、蓖麻RcCS1(C)和乌桕TsCS(D)CS的三维高级结构预测 蓝色. α区;红色. 金属离子结合区DDXXD;橙色. 金属离子结合区(N/D)DXX(S/T)XXXE;绿色. β区;紫色. 第一个α螺旋Fig.7 Predicted three-dimensional structure of JcCS1(A),EeCS(B),RcCS1(C) and TsCS(D) Blue. α domain; Red. Metal-binding motif DDXXD; Orange. Metal-binding motif (N/D)DXX(S/T)XXXE; Green. β domain; Purple. The first α helix
利用ProCheck[27]对建模结果进行监测,计算出Ramachandran图。Ramachandran图是反映立体化学质量的参数,它通过分析Phi(φ)角和Psi(ψ)角的分布方式大致评估模拟的结构与自然结构相同程度[11]。如果预测的蛋白质残基二面角(90%)位于黄色核心区域,则表明其空间结构稳定[16]。如图8所示检测麻风树(JcCS 1)、乳浆大戟、蓖麻(RcCS 1)和乌桕CS蛋白质残基的二面角有90%以上位于黄色区域,表明其有稳定的空间构象。对其余CS蛋白质残基的二面角进行预测,只有JcCS 4蛋白质残基的二面角为89.0%,略小于90%,存在不够稳定的可能性,其他均有稳定空间构象。

图8 麻风树JcCS1(A)、乳浆大戟EeCS(B)、蓖麻RcCS1(C)和乌桕TsCS(D)CS Swiss-Model三维建模的Ramachandran图Fig.8 Ramachandran map of JcCS1(A),EeCS(B),RcCS1(C) and TsCS(D) by using Swiss-Model method
3 讨论
作为一种潜在的可工业化开发的生物能源植物,麻风树自身毒性对其开发有很大限制,而它的毒性有一部分原因是由于其含有波酯类化合物。佛波酯类化合物的母核为巴豆烷,巴豆烷合成途径中蓖麻烯合酶是一个关键酶,对它的了解有助于麻风树的去毒研究和进一步的开发利用。例如,可以在后续研究中可以通过敲除麻风树的CS基因,来试验是否可以帮助其完成去毒。
通过生物信息学分析,本文发现麻风树等13个CS都属于酸性蛋白质,等电点在5.0~6.1,通过亲水性/疏水性分析发现CS为亲水性蛋白,为该蛋白的成功分离提供一定理论依据。由于CS在细胞中的底物GGPP主要存在于叶绿体当中,其叶绿体导肽的存在与否将是行使其功能的关键[4],而预测数据显示只有EeCS、JcCS1、RcCS2、RcCS3、RcCS4及TcCS具有叶绿体导肽,通过ChloroP 1.1验证发现,EeCS、JcCS1、JcCS2、RcCS3、RcCS4及TcCS具有叶绿体导肽,两者数据在对JcCS2和RcCS2两个蛋白的预测有些出入。分析原因可能是由于现在导肽数据库蛋白质数量有限导致预测结果并不完全准确,还需要进一步实验验证。信号肽和扩膜结构域的预测都显示CS是在细胞内行使功能,这符合了我们对CS的预期判断。对CS进行的二级结构和三级结构分析显示,CS是Ⅰ类萜类合酶,这为该酶的体外活性检测提供了一定理论依据。本研究初步分析预测了来自麻风树等物种的13个CS基因的理化性质和结构特点,为进一步研究CS的功能、探索佛波酯类化合物合成途径以及麻风树的开发利用奠定了一定的理论基础。
1.Adolf W,Opferkuch H J,Hecker E.Irritant phorbol derivatives from four Jatropha species[J].Phytochemistry,1984,23(1):129-132.
2.Gustafson K R,Cardellina J H,McMahon J B,et al.Non-promoting phorbol from the Samoan medicinal plant,Homalanthus nutans,inhibits cell killing by HIV-1[J].J Med Chem,1992,35(11):1978-1986.
3.Schmidt R J.The biosynthesis of tigliane and related diterpenoids:an intriguing problem[J].Bot J Linn Soc,1987,94(1):221-230.
4.Kirby J,Nishimoto M,Park J G,et al.Cloning of casbene and neocembrene synthases from Euphorbiaceae plants and expression in Saccharomyces cerevisiae[J].Phytochemistry,2010,71(13):1466-1473.
5.Sato S,Hirakawa H,Isobe S,et al.Sequence Analysis of the Genome of an Oil-Bearing Tree[J].Jatropha curcas L.DNA Res,2011,18(1):65-76.
6.徐建华,朱家勇.生物信息学在蛋白质结构与功能预测中的应用[J].医学分子生物学杂志,2005,2(3):227-232.
Xu J H,Zhu J Y.Bioinformatics and Its Application on Protein Structure and Function Prediction[J].J Med Mol Biol,2005,2(3):227-232.
7.许忠能.生物信息学[M].北京:清华大学出版社,2008:3-10.
Xu Z N.Bioinformatics[M].Beijing:Tsinghua University Press,2008:3-10.
8.Gasteiger E,Gattiker A,Hoogland C,et al.ExPASy:the proteomics server for in-depth protein knowledge and analysis[J].Nucl Acids Res,2003,31(13):3784-3788.
9.Tamura K,Dudley J,Nei M,et al.MEGA4:Molecular evolutionary genetics analysis(MEGA) software version 4.0[J].Mol Biol Evol,2007,24(8):1596-1599.
10.翟中和,王喜忠,丁明孝.细胞生物学[M].北京:高等教育出版社,2000:79-240.
Zhai Z H,Wang X Z,Ding M X.Cell biology[M].Beijing:Higher Education Press,2000:79-240.
11.董娇,周军,辛培尧等.不同植物LDOX/ANS基因的生物信息学分析[J].基因组学与应用生物学,2010,29(5):815-822.
Dong J,Zhou J,Xin P Y,et al.Bioinformatics Analysis of LDOX/ANS Gene in Different Plants[J].Genomics and Applied Biology,2010,29(5):815-822.
12.Emanuelsson O,Nielsen H,Brunak S,et al.Predicting subcellular localization of proteins based on their N-terminal amino acid sequence[J].J Mol Biol,2000,300(4):1005-1016.
13.Emanuelsson O,Nielsen H,Heijne G V.ChloroP,a neural network-based method for predicting chloroplast transit peptides and their cleavage sites[J].Protein Science,1999,8(5):978-984.
14.Bendtsen J D,Nielsen H,Heijne G V,et al.Improved prediction of signal peptides:SingalP 3.0[J].J Mol Biol,2004,340(4):783-795.
15.Iked A M,Arai M,Lao D M.Transmembrane topology prediction methods:a reassessment and improvement by a consensus method using a dataset of experimentally characterized transmembrane topologies[J].In Silico Bio,2002,2(1):19-33.
16.薛庆中.DNA和蛋白质序列数据分析工具[M].第2版.北京:科学出版社,2009:72-109.
Xue Q Z.Data analysis tools of DNA and Protein[M].2ed edition.Beijing:Science Press,2009:72-109.
17.Kyce J,Doolittle R F.A simple method for displaying the hydropathic character of a protein[J].J Mol Biol,1982,157(6):105-132.
18.Geourjon C,Deleage G.SOPMA:Significant improvement in protein secondary structure prediction by consensus prediction from multiple alignments[J].Bioinformatics,1995,11(6):681-684.
19.王镜岩,朱圣庚,徐长发.生物化学[M].第3版.北京:高等教育出版社,2002.
Wang J Y,Zhu S G,Xu C F.Biochemistry[M].The third edition.Beijing:Higher Education Press,2002.
20.薛永常,聂会忠,刘长斌.木质素合酶C3H基因的生物信息学分析[J].生物信息学,2009,7(1):13-17.
Xue Y C,Nie H Z,Liu C B.Bioinformatics analysis on C3H in different plants[J].Bioinformatics,2009,7(1):13-17.
21.Schultz J,Milpetz F,Bork P,et al.SMART,a simple modular architecture research tool:identification of signaling domains[J].Proc Natl Acad Sci,1998,95(11):5857-5864.
22.Bohlmann J,Steele C L,Croteau R.Monoterpene synthases from grand fir(Abiesgrandis):cDNA isolation,characterization, and functional expression of myrcene synthase,(-)-(4S)- limonene synthase,and(-)-(1S,5S)-pinene synthase[J].J Biol Chem,1997,272(35):21784-21792.
23.Lambert C,Leonard N,De Bolle X,et al.ESyPred3D:Prediction of proteins 3D structures[J].Bioinformatics,2002,18(9):1250-1256.
24.Cao R,Zhang Y H,Mann F M,et al.Diterpene cyclases and the nature of the isoprene fold[J].Proteins:Struct Funct Bioinf,2010,78(11):2417-2432.
25.Köksal M,Jin Y,Coates R M,et al.Taxadiene synthase structure and evolution of modular architecture in terpene biosynthesis[J].Nature,2011,469(6):116-120.
26.Wendt K U,Poralla K,Schulz G E .Structure and function of a squalene cyclase[J].Science,1997,277(19):1811-1815.
27.Laskowski R A,Macarthur M W,Moss D,et al.PROCHECK:A program to check the stereo chemical quality of protein structures[J].J Appl Cryst,1993,26(2):283-291.
The National Natural Science Foundation of China(31270705);National non-profit Research Institutions of Chinese Academy of Forestry(RIF2014-01)
introduction:LIU Hong-Wei(1987—),male,Dr.,Maily engagerd in the study of plant secondary metabolism.
date:2016-01-13
BioinformaticsAnalysisofCasbeneSynthaseGenesinDifferentPlants
LIU Hong-Wei1YANG Yan-Fang1XIONG Wang-Dan2WU Ping-Zhi2WU Guo-Jiang2QIU De-You1*
(1.State Key Laboratory of Forest Tree Genetics and Breeding,the Research Institute of Forestry,Chinese Academy of Forestry,Beijing 100091;2.Key Laboratory of Plant Resources Conservation and Sustainable Utilization,South China Botanical Garden,Chinese Academy of Sciences,Guangzhou 510650)
We used bioinformatics to study 13 casbene synthase (CS; EC 4.6.1.7) full-length gene sequences registered in the GenBank fromEuphorbiaesula,JatrophacurcasL.,RicinuscommunisandTriadicasebifera, and predicted the composition of nucleic acid and amino acid sequences, leader peptides, signal peptide, trans-membrane topological structure, hydrophobicity or hydrophilicity, the secondary and tertiary structure as well as the function domains. The 13 genes encoding ORFs were 1 647-1 845 bp, the molecular weight of the 13 predicted proteins were 63.0-70.8 kD, and the termination codons were TGA or TAA. The theoretical isoelectric points of the 13 proteins were lower than 7.0, which suggested that CS proteins were acidic. Leu was the most contented amino acid. By the homologous alignment of nucleic acid, CS genes were divided into two groups. The prediction of leading peptides showed that at least 6 CSs had leader peptide (chloroplast leader peptide mainly) in common. All 13 CSs had no signal peptide and trans-membrane topological structure in and the peptide chains were hydrophilicity. α-helix was the dominant secondary structure constructional element of the 13 proteins which contained two terpenoid synthases function domains.
tigliane;casbene synthase;bioinformatics
国家自然科学基金(31270705);中国林业科学研究院林业研究所中央级公益性科研院所基本科研业务费专项(RIF2014-01)
刘洪伟(1987—),男,博士研究生,主要从事植物次生代谢方向的研究。
* 通信作者:E-mail:qiudy@caf.ac.cn
2016-01-13
* Corresponding author:E-mail:qiudy@caf.ac.cn
S565.6
A
10.7525/j.issn.1673-5102.2016.04.017
