长白落叶松4CL基因单核苷酸多态性及其与木材材性的关联分析
2016-11-09王艳红贾庆斌张含国
王艳红 贾庆斌 张 磊 张含国*
(1.东北林业大学林木遗传育种国家重点实验室,哈尔滨 150040; 2.东北农业大学动物科学技术学院,哈尔滨 150030)
* 通信作者:E-mail:hanguozhang1@sina.com
长白落叶松4CL基因单核苷酸多态性及其与木材材性的关联分析
王艳红1,2贾庆斌1张 磊1张含国1*
(1.东北林业大学林木遗传育种国家重点实验室,哈尔滨 150040;2.东北农业大学动物科学技术学院,哈尔滨 150030)
为探讨长白落叶松4CL基因多态性与木材材性及生长性状的关系,本研究以10个种源的长白落叶松为实验材料,采用直接测序方法开展4CL基因多态性检测,共检测到64个SNPs,SNP频率为1/31 bp,多样性指数πT值为0.015 77,θW值为0.008 78。同义突变核苷酸多样性(πsyn=0.022 56)是非同义突变核苷酸多样性(πnonsyn=0.015 75)的1.4倍,推测长白落叶松4CL基因编码区在长白落叶松物种演化过程中受到纯化选择压力的作用。对频率大于10%的27个常见SNP的单位点和单倍型分别于长白落叶松木材材性和生长性状进行关联分析,检测到7个SNPs和6个单倍型的与木材材性和生长性状显著关联,表型贡献率为0.09%~2.08%。研究结果为基于分子标记的长白落叶松优良木材品质及生长性状定向辅助育种提供理论依据。
长白落叶松;木质素;4CL基因;SNPs;单倍型
长白落叶松(Larixolgensis)是温带和寒带山区非常重要的荒山造林树种和纸浆用材树种,具有生长快、适应性强、经济价值高等优点[1~2]。木材中木质素约占其干重的15%~36%,是仅次于纤维素的大分子有机物。主要分布在输导组织、机械组织和保护组织细胞次生壁中,起机械支持、水分运输及病虫害防御等重要作用[3~5]。在造纸业制浆造纸过程中,高木质素含量的木材会增加制浆造纸过程的成本与消耗,带来严重的环境污染[6],而木材中木质素的含量和构型将直接影响制浆造纸过程中发酵产物乙醇转化率和纸浆得率[7]。因此,木质素生物合成机制及木质素含量(组成)的基因调控一直是国内外关注和研究的热点[8]。
木质素的合成途径十分复杂[9],目前普遍认为可分为3个主要途径:(1)由植物光合作用初级产物葡萄糖生成苯丙氨酸、酪氨酸和色氨酸等的莽草酸途径;(2)从苯丙氨酸到肉桂酸及其酰基辅酶A酯的苯丙烷类代谢途径;(3)从肉桂酰辅酶A酯还原为木质素单体以及聚合生成木质素的木质素合成特异途径[10~11]。其中4-香豆酸辅酶A连接酶(4CL),为苯丙烷类代谢途径最后一个酶,也是该途径的限速酶[12],它催化羟基香豆酸生成相应的羟基硫酯。人们最早在拟南芥、烟草、美洲山杨三种植物进行了4CL表达活性的研究[13~15]。到目前为止,已从拟南芥、杨树、大豆、烟草等10多种植物中克隆出4CL基因[16]。大量研究表明,抑制4CL基因的表达可以降低木质素含量,且不影响植物生长。Hu等在抑制4CL基因表达的转基因美洲山杨中,发现转基因植株中木质素含量下降了45%,纤维素含量上升15%,木质素纤维素总量没变[15]。Li等将反义4CL基因转入美洲山杨中,发现木质素含量下降52%,S/G比率上升了64%,纤维素含量上升30%[17]。Emmanuel等年研究发现在火炬松中下调4CL基因会使木质素含量下降纤维素含量上升[18]。Tian等利用正义、反义和RNAi-4CL基因转入毛白杨中,结果表明转入反义4CL1基因的植株木质素含量下降了28.52%,且植物生长没有负面影响[19]。因此4CL基因是较为理想的用于改良造纸资源植物的目标基因。
单核苷酸多态性(single-nucleotide polymorphisms,SNPs)是真核生物中最常见的遗传变异形式,基于SNP多态性的关联分析研究对挖掘与表型性状显著关联的功能标记位点,提高重要性状早期测定的精确度和可靠性,缩短林木育种周期,制定林木育种方案具有非常重要的作用,因此基于SNP多态性的关联分析研究在较短时间内获得了快速的发展。目前,关联遗传学已在杨树[20~21]、桉树[22]和火炬松[23]等树种中取得了初步进展。Gonzalez-Martinez等在火炬松4CL基因中发现SNPM5与木质素含量相关[23]。Thumma等在桉树EniCOBL4A(COBRA-like gene)基因第5外显子上发现同义突变SNP与纤维素含量显著相关[24],Dillon等在辐射松PAL1(Phenylalanine ammonia lyase)基因上发现同义突变SNP(SNP60)与木材密度性状显著关联[25];国内,田佳星发现毛白杨PtUGDH(UDP-glucose dehydrogenase)基因5′UTR上的SNP7与毛白杨纤维素含量显著相关[20]。
本研究选用黑龙江省尚志市帽儿山镇1982年定植的长白落叶松第二次种源试验林为研究对象,共4个区组,10个种源240株个体。利用DNA直接测序法研究分析了长白落叶松4CL基因的遗传多样性,同时采用连锁不平衡分析构建了4CL基因中27个常见SNPs的单倍型,将单位点和单倍型分别于长白落叶松木材材性和生长性状进行关联分析,以期筛选出对长白落叶松重要经济性状具有显著效应的的SNPs,为长白落叶松的高效选育和分子标记数据库的建立、种质资源保护和利用提供依据。
1 材料与方法
1.1 植物材料
本实验材料来自黑龙江省尚志市帽儿山镇长白落叶松种源试验林(44°29′~45°34′N;127°17′~129°12′E),1980年在露水河、天桥岭等10个种源(地点)采种,1981年育苗,1982年造林,完全随机区组五次重复,每区组10个种源(1大石头、2露水河、3天桥岭、4白河、5和龙、6穆棱、7大海林、8小北湖、9白刀山、10鸡西桦木),每个种源60株,共3 000株。1995年进行隔行去行间伐,2001年进行隔1株去2株间伐。现该种源试验林共有500株个体。本实验从5个区组中选取了4个区组,每种源10个单株中选了6个个体,共240个个体为研究对象。于2011年6月底采取当年生嫩叶(松针),-20℃保存备用。
2010年对这240棵单株进行了表型性状:木质素(lignin,%)、纤维素(cellulose,%)、树高(height,m)、胸径(DBH,cm)、密度(density,g·cm-3)、材积(volume,m3)、冠幅(crown,m)、碳储量(CB,kg)、含碳率(CCR,%)的测定,建立表型数据库。木质素含量参照国家标准GB/T 2667.8-94进行测定[26];综纤维素含量参照国家标准GB/T 2667.10-95进行测定[27];含碳率利用德国耶拿分析仪器股份公司,碳元素分析仪multi EA 4000进行测定,碳储量根据含碳率进行计算[28]。
1.2 引物设计与基因克隆
根据兴安落叶松4CL基因(Accession No.EU280849.1)和火炬松4CL基因(Accession No.U12013.1)设计引物4CL a1、a2、b1、b2,扩增4CL基因片段,并克隆测序。利用NCBI数据库Nucleotide Blast功能和DNAMAN(http://www.mbio.ncsu.edu/dnaman/dnaman.html)等软件对测序结果进行同源性比对。根据比对结果重新设计引物。以240个单株的基因组DNA为模板,扩增4CL基因。序列拼接、氨基酸序列相似性比较利用DNAMAN软件;物种间分子系统进化树的构建利用MEGA 6.01软件[29]。上述引物合成及测序工作均由上海生工完成。
1.3 基因分型
同一基因不同个体序列用BioEdit软件(http://www.mbio.ncsu.edu/bioedit/bioedit.html)和Clustal软件(http://www.mbio.ncsu.edu/Clustal/Clustal.html)联合比对分析,结合每条序列测序峰值图,根据测序结果进行基因分型,纯合体为单峰,杂合子为套峰(图1)。基因型命名方式为:突变型用突变前后碱基命名,例如T到C的突变,则基因型表示为:TT、CC、TC型;插入缺失型用D(Dleate)、I(Insert)表示,即基因型表示为:DD、II、ID型。
图1 基因分型Fig.1 Genetype
1.4 核苷酸多样性分析
测得核苷酸序列用Dnaman和Bioedit软件进行分析,标计SNPs位点、计算SNPs转换和颠换的数量。采用DnaSP5.1软件(http://www.ub.edu/dnasp)估算个基因的核苷酸多样性和中性检验[30~31],同时计算沉默位点多样性(πsil)、同义突变多样性(πsyn)和非同义突变多样性(πnonsyn)。每个基因的单倍型数量(h)和单倍型多样性水平(Hd)在DnaSP5.1中采用Nei的方法进行[32]。
1.5 连锁不平衡程度分析(LD分析)
连锁不平衡程度分析利用Haploview4.2软件(http://www.broad.mit.edu/mpg/haploview.html),单倍型块的划分采用D′值95%置信区间在0.70~0.98的相邻SNP被归入同一个单倍型块。
1.6 关联分析
本研究采用分析软件JMP5.0的一般线性模型[33]和分析软件TESSAL5.0的GLM程序[21]分别进行基因型和表型性状的关联分析。将27个SNP单位点和25个单倍型分别与树木生长性状(树高、胸径、冠幅、材积)及木材材性性状(木质素、纤维素、密度、含碳率、碳储量)进行关联分析。单倍型构建采用滑窗方法,每3个SNPs构成一个窗口,每次向前滑动一个SNP(图2),每个单倍型用SAS9.1.3 HAPLOTYPE程序构建[33]。经试验,2、3、4个位点为一组没有显著区别,所以我们最终选择3个位点为一组(图2)。

图2 4CL基因单倍型构建Fig.2 Haplotypes constructed in 4CL gene
2 结果
2.1 相似性比较与进化树分析
根据兴安落叶松、火炬松4CL基因设计引物,扩增长白落叶松总DNA,得到大小约为584、403、560和595 bp的四条特异性扩增条带,经序列拼接得到片段大小为2 009 bp的4CL基因DNA序列,由142 bp的5′UTR,993 bp的外显子1(exon 1),82 bp的内含子(intron),620 bp的外显子2(exon 2)和172 bp的3′UTR组成。经分析,4CL基因含有大小为1 613 bp的完整开放阅读框,可编码含有537个氨基酸残基的蛋白质。将长白落叶松4CL基因氨基酸序列与GenBank中已报道的其他物种相应序列对位排列比较(图3)。可以清楚地看出,长白落叶松与火炬松(Pinustaeda)、毛果杨(Populustrichocarpa)、棉花(Gossypiumhirsutum)、大豆(Glycinemax)、苜蓿(MedicagotruncTUBAla)、拟南芥(Arabidopsisthaliana)4CL的结构高度一致,都具有共有基序BoxⅠ和BoxⅡ结构[34](图3)。

图3 长白落叶松4CL与其它物种4CL氨基酸序列比对结果 Box结果标为灰色Fig.3 Alignment of deduced amino acid of 4CL from L.olgensis and other species Box is labelled in grey.
分子进化树分析表明,4CL氨基酸由极点向2个方向发生了进化,所有的裸子植物被聚类到第一分支上,被子植物中的双子叶植物被聚类在第二分支上,唯独被子植物中的单子叶水稻(Oryzasativa)单独在一个分支上,第一分支分为2个亚分支,第一亚分支分为三个部分,落叶松(Larixolgensis)和花旗松(Pseudotsugamenziesii)被聚类到第一部分,第二部分为云杉(Piceasmithiana),第三部分包括火炬松(Pinustaeda)和华山松(Pinusarmandii),;第二亚分支上包括雪松(Cedrusatlantica)、云南油杉(Keteleeriaevelyniana)、冷衫(Abiesholophylla)、加拿大铁杉(Tsugacanadensis),而银杉(Cathayaargyrophylla)不属于任何亚分支,为单独一个亚分支。第二分支上包括双子叶植物大豆(Glycinemax)、拟南芥(Arabidopsisthaliana)、毛果杨(Populustrichocarpa)、苜蓿(MedicagotruncTUBAla)、黄瓜(Cucumissativus),说明长白落叶松与花旗松的亲缘关系较近(图4)。
2.2 SNPs的发现
经Bioedit软件对240个测序结果比对,共发现64个SNPs出现频率超过1%,平均每31 bp序列出现1个SNP;其中出现频率超过10%的常见SNP27个。这27个SNP中共发生16次转换突变,占总数的59.3%,11次颠换突变,占总数的40.7%,非同义突变6次,同义突变19次(表1)。

图4 各物种的4CL氨基酸序列UPGMA系统进化树Fig.4 UPGMA phylogenetic tree analysis based on amino acid sequence of 4CL from different species

基因Gene长度LSNP数SNPs常见SNP数CommonSNPsSNP频率Frequency(bp)编码区SNPCodingregions静默SNPSilentSNP非同义SNP数Non-synonSNPs同义SNP数SynonSNPs颠换Transversion转换Transition4CL200964273125216191116

表2 长白落叶松4CL基因核苷酸多样性

图5 4CL基因连锁不平衡分析Fig.5 The LD analysis of 4CL
2.3 多态性检测结果及基因型频率分析
以长白落叶松4CL基因中发现的64个SNP为基础计算核苷酸多样性,发现其具有较高的SNP多态性,πT值为0.015 77,θW值为0.008 78(表2)。平均沉默多样性(πsil=0.006 13)和非同义多样性(πnonsyn=0.015 75)水平均低于平均同义多样性(πsyn=0.022 56)。同义突变核苷酸多样性是非同义突变核苷酸多样性的1.4倍,推测长白落叶松4CL基因编码区在长白落叶松物种演化过程中受到纯化选择压力的作用。
2.4 连锁不平衡分析
Haploview4.2分析结果表明4CL基因SNP间连连锁不平衡程度较强,R2平均值为0.4826,27个SNP标记分为4个单倍型模块,分别为模块SNP1-2,SNP3-9,SNP13-14和SNP15-27(图5)。
2.5 单位点SNP与木材材性、生长性状关联分析
利用分析软件JMP5.0对单个位点与表型性状进行关联分析,共发现14个关联位点与表型性状显著关联,详见表3:其中SNP32、SNP41、SNP44、SNP45、SNP47、SNP51和SNP55不同基因型与木质素含量性状显著关联;SNP45不同基因型与胸径性状显著关联;SNP41不同基因型与树高性状显著关联;SNP47和SNP55与密度性状显著关联;SNP32、SNP41和SNP51不同基因型与碳储量性状显著关联。多重比较分析结果详见图6。
2.6 4CL单倍型与木材材性、生长性状关联分析
将构建的25个单倍型与长白落叶松木材材性及生长性状进行关联分析,共发现6个单倍型的与表型性状显著关联,分别为:窗口SNP51-53的单倍型多态性与长白落叶松木质素含量性状显著关联,窗口SNP41-43的单倍型多态性与长白落叶松树高性状显著关联,窗口SNP47-49的单倍型多态性与长白落叶松密度显著关联,窗口SNP32-34、SNP40-42和SNP51-53的单倍型多态性与长白落叶松碳储量显著关联(表4)。
表34CL基因单个SNP位点与长白落叶松木材材性及生长性状关联分析(P值)
Table3Associationanalysisofsingle-nucleotidepolymorphismsinthe4CLgenewithwoodqualityandgrowthtraitsinL.olgensis(p-value)

标记Marker性状TraitP值Pvalue表型贡献率Phenotypicvariance(%)SNP32s木质素Lignin0.0401*1.98SNP41s木质素Lignin0.0347*2.73SNP44ns木质素Lignin0.0462*1.48SNP45s木质素Lignin0.0410*1.33SNP47s木质素Lignin0.0462*1.31SNP51s木质素Lignin0.0407*2.08SNP55ns木质素Lignin0.0460*0.56SNP45s胸径DBH0.0494*2.05SNP41s树高Height0.0422*0.35SNP47s密度Density0.0242*1.53SNP55ns密度Density0.0407*1.72SNP32s碳储量Carbonstorage0.0442*0.37SNP41s碳储量Carbonstorage0.0456*0.43SNP51s碳储量Carbonstorage0.0477*0.09
注:*表示影响显著P<0.05。
Note:*Significant at theP<0.05 level.
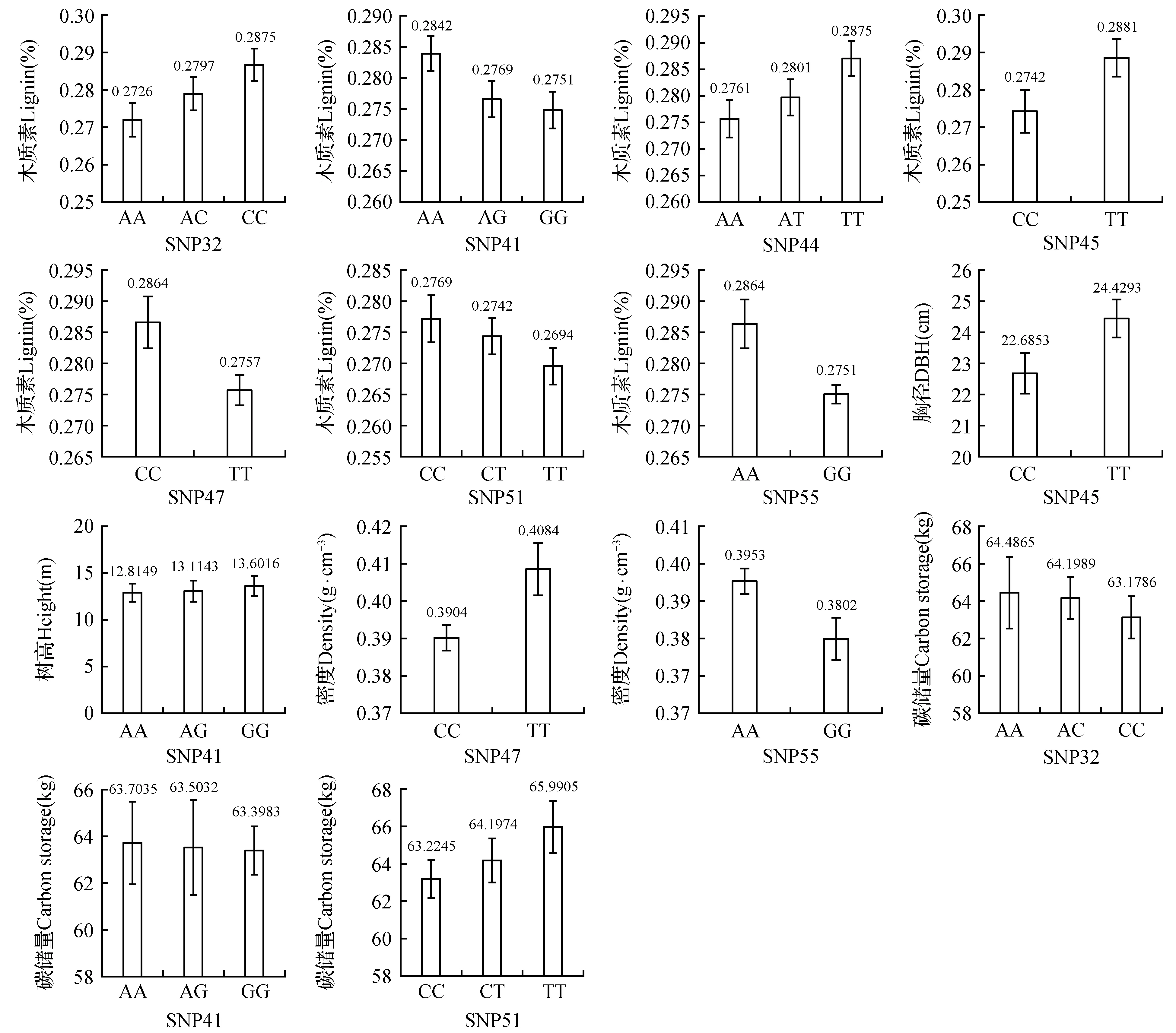
图6 4CL基因SNPs对长白落叶松木材材性及生长性状的基因型效应Fig.6 SNPs genotypic effects on wood properties and growth traits of L.olgensis
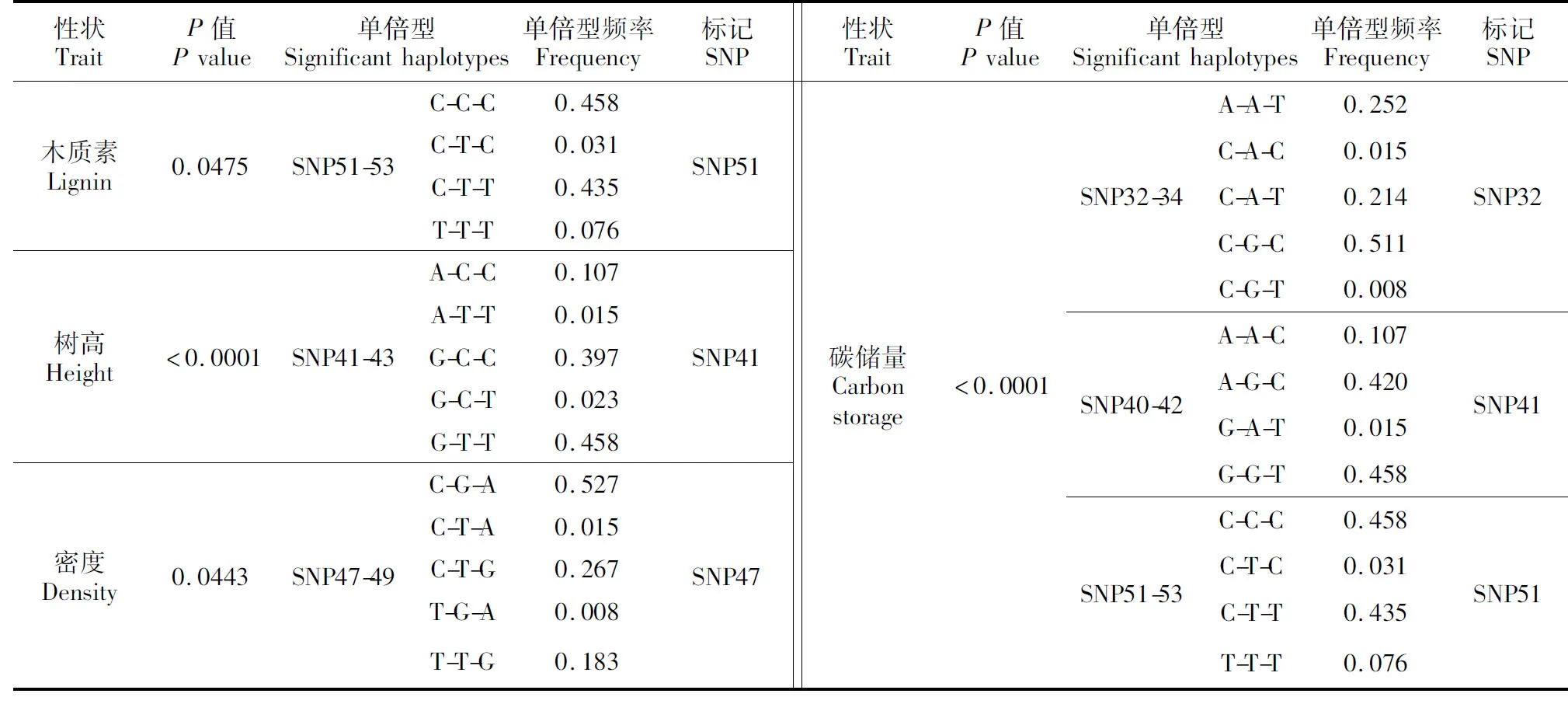
性状TraitP值Pvalue单倍型Significanthaplotypes单倍型频率Frequency标记SNP性状TraitP值Pvalue单倍型Significanthaplotypes单倍型频率Frequency标记SNP木质素Lignin0.0475SNP51-53C-C-C0.458C-T-C0.031C-T-T0.435T-T-T0.076SNP51树高Height<0.0001SNP41-43A-C-C0.107A-T-T0.015G-C-C0.397G-C-T0.023G-T-T0.458SNP41密度Density0.0443SNP47-49C-G-A0.527C-T-A0.015C-T-G0.267T-G-A0.008T-T-G0.183SNP47碳储量Carbonstorage<0.0001SNP32-34SNP40-42SNP51-53A-A-T0.252C-A-C0.015C-A-T0.214C-G-C0.511C-G-T0.008A-A-C0.107A-G-C0.420G-A-T0.015G-G-T0.458C-C-C0.458C-T-C0.031C-T-T0.435T-T-T0.076SNP32SNP41SNP51
3 讨论
3.1 多态性分析
4-香豆酸辅酶A连接酶(4-coumarate:CoA ligase,4CL)基因,为木质素苯丙烷类代谢途径中最后一个酶,也是该途径限速酶[12],它催化羟基香豆酸生成相应的羟基硫酯。本研究从10个种源240个单株中克隆得到候选基因目的片段,经DNASP软件分析发现长白落叶松具有较高多态性,πT值为0.015 77,θW值为0.008 78。与前人研究相比,长白落叶松4CL基因SNP的密度稍高于玉米(π=0.010 25)[35]。单倍型多样性(HD=0.921)也高于其他树种的观测值,如Pot等在辐射松(HD=0.425)[36],Gonzalez-Martinez等在火炬松(HD=0.68)[23],Mandrou等在尾叶桉(HD=0.853)中的发现[37]。说明长白落叶松实验群体10个种源间存在遗传结构的差异性。
3.2 关联分析
目前,林木中关于木质素合成相关基因的关联分析的研究有很多,但国内针对长白落叶松4CL基因关联分析的报道很少。本研究对频率大于10%的27个常见SNP的单位点和单倍型分别于长白落叶松木材材性和生长性状进行关联分析,检测到7个SNP(32、41、44、45、47、51和55)位点与木材材性和生长性状显著关联,其中4个SNP(32、41、47、和51)所在单倍型同样与表型性状显著关联。
SNP51与木质素含量和木材碳储量显著关联,表型贡献率为2.08%和0.09%,拥有TT基因型的个体比CC基因型个体木质素含量更低、碳储量更高。与前人的研究相比,Emmanuel等年研究发现在火炬松中下调4CL基因会使木质素含量下降而纤维素含量上升[18],Tian研究发现在杨树中上调或下调4CL1基因可以改变木质素含量和组成[19],Gonzalez-Martinez等年在4CL基因中发现SNPM5与木质素含量相关[23],本研究在关联性上与以上报道大体一致。
SNP47与木材密度显著相关,表型贡献率为2.50%,其TT基因型密度(0.408 4±0.006 6 g·cm-3)显著高于CC基因型密度含量(0.390 4±0.003 2 g·cm-3)。与前人的研究相比,Gonzalez-Martinez等在火炬松中发现4CL基因的SNPM3和SNPM7与早材的密度相关联[23]。以往的研究也在针叶树其他参与木质素合成的基因中发现与木材密度相关联的SNPs。Yu在火炬松杂交树中发现CAD-n1基因与木材密度相关联的[38],同样González-Martínez等对北美重要栽培树种火炬松四个基因(CAD,sams-2,lp3-1和a-tubulin)与不同木材材性性状进行了关联分析,找到9个SNP与木材材性性状相关,其中位于CAD基因的SNPM28与早材密度相关联[23,39];Dillon年在辐射松中发现CAD基因和COMT2基因同样与早材和晚材的密度相关联[25],作者在长白落叶松CAD基因中发现SNP7和SNP15两个SNP与木材密度显著关联[32]。
SNP41与树高和碳储量性状的显著关联,表型贡献率分别为0.35%和0.43%,多重比较分析结果表明,SNP41的GG基因型树高(13.601 6±0.181 1 m)显著高于AA基因型树高(12.814 9±0.487 0 m),AA基因型木材碳储量(63.703 5±1.701 7 kg)显著高于GG基因型木材碳储量(63.398 3±1.004 9 kg)。而SNP32与碳储量性状显著关联,表型贡献率为0.37%;多重比较分析结果表明SNP32的AA基因型木材碳储量(64.489 5±1.892 5 kg)显著高于CC基因型木材碳储量(63.178 6±1.090 9 kg)。
上面四个SNP均为同义突变,尽管同义突变不会导致氨基酸的改变,但能通过其单核苷酸多态性的变化导致翻译提前终止从而引起一系列的生物学效应,从而改变基因的表达力间接影响蛋白表达[40~41]。Thumma等在EniCOBL4A基因外显子5上的同义突变SNP与纤维素含量显著相关[24],Dillon等在辐射松PAL1基因上发现同义突变SNP(SNP60)与木材密度性状显著关联[25]。说明SNPs同义突变在与表型性状的关联分析中是有意义的。
长白落叶松为落叶松人工林主栽树种,广泛分布于黑龙江省东南部,吉林东部长白山地区以及辽宁省。长白落叶松人工林具有生长迅速、成林快、适应性广等优点,是我国东北地区重要造林、纸浆材和建筑材树种。候选基因的选择对优良木材材性及生长性状的培育具有重要意义,本研究将4CL基因作为选育有利于造纸工业脱木质素的长白落叶松优良植株的重要候选基因,对基因的多态性及关联分析将为基于分子标记的长白落叶松优良木材品质定向辅助育种提供理论依据。
1.Zhu J,Yang K,Yan Q,et al.Feasibility of implementing thinning in even-agedLarixolgensisplantations to develop uneven-aged larch-broadleaved mixed forests[J].Journal of Forest Research.2010,15(1):71-80.
2.Guan C,Zhang H,Zhang L,et al.Construction of Genetic Linkage Maps of Larch[J].Biotechnology & Biotechnological Equipment,2014,25(1):2197-2202.
3.Mandrou E,Hein P R G,Villar E,et al.A candidate gene for lignin composition in Eucalyptus:cinnamoyl-CoA reductase(CCR)[J].Tree Genetics & Genomes.2012,8(2):353-364.
4.Zapata-Valenzuela J,Isik F,Maltecca C,et al.SNP markers trace familial linkages in a cloned population ofPinustaedaprospects for genomic selection[J].Tree Genetics & Genomes,2012,8(6):1307-1318.
5.Trabucco G M,Matos D A,Lee S J,et al.Functional characterization of cinnamyl alcohol dehydrogenase and caffeic acid O-methyltransferase inBrachypodiumdistachyon[J].BMC Biotechnol.2013,13:61.
6.Xu Y,Thammannagowda S,Thomas T P,et al.LtuCAD1 Is a Cinnamyl Alcohol Dehydrogenase Ortholog Involved in Lignin Biosynthesis inLiriodendrontulipiferaL.,a Basal Angiosperm Timber Species[J].Plant Molecular Biology Reporter.2013,31(5):1089-1099.
7.Sassner P,Galbe M,Zacchi G.Steam pretreatment ofSalixwith and without SO2impregnation for production of bioethanol[Z].Appl Biochem,2005:121-124,1101-1117.
8.Sibout R.Ginnamyl Ulcohol Dehydrogenase-c and-D Are the Primary Genes Involved in Lignin Biosynthesis in the Floral Stem ofArabidopsis[J].THE PLANT CELL ONLINE,2005,17(7):2059-2076.
9.Matsui N,Chen F,Yasuda S,et al.Conversion of guaiacyl to syringyl moieties on the cinnamyl alcohol pathway during the biosynthesis of lignin in angiosperms[J].Planta,2000,210(5):831-835.
10.Boerjan W,Ralph J,Baucher M.Lignin biosynthesis[J].Annu Rev Plant Biol,2003,54:519-546.
11.Baucher M.Lignin:Genetic Engineering and Impact on Pulping[J].Critical Reviews in Biochemistry and Molecular Biology,2003,38(4):305-350.
12.倪志勇,王娟,吕萌,等.棉花4-香豆酸辅酶A连接酶基因克隆及原核表达[J].西北植物学报,2010(03):429-436.
13.Lee D,Meyer K,Chapple C,et al.Antisense suppression of 4-coumarate:coenzyme A ligase activity inArabidopsisleads to altered lignin subunit composition[Z].Planta,1997:9,1985-1998.
14.Kajita S,Hishiyama S,Tomimura Y,et al.Structural Characterization of Modified Lignin in Transgenic Tobacco Plants in Which the Activity of 4-Coumarate:Coenzyme A Ligase Is Depressed[J].Plant Physiol,1997,114(3):871-879.
15.Hu W J,Harding S A,Lung J,et al.Repression of lignin biosynthesis promotes cellulose accumulation and growth in transgenic trees[J].Nat Biotechnol,1999,17(8):808-812.
16.宋晓丹,饶国栋,蒋湘宁,等.杨树木质部特异性定位表达4CL1启动子的结构与表达特性[J].成都大学学报:自然科学版,2008,27(4):269-272.
17.Li L,Zhou Y,Cheng X,et al.Combinatorial modification of multiple lignin traits in trees through multigene cotransformation[J].Proc Natl Acad Sci USA,2003,100(8):4939-4944.
18.Emmanueljohnson T A.Increasing cellulose production and transgenic plant growth in forest tree species[J].Forestry Research,2005,16(1):67-72.
19.Tian X,Xie J,Zhao Y,et al.Sense-,antisense- and RNAi-4CL1 regulate soluble phenolic acids,cell wall components and growth in transgenicPopulustomentosaCarr[J].Plant Physiol Biochem,2013,65:111-119.
20.Tian J,Du Q,Li B,et al.Single-nucleotide polymorphisms in the 5′ UTR of UDP-glucose dehydrogenase(PtUGDH) associate with wood properties inPopulustomentosa[J].Tree Genetics & Genomes,2014,10(2):339-354.
21.Xu B,Tian J,Du Q,et al.Single nucleotide polymorphisms in a cellulose synthase gene(PtoCesA3) are associated with growth and wood properties inPopulustomentosa[Z].Planta,2014:240,1269-1286.
22.Thumma B R,Nolan M F,Evans R,et al.Polymorphisms in cinnamoyl CoA reductase(CCR) are associated with variation in microfibril angle inEucalyptusspp[J].Genetics,2005,171(3):1257-1265.
23.Gonzalez-Martinez S C,Wheeler N C,Ersoz E,et al.Association genetics inPinustaedaL.I.Wood property traits[J].Genetics,2007,175(1):399-409.
24.Thumma B R,Matheson B A,Zhang D,et al.Identification of a Cis-Acting Regulatory Polymorphism in a Eucalypt COBRA-Like Gene Affecting Cellulose Content[J].Genetics.2009,183(3):1153-1164.
25.Dillon S K,Nolan M,Li W,et al.Allelic Variation in Cell Wall Candidate Genes Affecting Solid Wood Properties in Natural Populations and Land Races ofPinusradiata[Z].Genetics,2010:185,1477-1487.
26.Kirk T K,Obst J R.Lignin determination[M].London:Academic Press,1988,161:87-101.
27.Porth I,Klapste J,Skyba O,et al.Populustrichocarpacell wall chemistry and ultrastructure trait variation,genetic control and genetic correlations[J].New Phytol,2013,197(3):777-790.
28.Zhu H Y,Weng Y H,Zhang H G,et al.Comparing fast- and slow-growing provenances ofPiceakoraiensisin biomass,carbon parameters and their relationships with growth[J].Forest Ecology and Management,2013,307:178-185.
29.Kumar S,Tamura K,Nei M.MEGA3:Integrated software for Molecular Evolutionary Genetics Analysis and sequence alignment[J].Brief Bioinform,2004,5(2):150-163.
30.Tajima F.Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J].Genetics,1989,123(3):585-595.
31.Watterson G A.On the number of segregating sites in genetical models without recombination[J].Theoretical Population Biology,1975,7(2):256-276.
32.Wang Y,Jia Q,Zhang L,et al.Allelic Variation in Cinnamyl Alcohol Dehydrogenase(LoCAD) Associated with Wood Properties ofLarixolgensis[Z].Forests,2015:6,1649-1665.
33.Zhang H,Liu S H,Zhang Q,et al.Fine-mapping of quantitative trait loci for body weight and bone traits and positional cloning of the RB1 gene in chicken[J].Journal of Animal Breeding and Genetics,2011,128(5):366-375.
34.Hu W J,Kawaoka A,Tsai C J,et al.Compartmentalized expression of two structurally and functionally distinct 4-coumarate:CoA ligase genes in aspen(Populustremuloides)[J].Proc Natl Acad Sci USA,1998,95(9):5407-5412.
35.Andersen J R,Zein I,Wenzel G,et al.Characterization of phenylpropanoid pathway genes within European maize(ZeamaysL.) inbreds[Z].BMC Plant Bioligy,2008:8,2.
36.Pot D,Mcmillan L,Echt C,et al.Nucleotide variation in genes involved in wood formation in two pine species[J].New Phytol,2005,167(1):101-112.
37.Mandrou E,Denis M,Plomion C,et al.Nucleotide diversity in lignification genes and QTNs for lignin quality in a multi-parental population ofEucalyptusurophylla[J].Tree Genetics & Genomes,2014,10(5):1281-1290.
38.Yu Q,Li B,Nelson C D,et al.Association of the cad-n1 allele with increased stem growth and wood density in full-sib families of loblolly pine[Z].Tree Genetics Genomes,2006:2,98-108.
39.Thumma B R.Polymorphisms in Cinnamoyl CoA Reductase(CCR) Are Associated With Variation in Microfibril Angle inEucalyptusspp.[J].Genetics,2005,171(3):1257-1265.
40.Tian J,Chang M,Du Q,et al.Single-nucleotide polymorphisms in PtoCesA7 and their association with growth and wood properties inPopulustomentosa[J].Molecular Genetics and Genomics,2014,289(3):439-455.
41.Xu N,Loflin P,Chen C Y,et al.A broader role for AU-rich element-mediated mRNA turnover revealed by a new transcriptional pulse strategy[Z].Nucleic Acids Research,1998:26,558-565.
AllelicVariationin4-coumarate:CoAligase(4CL)AssociatedwithWoodPropertiesofLarixolgensis
WANG Yan-Hong1,2JIA Qing-Bin1ZHANG Lei1ZHANG Han-Guo1*
(1.Key Laboratory of Forest Tree Genetic Improvement and Biotechnology,Northeast Forestry University,Harbin 150040;2.College of Animal Science and Technology,Northeast Agricultural University,Harbin 150030)
We used 10 provenances ofLarixolgensisto study the effects of polymorphism of 4CL gene on wood quality and growth traits inL.olgensis. The polymorphism in 4CL gene was identified using DNA sequencing, 64 mean SNPs were identified,the median SNP frequency was one site per 31 bp,and the average nucleotide diversity for the sequenced regions was calculated to beπT=0.015 77 andθW=0.008 78. The diversity level of synonymous nucleotide sbustituons(πsyn=0.022 56) was 1.4 of nonsynoymous nucleotide substitutions(πnonsyn=0.015 75), suggesting that the gene misht be evolved under purifying selection at the synonoymous sites of the coding region inL.olgensis. The association between the 27 common SNPs(frequency>10%), haplotypes and wood quality and growth traits were analyzed respectively, indicating that 7 SNPs and 6 haplotypes were significantly associated with wood quality and growth traits, explaining between 0.09%-2.08% of the phenotypic variance. Ourstudy provided an important genetic foundation for molecular marker-assistant selection breeding programs with the goals of improving the wood quality and growth products inL.olgensis.
Larixolgensis;lignin;4CL gene;SNPs;haplotype
国家863课题“落叶松优质、抗逆基因工程育种”(2013AA102704-04)
王艳红(1980—),女,博士研究生,主要从事林木遗传育种方面的研究。
2015-09-29
S791.22
A
10.7525/j.issn.1673-5102.2016.02.013
