腾格里沙漠沙坡头地区土壤微生物多样性分析
2016-11-02李靖宇张燕灵
李靖宇,张 琇,孙 敏,张燕灵
(北方民族大学生物科学与工程学院,宁夏 银川 750021)
腾格里沙漠沙坡头地区土壤微生物多样性分析
李靖宇,张 琇①,孙 敏,张燕灵
(北方民族大学生物科学与工程学院,宁夏 银川 750021)
为了揭示沙坡头沙漠固沙区与流沙区不同植物根际微生物群落结构的组成、丰度和多样性对沙漠极端环境的响应,采用DNA提取试剂盒提取土壤总DNA,对细菌群落编码16S rRNA的基因(16S rDNA)的V4~V5区进行MiSeq测序,分析各样品中细菌群落结构的组成、丰度以及多样性指标,通过非度量多维尺度(NMDS)法和文氏(Venn)图解释微生物群落结构对环境的响应。固沙区与流沙区不同土壤样品微生物群落在门水平上主要以变形细菌门(Proteobacteria)、放线菌门(Actinobacteria)、酸杆菌门(Acidobacteria)和蓝细菌门(Cyanobacteria)为优势类群。在属水平上,不同样品优势类群差异明显。11个样品在NMDS纵轴方向上按照固沙区与流沙区分为2大组,说明微生物类群对人工固沙做出了环境响应。文氏图分析结果表明,尽管不同样品之间存在一定差异,但存在共有微生物类群,可能在沙坡头沙漠生态系统中发挥着核心作用。蓝细菌以及根瘤菌在沙生植物固沙过程中对于提供营养物质等生物地球化学循环起着重要作用。
微生物群落;极端环境;沙坡头沙漠;MiSeq测序
土地沙漠化面积的迅速扩展,造成环境退化和巨大的经济损失,使之成为全球广泛关注的热点[1]。沙漠化也是我国北方干旱、半干旱及部分半湿润地区由于人地关系不协调所造成的以风沙活动为主要标志的土地退化[2]。我国沙漠及其边缘地区的居民积累了丰富的治沙和沙地开发利用经验,建立了“以固为主、固阻结合、机械固沙和生物固沙并举”的防沙体系,开展了腾格里沙漠地区无灌溉固沙植物种植和综合防沙体系建设的开创性工作[1,3]。沙生植物对我国北部、西北部荒漠化防治起到了极其重要的作用,是维系沙漠植被生态系统的重要保障[4-6]。沙生植物有利于土壤养分、水分的保持及土壤的形成、发育,沙地灌丛具有肥岛效应[7]。在这个微环境中,根系分泌物为微生物提供重要的营养和能量物质,其成分和数量影响着根际微生物的种类和繁殖,根际微生物的代谢作用也影响根际土壤中的物质转化[8]。土壤微生物作为生态系统中重要组分之一,推动着生态系统的能量流动和物质循环,在维持生态系统整体服务功能方面发挥着重要作用。自然界中可培养的微生物种类仅占微生物总数的0.1%~10%[9-11]。
依赖于聚合酶链式反应(polymerase chain reaction,PCR)的核酸分析技术可检测某一基因的整个微生物群落结构和功能群的多样性信息,在微生物群落结构变化与环境变化相互作用等诸多领域发挥着传统分析方法无可替代的作用[12];特别是高通量测序技术的诞生,是基因组学研究领域一个具有里程碑意义的事件,在揭示微生物基因信息方面的技术优势远远超出了克隆文库所涵盖的信息量,正是这种前所未有的能够更加真实反映环境中微生物组成技术的应用极大地推动了人们对微生物生态学的认识[13]。笔者以宁夏中卫市沙坡头沙区土壤为研究对象,试图在沙漠科学研究中引入高通量测序技术,对沙生植物根际土壤微生物多样性进行深入探讨。
1 材料与方法
1.1 样点位置与样品采集
研究区域设在腾格里沙漠东南缘的宁夏中卫市沙坡头地区(37°32′ N,105°02′ E)。该地区年均气温10.0 ℃,低温极值-25.1 ℃,高温极值38.1 ℃,全年日照时数3 264 h,1956—2012年年均降水量186.2 mm,年潜在蒸发量3 000 mm,年均风速2.9 m·s-1,年均沙暴天数59 d[14]。
沙漠土壤样品按流沙区(A系列)与固沙区(B系列)分别取样,土壤类型以风沙土为主[15],于2014年10月3日采集土壤样品,按5点法随机选取沙生植物根系0~30 cm深度土壤,轻轻抖落根表面的土壤作为根际土壤样品[16]。A系列包括无植被对照组A0以及不同植被A1、A2、A3和A4这5个样品,其中A1植被为细枝岩黄芪(Hedysarumscoparium),A2为沙蓬(Agriophyllumsquarrosum),A3为百花蒿(Stilpnolepiscentiflora),A4为雾冰藜(Bassiadasyphylla);B系列包括固沙区不同植被B1、B2、B3、B4这4个样点以及不同于A系列沙生植物的样点(记为B5和B6),其中B1植被为细枝岩黄芪,B2为沙蓬,B3为百花蒿,B4为雾冰藜,B5为刺沙蓬(Salsolatragus),B6为中间锦鸡儿(Caraganaintermedia),具体信息见表1。
表1 不同样点的基本信息
Table 1 Basic information of the sampling sites
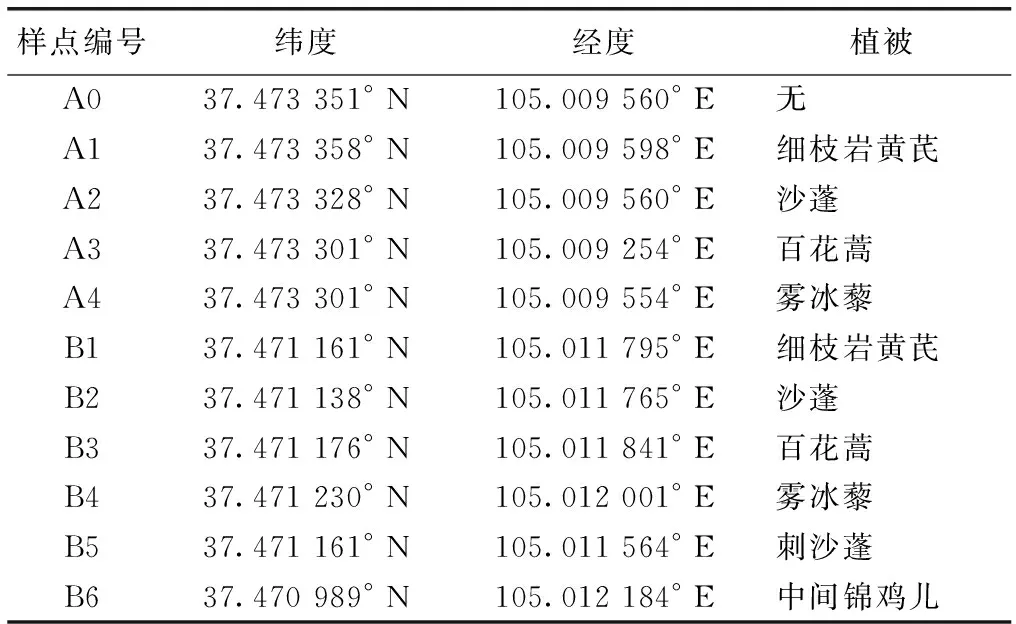
样点编号纬度经度植被A037.473351°N105.009560°E无A137.473358°N105.009598°E细枝岩黄芪A237.473328°N105.009560°E沙蓬A337.473301°N105.009254°E百花蒿A437.473301°N105.009554°E雾冰藜B137.471161°N105.011795°E细枝岩黄芪B237.471138°N105.011765°E沙蓬B337.471176°N105.011841°E百花蒿B437.471230°N105.012001°E雾冰藜B537.471161°N105.011564°E刺沙蓬B637.470989°N105.012184°E中间锦鸡儿
样品A系列为流沙区,B系列为固沙区。
1.2 样品基因组总DNA提取
沙漠土壤样品微生物DNA参照E. Z. N. A.®Soil DNA Kit提取试剂盒(Omega Bio-tek,Norcross,GA,U. S.)的说明书进行提取。
1.3 16S rDNA V4~V5区Illumina MiSeq测序
PCR采用TransGen AP221-02:TransStart Fastpfu DNA聚合酶,20 μL反应体系:2.0 μL 2.5×10-3mol·L-1脱氧核苷酸(dNTPs),0.8 μL 0.5×10-5mol·L-1515F引物(5′-GTG CCA GCM GCC GCG G-3′),0.8 μL 0.5×10-5mol·L-1907R引物(5′-CCG TCA ATT CMT TTR AGT TT-3′),4.0 μL 5×FastPfu 缓冲液,10 ng模板以及 0.4 μL Fast Pfu聚合酶,最后加双蒸水(ddH2O)到20 μL。
反应条件:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸 45 s,27个循环;72 ℃延伸10 min。
获得的PCR产物进行20 g·L-1琼脂糖凝胶电泳,使用AXYGEN 公司的AxyPrep DNA凝胶回收试剂盒切胶回收PCR产物,Tris-HCL洗脱;使用Promega公司的QuantiFluorTM-ST蓝色荧光定量系统进行检测。将每个样品等比例混合,然后根据Illumina MiSeq测序平台的标准流程进行双端测序。
1.4 测序数据分析
Miseq测序得到的是双端序列数据,首先根据双端读长(paired-end reads,PE reads)之间的重叠关系,将成对的读长(reads)拼接(merge)成一条序列,同时对读长的质量和拼接的效果进行质控过滤,根据序列首尾两端的条形码(barcode)和引物序列区分样品得到有效序列,并校正序列方向。过滤读长尾部质量值20以下的碱基,设置50 bp的窗口,如果窗口内的平均质量值低于20,从窗口开始截去后端碱基,过滤质控后50 bp以下的读长;根据双端读长之间的重叠关系,将成对读长拼接成一条序列,最小重叠长度为10 bp;拼接序列的重叠区允许的最大错配比率为0.2,筛选不符合序列;根据序列首尾两端的条形码和引物区分样品,并调整序列方向,条形码允许的错配数为0,最大引物错配数为2;且去掉包含模糊碱基的序列。
用UPARSE(version 7.1,http:∥drive5.com/uparse/)软件聚类生成操作分类单元(operational taxonomic units,OTUs),相似度为97%。再用UCHIME软件鉴别嵌合体序列,并将之去除。用silva (SSU115) 16S rRNA数据库进行分类学比对,算法为RDP Classifier (http:∥rdp.cme.msu.edu/),置信阈值为70%。根据BOWEN等[17]的研究结果,将相对丰度大于1%的类群作为优势类群。使用97%相似度的OTUs,利用mothur软件做稀释性曲线(rarefaction)分析,利用R语言工具制作曲线图。
2 结果与分析
2.1 测序数据概况
为了比较流沙区和固沙区不同植物根际微生物群落结构的差异,试验共设计取样11个土壤样品,通过高通量测序16S rDNA V4~V5区获得微生物群落结构的组成。根据YOUSSEF等[18]报道,V4~V5区序列对微生物多样性的估计与全长16S rDNA序列得到的微生物多样性最接近。因此,选择V4~V5区序列进行群落结构和多样性分析,共获得有效序列416 372条,其中A0包含31 064条,A1包含43 650条,A2包含44 993条,A3包含35 080条,A4包含32 638条,B1包含42 796条,B2包含37 590条,B3包含41 707条,B4包含35 100条,B5包含30 672条,B6包含41 082条,平均长度为396 bp。为了得到更高质量的优化序列,根据UPAESE软件聚类,在聚类过程中会有单条序列和部分嵌合体被去除[19]。最后A0包含18 999条,A1包含29 740条,A2包含20 909条,A3包含21 198条,A4包含19 531条,B1包含26 707条,B2包含23 630条,B3包含23 657条,B4包含20 375条,B5包含17 392条,B6包含31 335条。基于UPAESE软件处理后的更高准确度的序列进行后续的稀释性曲线、OTUs聚类等分析。根据稀释性曲线(图1),测序深度基本可以真实反映沙漠环境中微生物群落结构的实际情况。

样品A系列为流沙区,B系列为固沙区。A0为无植被对照;A1、B1植被为细枝岩黄芪;A2、B2为沙蓬;A3、B3为百花蒿;A4、B4为雾冰藜;B5为刺沙蓬;B6为中间锦鸡儿。
2.2 细菌群落结构的组成、丰度与多样性
OTUs分析表明,在相似性为0.97水平上,A0样品包含1 418个OTUs,A1包含1 263个OTUs,A2包含1 564个OTUs,A3包含1 481个OTUs,A4包含1 515个OTUs,B1包含1 574个OTUs,B2包含1 115个OTUs,B3包含1 328个OTUs,B4包含1 286 个OTUs,B5包含1 368个OTUs,B6包含1 679个OTUs(表2)。
由图2可见,在门水平上,主要涵盖了变形细菌门(Proteobacteria)、放线菌门(Actinobacteria)、酸杆菌门(Acidobacteria)、蓝细菌门(Cyanobacteria)、绿弯菌门(Chloroflexi)、拟杆菌门(Bacteroidetes)、浮霉状菌门(Planctomycetes)、厚壁菌门(Firmicutes)、芽单胞菌门(Gemmatimonadetes)、Armatimonadetes、绿菌门(Chlorobi)、异常球菌-栖热菌门(Deinococcus-Thermus)、硝化刺菌门(Nitrospirae)和疣微菌门(Verrucomicrobia)。其中,变形细菌门、放线菌门、酸杆菌门和蓝细菌门为优势类群。从A0到B6的11个样品中,变形细菌门的相对丰度为20.30%~39.77%,放线菌门的相对丰度为10.69%~38.90%,酸杆菌门的相对丰度为4.13%~19.61%,蓝细菌门的相对丰度为0.09%~29.44%。
表2 不同样点细菌群落的丰度和多样性指数
Table 2 Richness and diversity index of bacterial community relative to sampling sites

样点编号读长平均长度/bp 操作分类单元距离覆盖度/%Chao1指数ACE指数辛普森指数香农指数A018999396 14180.0398.0473171917120.02105.53A129740396 12630.0398.9610148615360.01315.34A226909396 15640.0398.5470187418810.01565.62A321198395 14810.0398.1319184018120.01635.58A419531395 15150.0398.0032184818290.01035.91B126707396 15740.0398.5772186618830.01295.70B223630396 11150.0398.7601141413710.02205.18B323657396 13280.0398.6854160015730.01255.68B420375396 12860.0398.4098155015650.01445.64B517392396 13680.0398.0221163416490.00935.86B631335396 16790.0398.8767193119260.01155.80
样品A系列为流沙区,B系列为固沙区。A0为无植被对照;A1、B1植被为细枝岩黄芪;A2、B2为沙蓬;A3、B3为百花蒿;A4、B4为雾冰藜;B5为刺沙蓬;B6为中间锦鸡儿。
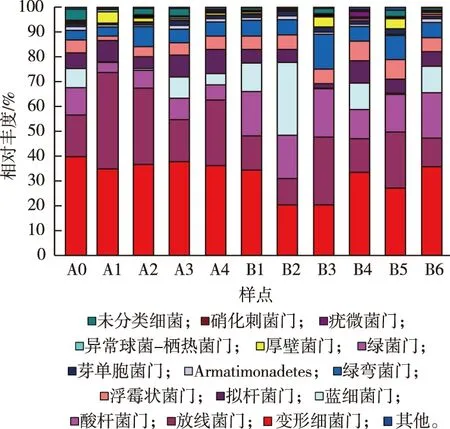
样品A系列为流沙区,B系列为固沙区。A0为无植被对照;A1、B1植被为细枝岩黄芪;A2、B2为沙蓬;A3、B3为百花蒿;A4、B4为雾冰藜;B5为刺沙蓬;B6为中间锦鸡儿。
在属水平上,不同样品优势类群差异明显。其中A0优势类群为微枝形杆菌属(Microvirga)(11.66%)和微鞘藻属(Microcoleus)(7.37%);A1优势类群为诺卡氏菌属(Nocardioides)(13.66%)、节细菌属(Arthrobacter)(4.63%)、微枝形杆菌属(4.22%)、博斯氏菌属(Bosea)(4.04%)和新鞘氨醇杆菌属(Novosphingobium)(4.01%);A2优势类群为诺卡氏菌属(10.01%)、节细菌属(4.78%)和微枝形杆菌属(4.08%);A3优势类群为微枝形杆菌属(9.00%)、博斯氏菌属(5.84%)、微鞘藻属(5.43%)和Rubellimicrobium(5.08%);A4优势类群为微枝形杆菌属(7.64%)、博斯氏菌属(4.35%)、诺卡氏菌属(3.42%)、Rubellimicrobium(3.08%)和微鞘藻属(3.04%);B1优势类群为微鞘藻属(5.91%)、微枝形杆菌属(5.25%)、博斯氏菌属(3.34%)和席藻属(Phormidium)(3.31%);B2优势类群为微鞘藻属(11.13%)、Blastocatella(10.51%)和微枝形杆菌属(3.42%);B3优势类群为诺卡氏菌属(8.74%);B4优势类群为马西亚菌属(Massilia)(4.73%)、Bryobacter(3.12%)和德沃斯氏菌属(Devosia)(3.06%);B5优势类群为诺卡氏菌属(4.08%);B6优势类群为微鞘藻属(5.40%)、微枝形杆菌属(4.82%)和席藻属(3.13%)。
多样性指数Chao1、Ace是生态学中估计物种总数的常用指数。从A0到B6的11个样品中,Chao1指数范围为1 414~1 931,Ace指数范围为1 371~1 926,结果基本一致。辛普森指数是用来估算样品中微生物多样性的指数之一,其值越大,说明群落多样性越低。从A0到B6的11个样品中,其指数范围为0.009 3~0.022 0。香农指数是用来估算样品中微生物多样性的指数之一,它与辛普森多样性指数常用于反映alpha多样性指数,其值越大,说明群落多样性越高,从A0到B6的11个样品中,其指数范围为5.18~5.91(表2)。
2.3 流沙区与固沙区植物根际微生物群落的比较
在流沙区设置A1~A4这4个样点,固沙区设置B1~B4这4个样点,其中A1与B1、A2与B2、A3与B3、A4与B4植被类型相同,对其根际土壤微生物多样性进行分析比较,结果见图2。流沙区与固沙区相同植被根际微生物群落有明显差异,这种差异主要体现在某些种群的相对丰度上。在属的水平上,其优势类群差异较为明显。
A1与B1在优势类群上的差异主要体现在:节细菌属,A1相对丰度为4.63%,B1为1.15%;微鞘藻属,A1相对丰度为0%,B1为5.91%;诺卡氏菌属,A1相对丰度为13.66%,B1为0.96%;新鞘氨醇杆菌属,A1相对丰度为4.01%,B1为0.30%;席藻属,A1相对丰度为2.80%,B1为2.11%。
A2与B2在优势类群上的差异主要体现在:节细菌属,A2相对丰度为4.78%,B2为0.06%;Blastocatella,A2相对丰度为0.36%,B2为10.51%;微鞘藻属,A2相对丰度为0.01%,B2为11.13%;诺卡氏菌属,A2相对丰度为10.01%,B2为0.16%。
A3与B3在优势类群上的差异主要体现在:节细菌属,A3相对丰度为3.42%,B3为0.94%;博斯氏菌属,A3相对丰度为5.84%,B3为0.30%;微鞘藻属,A3相对丰度为5.43%,B3为0.03%;微枝形杆菌属,A3相对丰度为8.96%,B3为1.89%;诺卡氏菌属,A3相对丰度为0.83%,B3为8.74%;Rubellimicrobium,A3相对丰度为5.08%,B3为0.05%。
A4与B4在优势类群上的差异主要体现在:节细菌属,A4相对丰度为2.94%,B4为0.20%;博斯氏菌属,A4相对丰度为4.35%,B4为2.55%;Bryobacter,A4相对丰度为1.20%,B4为3.12%;马西亚菌属,A4相对丰度为0.63%,B4为4.73%;微鞘藻属,A4相对丰度为3.04%,B4为0.79%;微枝形杆菌属,A4相对丰度为7.64%,B4为2.80%;诺卡氏菌属,A4相对丰度为3.42%,B4为0.84%;Rubellimicrobium,A4相对丰度为3.08%,B4为0.57%。
为了进一步说明流沙区和固沙区相同植被根际微生物群落的差异,设置了无植被流动沙丘作为对照组。以高通量测序数据为基础,通过文氏图反映流沙区和固沙区相同植物根际微生物群落结构的差异,并发现这些相似环境中的“核心微生物群落”[20]。分析可知,在OTUs组成上,A0、A1、B1之间既有共同的微生物类群,也存在不同的微生物类群。A0、A2、B2,A0、A3、B3,A0、A4、B4情况相似。由图3可以看出,沙漠环境中,植被根际微生物类群差异明显,无植被与有植被情况下微生物类群差异明显,固定沙丘和流动沙丘微生物类群差异明显。在沙漠这种极端环境中,微生物群落变化较大,但核心类群较为明显,它们可能在生物地球化学循环中发挥着重要的作用。

数值表示可操作分类单元(OTUs)数。
2.4 流沙区与固沙区不同样品微生物群落的聚类分析
非度量多维尺度(NMDS)法是一种将多维空间的研究对象(样本或变量)简化到低维空间进行定位、分析和归类,同时又保留对象间原始关系的数据分析方法。该方法的特点是根据样本中包含的物种信息,以点的形式反映在多维空间上,而对不同样本间的差异程度,则通过点与点间的距离体现,最终获得样本的空间定位点图。由图4可知,11个不同样品在NMDS纵轴方向上基本分为2大类,即固沙区与流沙区2大组,这与实际情况相符,说明微生物类群对人工固沙做出了环境响应。
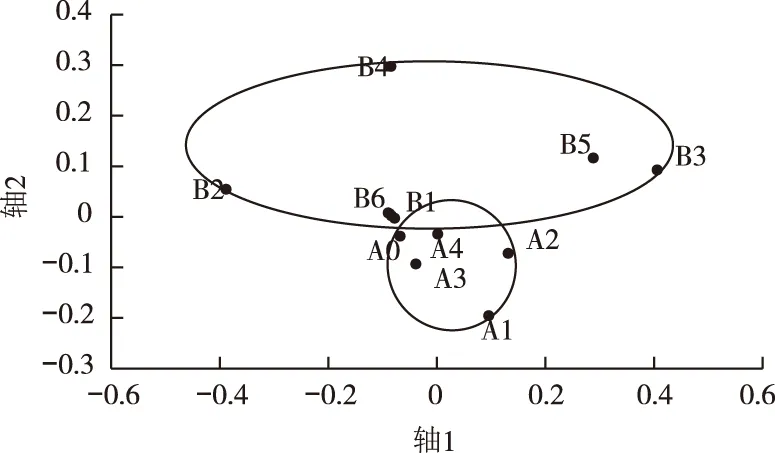
样品A系列为流沙区,B系列为固沙区。A0为无植被对照;A1、B1植被为细枝岩黄芪;A2、B2为沙蓬;A3、B3为百花蒿;A4、B4为雾冰藜;B5为刺沙蓬;B6为中间锦鸡儿。
3 讨论
沙漠生态系统蒸发量大于降水量,日温差变化剧烈,土壤中营养物质匮乏,严酷的自然条件限制了许多植物的生存,只有为数不多的超旱生植物稀疏分布,生物种类多样性低,群落结构简单,自动调节能力差,干旱的限制使荒漠生态系统成为地球上各类生态系统中生产力最低、最脆弱的生态系统之一。那么,微生物群落在这个脆弱的生态系统中发挥着怎样的作用?笔者从解析微生物群落结构入手,采用宏基因组学技术手段最大限度地反映环境中微生物群落结构的真实情况。研究采用MiSeq测序技术,针对沙坡头沙漠流沙区和固沙区不同植物根际微生物群落结构和多样性进行了深度测序,11个土样分别获得17 392~31 335条高质量序列,覆盖度数据表明测序结果基本可以反映土壤样品微生物群落结构的真实情况。沙坡头沙漠土壤中微生物多样性较高,类群丰富。流沙区和固沙区相同植物根际微生物群落结构和组成差异明显。文氏图分析结果表明,尽管不同样品之间存在一定的差异,但是存在共有微生物类群;共有微生物类群可能在沙漠生态系统中发挥着核心作用,差异微生物类群可能是由于流沙与固沙的状态不同等因素造成的。尽管微生物类群存在明显差异,但在较大尺度上,流沙区样品相似性较高,聚为1类;固沙区样品相似性较高,聚为1类。NMDS分析结果间接表明流沙与固沙过程对微生物类群结构影响明显;根际环境随着土壤类型、植物种类和生育阶段存在很大的时空变异性。
微生物的分布、多样性和群落组成差异主要取决于环境和生物因素[21-23]。在大的空间尺度上,微生物群落结构与土壤变量密切相关,如土壤pH值[24-25]和含水量[26]。在局部尺度上,植物群落通过根际交互作用来影响土壤微生物群落[27-28]。干旱环境中,细菌的多样性被认为是由非生物因素(如温度的剧烈变化、紫外线的强烈辐射、土壤含水量和营养差)以及生物因素(如植物的丰度和物种组成[12])所影响。在营养贫乏的沙漠土壤中,植物不仅提供营养物质丰富的栖息地,还会进一步对土壤微生物施加选择性的影响[12]。在水和养分有限的环境中,肥岛效应假说认为,由于营养物在根和土壤界面处的积累,根际细菌多样性应大于根围[12]。笔者的测序结果表明,流沙区无植被土壤样品的微生物多样性同样很高,不同样品微生物群落组成和多样性的差异主要源于不同植物根际土壤。可能不同植物对微生物群落结构以及组成存在较大的选择性影响,在较大的尺度上体现在固沙区与流沙区相同植物根际微生物群落间的差异。LIU等[29]报道古尔班通古特沙漠微生物群落的空间分布与沙丘地貌相关。由此推测,沙坡头沙漠中流沙区与固沙区不同植被根际土壤微生物类群差异明显,除了与不同植物的属性相关外,主要与沙丘的地貌密切相关。此外,在荒漠生态系统,各种物理扰动可能会显著影响土壤微生物群落的时空分布,如沙埋、动物践踏和野火焚烧[29]。
沙坡头沙漠微生物群落组成在门的水平上以变形细菌门、放线菌门、酸杆菌门、蓝细菌门为优势类群,11个样品在门水平上组成相似;这与CRITS-CHRISTOPH等[30]报道的智力阿塔卡马沙漠优势类群以放线菌门(72%~88%)最多,其次为酸杆菌门(3.8%~6.6%)和变形细菌门(2.2%~9.2%)的组成相似。但沙坡头沙漠与阿塔卡马沙漠优势类群的丰度存在差异,主要体现在沙坡头沙漠以变形细菌门为主要优势类群,阿塔卡马沙漠以放线菌门为主要优势类群。阿塔卡马沙漠在副热带高气压带下沉气流、离岸风和秘鲁寒流综合影响下,成为世界最干燥的地区之一;我国的沙漠大多数分布于干旱区,属中温带典型的大陆性气候,是干旱气候与丰富沙源条件下的产物[1]。分析认为,大环境的不同造成微生物群落结构的组成和优势类群的相对丰度不同,这也说明了微生物群落结构的生境异质性,特别是沙坡头沙漠蓝细菌门类群的出现,更体现出这2种环境的差异。
在属水平上,沙坡头不同样品优势类群差异明显,这种差异主要体现在博斯氏菌属、德沃斯氏菌属、马西亚菌属、微枝形杆菌属、新鞘氨醇杆菌属、Rubellimicrobium(变形细菌门);节细菌属、诺卡氏菌属(放线菌门);Blastocatella、Bryobacter(酸杆菌门);席藻属、微鞘藻属(蓝细菌门)。其中博斯氏菌属和微枝形杆菌属属根瘤菌(Rhizobiales)目,与固氮密切相关[31]。博斯氏菌属的一些种能够氧化还原态的无机硫化合物,有的能以CO2作为唯一碳源进行生长[32],这些生理生化特性对于营养贫瘠的沙漠环境具有重要的意义。来自于土壤生物结皮的Microvirgasp. BSC39的基因组测序结果表明,其具有趋化和胞外多糖合成能力,有利于生物膜的形成[33]。Microvirgalotononidis是最近被描述的一个根瘤菌菌种,能够有效地进行固氮,具有一些与其他根瘤菌不同的性状,含有色素,且能够在45 ℃高温条件下生长[34]。蓝细菌是一类进化历史悠久、革兰染色阴性、无鞭毛、含叶绿素和藻蓝素(但不形成叶绿体)、能进行产氧性光合作用的大型原核微生物。此外,许多丝状蓝细菌在缺乏无机氮化合物(如硝酸盐或铵盐)的极端寡营养条件下,能够形成异形胞,进行固氮作用[35]。蓝细菌是自然界中生态适应范围最广的一类生物。各种盐度、各种温度、各种压力下都有蓝细菌的存在[36]。蓝细菌通常作为沙漠生态系统的先锋和土壤生物结皮的优势类群,成为最初的生产者,固定碳并增加土壤有机物质,通过提供和再循环关键营养物质促进后续维管植物的生长,且在这些生态系统的氮平衡过程中发挥重要作用[37]。具鞘微鞘藻(Microcoleusvaginatus)是蓝藻门中一种广泛分布的陆生丝状藻类,同时也是荒漠干旱地区藻结皮的优势物种,作为荒漠植被演替过程中的先锋拓殖生物,具鞘微鞘藻能在条件极其恶劣的环境下生长、繁殖,并利用藻丝体对沙粒的捆绑、黏结作用使松散的沙面形成一个有机的整体,增强荒漠土壤表面的团聚力,对藻结皮的形成和发育过程产生重要的生态学效应[38-39]。
致谢: 感谢内蒙古大学赵利清教授对沙生植物鉴定给予的无私帮助。
[1] 王涛,赵哈林.中国沙漠科学的五十年[J].中国沙漠,2005,25(2):145-165.
[2] 王涛,朱震达.我国沙漠化研究的若干问题:1.沙漠化的概念及其内涵[J].中国沙漠,2003,23(3):209-214.
[3] 吴正.中国沙漠与治理研究50年[J].干旱区研究,2009,26(1):1-7.
[4] 潘伯荣.沙漠里的生命演绎:沙生植物[J].森林与人类,2006(6):70-73.
[5] 周芸芸,冯金朝,袁飞,等.我国沙生植物遗传多样性研究综述[J].中央民族大学学报(自然科学版),2011,20(3):5-11.
[6] POINTING S B,BELNAP J.Microbial Colonization and Controls in Dryland Systems[J].Nature Reviews,2012,10(8):551-562.
[7] 苏永中,赵哈林,张铜会.几种灌木、半灌木对沙地土壤肥力影响机制的研究[J].应用生态学报,2002,13(7):802-806.
[8] MORGAN J A W,BENDING G D,WHITE P J.Biological Costsand Benefits to Plant-Microbe Interactions in the Rhizosphere[J].Journal of Experimental Botany,2005,56(417):1729-1739.
[9] BROCK T D.The Study of Microorganismsinsitu:Progress and Problems[M]∥FLETCHER M,GRAY T R G,JONES J G.Forty-First Symposium of the Society for General Microbiology.University of St. Andrews:Cambridge University Press,2005:1-17.
[10]AMANN R I,LUDWIG W,SCHLEIFER K H.Phylogenetic Identification andinsituDetection of Individual Microbial Cells Without Cultivation[J].Microbiological Reviews,1995,59(1):143-169.
[11]DANIEL R.The Metagenomics of Soil[J].Nature Reviews,2005,3(6):470-478.
[12]ANDREW D R,FITAK R R,MUNGUIA-VEGA A,etal.Abiotic Factors Shape Microbial Diversity in Sonoran Desert Soils[J].Applied and Environmental Microbiology,2012,78(21):7527-7537.
[13]HOLLISTER E B,ENGLEDOW A S,HAMMETT A J M,etal.Shifts in Microbial Community Structure Along an Ecological Gradient of Hypersaline Soils and Sediments[J].The ISME Journal,2010,4(6):829-838.
[14]李新荣,张志山,谭会娟,等.我国北方风沙危害区生态重建与恢复:腾格里沙漠土壤水分与植被承载力的探讨[J].中国科学:生命科学,2014,44(3):257-266.
[15]樊宝丽,马全林,张德魁,等.腾格里沙漠南缘3种优势植物种子出苗对土壤类型和沙埋的响应[J].干旱区研究,2011,28(6):1038-1044.
[16]顾美英,徐万里,茆军,等.新疆绿洲农田不同连作年限棉花根际土壤微生物群落多样性[J].生态学报,2012,32(10):3031-3040.
[17]BOWEN J L,MORRISON H G,HOBBIE J E,etal.Salt Marsh Sediment Diversity:A Test of the Variability of the Rare Biosphere Among Environmental Replicates[J].The ISME Journal,2012,6(11):2014-2023.
[18]YOUSSEF N,SHEIK C S,KRUMHOLZ L R,etal.Comparison of Species Richness Estimates Obtained Using Nearly Complete Fragments and Simulated Pyrosequencing-Generated Fragments in 16S rRNA Gene-Based Environmental Surveys[J].Applied and Environmental Microbiology,2009,75(16):5227-5236.
[19]EDGAR R C.UPARSE:Highly Accurate OTU sequences From Microbial Amplicon Reads[J].Nature Methods,2013,10(10):996-1000.
[20]SHADE A,HANDELSMAN J.Beyond the Venn Diagram:The Hunt for a Core Microbiome[J].Environmental Microbiology,2012,14(1):4-12.
[21]KUSKE C R,TICKNOR L O,MILLER M E,etal.Comparison of Soil Bacterial Communities in Rhizosphere of Three Plant Species and the Interspaces in an Arid Grassland[J].Applied and Environmental Microbiology,2002,68(4):1854-1863.
[22]ROESCH L F,FULTHORPE R R,RIVA A,etal.Pyrosequencing Enumerates and Contrasts Soil Microbial Diversity[J].The ISME Journal,2007,1(4):283-290.
[23]FIERER N,LENNON J T.The Generation and Maintenance of Diversity in Microbial Communities[J].American Journal of Botany,2011,98(3):439-448.
[24]FIERER N,JACKSON R B.The Diversity and Biogeography of Soil Bacterial Communities[J].Proceedings of the National Academy of Sciences,2006,103(3):626-631.
[25]FULTHORPE R R,ROESCH L F W,RIVA A,etal.Distantly Sampled Soils Carry Few Species in Common[J].The ISME Journal,2008,2(9):901-910.
[26]ANGEL R,SOARES M I M,UNGAR E D,etal.Biogeography of Soil Archaea and Bacteria Along a Steep Precipitation Gradient[J].The ISME Journal,2010,4(4):553-563.
[27]BERG G,SMALLA K.Plant Species and Soil Type Cooperatively Shape the Structure and Function of Microbial Communities in the Rhizosphere[J].FEMS Microbiology Ecology,2009,68(1):1-13.
[28]BUÉE M,DE BOER W,MARTIN F,etal.The Rhizosphere Zoo:An Overview of Plant-Associated Communities of Microorganisms,Including Phages,Bacteria,Archaea,and Fungi,and Some of Their Structuring Factors[J].Plant and Soil,2009,321(1/2):189-212.
[29]LIU R,LI K,ZHANG H,etal.Spatial Distribution of Microbial Communities Associated With Dune Landform in the Gurbantunggut Desert,China[J].Journal of Microbiology,2014,52(11):898-907.
[30]CRITS-CHRISTOPH A,ROBINSON C K,BARNUM T,etal.Colonization Patterns of Soil Microbial Communities in the Atacama Desert[J].Microbiome,2013,1:28.
[31]BHATTACHARJEE R,DEY U.Biofertilizer,a Way Towards Organic Agriculture:A Review[J].African Journal of Microbiology Research,2014,8(24):2332-2342.
[32]OUATTARA A S,ASSIH E A,THIERRY S,etal.Boseaminatitlanensissp. nov.,a Strictly Aerobic Bacterium Isolated From an Anaerobic Digester[J].International Journal of Systematic and Evolutionary Microbiology,2003,53(5):1247-1251.
[33]BAILEY A C,KELLOM M,PORET-PETERSON A T,etal.Draft Genome Sequence ofMicrovirgasp. Strain BSC39,Isolated From Biological Soil Crust of Moab,Utah[J].Genome Announc,2014,2(6):1197-1200.
[34]REEVE W,ARDLEY J,TIAN R,etal.Genome Sequence of the Listia Angolensis MicrosymbiontMicrovirgalotononidisStrain WSM3557T[J].Standards in Genomic Sciences,2014,9(3):540-550.
[35]倪雪姣,齐兴娥,顾燕玲,等.天山乌鲁木齐河源一号冰川表面粉尘蓝细菌群落结构及其系统发育[J].微生物学报,2014,54(11):1256-1266.
[36]姜红霞,吴亚生,蔡春芳.重庆老龙洞二叠系-三叠系界线地层中的管状蓝细菌化石及其意义[J].科学通报,2008,53(7):807-814.
[37]RAJEEV L,DA ROCHA U N,KLITGORD N,etal.Dynamic Cyanobacterial Response to Hydration and Dehydration in a Desert Biological Soil Crust[J].The ISME Journal,2013,7(11):2178-2191.
[38]郑云普,张丙昌,赵建,等.具鞘微鞘藻在荒漠藻结皮形成过程中的作用[J].生态学报,2010,30(6):1655-1664.
[39]张丙昌,张元明,赵建成,等.古尔班通古特沙漠生物结皮不同发育阶段中藻类的变化[J].生态学报,2009,29(1):9-17.
(责任编辑: 许 素)
Analysis of Soil Microbial Diversity in Shapotou Area of Tengger Desert.
LI Jing-yu, ZHANG Xiu, SUN Min,ZHANG Yan-ling
(College of Biological Science & Engineering, Beifang University of Nationality, Yinchuan 750021, China)
Community composition, abundance and diversity of soil microbe in different vegetation rhizospheres and their responses to desert extreme environments in an artificial sand fixed area and a natural bare sand area in the Shapotou Desert, were studied. In the study, soil total DNA was extracted with the aid of the E.Z.N.A.®Soil DNA Kit, V4-V5 sections of the 16S rDNA of the soil bacterial community analyzed with a MiSeq pyrosequencer for community composition, abundance and diversity of the soil bacteria, and responses of the soil microbial community in structure to changes in environment explained with the NMDS and Venn diagrams. In the soil samples, no matter from which area, Proteobacteria, Actinobacteria, Acidobacteria and Cyanobacteria were dominant groups in the soil microbial community on the phylum level, while the soil samples from the two different areas differed sharply in dormancy of groups on the genus level. The 11 soil samples were divided into two groups along the vertical axis of the NMDS in accordance with the artificial sand fixed area and the natural bare sand area, indicating that the microbial community responded to changes in the environment, i.e. sand fixation. The Venn diagram analysis indicates that although some differences are found between different soil samples,groups of microbes common in the two groups of soil samples exist and are thought to play a core role in the Shapotou Desert ecosystem. Cyanobacteria and Rhizobia are the two species of soil microbes that play an important role in biogeochemical cycling, like providing nutrients to psammophytes in sand-fixing process.
microbial community; extreme environment; Shapotou Desert; MiSeq sequencing
2015-09-24
北方民族大学引进人才科研启动项目(44/4400302502);宁夏自然科学基金(NZ15098);国家自然科学基金(31460212)
X172;Q93
A
1673-4831(2016)05-0780-08
10.11934/j.issn.1673-4831.2016.05.014
李靖宇(1986—),男,内蒙古包头人,讲师,博士,从事环境微生物学研究。E-mail: lijingyu1986@126.com
① 通信作者E-mail: zhangxiu101@aliyun.com
