番茄质膜H+-ATPase家族基因的鉴定和表达分析
2016-10-27刘健健刘俊丽季敏杰陈家栋杨晓峰陈爱群
刘健健,刘俊丽,季敏杰,陈家栋,杨晓峰,陈爱群
(南京农业大学 资源与环境科学学院,江苏 南京210095)
番茄质膜H+-ATPase家族基因的鉴定和表达分析
刘健健,刘俊丽,季敏杰,陈家栋,杨晓峰,陈爱群
(南京农业大学 资源与环境科学学院,江苏 南京210095)
植物质膜H+-ATPase(Ec3.6.1.3)是一类普遍存在于细胞质膜上通过水解三磷酸腺苷(ATP)产生能量,将细胞质中的氢离子(H+)逆浓度泵出细胞的运输蛋白。植物中的质膜H+-ATPase由一个多基因家族所编码,其功能涉及到植物生长发育的多个生理过程。通过对全基因组检索在茄科Solanaceae植物番茄Solanum lycopersicum中共鉴定到8个编码质膜H+-ATPase的同源基因(LHA1~8)。生物信息学分析显示:这8个LHA基因具有较高的序列相似性和较为保守的外显子/内含子结构特征。实时荧光定量聚合酶链式反应(qRT-PCR)分析显示,LHA1~4在所有被检测的组织器官中都有表达,LHA5~7几乎只在花器官中高量表达,而LHA8在正常培养和养分(氮、磷、钾和镁)缺乏以及高盐胁迫处理条件下几乎都不表达,但能够在被菌根真菌侵染的根系中强烈表达。将一段2 669 bp的LHA8的启动子融合GUS报告基因转入到烟草Nicotiana tabacum中发现,GUS基因几乎只在被菌根真菌菌丝侵入形成丛枝的根系细胞中特异性表达。图5表2参22
分子生物学;番茄;质膜H+-ATPase;表达模式;菌根真菌
H+-ATPase是广泛存在于植物质膜和各种内膜系统中的一种功能蛋白,在细胞代谢过程中起非常关键的作用[1]。目前,被报道的H+-ATPase按结构可分为P型、F型和V型。其中F型H+-ATPase位于叶绿体内囊体膜和线粒体内膜上,V型H+-ATPase定位于液泡膜上,质膜H+-ATPase属于P型ATPase[2]。质膜H+-ATPase能够通过水解三磷酸腺苷(ATP)产生能量,将细胞质中的氢离子(H+)逆浓度泵出细胞(也称为质膜质子泵),其功能主要是在细胞膜两侧产生氢离子(H+)浓度梯度和膜电位,为一系列次级转运体和通道蛋白跨质膜转运各种离子和小分子代谢产物提供驱动力和能量[3]。一系列的实验结果证明:植物质膜H+-ATPase能够参与胞内酸碱度的调节、离子平衡、细胞的伸长生长、气孔的开闭等多种生理过程[4]。植物中的质膜H+-ATPase几乎都是由一个多基因家族所编码,且不同的成员在表达上具有一定的特异性和部分重叠性[5]。例如,在模式植物拟南芥Arabidopsis thaliana中共鉴定到11个编码质膜H+-ATPase的同源基因(AHA1~11)[6]。其中,AHA1和AHA2在所有的组织器官中都有表达,其表达模式趋向于组成型[7];AHA3主要在维管组织和生殖器官中表达[8];AHA6,AHA8和AHA9几乎只在花器官中表达[9];AHA10主要在发育中的种子种皮的内膜上表达[10]。这些结果暗示了在长期的进化过程中,不同的质膜H+-ATPase基因在植物发育的不同阶段所分化形成的相对保守性和/或特异性的生理功能。植物质膜H+-ATPase活性在基因表达水平的调节还受到激素(如吲哚-3-乙酸等)和环境(如盐害、病菌侵染、菌根真菌共生)因素的影响[11]。番茄 Solanum lycopersicum作为茄科Solanaceae作物中的一员,不仅是一种重要的经济作物,而且由于其基因组相对比较简单(大部分品种都为二倍体),以及全基因组测序和组装工作已经接近完成,也已经成为生物学和遗传学研究的一个理想的模式作物[12]。根据前期研究关于Southern印记杂交的结果推测,番茄中可能存在着至少7个编码质膜H+-ATPase的基因LHA1~7[13]。现有研究仅对其中的3个基因(LHA1,LHA2和LHA4)进行了全长cDNA序列的克隆和表达调控分析,而其他4个可能的LHA基因只报道了约200 bp的DNA序列[14]。本研究通过对番茄全基因组序列文库和EST/cDNA文库进行检索,共获得了8个具有完整编码区的质膜H+-ATPase基因LHA1~8,其中LHA8为从未报道过的新基因。对这8个基因的序列结构特征、进化关系以及组织表达模式做进一步分析,可为将来深入研究番茄质膜H+-ATPase基因家族的生物学功能奠定工作基础。
1 材料与方法
1.1供试材料
番茄微型模式品种Solanum lycopersicum‘Micro-Tom’,成熟期为70~80 d,株高约为15 cm。
1.2试验设计
番茄种子用体积分数为10%的过氧化氢溶液表面消毒10 min,自来水冲洗后置灭过菌的石英沙于25℃培养箱中萌发至真叶完全展开,用1/2浓度的营养液和完全浓度的营养液各培养1周后移栽进行后续实验处理。营养液的配方及浓度为:1.00 mmol·L-1硝酸铵(NH4NO3),2.00 mmol·L-1硝酸钾(KNO3),0.50 mmol·L-1硝酸钙(Ca(NO3)2),1.00 mmol·L-1磷酸二氢钠(NaH2PO4),0.25 mmol·L-1氯化钙(CaCl2),0.50 mmol·L-1硫酸镁(MgSO4),20.00 μmol·L-1乙二胺四乙酸铁钠(Fe-EDTA),9.00 μmol·L-1氯化锰(MnCl2),46.00 μmol·L-1硼酸(H3BO3),8.00 μmol·L-1硫酸锌(ZnSO4),3.00 μmol·L-1硫酸铜(CuSO4),0.03 μmol·L-1钼酸铵((NH4)6Mo7O24)。pH值调至pH 5.8。
养分缺乏和高盐处理:试验共设计4个养分缺乏(氮、磷、钾和镁)和1个高盐(氯化钠)胁迫处理。对照处理为完全营养液培养;养分缺乏处理中的磷、钾和镁的浓度分别降低到对照处理的1/20,其他养分元素浓度不变;高盐处理为在完全营养液中额外加入200 mmol·L-1的氯化钠。生物学重复3个·处理-1。
接种菌根真菌盆栽试验:试验前将洗净的石英砂灭菌后装到3 L的塑料盆中,种番茄小苗3株·盆-1,苗根部接入2.0 g·株-1左右的丛枝菌根真菌Rhizophagus irregularis孢子菌剂,共设3个生物学重复。
1.3实时荧光定量聚合酶链式反应(qRT-PCR)分析
取约2 μg的植物组织样品的总RNA,严格按照Takara公司的PrimeScript RT reagent Kit With cD-NA Eraser反转录试剂盒的操作说明进行cDNA合成及后续的实时荧光定量PCR分析(ABI plus real-time PCR system。表1)。内参基因为组成型表达的Actin基因[15]。

表1 番茄8个LHA基因及Actin的定量引物Table 1 Gene-specific primers used for real-time RT-PCR amplification of tomato LHA and Actin genes
1.4进化树分析
HA蛋白序列的比对分析和系统进化树的绘制分别由ClustalX 1.83和MEGA 4.0软件完成。构建进化树的算法采用邻接法(Neighbor-Joining)。
1.5启动子片段融合pBI121表达载体的构建
根据LHA8起始密码子ATG上游的启动子序列,设计5′端分别引入HindⅢ和BamHⅠ的正向引物(aagcttagaatccatcattggatcact)和反向引物(ggatccggtagctcaattgattgaaccc),以番茄基因组DNA为模板,进行PCR扩增,将扩增得到的2 669 bp的启动子片段克隆到pEASY-Blunt载体(北京全式金公司产品)。测序验证后用HindⅢ和BamHⅠ将启动子片段从克隆载体中切下,回收。同样将双元表达载体pBI121用HindⅢ和BamHⅠ将35S启动子片段切下并回收剩余的线性化的片段,与从克隆载体上切下的目的基因启动子片段通过 T4 DNA Ligase定向连接。将重组后的表达载体质粒用电击法转化至根癌农杆菌A-grobacterium tumefaciens感受态细胞(EHA105)中备用。
1.6烟草Nicotiana tabacum转基因
采用根癌农杆菌介导的烟草遗传转化方法(叶盘法)[16]。
1.7GUS染色
剪取不同转基因株系的根系浸入Magenta-GUS染液中,37℃反应6 h后在FAA固定液[V(甲醛)∶V(冰醋酸)∶V(70%乙醇)=1∶1∶18混合配制而成]中脱色固定10 h,镜检成像(BX50 OLYPUS显微镜)。
2 结果与分析
2.1番茄质膜H+-ATPase编码基因的鉴定及序列分析
为了确认番茄质膜H+-ATPase基因家族中的成员数目,我们以已报道的3个番茄质膜H+-ATPase基因LHA1,LHA2和LHA4的全长编码序列(CDS)作为查询序列,采用BLASTN和TBLASTN程序分别在番茄基因组数据库和EST/cDNA数据库中进行同源序列搜索。通过对所获得的序列进行相似性和结构域分析,最终筛选到8个具有完整编码区的LHA基因,其中7个与之前报道的LHA1~7序列完全一致,而另外一个(本研究中命名为LHA8)为从未报道过的新基因。序列分析显示,这8个LHA基因具有相近大小的开放阅读框(ORF),且编码的氨基酸序列之间具有很高的相似性(表2)。其中,LHA1和LHA2以及LHA5和LHA6之间的氨基酸序列相似性分别高达91.5%和98.3%。
2.2番茄质膜H+-ATPase编码基因的外显子/内含子结构分析
将8个LHA基因的编码序列(CDS)和DNA序列比对后分析发现,这些基因的编码区中都包含有多个内含子(图1)。其中LHA1,LHA2和LHA8含有的内含子最多,为20个;而LHA4含有的内含子最少,只有10个。对内含子在LHA基因中的分布位置进一步分析发现,这8个LHA基因具有较为保守的内含子/外显子结构特征,即各LHA基因中的不同内含子相对于外显子的位置较为一致。从图1中还可以得知,LHA基因在进化过程中存在着多处较为明显的内含子丢失(intron loss)现象,如LHA5,LHA6和LHA7相对于LHA1,LHA2和LHA8这3个基因而言在第5个和第7个内含子处出现了丢失,导致了相应位置的外显子发生了序列合并。
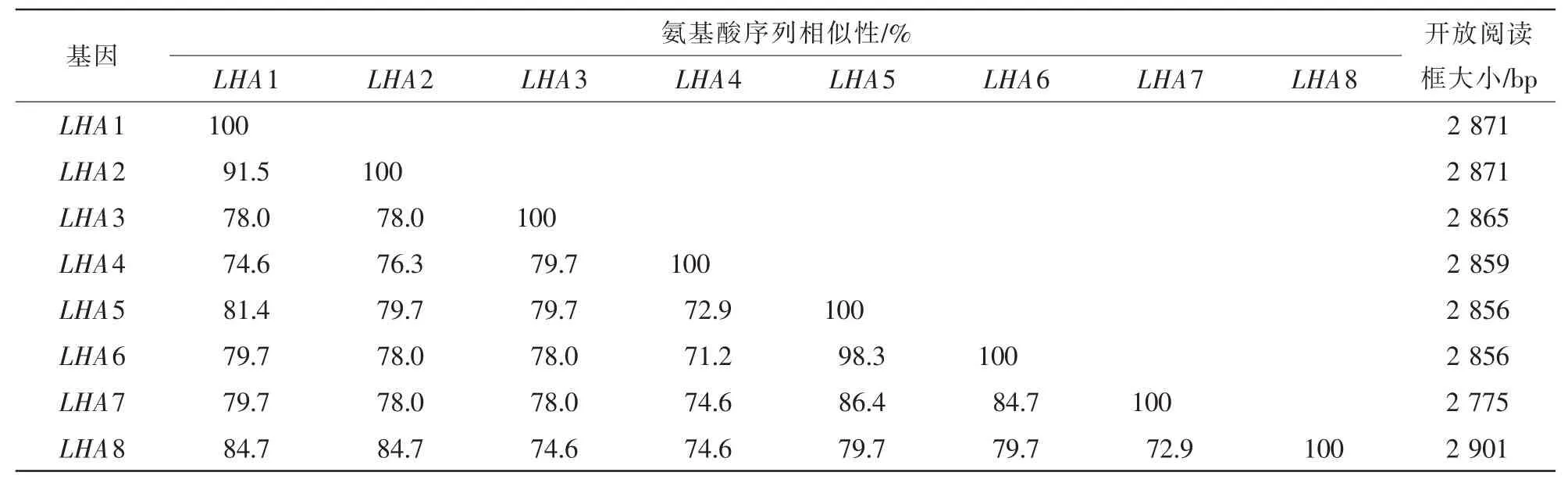
表2 番茄8个LHA基因的氨基酸序列相似性分析Table 2 Similarity matrix for the predicted amino acid sequences of the eight tomato LHA genes

图1 番茄LHA基因的外显子/内含子结构分析Figure 1 Exon/intron structures of the eight tomato LHA genes
2.3番茄质膜H+-ATPase家族基因的进化分析
对番茄LHA基因以及其他模式植物中同源基因序列进行的系统进化分析发现,植物质膜H+-ATPase基因家族可以大致分为5个亚家族(图2),与之前其他课题组所报道的结果类似。除了番茄LHA3孤立于这5个亚家族之外,其他7个LHA基因分布于4个亚家族中,其中LHA1和LHA2分布在第I亚家族,且聚在同一进化分枝的末端,暗示了LHA1和LHA2的进化关系较近,复制产生这2个同源基因的事件发生在茄科植物与其他双子叶植物分化之后。与此类似的是,聚集在第Ⅳ亚家族的LHA5,LHA6 和LHA7的进化关系也非常近。由于LHA5和LHA6之间的序列相似性非常高(表1),且外显子/内含子的结构完全一致(图1),暗示LHA5和LHA6可能是来源于同一个基因的复制,因此推测这2个基因可能具有相似的表达调控模式或行使相似的生物学功能。LHA4和LHA8分别处于第Ⅱ和第Ⅴ亚家族,与来自于其他双子叶和单子叶植物中的同源基因具有相对较近的进化关系,暗示了LHA4和LHA8的进化起源较早,至少出现在双子叶植物和单子叶植物分化之前。值得注意的是,番茄LHA基因的内含子/外显子结构特征能够很好地反应这些基因之间的进化关系,即在进化上具有较近关系的LHA同源基因,其内含子/外显子的结构特征也较为接近。
2.4番茄质膜H+-ATPase家族基因的组织表达模式分析
图3显示为番茄LHA基因的组织表达特征。结果显示:在正常培养条件下,除了LHA8在所有被分析的组织中都检测不到表达信号外,其他7个LHA基因都能在某些或某个组织中被检测到较为明显的表达。具体而言,LHA1,LHA2和LHA4在所有的组织中都有表达,但在果实中的表达都相对较低,其中LHA2在所有组织中的表达丰度相对较高。尽管LHA3在所有被检测的组织中也都有表达,但除了在花中有相对较高的表达之外,在其他组织中的表达都很微弱。LHA5,LHA6和LHA7这3个基因的表达模式比较特异,几乎只在花中有表达,其中LHA5和LHA6的表达尤为强烈(图3)。

图2 番茄LHA基因及其他植物中同源基因的进化分析Figure 2 Phylogenetic analysis of tomato LHA genes and other plant homologs
2.5LHA8的表达调控模式分析
鉴于LHA8为新报道的基因,且在正常培养的番茄中不能被检测到表达。为了验证该基因的表达是否受到其他环境因素调控,本研究检测了LHA8在不同养分(氮、磷、钾和镁)缺乏、高盐(氯化钠)胁迫以及接种菌根真菌处理条件下番茄根系中的表达水平(图4)。结果表明:LHA8在养分缺乏和高盐处理的番茄根系中也都检测不到表达,但是在接种菌根真菌处理的番茄根系中能够检测到强烈的表达(图4)。
为了更直观地证实LHA8在被菌根真菌侵染根系中的表达部位,我们构建了LHA8启动子片段(命名为pLHA8)融合GUS报告基因的双元表达载体pLHA8-GUS,并通过农杆菌介导的转基因方法转化到了烟草中。对阳性转基因烟草植株的GUS染色分析发现,LHA8的启动子能够驱动GUS报告基因在被菌根真菌侵染的根系中特异性表达,且表达部位主要集中在被菌根真菌菌丝侵入形成丛枝的细胞中表达,在未被菌丝侵染的细胞中检测不到GUS表达(图5)。
3结论与讨论
质膜H+-ATPase是植物细胞膜上非常重要的功能蛋白,被认为是植物细胞代谢和生命活动过程的主宰酶[5]。迄今为止,质膜H+-ATPase的活性已被证实与植物生长发育过程中的很多生理过程(如细胞伸长生长、气孔开闭、胞内pH调节、养分吸收等)密切相关[4]。鉴于质膜H+-ATPase功能的重要性,植物在漫长的进化过程中进化和保留了多个编码质膜H+-ATPase的同源基因。如在模式植物拟南芥中已被报道存在有11个质膜H+-ATPase的编码基因(AHA1~11)[7];在单子叶禾本科Gramineae水稻Oryza sativa中也已经被鉴定到10个具有转录活性的质膜H+-ATPase基因(OsA1~10)[6]。有意思的是,尽管番茄的基因组大小(约900 Mb)要比拟南芥(约125 Mb)和水稻的基因组(约460 Mb)要大得多[12],但通过对番茄全基因组序列搜索只获得了8个质膜H+-ATPase编码基因,序列分析表明,这些LHA基因具有较为保守的编码序列和外显子/内含子结构特征,其中新报道的LHA8与之前报道过的LHA1和LHA2的外显子/内含子结构完全一致。
前期有研究表明,植物中的质膜H+-ATPase基因在进化上可以被大致分为5个亚家族(Ⅰ~Ⅴ)[17]。对拟南芥、烟草等植物中质膜H+-ATPase基因的表达分析显示,处于第Ⅰ和第Ⅱ亚家族中的成员,其表达模式偏向于组成型,即几乎在所有的组织器官中都有表达;而处于其他几个亚家族中的成员的表达模式较为特异,只能在少数组织器官中有所表达[18]。在本研究中对番茄LHA基因的表达模式分析的结果也获得了类似的结论。LHA1,LHA2和LHA4在所有被检测的组织器官中都有表达,暗示了这几个基因在番茄植株中可能参与了一系列的生理过程。LHA5,LHA6和LHA7表达模式比较特异,几乎只在花中有表达,暗示了这几个基因的功能主要与花器官和生殖器官发育相关,且这些基因之间可能存在着一定程度的功能冗余。之前有研究显示,质膜H+-ATPase基因在转录水平上的表达受到很多因素(如养分缺乏、盐害、病菌侵害和菌根真菌共生等)的影响[11]。LHA8无论是在正常培养条件下,还是在养分缺乏和高盐处理中都不表达,但能够在菌根真菌侵染的根系中强烈表达。菌根真菌是土壤中一类属于球囊霉门Glomeromycota的能够与大多数植物根系形成互惠共生关系的真菌[19]。形成菌根共生体后,菌根真菌的一端(根内菌丝)定殖于根系皮层细胞内,另一端(根外菌丝)穿越根际养分耗竭区。植物根系借助于菌根真菌的根外菌丝可数十倍地扩展在土壤中的吸收空间,增加对土壤中养分(主要是磷)的吸收利用;作为交换,植物提供给菌根真菌多达20%的光合产物以维持其生长繁殖[20]。LHA8在接种菌根真菌的番茄根系中特异性表达,暗示了该基因可能参与了植物与菌根真菌共生过程中的养分和信号交换过程。最近在蒺藜苜蓿Medicago truncatula和水稻中也各发现了1个菌根真菌共生特异诱导表达的质膜H+-ATPase基因(MtHA1和OsHA1/OsA8)[21-22]。在水稻中突变OsHA1/OsA8能够显著影响水稻植株根系与菌根真菌共生,并抑制水稻对土壤中磷素营养的吸收[22]。从进化树上分析可知,LHA8与MtHA1和OsHA1/OsA8同处于第Ⅴ亚家族,暗示了LHA8在进化中保留了与MtHA1和OsHA1/OsA8相似的生物学功能。本研究通过对番茄质膜H+-ATPase基因家族各成员的鉴定、序列结构、进化关系和表达模式分析可以为将来深入研究质膜H+-ATPase在番茄植物生长发育过程中的作用机制提供理论依据。

图3 番茄LHA基因的表达模式分析Figure 3 Expression analysis of the tomato LHA genes

图4 LHA8在不同处理条件下番茄根系中的表达分析Figure 4 Expression analysis of LHA8 in roots of the tomato plants under different treatments
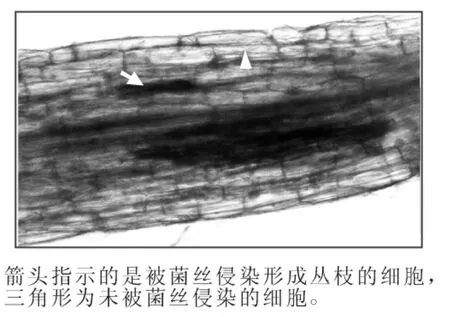
图5 LHA8启动子驱动GUS报告基因在转基因烟草接种菌根真菌的根系中的表达分析Figure 5 Expression analysis of the GUS reporter driven by LHA8 promoter in transgenic tobacco roots colonized by arbuscular mycorrhizal fungi
[1]MORSOMME P,BOUTRY M.The plant plasma membrane H+-ATPase:structure,function and regulation[J].Biochim Biophysic Acta Biomembr,2000,1465(1/2):1-16.
[2]KANCZEWSKA J,MARCO S,VANDERMEEREN C,et al.Activation of the plant plasma membrane H+-ATPase by phosphorylation and binding of 14-3-3 proteins converts a dimer into a hexamer[J].Proc Natl Acad Sci USA,2005,102(33):11675-11680.
[3]SONDERGAARD T E,SCHULZ A,PALMGREN M G.Energization of transport processes in plants.Roles of the plasma membrane H+-ATPase[J].Plant Physiol,2004,136(1):2475-2482.
[4]ALSTERFJORD M,SEHNKE P C,ARKELL A,et al.Plasma membrane H+-ATPase and 14-3-3 isoforms of Arabidopsis leaves:evidence for isoform specificity in the 14-3-3/H+-ATPase interaction[J].Plant Cell Physiol,2004,45(9):1202-1210.
[5]GAXIOLA R A,PALMGREN M G,SCHUMACHER K.Plant proton pumps[J].FEBS Lett,2007,581(12):2204-2214.
[6]BAXTER I,TCHIEU J,SUSSMAN M R,et al.Genomic comparison of P-type ATPase ion pumps in Arabidopsis and rice[J].Plant Physiol,2003,132(2):618-628.
[7]HARUTA M,BURCH H L,NELSON R B,et al.Molecular characterization of mutant Arabidopsis plants with reduced plasma membrane proton pump activity[J].J Biol Chem,2010,285(23):17918-17929.
[8]ROBERTSON W R,CLARK K,YOUNG J C,et al.An Arabidopsis thaliana plasma membrane proton pump is essential for pollen development[J].Genetics,2004,168(3):1677-1687.
[9]LAN Ping,LI Wenfeng,LIN Wendai,et al.Mapping gene activity of Arabidopsis root hairs[J].Genome Biol,2013,14(16):1-20.
[10]BAXTER I R,YOUNG J C,ARMSTRONG G,et al.A plasma membrane H+-ATPase is required for the formation of proanthocyanidins in the seed coat endothelium of Arabidopsis thaliana[J].Proc Natl Acad Sci USA,2005,102(7):2649-2654.
[11]SCHALLER A,OECKING C.Modulation of plasma membrane H+-ATPase activity differentially activates wound and pathogen defense responses in tomato plants[J].Plant Cell,1999,11(2):263-272.
[12]SATO S,TABATA S,HIRAKAWA H,et al.The tomato genome sequence provides insights into fleshy fruit evolution[J].Nature,2012,485(7400):635-641.
[13]ROSEWARNE G M,SMITH F A,SCHACHTMAN D P,et al.Localization of proton-ATPase genes expressed in arbuscular mycorrhizal tomato plants[J].Mycorrhiza,2007,17(3):249-258.
[14]EWING N N,BENNETT A B.Assessment of the number and expression of P-Type H+-ATPase genes in tomato[J]. Plant Physiol,1994,106(2):547-557.
[15]CHEN Aiqun,CHEN Xiao,WANG Huimin,et al.Genome-wide investigation and expression analysis suggest diverseroles and genetic redundancy of Pht1 family genes in response to Pi deficiency in tomato[J].BMC Plant Biol,2014,14(1):61.doi:10.1186/1471-2229-14-61.
[16]HORSCH R B,FRY J E,HOFFMAN N L,et al.A simple and general method for transferring genes into plants[J]. Science,1985,227(4691):1229-1231.
[17]ARANGO M,GÉVAUDANT F,OUFATTOLE M,et al.The plasma membrane proton pump ATPase:the significance of gene subfamilies[J].Planta,2003,216(3):355-365.
[18]ZHU Yiyong,DI Tingjun,XU Guohua,et al.Adaptation of plasma membrane H+-ATPase of rice roots to low pH as related to ammonium nutrition[J].Plant Cell Environ,2009,32:1428-1440.
[19]LIAO Dehua,CHEN Xiao,CHEN Aiqun,et al.The characterization of six auxin-induced tomato GH3 genes uncovers a member,SlGH3.4,strongly responsive to arbuscular mycorrhizal symbiosis[J].Plant Cell Physiol,2015,56(4):674-687.
[20]SMITH S E,SMITH F A,JAKOBSEN I.Functional diversity in arbuscular mycorrhizal(AM)symbioses:the contribution of the mycorrhizal P uptake pathway is not correlated with mycorrhizal responses growth or total P uptake[J]. New Phytol,2004,162(2):511-524.
[21]KRAJINSKI F,COURTY P E,SIEH D,et al.The H+-ATPase HA1 of Medicago truncatula is essential for phosphate transport and plant growth during arbuscular mycorrhizal symbiosis[J].Plant Cell,2014,26(4):1808-1817.
[22]WANG Ertao,YU Nan,BANO S A,et al.A H+-ATPase that energizes nutrient uptake during mycorrhizal symbioses in rice and Medicago truncatula[J].Plant Cell,2014,26(4):1818-1830.
Identification and expression analysis of tomato plasma membrane H+-ATPase family genes
LIU Jianjian,LIU Junli,JI Minjie,CHEN Jiadong,YANG Xiaofeng,CHEN Aiqun
(College of Resources and Environmental Sciences,Nanjing Agricultural University,Nanjing 210095,Jiangsu,China)
Plasma membrane(PM)H+-ATPase,which catalyzes ATP hydrolysis coupled with pumping out protons from cells and which is ubiquitously existent in all cell types of plants examined so far,has been characterized to be encoded by a multigene family involved in a number of plant physiological and developmental processes.The aim of this study is to identify and characterize the genes encoding PM H+-ATPase in Solanum lycopersicum(tomato).Through genomic sequence database hunting and bioinformatics analysis,eight putative PM H+-ATPase genes(named as LHA1-8)were identified,and their expression patterns were subsequently analysed by quantitative real-time reverse transcription polymerase chain reaction(qRT-PCR).Bioinformatics analysis revealed a high conservation among these paralogous genes in both coding sequences and Exon/Intron structures.Expression analysis showed that LHA1,LHA2,LHA3,and LHA4 were expressed in all tissues examined;whereas LHA5,LHA6,and LHA7 were predominantly expressed in flowers.LHA8,the newly identified gene in this study,with barely detectable transcripts in normal growth conditions or in nutrient(N,P,K,and Mg)-deficient conditions,was strongly induced in arbuscular mycorrhizal(AM)fungal-colonized roots.A 2 669 bp LHA8 promoter fragment could direct the β-glucuronidase(GUS)reporter expression specifically in transgenic tobacco roots colonized by AM fungi.The results provide new insights into the evolutionary conservation and functional divergence of the tomato PM H+-ATPase gene family during plant growth and development.[Ch,5 fig.2 tab.22 ref.]
molecular biology;Solanum lycopersicum(tomato);plasma membrane H+-ATPase;expression pattern;mycorrhizal fungi
S641.2;Q943.2
A
2095-0756(2016)05-0734-08
10.11833/j.issn.2095-0756.2016.05.002
2015-06-27;
2015-08-06
国家自然科学基金资助项目(31372121)
刘健健,从事丛枝菌根共生的分子机制研究。E-mail:jianjian2013103114@126.com。通信作者:陈爱群,副教授,博士,从事植物与微生物互作的分子机制研究。E-mail:chenaq8@163.com
浙 江 农 林 大 学 学 报,2016,33(5):734-741
Journal of Zhejiang A&F University
