鸡长链非编码RNA发掘及组织特异性表达分析
2016-10-26李辉张继扬杜志强
李辉,张继扬,杜志强
(农业部鸡遗传育种重点实验室,黑龙江省普通高等学校动物遗传育种与繁殖重点实验室,东北农业大学动物科学技术学院,哈尔滨 150030)
鸡长链非编码RNA发掘及组织特异性表达分析
李辉,张继扬,杜志强
(农业部鸡遗传育种重点实验室,黑龙江省普通高等学校动物遗传育种与繁殖重点实验室,东北农业大学动物科学技术学院,哈尔滨150030)
长链非编码RNA(Long non-coding RNA,lncRNA)是一类长度大于200个核苷酸、缺少蛋白编码功能的RNA。lncRNA可从多层面调控基因表达,影响表型性状。鸡作为重要经济动物和模式生物,lncRNA研究相对滞后。为加快鸡lncRNA研究进展,利用公共数据库(如NCBI-SRA等)中鸡高通量转录组测序(RNA-seq)数据,通过生物信息学方法发掘8 040条鸡lncRNA,发现大量组织特异性表达lncRNA,为鸡lncRNA功能研究奠定基础。
鸡;RNA-seq;lncRNA;组织特异性表达
网络出版时间2016-7-20 16:39:50[URL]http://www.cnki.net/kcms/detail/23.1391.S.20160720.1639.014.html
李辉,张继扬,杜志强.鸡长链非编码RNA发掘及组织特异性表达分析[J].东北农业大学学报,2016,47(7):40-47.
Li Hui,Zhang Jiyang,Du Zhiqiang.Identification of long non-coding RNAs and their tissue-specific expression analysis in chickens[J].Journal of Northeast Agricultural University,2016,47(7):40-47.(in Chinese with English abstract)
长链非编码RNA是新发现的一类长度大于200个核苷酸,缺乏蛋白编码功能,由RNA聚合酶II或RNA聚合酶III转录生成的RNA,可在多层面参与转录后调控和表观调控等生物学过程[1-3]。长链非编码RNA是RNA聚合酶转录过程副产物,不具备生物学功能。近年研究发现lncRNA可作为miRNA的吸附“海绵体”、转录因子引导者、顺式调控元件以及蛋白支架等,与miRNA、mRNA、DNA及蛋白等相互作用,参与基因表达调控、染色体沉默、蛋白质合成等诸多生物学过程[4-6]。同时,与蛋白编码基因相比lncRNA保守性较差,表现明显组织特异性[5,7]。Cabili等对人的24种组织或细胞进行lncRNA特异性表达分析[7],发现约有78%lncRNA表现组织或细胞特异性,约19%蛋白编码基因表现组织或细胞特异性。Li等发现小鼠肝脏特异性富集lncLSTR(Liver-specific triglyceride regulator lncRNA)影响小鼠脂质代谢,表明组织特异性表达的lncRNA可能在特定组织中发挥重要生物学功能[8]。
随着二代测序技术兴起,动物转录组学研究发展迅速。大量动物高通量转录组测序(RNA-seq)数据公布。基于公共数据库(如NCBI-SRA数据库)中RNA-seq数据,通过生物信息学手段,已成功在人和鼠等动物中发掘大量lncRNA[1-3]。如Paralkar等利用NCBI-SRA数据库中人RNA-seq数据,发掘1 109条lncRNA[3];Lv等利用NCBI-SRA数据库中鼠RNA-seq数据,发掘29 837条lncRNA[1]。鸡作为重要经济动物和模式生物,具有不可替代经济价值和独特生物学特性。然而,国内外鸡lncRNA功能研究鲜见报道[9]。本研究利用NCBISRA数据库中鸡RNA-seq数据,通过生物信息学手段初步探讨鸡lncRNA序列结构特征和组织表达特异性,为鸡lncRNA功能研究奠定基础。
1 材料与方法
1.1材料
1.1.1RNA-Seq数据
本研究所用数据主要来源于NCBI-SRA、Ensembl等公共数据库;其中RNA-seq数据(见表1)来源于NCBI-SRA(http://www.ncbi.nlm.nih.gov/ sra/?term=)数据库,共计7组109个鸡RNA-seq数据样本,包括大脑、小脑、下丘脑、垂体、坐骨神经、心脏、肺脏、肝脏、肾脏、脾脏、脾胃、胸肌、骨骼肌、睾丸、结肠、脂肪等16种组织的RNA-seq数据及P1(原条期,Primitive streak)、HH(胚胎发育期,Hamburger Hamilton stages)6、HH11、HH14、HH16、HH19、HH28、HH38等8种不同时期鸡胚胎RNA-seq数据(见表1)。鸡基因组信息文件(ftp://ftp.ensembl.org/pub/release-75/ fasta/gallus_gallus/dna/)和鸡基因组注释文件(ftp:// ftp.ensembl.org/pub/release-75/gtf/gallus_gallus)均来源于Ensembl数据库。

表1 发掘鸡lncRNA的RNA-Seq数据Table 1Public RNA-seq data used for lncRNA identification
1.1.2生物信息学软件
FastQC,RNA-seq数据质量控制;Cutadapt,RNA-seq数据序列接头(Adaptor)剪切等;Tophat2,短序列比对[10];Bowtie2,建立索引文件[10];Cufflinks软件包,组装转录本、估计转录本丰度、并检测样本间差异表达及可变剪接[10];gffread,翻译基因组注释文件[10];CPC(Coding Potential Calculator),编码潜能预测[11];Blastn(v2.2.26),序列比对,lncRNA数据集合并。
1.2方法
1.2.1lncRNA发掘
利用鸡RNA-seq数据通过生物信息学手段发掘鸡lncRNA[1-3,12],具体流程如下:
①RNA-seq数据预处理。使用FastQC软件质量检测表1中的RNA-seq数据,Cutadpt等软件去除reads中adaptor和不稳定的序列等。
②与参考基因组比对。使用TopHat2软件,以Ensembl数据库中鸡的基因组文件和基因组注释文件为参考信息作有参比对。
③转录本拼接。Cufflinks软件包利用Tophat2软件比对结果作转录本拼接,定量分析转录本。
④转录本筛选。通过对比已注释基因组信息,筛选cufflinks软件包cuffcompare模块分析结果(merged.gtf文件)中class_code为“u”的转录本(即基因组未注释的转录本),同时通过Cufflinks计算每条转录本reads覆盖度,剔除reads覆盖度小于3的未注释转录本。
⑤设置长度和外显子数目阈值。筛选长度≥200个核苷酸和外显子数目≥2的未注释转录本作后续分析。
⑥编码潜能预测。将class_code为“u”(即基因组未注释的转录本)、reads覆盖度大于等于3、长度大于等于200个核苷酸且外显子数目≥2的转录本序列信息输入CPC软件,编码潜能评估得到编码潜能评分,即CPC评估得分。CPC评估得分小于等于-1的转录本判定为lncRNA。
⑦合并lncRNA数据集。使用Blastn软件,设置阈值e-value小于等于1e-11,删除重复项,合并7组RNA-seq数据中lncRNA[13]。
1.2.2组织特异性表达分析
以Cufflinks软件包中Cuffdiff(http://coletrapnelllab.github.io/cufflinks/cuffdiff/index.html)软件对lncRNA作定量分析,得到各样本中lncRNA表达量信息,并以FPKM(Tragments per kilobaseof exon per million fragments mapped)为单位输出表达量结果。
lncRNA组织特异性分析以lncRNA组织特异性表达值(The tissue specificity,Ts值)表示,即一种组织中某条lncRNA表达量(FPKM)占所有组织中该lncRNA表达量总和的比例[2]。
2 结果与分析
2.1全基因组范围内发掘鸡lncRNA
lncRNA是一类长度大于200个核苷酸、缺少蛋白编码功能的RNA分子。本研究基于lncRNA长度、缺少蛋白编码功能特点,综合lncRNA筛选方法,设置一系列严格lncRNA筛选条件。通过以下五个步骤利用NCBI-SRA数据库中7组109个鸡的RNA-seq数据发掘鸡lncRNA,并作后续lncRNA特征分析。
①与参考基因组比对。采用Bowtie2和Tophat2等软件对7组109个鸡RNA-seq数据样本作有参比对,得到各组RNA-seq数据比对到参考基因组reads数。
②转录本拼接。采用Cufflinks软件对Tophat2等软件比对结果作转录本拼接,各组RNA-seq数据得到转录本数。
③转录本筛选。通过筛选cuffcompare模块分析结果中class_code为“u”以及reads覆盖度≥3的基因组未注释转录本,得到各组RNA-seq数据的未注释转录本数。
④设置长度和外显子数目阈值。筛选长度≥200个核苷酸未注释转录本,同时为有效避免假阳性错误,筛选外显子数目≥2未注释转录本作后续分析,得到转录本过滤结果。
⑤编码潜能预测。具有编码潜能与否是判定转录本是否为lncRNA关键步骤。将class_code为“u”、reads覆盖度≥3、长度≥200个核苷酸且外显子数目≥2未注释转录本序列信息输入CPC软件,编码潜能评估得到编码潜能评分,即CPC评估得分。CPC评估得分≤-1的未注释转录本判定为lncRNA。
最终得到7组鸡lncRNA数据集(见表2),共计11 180条lncRNA。使用Blastn软件,设置阈值evalue<=1e-11[13],合并7组lncRNA数据集,得到8 040条鸡lncRNA。
2.2鸡lncRNA序列结构分析
为了解鸡lncRNA基本序列结构特征,分析所发掘8 040条鸡lncRNA染色体分布、序列长度、外显子数量等基本序列结构特征,并与Ensembl数据库中已注释的15 675条鸡蛋白编码基因(ftp://ftp. ensembl.org/pub/release-75/gtf/gallus_gallus)相关序列结构特征作比较。
2.2.1鸡lncRNA染色体分布
为了解鸡lncRNA参考基因组分布状况,统计鸡lncRNA在不同染色体上分布。8 040条鸡lncRNA染色体分布情况(见图1),结果表明鸡lncRNA在各染色体(1~28号染色体,Z和W染色体)均有分布,其中1~5号大染色体分布较多,占总数59.02%;W染色体上lncRNA分布最少,仅占0.05%。该结果与鸡蛋白编码基因在各染色体上的分布相似(见图2),即较大染色体分布更多lncRNA[25]。
表2 鸡lncRNA发掘结果Table 2Information on lncRNAs identified in chickens

图1 鸡lncRNA染色体分布Fig.1Chromosomal distribution of chicken lncRNAs
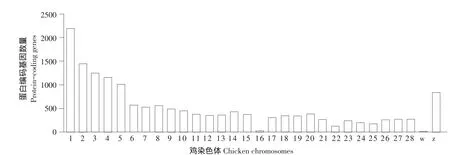
图2 鸡蛋白编码染色体分布Fig.2Chromosomal distribution of chicken protein coding genes
2.2.2鸡lncRNA序列长度特征
为了解鸡lncRNA序列长度(lncRNA各外显子序列长度之和)特征,统计鸡lncRNA序列长度。8 040条lncRNA序列长度分布情况见图3,结果表明lncRNA序列长度多集中在500~2 000 bp,占lncRNA总数61.3%。15 675条鸡蛋白编码基因序列长度分布情况见图4,结果表明蛋白编码基因序列长度多集中在500~3 000 bp,占蛋白编码基因总数63.6%。分别计算鸡lncRNA和鸡蛋白编码基因序列平均长度,发现鸡lncRNA序列平均长度为1 461 bp,鸡蛋白编码基因序列序列平均长度为2 592 bp。
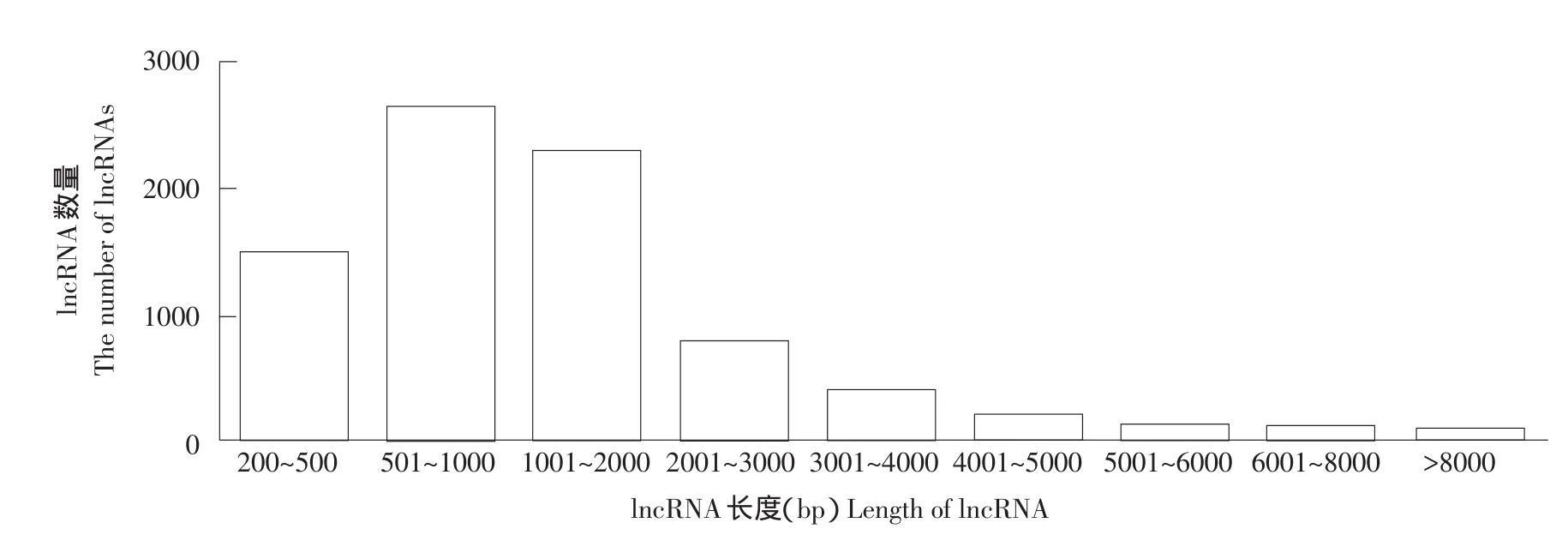
图3 lncRNA长度分布Fig.3Length distribution of lncRNAs

图4 蛋白编码基因长度分布Fig.4Length distribution of Protein-coding genes
2.2.3鸡lncRNA外显子数特征
为了解鸡lncRNA外显子数状况,统计鸡lncRNA外显子数,并与鸡蛋白编码基因外显子数比较。8 040条lncRNA外显子数分布情况见图5,结果表明lncRNA外显子数以2个居多,约占lncRNA总数60%,大于7个外显子的lncRNA较少,仅占1%。鸡蛋白编码基因与鸡lncRNA的外显子数特征区别明显,鸡蛋白编码基因以大于7个外显子居多,占蛋白编码基因总数55%(见图6);仅有6%蛋白编码基因外显子数目为2。
2.3鸡lncRNA组织特异性表达分析
研究表明,哺乳动物lncRNA有明显组织表达特异性[5,7]。为了解鸡lncRNA组织表达特异性,本研究选取一组包含原鸡12种组织RNA-seq数据(ERP003988)(包括脂肪、神经、肾脏、小脑、下丘脑、心脏、脾脏、肺脏、肝脏、大脑、脾胃、胸肌等组织,每种组织包含2个RNA-Seq数据样本),特异性分析鸡lncRNA组织表达。
2.3.1鸡各组织lncRNA数量
本组RNA-seq数据共发掘出3 346条鸡lncRNA。本研究首先统计该组RNA-seq数据中每种组织中lncRNA数量,发现每种组织中检测lncRNA数量不同(见表3)。
其中,肾脏组织中lncRNA数量最多,为1 143条;其次是坐骨神经、小脑、下丘脑、大脑、心脏、肝脏、肺脏、脂肪、脾脏、腺胃,胸肌;胸肌中lncRNA数量最少,仅189条(见表3)。
2.3.2鸡lncRNA组织表达特异性分析
为初步了解鸡lncRNA组织表达特异性,使用Cuffdiff软件计算每种组织中每条lncRNA表达量。依据文献提供lncRNA组织特异性分析方法,计算每种组织中每条lncRNA组织表达特异性值(The tissue specificity,Ts值),并统计每个组织中特异性表达lncRNA数量。
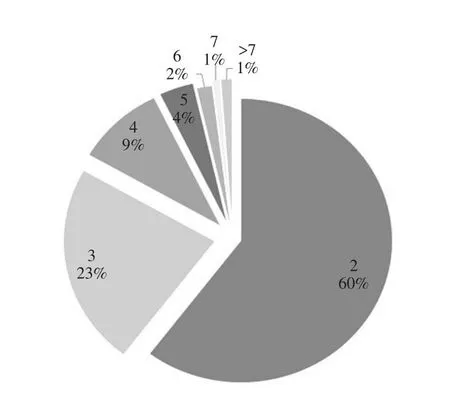
图5 lncRNA外显子数目Fig.5Distribution of exon numbers of lncRNAs
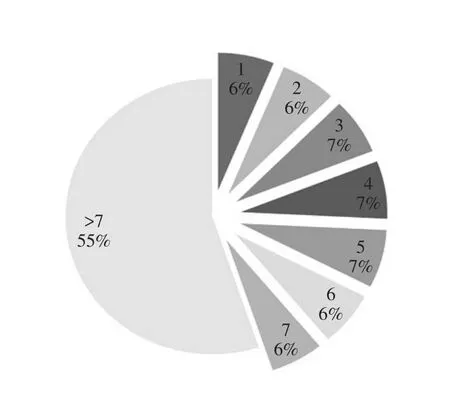
图6 蛋白编码基因外显子数目Fig.6Distribution of exon numbers of protein-coding genes

表3 鸡各组织lncRNA数Table 3Total number of lncRNAs
lncRNA组织特异性表达值(Ts值)是指一种组织中lncRNA表达量(FPKM)占所有12种组织中相同lncRNA表达量总和比例。当某组织样本中一条lncRNA的Ts值等于1时,表示这条lncRNA仅在此组织样本中表达,即此lncRNA表现较强组织表达特异性;当某组织样本中一条lncRNA的Ts值在0.8~1.0之间时,表明此条lncRNA在此组织样本中表达量远高于其他组织样本中此lncRNA表达量之和,即此lncRNA表现较强组织表达特异性。
表达分析原鸡12种组织3 346条lncRNA组织特异性,结果如下(见表4):坐骨神经、肾脏、小脑和下丘脑中组织特异性表达lncRNA数量较多,其次是心脏、脾脏、肺脏、脂肪、大脑、肝脏、腺胃,最后是胸肌组织。其中,坐骨神经特异表达lncRNA占坐骨神经lncRNA总数比例最大,约34.30%;其次脾脏、腺胃、肺脏、肾脏、下丘脑、小脑、肌肉、心脏、脂肪、大脑,最低的是胸肌组织。
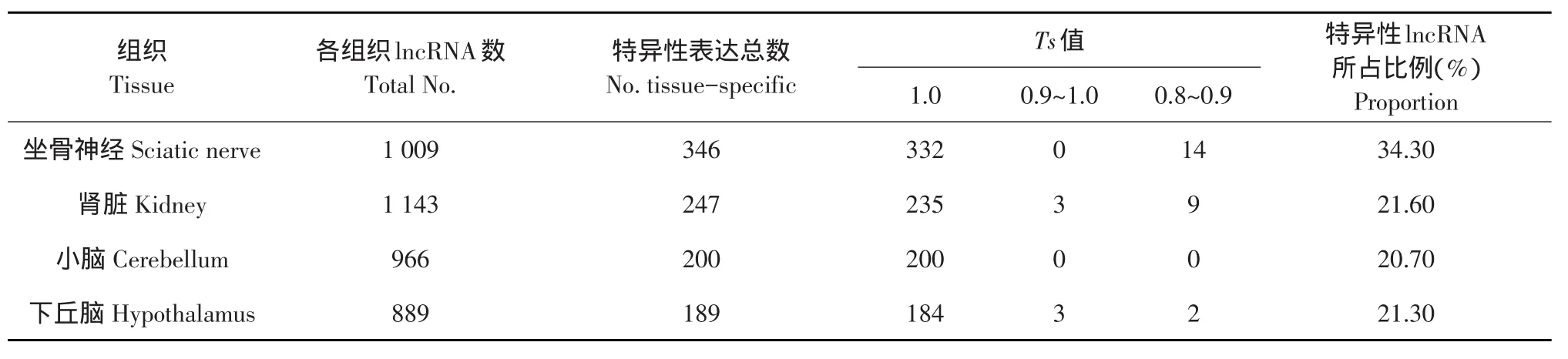
表4 各组织lncRNA特异性表达数量Table 4The number of lncRNAs with tissue-specific expression patterns
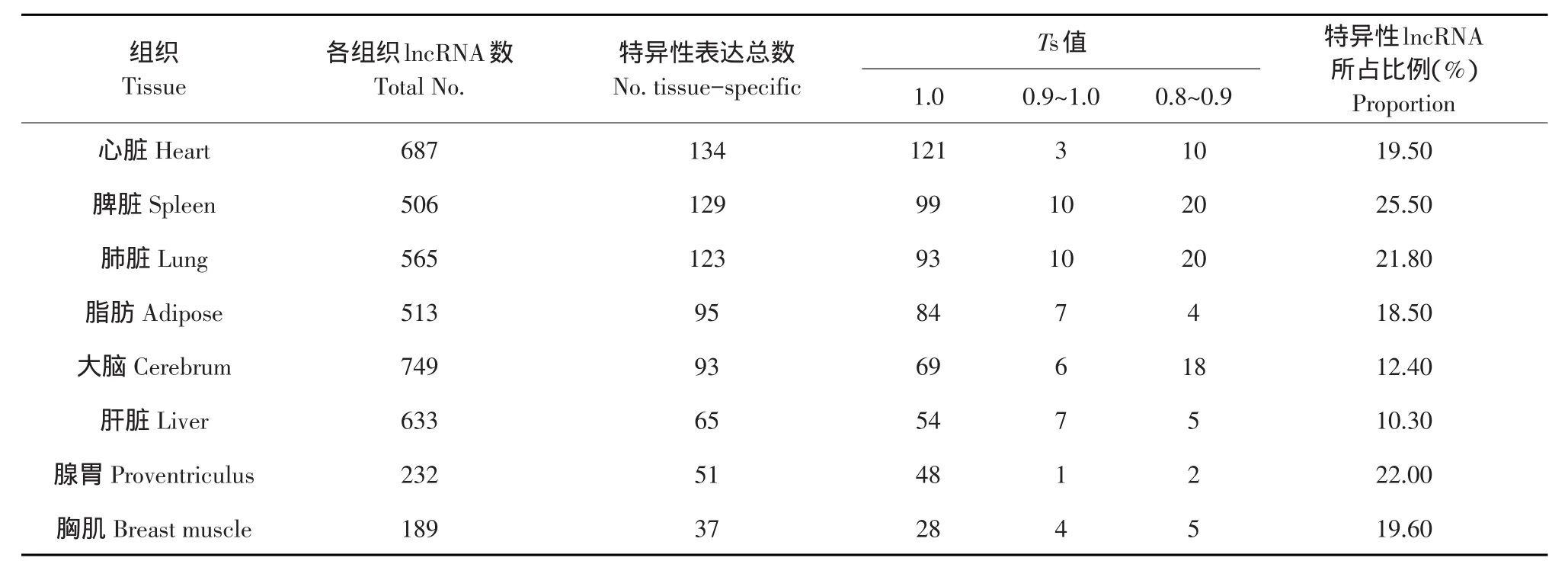
续表
3 讨论与结论
随着二代测序技术发展,大量动物转录组数据不断公布。基于公共数据库(如NCBI-SRA)中RNA-seq数据,通过生物信息学手段已成功在人和小鼠等哺乳动物中发掘出大量lncRNA[1-3]。发掘lncRNA生物信息学流程较为固定,一般由以下5步组成:①RNA-seq数据质控;②reads比对、转录本拼装;③发掘基因组未注释的转录本;④去除长度小于200个核苷酸的未注释转录本;⑤编码潜能预测,获得候选lncRNA集合。其中,转录本编码潜能预测是lncRNA生物信息学发掘关键步骤。本研究所用的转录本编码潜能预测软件CPC为目前国内外文献报道中常见编码潜能预测软件[1,12,14-15]。CPC是一种蛋白质编码潜能计算工具,将转录本与已知蛋白数据库作blastx比对,依据转录本各个编码框生物学序列特征等,通过支持向量机分类器评估转录本编码潜能,编码潜能预测准确性较高[11]。本研究利用公共数据库中鸡RNASeq数据,通过生物信息学手段在全基因范围内发掘出8 040条lncRNA。Paralkar等利用NCBISRA数据库中人的RNA-seq数据[3],发掘1 109条lncRNA;Lv等利用NCBI-SRA数据库中鼠RNA-seq数据,发掘29 837条lncRNA[1];Zhou等利用NCBI-SRA数据库中猪RNA-seq数据,发掘6 621条lncRNA[12];鸟类中利用RNA-seq数据发掘lncRNA报道较少。
二代测序虽然能够有效捕捉lncRNA序列信息,但如何精准预测lncRNA基因结构和编码潜能仍存在困难[18-19]。在lncRNA基因结构和编码潜能预测方面:首先,由于二代测序技术测序深度、错误及偏好性等,导致转录本拼装阶段易产生部分转录本和假阳性转录本,影响lncRNA识别;其次,如何鉴定转录本的编码潜能性,准确区分蛋白编码基因和非编码基因,尚无准确生物学判定标准。因此,目前研究者倾向于生物信息学预测后利用分子生物学试验验证,两种方法互补研究lncRNA生物学功能[8,20]。
Billerey等研究发现[16],牛lncRNA在各染色体均有分布,且1号染色体分布最多而在X染色体分布最少,表现出与牛蛋白编码基因相似染色体分布状况。Ponjavic等研究发现[17],鼠lncRNA除不具备蛋白编码潜能外,lncRNA在序列结构上与mRNA也不同,如lncRNA平均长度小于蛋白编码基因平均长度且外显子数普遍少于蛋白编码基因外显子数。本研究表明,鸡lncRNA序列结构特征与哺乳动物lncRNA序列结构特征类似,即鸡lncRNA在染色体分布与鸡蛋白编码基因染色体分布无明显区别,鸡lncRNA平均长度小于鸡蛋白编码基因平均长度且外显子数普遍少于鸡蛋白编码基因外显子数。
随着lncRNA研究深入,越来越多证据表明组织特异性表达的lncRNA在特定组织发挥重要生物学功能[7-8]。Cabili等分析人24种不同组织或细胞lncRNA表达特异性[7],发现每种组织或细胞中均存在特异性表达lncRNA。Li等干扰小鼠肝脏中特异性表达的lncLSTR,发现可影响小鼠肝脏中血浆甘油三酯水平[8],表明组织特异性表达lncRNA可能在特定组织中发挥重要生物学功能。本研究分析原鸡12种组织中lncRNA组织表达特性,发现坐骨神经、肾脏、小脑和下丘脑组织表达特异性lncRNA较多,其次是心脏、脾脏、肺脏、脂肪、大脑、肝脏、腺胃,胸肌组织中最少,表明原鸡12种组织中均存在特异性表达lncRNA,可能在组织中发挥重要生物学功能。
[1]Lv J,Cui W,Liu H,et al.Identification and characterization of long non-coding RNAs related to mouse embryonic brain development from available transcriptomic data[J].PLoS One,2013,8(8):71152.
[2]Tsoi L C,Iyer M K,Stuart P E,et al.Analysis of long non-coding RNAshighlightstissue-specificexpressionpatternsand epigenetic profiles in normal and psoriatic skin[J].Genome Biology,2015,16(1):24.
[3]Paralkar V R,Mishra T,Luan J,et al.Lineage and speciesspecific long noncoding RNAs during erythro-megakaryocytic development[J].Blood,2014,123(12):1927-1937.
[4]St L G,Wahlestedt C,Kapranov P.The Landscape of long noncoding RNA classification[J].Trends Genet,2015,31(5):239-251.
[5]Chen Z.Progress and prospects of long noncoding RNAs in lipid homeostasis[J].Mol Metab,2016,5(3):164-170.
[6]Blythe A J,Fox A H,Bond C S.The ins and outs of lncRNA structure:How,why and what comes next?[J].Biochim Biophys Acta,2016,1859(1):46-58.
[7]Cabili M N,Trapnell C,Goff L,et al.Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses[J].Genes Dev,2011,25(18):1915-1927.
[8]Li P,Ruan X,Yang L,et al.A liver-enriched long non-coding RNA,lncLSTR,regulates systemic lipid metabolism in mice[J]. Cell Metab,2015,21(3):455-467.
[9]Li T,Wang S,Wu R,et al.Identification of long non-protein coding RNAs in chicken skeletal muscle using next generation sequencing[J].Genomics,2012,99(5):292-298.
[10]Trapnell C,Roberts A,Goff L,et al.Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J].Nat Protoc,2012,7(3):562-578.
[11]Kong L,Zhang Y,Ye Z Q,et al.CPC:assess the protein-coding potential of transcripts using sequence features and support vector machine[J].Nucleic Acids Res,2007,35(Web Server issue): W345-W349.
[12]Zhou Z Y,Li A M,Adeola A C,et al.Genome-wide identification of long intergenic noncoding RNA genes and their potential association with domestication in pigs[J].Genome Biol Evol,2014,6(6):1387-1392.
[13]Billerey C,Boussaha M,Esquerre D,et al.Identification of large intergenic non-coding RNAs in bovine muscle using next-generation transcriptomic sequencing[J].BMC Genomics,2014,15:499.
[14]Li L,Eichten S R,Shimizu R,et al.Genome-wide discovery and characterization of maize long non-coding RNAs[J].Genome Biol,2014,15(2):R40.
[15]Weikard R,Hadlich F,Kuehn C.Identification of novel transcripts and noncoding RNAs in bovine skin by deep next generation sequencing[J].BMC Genomics,2013,14:789.
[16]Billerey C,Boussaha M,Esquerre D,et al.Identification of large intergenic non-coding RNAs in bovine muscle using next-generation transcriptomic sequencing[J].BMC Genomics,2014,15:499.
[17]Ponjavic J,Ponting C P,Lunter G.Functionality or transcriptional noise?Evidence for selection within long noncoding RNAs[J]. Genome Res,2007,17(5):556-565.
[18]孙磊,张林,刘辉.基于RNA-Seq的长非编码RNA预测[J].生物化学与生物物理进展,2012(12):1156-1166.
[19]王曦,汪小我,王立坤,等.新一代高通量RNA测序数据的处理与分析[J].生物化学与生物物理进展,2010(8):834-846.
[20]Zhao X Y,Li S,Wang G X,et al.A long noncoding RNA transcriptional regulatory circuit drives thermogenic adipocyte differentiation[J].Mol Cell,2014,55(3):372-382.
Identification of long non-coding RNAs and their tissue-specific expression analysis in chickens
LI Hui,ZHANG Jiyang,DU Zhiqiang
(Key Laboratory of Chicken Genetics and Breeding,Ministry of Agriculture,Key Laboratory of Animal Genetics, Breeding and Reproduction,Education Department of Heilongjiang Province,School of Animal Science and Technology,Northeast Agricultural University,Harbin 150030,China)
Long non-coding RNAs(lncRNAs),one class of functional RNAs longer than 200 nucleotides,lack of protein coding potential,are involved in a diverse array of biological processes. LncRNA can affect the expression of phenotype,by regulating gene expression in multiple ways. Besides its great economical value,chicken is widely used as an excellent animal model,however, chicken lncRNA research was still lagging behind.Here,we collected public RNA-Seq data sets of chickens from the NCBI-SRA database.Bioinformatics pipeline and methods were then established for the identification and tissue-specific expression analysis of lncRNAs in chickens.In total 8 040 transcripts were identified as putative lncRNAs,and chicken lncRNAs tend to have higher expression levels in neuronal tissues than other tissues.The findings lay the foundation for the further functional analysis of chicken lncRNAs.
chicken;RNA-seq;lncRNA;tissue-specific expression
S831.2
A
1005-9369(2016)07-0040-08
2016-05-23
国家863计划项目(2013AA102501);现代农业产业技术体系建设专项资金资助项目(CARS-42);国家自然科学基金(31472088)作者简介:李辉(1963-),男,教授,博士,博士生导师,研究方向为动物遗传育种。E-mail:lihui@neau.edu.cn
