白草种子基因组DNA五种提取方法的比较*
2016-10-25郝豆豆田志华雷鸣武俊喜拉多张勇群
郝豆豆,田志华,雷鸣,武俊喜,拉多,张勇群
(1.西藏大学,西藏 拉萨850000;2.中国科学院地理科学与资源研究所,西藏 拉萨850000;3.西藏自治区人民政府驻成都办事处医院,四川 成都610000)
白草种子基因组DNA五种提取方法的比较*
郝豆豆1,田志华1,雷鸣1,武俊喜2,拉多1,张勇群3
(1.西藏大学,西藏拉萨850000;2.中国科学院地理科学与资源研究所,西藏拉萨850000;3.西藏自治区人民政府驻成都办事处医院,四川成都610000)
用5种方法(传统CTAB法、改良CTAB法、高盐低pH法、改良SDS法、试剂盒法)对白草种子基因组DNA进行提取,分别用紫外分光光度计法、琼脂糖凝胶电泳法、PCR扩增检测提取的DNA的质量。结果表明,传统CTAB法提取的DNA浓度低,含有较多杂质,抑制PCR反应;改良SDS法和改良CTAB法提取的DNA纯度低,不符合检测要求;高盐低pH法和试剂盒法提取的DNA纯度高、完整性好,但试剂盒法成本高、DNA得率低。所以高盐低pH法为5种方法中最经济、效果最好的白草种子DNA提取方法。
白草;基因组DNA;DNA提取方法;高盐低pH法
白草(Pennisetumflaccidum)属禾本科(Gramineae)狼尾草属(Pennisetum),多年生沙生草本植物,多生长于道路两旁、固定半固定沙地、沙丘间荒地、农田落荒地及林缘等地,海拔2 900~4 500m[1]。为沙质草地的优势种,具有很强的固沙作用,也是牲畜喜食的优良牧草,在农田和苗圃是常见的杂草,广泛分布于中国西北部各省区[2~4]。
白草因固沙作用而具有极高的生态价值,随着分子生物学的飞速发展,从基因方面来研究白草的各种优良性状很有发展前景,提取高质量的基因组DNA是开展一系列分子生物实验的前提。常用的DNA提取方法有:CTAB(Hexadecyl trimethyl ammouium Bromide,十六烷基三甲基溴化铵)法[5]、SDS(odecyl sulfate sodium salt,十二烷基硫酸钠)法[6]、高盐低pH法[7]、尿素法[8]、异硫氰酸胍法[9]等。这些方法各有优缺点,不同的实验材料适合的方法不同,需要根据材料中的成分对提取方法进行选择,而且为了得到更好的DNA提取效果,还需要在前人研究经验的基础上对一些方法做适当的改进。本文参考了前人的研究,采用5种方法提取白草种子基因组DNA,综合比较各个因素,得出了其中的最佳提取方法,为后续的白草分子生物实验提供科学依据。
1 材料与方法
1.1材料
白草种子于2007年9月27日采自西藏自治区山南乃东,农田落荒地。
主要试剂有无水乙醇、氯仿、异戊醇、异丙醇、CTAB、PVP、DTT、Tris、EDTA、SDS、λ-marker、marker-D、核苷酸染料、loading buffer、Agarose M、Taq酶、BPB、dNTP、MgCl2、10×Taq酶配套缓冲液〔均购自生工生物工程(上海)有限公司〕;主要仪器有Eppendorf 5417R型高速冷冻离心机、超低温冰箱、水浴锅-CD3192型恒温控制器、研钵、1.5mL离心管、不同量程的移液器及相应的枪头(购自上海生物公司)、核酸蛋白测定仪(Eppendorf BioPhotometer plus)、WD-9413B凝胶成像分析仪(购自北京市六一仪器厂)、电泳槽(购自BIO-RAD公司)、PCR-T100(购自BIO-RAD公司)。
1.2DNA提取方法
将大量的白草种子在粉碎机中磨成粉,4℃中干燥保存。
1.2.1传统CTAB法[10]
称取50mg干粉置于1.5mL离心管中,加入600μL、65℃预热的CTAB提取液〔0.1mol/L Tris-HCl(pH 8.0)、3%PVP、2%β-巯基乙醇、0.025mol/L EDTA(pH 8.0)、2%CTAB〕,充分混匀后于65℃中保温30min,不时轻轻颠倒;取出,稍冷却后加入等体积的氯仿/异戊醇(24︰1),震荡至出现乳浊状,静置分层;12 000rpm、4℃离心10min,取上清液加入1/3体积无水乙醇轻微混匀后,加入等体积氯仿/异戊醇(24︰1)混匀,立即以10 000rpm离10min,取上清液加等体积的-20℃预冷的异丙醇;-20℃静置30min,14 000rpm、4℃离心23min;70%乙醇洗涤沉淀2次,风干后溶于30μL TE,标记后于-20℃保存备用。
1.2.2改良SDS法
称取50mg干粉置于1.5mL离心管中,加入600μL冰上预冷的提取液〔0.05mol/L Tris·HCl(pH8.0)、3%可溶性PVP、2%β-巯基乙醇、0.05mol/L EDTA(pH8.0)〕充分混匀后,冰上放置10min;6 000rpm离心5min,弃上清液,沉淀加入500μL、65℃预热的裂解缓冲液〔0.1mol/L Tris·HCl(pH8.0)、0.02mol/L EDTA(pH8.0)、0.5mol/L NaCl、1.5%SDS〕,65℃水浴30min,不时轻轻颠倒;取出,稍冷却后加入500μL氯仿/异戊醇(24︰1),颠倒成乳浊状;10 000rpm、4℃离心10min,重复2次;取上清液后加入1/4体积乙醇和1/3体积5mol/L NaAc(pH4.8),立即10 000rpm离心10min;取上清液后加入等体积-20℃预冷的异丙醇;置于-20℃静置30min,14 000rpm、4℃离心23min;70%乙醇洗涤沉淀2次,风干后溶于30μL TE,标记后于-20℃保存备用。
1.2.3高盐低pH值法
称取50mg干粉置于1.5mL离心管中,加入600μL、65℃预热提取液〔0.1mol/L NaAc、3%可溶性PVP、2%β-巯基乙醇、0.025mol/L EDTA(pH8.0)、1.5%SDS〕,充分混匀后于65℃中保温30min,不时轻轻颠倒;4℃、10 000rpm离心10min,取上清液后加入2/3倍体积的2.5mol/L KAC溶液(pH8.0),0℃放置15min;10 000rpm、4℃离心10min;取上清液加入等体积的氯仿/异戊醇(24︰1),震荡至出现乳浊状,静置分层后,10 000rpm、4℃离心10min,取上清液加入等体积的-20℃预冷的异丙醇;-20℃静置30min,14 000rpm、4℃离心23min;70%乙醇洗涤沉淀2次,风干后溶于30μL TE,标记后于-20℃保存备用。
1.2.4改良CTAB法[11]
称取50mg干粉置于1.5mL离心管中,加入750μL 65℃预热的提取缓冲液〔0.1mol/L Tris-HCl(pH8.0)、0.1mol/L EDTA(pH8.0)、0.5mol/L NaCl、2% CTAB、3% PVP、2% DTT〕,65℃水浴2min,不时颠倒混匀;稍冷却后,加等体积的苯酚/氯仿/异戊醇(25︰24︰1),震荡混匀,离心(4℃、12 000rpm、10min);取上清液,加等体积的氯仿/异戊醇(24︰1);震荡混匀后,离心(4℃、12 000rpm、10min);取上清液,重复1次,加等体积的异丙醇(-20℃预冷),轻轻颠倒混匀,-20℃静置30min;4℃、12 000rpm、20min离心,弃上清液;沉淀用70%乙醇洗涤2次,风干后溶于30μL TE,标记后于-20℃保存备用。
1.2.5Ezup柱式试剂盒法
基因组提取试剂盒购于生工生物工程(上海)有限公司,按照说明书进行操作。
1.3DNA检测方法
1.3.1紫外分光光度计法
各取2μL 5种方法提取出来的白草基因组DNA样品稀释至100μL,使用Eppendorf BioPhotometer plus核酸蛋白测定仪测定各样品的A260/A280、A260/A230及各DNA样品中基因组DNA的浓度。通过260nm和280nm、260nm和230nm的紫外吸收值的比值,来判断提取的基因组DNA的纯度。观察并记录各项数据。
1.3.2琼脂糖凝胶电泳
配制0.8%的琼脂糖凝胶,用λDNA作为对照,取4μL DNA原液与1μL的核酸染料混合,点入凝胶点样孔,在TAE缓冲液中以110V的电压电泳50min,并用凝胶成像系统(北京六一仪器)进行拍照并保存。
1.3.3PCR扩增
将上述实验中所提得的总DNA稀释至75ng/μL左右,核糖体基因为引物,配制25μL的反应体系(ddH2O:15.8μL,Buffer:2μL,MgCl2:2μL,dNTP:2μL,ITS2F(ATGCGATACTTGGTGTGAAT):1μL,ITS2R(GACGCTTCTCCAGACTACAAT):1μL,Taq酶:0.2μL,模板DNA:1μL。
PCR反应程序94℃预变性5min,94℃变性0.5min,56℃退火0.5min,72℃延伸45s,共40个循环;最后72℃延伸10min。将PCR扩增产物经1.5%琼脂糖凝胶电泳(110V、50min)分离,在凝胶电泳成像仪上观察,并保存图像。
2 结果与分析
2.1紫外分光光度计检测结果
由表1可知,通过每种方法的3次平行试验,试剂盒法提取的DNA浓度的平均值最低,说明此种方法提取的DNA的量最少;传统CTAB法提取的DNA的A260/280和A260/230值最低,说明DNA中有大量的蛋白质、酚类、小分子物质的污染;其次是改良CTAB法、改良SDS法、高盐低pH法、试剂盒法;高盐低pH法提取的DNA的A260/280平均值接近1.8,说明蛋白质、酚类物质去除干净,A260/230平均值小于2.0,说明有小分子物质的污染;试剂盒法提取的DNA的A260/280平均值大于2.0,说明DNA中有RNA,A260/230的平均值大于2.0,说明小分子物质去除干净。
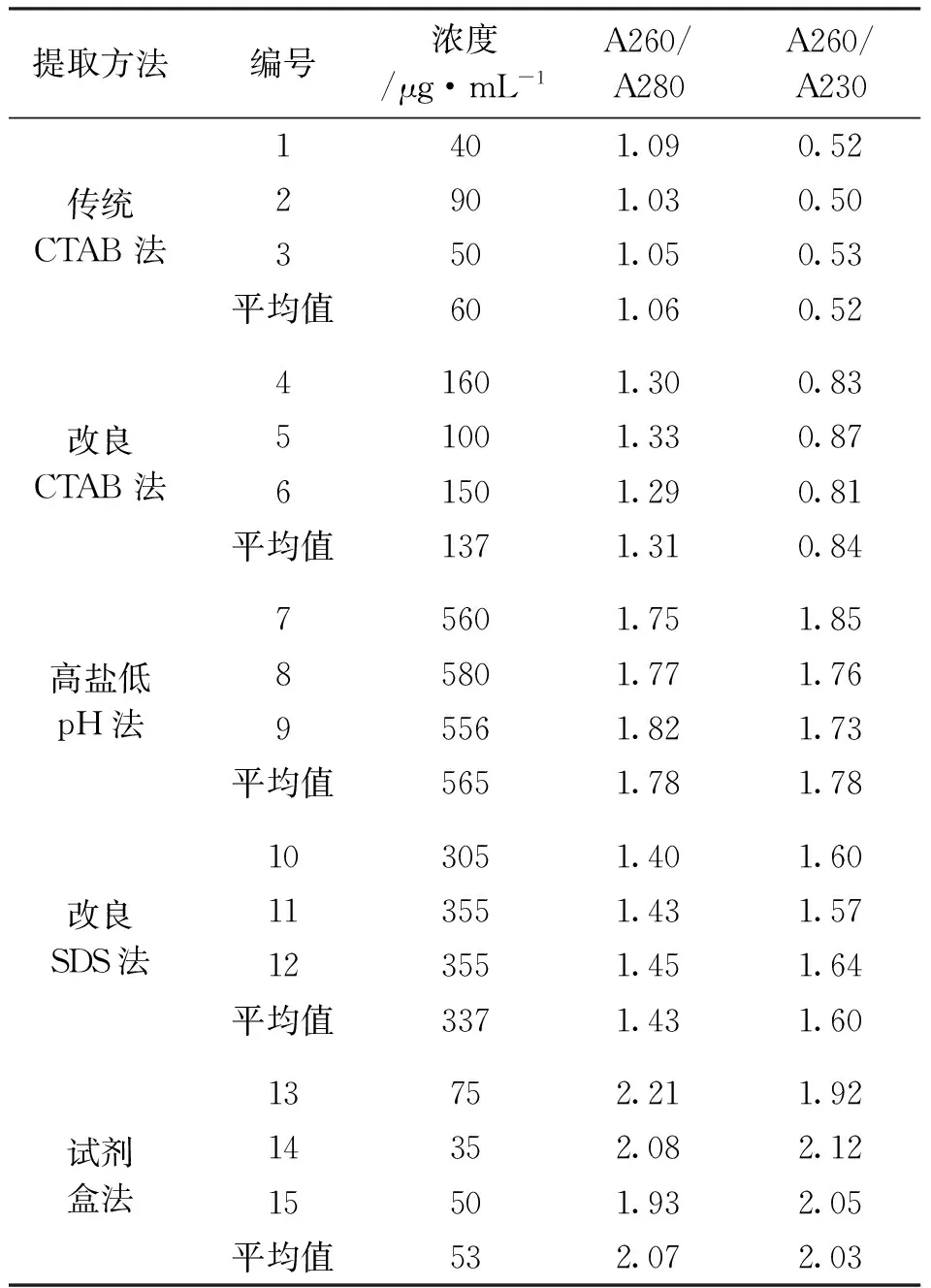
表1 5种方法提取的白草基因组DNA的浓度和纯度
2.2琼脂糖凝胶电泳结果
如图1所示,用传统CTAB法提取的白草种子DNA没有电泳条带,说明这种方法不适合提取白草种子DNA;改良CTAB法和试剂盒法提取的DNA的浓度低,电泳条带颜色浅;改良SDS法和高盐低pH法提取的DNA浓度高,所以电泳条带宽,且条带颜色亮;改良SDS法提取的DNA质含量多,所以点样孔中残留的物质也较多。
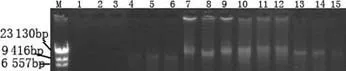
图15种方法提取的白草基因组DNA电泳图谱
注:M为λDNA,1、2、3为传统CTAB法,4、5、6为改良CTAB法,7、8、9为改良SDS法,10、11、12为高盐低pH法,13、14、15为试剂盒法。
Fig.1Electrophoretogram of genomic DNA extracted by 5 methods fromPennisetumflaccidum
2.3PCR扩增结果
以5种方法提取的DNA为模板,核糖体基因为引物进行PCR扩增(图2),除了传统CTAB法提取的DNA没有扩增产物外,其他4种方法都可以得到扩增产物,条带单一明亮,未出现非特异性条带,说明满足PCR扩增的要求。
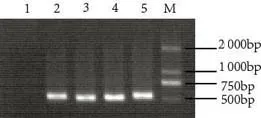
图2 5种方法提取的白草基因组DNA的PCR扩增电泳图谱
3 结论与讨论
在分子生物学实验中,模板DNA既要保证一定的量又要保证一定的纯度,才能保证后续的PCR反应成功[12]。植物细胞不同于动物细胞,不仅具有细胞壁,还含有大量的多糖、脂质、蛋白质、色素、酚类物质等次级代谢产物,给植物基因组DNA提取和纯化带来许多困难。提取植物基因组DNA的常用试剂CTAB(十六烷基三甲基溴化铵)和SDS(十二烷基磺酸钠)都可以破坏细胞膜,以利于细胞内容物的释放。CTAB可以与核酸结合,使核酸与蛋白质分离;SDS可以与多糖、蛋白质结合,通过离心而去除蛋白质[13];PVP能络合多酚类物质,防止其氧化成醌[14]。DNA的纯度根据A260/280和A260/230的值判断,纯度高的DNA,其A260/280≈1.8,A260/230>2.0。若A260/280<1.6,说明DNA中存在酚或蛋白质,若A260/280>1.9,说明DNA中有RNA的污染[15];若A260/230<2.0,说明DNA样品中有小分子物质的污染,如盐类、氨基酸等。
本文中传统CTAB法提取的白草种子DNA的A260/A280的值远低于标准值,DNA中有大量的杂质,琼脂糖凝胶电泳中没有条带及PCR扩增中也没有扩增产物,所以此种方法不适合提取白草种子DNA;改良CTAB法提取的DNA浓度低,且有较多的多糖、蛋白质、小分子物质等的污染,改良SDS法提取的DNA也有大量的杂质污染,但是这2种方法提取的DNA都可以进行PCR扩增。DNA提取过程中应该尽量简化操作步骤,这样可以减少对基因组的损伤和操作过程中污染的可能性[16]。这2种方法的操作步骤较繁琐,用到的有机试剂较多,耗时长,成本高,所以这2种方法也不适合提取白草种子DNA;试剂盒法提取的DNA所含杂质少,耗时最短,但是成本最高,提取的DNA浓度低;高盐低pH法提取的DNA浓度高,A260/A280接近1.8,说明没有蛋白质多糖等的污染,有少量的小分子物质的污染,提取过程相对简单,成本低。所以用高盐低pH法是提取白草种子DNA的最佳方法。
[1]吴征镒.西藏植物志[M].北京:科学出版社,1983:301-303.
[2]任安芝,高玉葆,王金龙.农牧交错区弃耕地生境中白草无性系的形态可塑性研究[J].中国沙漠,2000,20(增刊):33-37.
[3]李海燕,杨允菲.科尔沁草原区火电厂储灰池植被恢复过程中白草种群分蘖株的年龄结构[J].生态学报,2014,34(20):5907-5914.
[][]
[4]任安芝,高玉葆,梁宇,等.白草和赖草无性系生长对干旱胁迫的反应[J].中国沙漠,1999,19(增刊1):30-34.
[5]孔德政,刘艺平,田云芳.改良CTAB法对碗莲叶片基因组DNA提纯效果的影响[J].沈阳农业大学学报,2005,36(4):428-431.
[6]候泽菁,常廼滔.用改良SDS法提取适于PCR扩增的小麦基因组DNA[J].江苏农业科学,2014,42(5):49-51.
[7]许理文,王风格,赵久然.高盐低pH值法提取玉米基因组DNA的研究[J].玉米科学,2009,17(1):59-61,70.
[8]雷开荣,石春焱,李明顺.一种玉米叶片基因组DNA快速提取新方法的初步研究[J].华北农学报,2006,21(2):10-12.
[9]曹文波,郑璐璐,谢文海.一种提取植物基因组DNA的方法-改良尿素法[J].华中师范大学学报,2008,42(3):448-451.
[10]汤洁,刘连生,许娜娜,等.绞股蓝叶片DNA提取方法的比较研究[J].安徽农业大学学报,2008,35(3):445-448.
[11]张勇群,石梦菲,德吉,等.菊叶香黎(Chenopodiumfoetidum)基因组DNA的提取方法研究[J].西藏大学学报,2014,29(1):13-15,70.
[12]翟永功,马建岗,赵文明.12种作物种子DNA的提取与纯化[J].西北大学学报,1998,28(1):64-67.
[13]赵姝华,王富德,张世苹,等.提取、纯化植物DNA方法的比较[J].国外农学-杂粮作物,1998,18(2):35-38.
[14]邓力超,邱道寿,屠乃美,等.PVP对烟草基因组DNA提取的影响[J].广东农业科学,2009(5):37-40.
[15]王钰婷,汪泰初,李瑞雪,等.3种桑蟥基因组DNA提取方法比较研究[J].农学学报,2014,4(12):95-98.
[16]戴军,汪海,朱莉,等.1种适用于转基因玉米PCR检测的DNA快速提取方法[J].安徽农业科学,2014,42(12):3494-3496.
Comparison of 5 DNA Extraction Methods from Pennisetum flaccidum Seed
HAO Dou-dou1,TIAN Zhi-hua1,LEI Ming1,WU Jun-xi2,LA duo1,ZANG Yong-qun3
(1.Tibet University,Lhasa Xizang 850000,P.R.China;2.Institute of Geographical Sciences,Lhasa Xizang 850000,P.R.China;3.Hospital of Tibetan Autonomous Region Government Chengdu Office,Chengdu Sichuan 610000,P.R.China)
The objective of this study was to explore a suitable method for genomic DNA extraction fromPennisetumflaccidumseed by comparing five DNA extraction methods ( traditional CTAB,improved CTAB,improved SDS,high salt and low pH,kit method).The quality of extracted DNA was examined with ultraviolet spectrophotometry,agarose gel electrophoresis and PCR amplification,respectively.The results showed that the DNA extracted by traditional CTAB contained many PCR inhibitors;the purity of DNA extracted by improved CTAB and SDS did not meet the demand of quality standard of DNA in molecular experiment.High salt and low pH and kit method could extract high-quality and good integrity DNA,but kit method cost too much and the yield of DNA was low.Therefore,high salt and low pH was the most suitable and low cost method of these 5 methods.
Pennisetumflaccidum;genomic DNA;DNA extraction methods;high salt and low pH method
10.16473/j.cnki.xblykx1972.2016.03.014
2015-08-07
湖南师范大学蛋白质与发育生物学教育部重点实验室(2014年)开放课题(2015DF03),西藏自治区科技厅自然科学基金项目(2015ZR-13-5)。
郝豆豆(1992-),女,硕士研究生,主要从事植物学研究。E-mail:31724478@qq.com
简介:张勇群(1976-),女,博士,副教授,硕士生导师,主要从事藏药材基因与功能研究。E-mail:yongqunzhang@yahoo.com
Q 943.2
A
1672-8246(2016)03-0081-05
