软体动物牡蛎肌联蛋白一级结构的预测
2016-10-24包玉龙张三润徐宋瑶周好乐
包玉龙,张三润,徐宋瑶,周好乐
(内蒙古医科大学基础医学院,内蒙古 呼和浩特 010110)
软体动物牡蛎肌联蛋白一级结构的预测
包玉龙,张三润,徐宋瑶,周好乐
(内蒙古医科大学基础医学院,内蒙古 呼和浩特 010110)
旨在预测牡蛎肌联蛋白的基因exon-intron结构,并确定其一级结构及结构域。利用生物信息学方法,从牡蛎基因组数据中预测肌联蛋白基因的exon-intron结构,并进一步预测编码肌联蛋白的mRNA组成,从而确定该蛋白的一级结构,并检测其结构域;与同源蛋白进行序列比对,制作系统进化树以了解其在系统进化中的位置。结果表明,该蛋白基因在基因组中横跨200 kb的序列区域,预测编码肌联蛋白的mRNA大约由74 kb组成,推测相对分子质量约为2 700 ku;该蛋白由Ig、Fn、kinase结构域,PEVK结构域和unique序列组成;在系统发育方面,该蛋白归属于原口动物集团。牡蛎肌联蛋白是一种新型的肌联蛋白样蛋白。
生物信息学;基因组;肌联蛋白;同源蛋白
肌联蛋白是相对分子质量约为300万ku的巨大弹性蛋白质[1],由1个分子连接肌小节的Z线到M线[2-3]。肌联蛋白的弹性与肌小节的结构维持和被动张力有关[4],该种弹性性质与存在于肌小节I带区域的免疫球蛋白(immunoglobulin,Ig)结构域、PEVK 区域、unique 序列有关[2,5-7]。 在无脊椎动物横纹肌中,从其一级结构和抗体交叉反应方面来看,存在很多类似肌联蛋白的肌联蛋白样蛋白(connectin-like protein),如线虫 TTN-1、小龙虾凸出蛋白、昆虫凸出蛋白等[8-12]。
在软体动物平滑肌中,当细肌丝和粗肌丝相互作用产生收缩后,存在一种叫做捕捉(catch)的结构,它能利用少量能量维持长时间的张力。该种状态与一种相对分子质量为53万ku的肌联蛋白样蛋白——颤搐蛋白(twitchin)有关。贻贝颤搐蛋白的一级结构已被确定,它是由24个Ig结构域和15个纤连蛋白 3 型(fibronectin type 3,Fn3)结构域组成,在C末端附近存在1个激酶结构域;在肌小节中定位于粗肌丝,与肌球蛋白等粗肌丝组成蛋白相结合[13-14]。
到目前为止,软体动物的肌联蛋白样蛋白,除了颤搐蛋白以外尚未有报道。关于软体动物横纹肌收缩及舒展时肌小节结构维持的机制尚不明确。在对牡蛎平滑肌全蛋白提取液进行SDS-PAGE检测时发现,在相对分子质量为3 500 ku(肌联蛋白)的位置出现条带,该蛋白可能是存在于软体动物中的新型肌联蛋白样蛋白,对软体动物横纹肌的结构维持与被动张力的产生起到决定性作用。笔者通过生物信息学的方法预测牡蛎高分子肌联蛋白的一级结构并检测其结构域,同时利用激酶结构域氨基酸序列制作系统发育树,以了解其在系统发育中的地位。
1 材料与方法
1.1 SDS-PAGE 试验 根据 Laemmli[15]报道的方法进行SDS-PAGE试验,使用无浓缩胶的2%~6%的梯度凝胶,凝胶染色使用考马斯亮蓝染色。
1.2 肌联蛋白基因序列检索 对牡蛎肌联蛋白进行检索时,使用人肌联蛋白[7]的N末端2个Ig结构域和C末端激酶结构域氨基酸序列,在OysterBase (http://www.oysterdb.com/FrontHomeAction.do?method=home)主页上公开的牡蛎基因组数据库 oyster_genome_v1[16]中进行 TBLASTN 检索,查找肌联蛋白的类似序列。检索得到牡蛎基因组序列片段「scaffold556」的碱基序列有较高同源性,将其用于详细的检索。
1.3 蛋白结构域(domain)和肌联蛋白基因exonintron结构的预测 使用GENETYX version 4软件将得到的基因组序列「scaffold556」的480 kb碱基序列打开,并将该序列翻译成3行氨基酸序列。将得到的氨基酸序列保存为Microsoft Word文档。然后,使用 SMART(http://smart.embl-heidelberg.de/)程序进行蛋白的结构域预测[8]。按照结构域序列间的内含子以碱基gt开始、以碱基ag终止的原则对牡蛎肌联蛋白基因的exon-intron结构进行预测。将这些结构域合并后,得到肌联蛋白的结构域结构[2]。
1.4 同源性分析 使用DDBJ(DNA Data Bank of Japan)在主页上提供的CLUSTALW(http://clustalw.ddbj.nig.ac.jp/top-j.html)程序及GENETYX软件进行同源性分析[7,17]。
1.5 系统发育树分析 使用不同物种的肌联蛋白样蛋白的激酶结构域氨基酸序列,在由DDBJ(DNA Data Bank of Japan)主页提供的CLUSTALW(http://clustalw.ddbj.nig.ac.jp/top-j.html)程序中进行同源性分析,并得到系统进化树文件。使用Tree view[18]软件对进化树进行分析,了解同源蛋白间的系统发育关系。
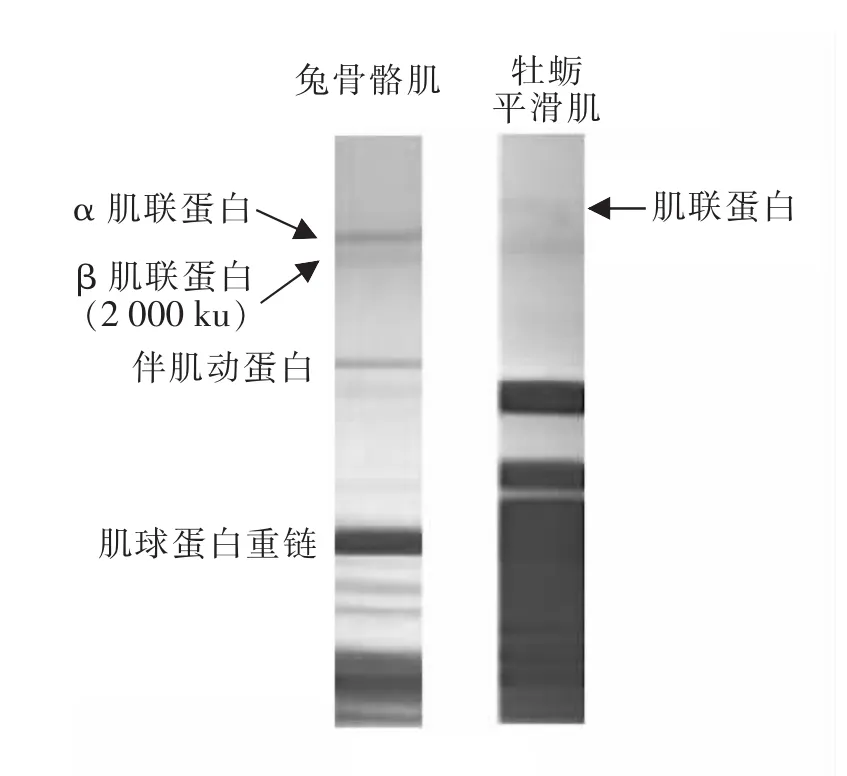
图1 牡蛎平滑肌全蛋白提取物的SDS-PAGE结果
2 结果与分析
2.1 SDS-PAGE试验 利用2%~6%的梯度凝胶对牡蛎平滑肌全蛋白提取物进行电泳分离。结果表明,在相对分子质量为3 500 ku左右的位置上出现了条带(见图1)。将相对分子质量约为3 500 ku的蛋白视为牡蛎肌联蛋白。
2.2 肌联蛋白基因的exon-intron结构 在基因组数据库中进行BLAST和SMART检索,得到的肌联蛋白基因的exon-intron结构见图2。由图2可知,肌联蛋白基因在基因组数据库中横跨200 kb的序列区间。该基因所有外显子长度之和约为74 kb,推测相对分子质量约为2 700 ku。
2.3 肌联蛋白的结构域结构 将得到的exon-intron结构中的所有外显子合并后,获得了牡蛎肌联蛋白的结构域结构(见图3)。由图3可知,牡蛎肌联蛋白由62个Ig结构域、48个Fn结构域、7个Spectrin结构域、5个 PEVK结构域和1个kinase结构域组成。
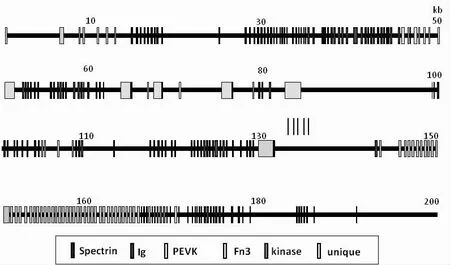
图2 牡蛎肌联蛋白基因的exon-intron结构
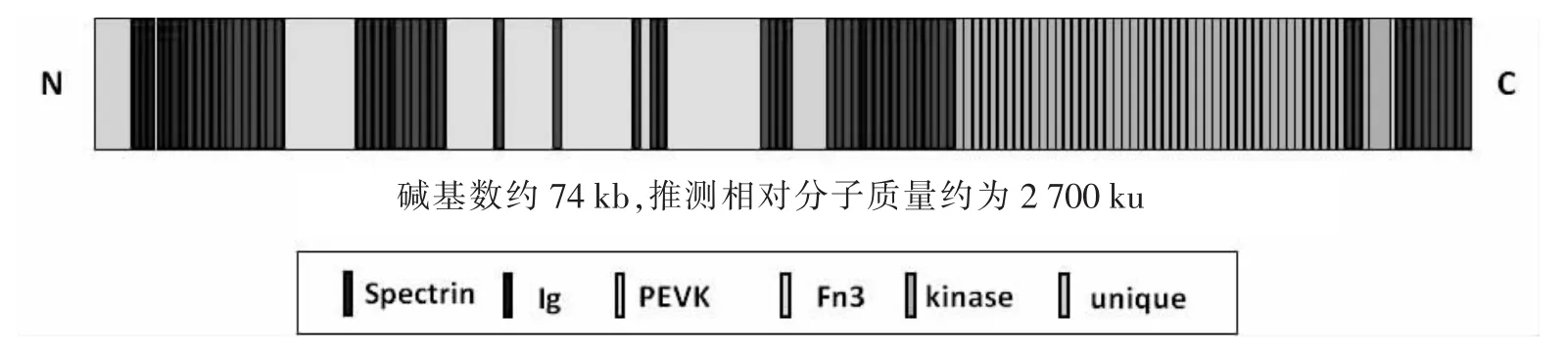
图3 牡蛎肌联蛋白的结构域结构

表1 牡蛎肌联蛋白激酶结构域与脊椎动物和无脊椎动物肌联蛋白激酶结构域的同源性

图4 各类动物肌联蛋白样蛋白激酶结构域系统发育分析
2.4 同源性及系统发育分析 所预测的结构域中在C末端存在1个激酶结构域,其氨基酸数目为255个,与其他肌联蛋白样蛋白的激酶结构域氨基酸数目保持一致。在氨基酸水平上计算与不同物种激酶结构域的同源性,结果表明,牡蛎肌联蛋白激酶结构域与脊椎动物肌联蛋白激酶结构域同源性约为40%,与无脊椎动物的同源性约为50%(见表1)。利用激酶结构域的氨基酸序列进行分组系统发育分析,结果表明,牡蛎肌联蛋白归属于原口动物集团(见图 4)。
3 讨论
该研究中得到的编码牡蛎肌联蛋白的mRNA长度约为74 kb,推测相对分子质量约为2 700 ku。牡蛎平滑肌SDS-PAGE结果表明,在相对分子质量约为3 500 ku左右的位置出现了条带,这与推测的相对分子质量间存在差异,这可能与序列中存在的5个PEVK结构域有关。此外,由于该研究只是通过基因组数据对其进行预测分析,因此,一些外显子可能未被预测出来,需要今后进一步利用PCR试验来验证。
从该基因的结构域结构来看,N末端存在连续的Ig结构域和PEVK结构域,C末端具有激酶结构域,这一特点与脊椎动物肌联蛋白的结构域结构非常相似。但该结构域结构中C末端存在47个连续的Fn结构域,这在以往已知的肌联蛋白样蛋白中从未发现。该蛋白结构域结构中不存在人肌联蛋白所具有的由Ig和Fn组成的超级重复序列,另外,由Ig和PEVK结构域构成的区域较长,占整体的一半以上。这些特点表明,牡蛎肌联蛋白是一种与之前发现的肌联蛋白样蛋白的结构域结构不同的新型肌联蛋白样蛋白。
[1]MARUYAMA K.Connectin,an elastic filamentous protein of striated muscle[J].Int Rev Cytol,1986,104:81-114.
[2]MARUYAMA K.Connectin,an elastic protein of striated muscle[J].Biophys Chem,1994,50(1/2):73-85.
[3]KÖTTER S,ANDRESEN C,KRÜGER M.Titin:central player of hypertrophic signaling and sarcomeric protein quality control[J].Biol Chem,2014,395(11):1341-1352.
[4]NEIVA-SOUSA M,ALMEIDA-COELHO J,FALCÃOPIRES I,et al.Titin mutations:the fall of Goliath[J].Heart Fail Rev,2015,20(5):579-588.
[5]LINKE W A,HAMDANI N.Gigantic business:titin properties and function through thick and thin [J].Circ Res,2014,114(6):1052-1068.
[6]GRITSYNA IUV,SALMOV N N,VIKHLIANTSEV I M,et al.Changes in gene expression and content of titin(connectin)in striated muscles of chronically etihanol-fed rats[J].Mol Biol(Mosk),2013,47(6):996-1003.
[7]OHTSUKA S,HANASHIMA A,KUBOKAWA K,et al.Amphioxus connectin exhibits merged structure as invertebrate connectin in I-band region and vertebrate connectin in A-band region[J].J Mol Biol,2011,409(3):415-426.
[8]FORBES J G,FLAHERTY D B,MA K,et al.Extensive and modular intrinsically disordered segments in C.elegans TTN-1 and implications in filament binding,elasticity and oblique striation[J].J Mol Biol,2010,398(5):672-689.
[9]YUAN C C,MA W,SCHEMMEL P,et al.Elastic proteins in the flight muscle of Manduca sexta [J].Arch Biochem Biophys,2015,568:16-27.
[10]AYME-SOUTHGATE A J,TURNER L,SOUTHGATE R J.Molecular analysis of the muscle protein projectin in Lepidoptera[J].J Insect Sci,2013(13):88.
[11]AYME-SOUTHGATE A,PHILIPP R A,SOUTHGATE R J.Projectin PEVK domain,splicing variants and domain structure in basal and derived insects[J].Insect Mol Biol,2011,20(3):347-356.
[12]HU D H,MATSUNO A,TERAKADO K,et al.Projectin is an invertebrate connectin (titin):isolation from crayfish claw muscle and localization in crayfish claw muscle and insect flight muscle [J].J Muscle Res Cell Motil,1990,11(6):497-511.
[13]FUNABARA D,WATANABE D,SATOH N,etal.Genome-wide survey of genes encoding muscle proteins in the pearl oyster,Pinctada fucata [J].Zoolog Sci,2013,30(10):817-825.
[14]BUTLER T M,SIEGMAN M J.A force-activated kinase in a catch smooth muscle [J].J Muscle Res Cell Motil,2011,31(5/6):349-358.
[15]LAEMMLI U K.Cleavage of structural proteins during the assembly of the head of bacteriophage T4 [J].Nature,1970,227(5259):680-685.
[16]ZHANG G,FANG X,GUO X,et al.The oyster genome reveals stress adaptation and complexity of shell formation[J].Nature,2012,490(7418):49-54.
[17]HANASHIMA A,OGASAWARA M,NOMIYA Y,et al.Genomic-and protein-based approaches for connectin(titin)identification in the ascidian Ciona intestinalis[J].Methods,2012,56(1):18-24.
[18]PAGE R D.TreeView:an application to display phylogenetic trees on personal computers [J].Comput Appl Biosci,1996,12(4):357-358.
Prediction of Primary Structure of Oyster Connectin
BAO Yu-long,ZHANG San-run,XU Song-yao,ZHOU Hao-le
(Basic Medical College,Inner Mongolia Medical University,Hohhot 010110,China)
The aim of the present study was to predict the exon-intron structure of gene coding connectin in oyster and to determine its primary structure and domains.The exon-intron structure of gene coding connectin was deduced based on oyster genomic data by using bioinformatics method;the mRNA composition of coding regions for connectin in oyster was predicted;the primary structure of oyster connectin was determined and its domains were detected;phylogenetic tree was constructed by comparing amino acid sequences of homologous protein associated with connectin from different species.The results showed that the gene coding connectin in oyster genome covered 200 kb,the connectin-coding mRNA was about 74 kb,and the relative molecular weight of oyster connectin was deduced as 2 700 ku;oyster connectin was composed of Ig,Fn,and kinase domain,PEVK domain and unique sequences;the oyster connectin phylogenetically belonged to protostome group.Oyster connectin is a new type of connectin-like protein.
bioinformatics;genome;connectin;homologous protein
Q516
A文章顺序编号1672-5190(2016)12-0005-04
2016-11-15
项目来源:内蒙古自然科学基金项目(2014MS08106)。
包玉龙(1976—),男,讲师,博士,主要从事分子细胞生物学研究工作。
周好乐(1962—),女,教授,主要从事动物遗传学研究工作。
(责任编辑:赵俊利)
