中哈边境阿拉山口口岸输入性猫栉首蚤指名亚种线粒体COI和COII基因序列分析
2016-10-24王安东尹小平田延河张江国
罗 丹,王安东,尹小平,田延河,梁 臻,巴 特,张江国
中哈边境阿拉山口口岸输入性猫栉首蚤指名亚种线粒体COI和COII基因序列分析
罗丹1,王安东2,3,尹小平3,田延河3,梁臻3,巴特3,张江国3
目的分析阿拉山口口岸地区输入性猫栉首蚤指名亚种细胞色素C氧化酶I(COI)和II(COII)基因特征和系统进化关系。方法从2014年1月入境集装箱死猫体表采集蚤类样本,形态学鉴定完毕后提取DNA,PCR 扩增COI和COII基因并测定序列,使用Mega 6.0通过ML法构建系统发育树。结果猫栉首蚤指名亚种COI和COII基因富含A+T,碱基突变多为置换突变,无移码,缺失和插入突变;Blast显示与澳大利亚猫栉首蚤指名亚种同源性较高(99%)。结论COI基因序列存在足够的变异能够区分亲缘关系很近的种类,为外来或新发现的蚤种的鉴别提供了分子水平的技术依据。
猫栉首蚤指名亚种;细胞色素 C 氧化酶亚基 I(COI);细胞色素 C 氧化酶亚基 II(COII); 聚合酶链式反应;序列分析;中哈边境
阿拉山口口岸是中国与哈萨克斯坦的边境国家一类口岸,包括铁路口岸,公路口岸,此外还有原油管道运输,是第二座亚欧大陆桥中国段西部的桥头堡。阿拉山口口岸2013年全年过货量达2 982.9万吨[1]。近年来我国口岸在入境国际交通工具、集装箱和货物中发现并捕获大量输入性医学媒介生物[2]。2014年,我们从哈萨克斯坦入境集装箱一死猫体表采集到蚤类样本,经形态学鉴定为猫栉首蚤指名亚种。本研究使用线粒体COI和COII基因对输入性猫栉首蚤指名亚种进行基因序列分析,分析系统进化关系,为口岸地区建立外来或新发现蚤种分子鉴定方法奠定基础。
1 材料与方法
1.1样本采集2014年从一入境集装箱死猫体表采集蚤类样本,置于95%无水乙醇中,以备后续研究。
1.2形态学鉴定依据陆宝麟蚤的鉴别方法[3],利用体式显微镜对采集的蚤类样本进行形态学鉴定。
1.3分子生物学鉴定
1.3.1DNA提取按照DNeasy○RBlood & Tissue kit(QIAGEN)说明书提取基因组DNA(5只/1组),产物于-20 ℃保存备用。
1.3.2线粒体COI和COII基因克隆PCR扩增引物、反应体系及条件参考A. L. Lawrence的报道,引物合成及PCR产物测序由生工生物工程(上海)股份有限公司完成。
1.3.3序列分析与系统树构建PCR产物送生工生物工程(上海)股份有限公司进行测序,测序结果登录GenBank进行Blast(http://blast.ncbi.nlm.nih.gov)分析比对。 通过Mega 6.0 软件进行遗传进化树和遗传距离分析。
表1PCR反应引物和扩增条件
Tab.1Primers of COI and COII sequences and reaction condition of PCR

目的基因(Gene)引物名称(Premer)序列(5'-3')(Sequence)片段大小(bp)(Fragment)预变性(℃,S)(InitialDenaturation)变性(℃,S)(Denaturation)退火(℃,S)(Anneaking)延伸(℃,S)(Extension)循环数(Cycle)总延伸(℃,S)(FinalExtension)参考文献(Reference)COILCO1490GGTCAACAAATCATA-AAGATATTGG60195,6095,1555,1572,103572,300[4]HC02198TAAACTTCAGGGT-GACCAAAAAATCACOIIF-LeuTCTAATATG-GCAGATTAGTGC72795,18094,3042,3072,153772,300R-LysGAGACCAGTACTT-GCTTTCAGTCATC
2 结 果
2.1蚤采集与鉴定结果现场在猫体表采集蚤类标本75只,随机选取其中40只进行形态学鉴定。经体式显微镜观察,所选取标本均为猫栉首蚤指名亚种。
2.2线粒体COI和COII基因扩增结果经过PCR扩增猫栉首蚤指名亚种线粒体COI和COII基因,产物经1.5%琼脂糖凝胶电泳分析,可得到601 bp和727 bp左右的核酸片段,与预期片段大小相符。
2.3线粒体COI和COII基因组成分析该猫栉首蚤指名亚种COⅠ基因片段 A、C、G、T的平均碱基含量分别为31.0%,16.2%,14.9%,37.8%,碱基 A+T含量是68.8%,有4个碱基置换突变位点(3个T-C转换,1个A-G转换),无移码,缺失和插入突变;COII基因片段 A、C、G、T的平均碱基含量分别为35.8%; 12.1%; 9.5%; 42.6%,碱基 A+T含量是78.4%,有2个碱基置换突变(1个t-a颠换,1个转换g-a),无移码,缺失和插入突变。
2.4Blast分析猫栉首蚤指名亚种COI基因与Ctenocephalidesfelis(JN008917,澳大利亚)具有99% (621/629)的同源性,与Ctenocephalidesfelisfelis(KP684201,捷克)具有100% (592/592)的同源性;与Ctenocephalidesfelisfelis(KP687808,印度)具有99% (582/587)的同源性;与Ctenocephalidesfelisstrongylus(KF684875,塞舌尔)具有98% (582/592)的同源性,与Ctenocephalidescanis(KP684211,捷克)具有92% (596/647)的同源性,与Ctenocephalidesorientis(KF684867,泰国)具有92% (546/591)的同源性。COII基因显示与Ctenocephalidesfelisstrongylus(KF684933,塞舌尔)具有99% (704/709)的同源性,与Ctenocephalidesorientis(KF684922,泰国)具有99% (704/709)的同源性;与Ctenocephalidesfelisfelis(KT376424,澳大利亚)具有99% (703/709)的同源性;与Ctenocephalidesfelisfelis(HQ696926,澳大利亚)具有99% (703/709)的同源性,与Ctenocephalidesfelis(JF966771,塞内加尔)具有99% (704/710)的同源性。
2.5系统进化树分析分子遗传进化树显示:该猫栉首蚤指名亚种COI基因序列与澳大利亚、捷克、塞舌尔三国猫栉首蚤指名亚种遗传距离在0.0052-0.0141之间,与Ctenocephalidesfelis(JN008917,澳大利亚)形成独立一支,与Ctenocephalidesfelisfelis(KP684201,捷克)形成姐妹支;其COII基因序列与澳大利亚、塞内加尔2国猫栉首蚤指名亚种遗传距离在0.0029-0.0057之间,其单独形成一支,与Ctenocephalidesfelisfelis(KT376424,澳大利亚)、Ctenocephalidesfelisstrongylus(KF684933,塞舌尔)和Ctenocephalidesfelisfelis(HQ696926,澳大利亚)形成姐妹支。
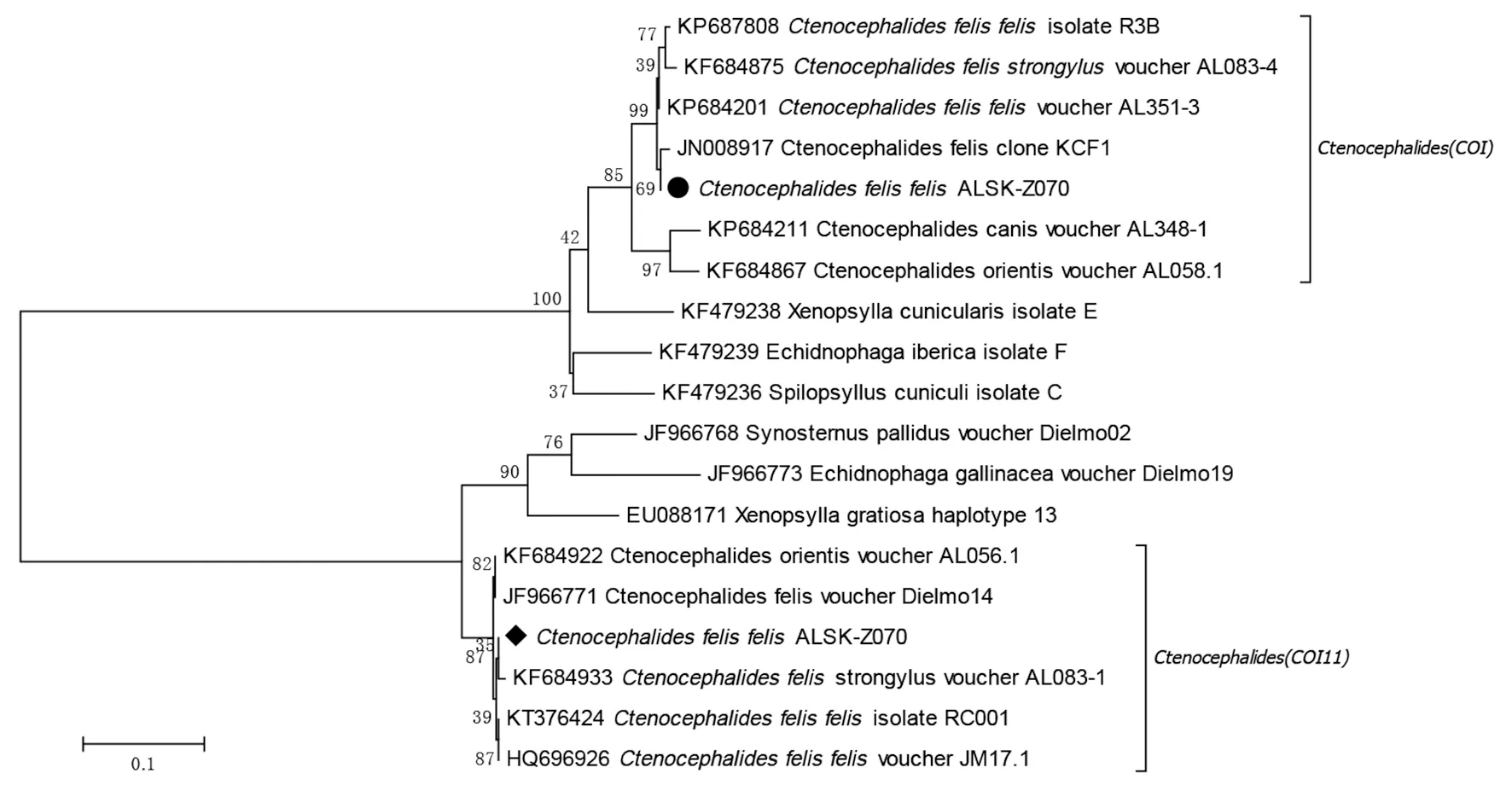
图1 基于COI基因进行系统进化树分析Fig.1 Phylogenetic tree of COI gene
表2猫栉首蚤指名亚种COI基因序列间遗传距离
Tab.2Estimates of evolutionary divergence between sequences of COI form Ctenocephalides felis felis
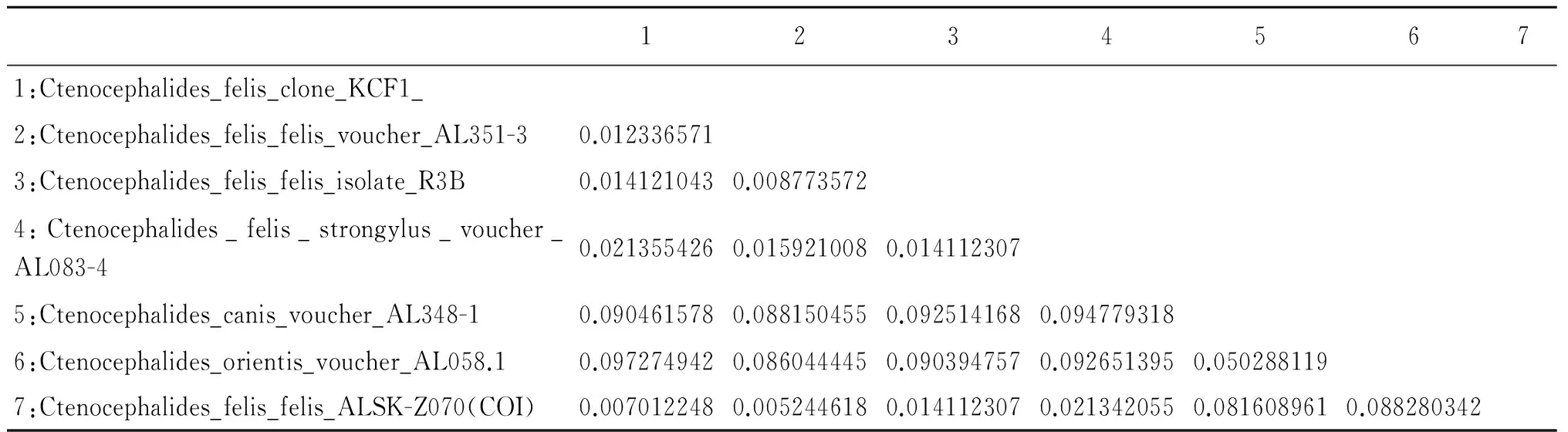
12345671:Ctenocephalides_felis_clone_KCF1_2:Ctenocephalides_felis_felis_voucher_AL351-30.0123365713:Ctenocephalides_felis_felis_isolate_R3B0.0141210430.0087735724:Ctenocephalides_felis_strongylus_voucher_AL083-40.0213554260.0159210080.0141123075:Ctenocephalides_canis_voucher_AL348-10.0904615780.0881504550.0925141680.0947793186:Ctenocephalides_orientis_voucher_AL058.10.0972749420.0860444450.0903947570.0926513950.0502881197:Ctenocephalides_felis_felis_ALSK-Z070(COI)0.0070122480.0052446180.0141123070.0213420550.0816089610.088280342
表3猫栉首蚤指名亚种COII基因序列间遗传距离
Tab.3Estimates of evolutionary divergence between sequences of COII Ctenocephalides felis felis
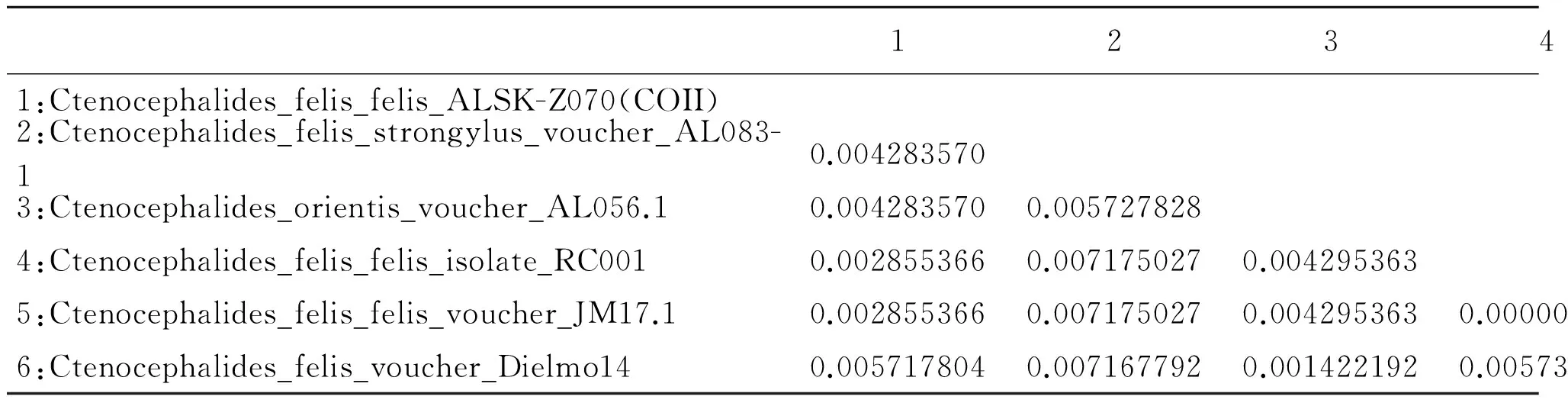
1234561:Ctenocephalides_felis_felis_ALSK-Z070(COII)2:Ctenocephalides_felis_strongylus_voucher_AL083-10.0042835703:Ctenocephalides_orientis_voucher_AL056.10.0042835700.0057278284:Ctenocephalides_felis_felis_isolate_RC0010.0028553660.0071750270.0042953635:Ctenocephalides_felis_felis_voucher_JM17.10.0028553660.0071750270.0042953630.0000000006:Ctenocephalides_felis_voucher_Dielmo140.0057178040.0071677920.0014221920.0057335900.005733590
3 讨 论
线粒体DNA是1个封闭的环状双链,它是由13个编码蛋白质的基因、2个rRNA基因(16S rRNA和12S rRNA)、22个tRNA基因和1个包含复制起点的控制区组成的[5-6]。与脊椎动物mtDNA相比,昆虫mtDNA富含更多A+T,C+G含量偏低[7]。本研究发现:COI基因碱基A+T含量是68.8%;COII基因碱基A+T含量则高达78.4%。蚊虫COI和COII基因序列间颠换频率高于转换频率[8-9],此次研究发现猫栉首蚤指名亚种COI基因碱基突变均为转换,而COII基因颠换与转换频相同,有别于蚊虫序列间碱基突变频率。
COI基因由于其氨基酸的演化速率较慢可以区分时间较远的种类,同属物种的COI基因序列又存在有足够的变异能够区分亲缘关系很近的种类,所以它被广泛应用于不同分类阶元层次上的分子系统学研究[10]。COII基因序列具有很高的趋异性,尤其是一些双翅目和膜翅目昆虫,COII基因序列和氨基酸序列均表现出高度的趋异性,众多研究表明COII基因在双翅目昆虫进化机制和系统重建研究上是一种有效的分子标记[9]。目前运用线粒体COI和COII基因研究昆虫系统进化的研究越来越多。张映梅等对我国44种重要蚤类进行DNA条形码初步研究,结果表明,同种个体之间的同源性达到99%以上,不同种之间的同源性最高仅为90%,且几乎所有蚤类都能被DNA条形码正确鉴定(种间序列差异大于2%),可以进行准确快速鉴定[11]。此次研究发现:猫栉首蚤指名亚种COI基因序列与Ctenocephalides felis strongylus(KF684875,塞舌尔)具有98%(582/592)的同源性,与Ctenocephalides orientis(KF684867,泰国)具有92%(546/591)的同源性,而COII基因显示与Ctenocephalidesfelis strongylus(KF684933,塞舌尔)具有99%(704/709)的同源性,与Ctenocephalides orientis(KF684922,泰国)具有99%(704/709)的同源性。由此可见,COI基因序列存在足够的变异能够区分亲缘关系很近的种类。
随着经济全球化的发展,世界各地的贸易越来越频繁,增加了媒介生物入侵的风险。此次发现该从哈萨克斯坦输入性猫栉首蚤指名亚种COI和COII基因序列均与澳大利亚猫栉首蚤指名亚种同源性较高(99%),由此我们推测,该输入性猫栉首蚤指名亚种与国际贸易往来有密切关系,进一步佐证媒介生物随交通工具入侵的风险。因此在国境口岸地区建立媒介生物的快速检测方法显得尤为重要,本研究为口岸快速准确地识别宿主和外来医学媒介生物的监控提供科学依据。
[1] Xu J,Mo SM,Zhang JG. Animal and plant species resources and quarantine at Alataw Pass[M]. Beijing: China Quality Control Publishing House,2015: 6-9.(in Chinese)
徐军,莫善明,张江国.阿拉山口口岸动植物物种资源与检疫[M].北京:中国质检出版社,2015:6-9.
[2] Li JC,Li DX,Nie WZ,et al. Construction of control systems on introduced medical vectors at frontier ports of China[J].J Inspect Quarant,2012(2): 1-5.(in Chinese)
李俊成,李德昕,聂维忠,等.中国口岸输入性医学媒介生物防控体系的建立[J].检验检疫学刊,2012(2):1-5.
[3] Lu BL. Classification and identification of important medical insects in China[M]. Zhengzhou: Henan Science and Technology Press,2003.(in Chinese)
陆宝麟.中国重要医学昆虫分类与鉴别[M].郑州:河南科学技术出版社,2003.
[4] Lawrence AL,Brown GK,Peters B,et al. High phylogenetic diversity of the cat flea (Ctenocephalides felis)at two mitochondrial DNA markers[J]. Med Vet Entomol,2014,28(3): 330-336.DOI:10.1111/mve.12051
[5] Clary DO,Wolstenholme DR. The mitochondrial DNA molecule of Drosophilayakuba: nucleotide sequence,geneorganization and geneticcode[J]. J Mol Evol,1985,22: 252-271.DOI: 10.1007/BF02099755
[6] Fauron CMR,Wolstenholme DR. Intraspectific diversity of nucleotide sequences within the adenine+thymine-richregion of mitochondrial DNA molecules of Drosophilasimulans[J]. Nucleic Acids Res,1980,8: 5391-5410.DOI:10.1093/nar/8.22.539
[7] Navajas M,Fournier D,Lagnel J,et al. Mitochondrial COI sequences in mites: evidence for variations in base composition[J]. Insect Mol Biol,1996,5(4): 281-285.DOI:10.1111/j.1365-2583.1996.tb00102.x
[8] Shi YX,Xiang DP,Li ZH,et al. COI Sequences analysis and molecular identification for five species of mosquitoes from Guangdong Port[J]. Chin Front Health Quarant,2008,31(2): 103-107. (in Chinese)
师永霞,相大鹏,李祖海,等.广东国境口岸不同蚊种COI序列分析和分子鉴定方法[J].中国国境卫生检疫杂志,2008,31(2):103-107.
[9] Huang ZH,Wang JF. Cloning and sequencing of Cytochrome cOxidase II(COII) gene of three species of mosquitoes[J]. Chin J Parasit Dis,2001,19(2): 90-92. (in Chinese)
黄朝晖,王金福.三种蚊虫COII基因的克隆与序列分析[J].中国寄生虫学与寄生虫病杂志,2001,19(2):90-92.
[10] Fu J,Zhang YC. Sequence analysis of mtDNA-COI gene and molecular phylogeny on twenty-seven species ofCoccinellids(Coleoptera: Coccinellidae)[J]. Entomotaxonomia,2006,28: 179-186.(in Chinese)
付景,张迎春.二十七种瓢虫mtDNA-COI基因序列分析及系统发育研究(鞘翅目:瓢虫科)[J].昆虫分类学报,2006,28:179-186.
[11] Wu W. Application of DNA barcoding technique in medical vector identification[J]. Anim Husb Feed Sci,2012,33(1): 22-23.(in Chinese)
吴薇. DNA条码技术在医学媒介生物鉴定中的应用前景展望[J]. 畜牧与饲料科学,2012,33(1):22-23.
COI and COII sequences analysis for an importedSiphonapterasample ofCtenocephalidesfelisfelisat Alataw Pass,China-Kazakhstan
LUO Dan1,WANG An-dong2,3,YIN Xiao-ping3,TIAN Yan-he3,LIANG Zhen3,BA Te3,ZHANG Jiang-guo3
(1.CollegeofMedicine,ShiheziUniversity,Shihezi832000,China;2.CollegeofAnimalScienceandTechnology,ShiheziUniversity,Shihezi832000,China;3.AlashankouEntry-ExitInspectionandQuarantineAuthority,Alashankou833418,China)
To analyze the molecular of cytochrome c oxidase subunit I (COI) and subunit II (COII) of mitochondrial DNA and the phylogenetic relationship ofCtenocephalidesfelisfelisat Alataw Pass,China-Kazakhstan,blood feedingSiphonapterawere collected from cat and DNA was extracted after morphological Identification. The COI and COII gene sequence were amplified. The phylogenetic trees was constructed by Mega 6.0 based on the nucleotide sequences ofSiphonapterapublished in the GenBank and the homology was analyzed. Result showed that the A+T in the amplified COI and COII fragments ofCtenocephalidesfelisfeliswere rich.Ctenocephalidesfelisfeliswere 99% closely related toCtenocephalidesfelisfelisin Australia. It’s suggested that COI nucleotide identification could be used to classify species ofSiphonapteraand provide molecular basis for identifying the foreignSiphonapteraspecies at Alataw Pass or other frontier ports.
Ctenocephalidesfelisfelis; COI; COII; PCR; sequence analysis; China-Kazakhstan
Yin Xiao-ping,Email: yxpciq@163.com
10.3969/j.issn.1002-2694.2016.09.005
尹小平,Email:yxpciq@163.com
1.新疆石河子大学医学院,石河子832000;
2.新疆石河子大学动物科技学院,石河子832000;
R384.3
A
1002-2694(2016)09-0789-04
2016-02-23;
2016-06-21
国家质检总局科技计划项目(No.2016LK264)资助
3.新疆阿拉山口检验检疫局,阿拉山口833418
Supported by the AQSIQ Science ang Technology Project (No.2016LK264)
猜你喜欢
——和田盘羊
