MALDI-TOF-MS鉴定布鲁氏菌方法建立和评价
2016-10-24张慧芳陈经雕毛玲玲崔步云张建中
汤 旭,肖 迪,张慧芳,陈经雕,毛玲玲,崔步云,张建中,姜 海
MALDI-TOF-MS鉴定布鲁氏菌方法建立和评价
汤旭1,肖迪2,张慧芳2,陈经雕3,毛玲玲4,崔步云2,张建中2,姜海2
目的为了快速、准确、便捷的检测和鉴定布鲁氏菌,本研究拟建立鉴定布鲁氏菌属的MALDI-TOF-MS方法,利用现场分离菌株对该方法进行特异性和敏感性评价。方法收集布鲁氏菌标准菌株和地方流行株,应用MALDI-TOF-MS采集图谱,获取独特的蛋白质指纹图谱,汇总成标准图谱,建立布鲁氏菌鉴定数据库。并用39株布鲁氏菌菌株,对建立的数据库进行验证。结果经数据库鉴定,39株布鲁氏菌菌株与数据库中布鲁氏菌的匹配分数全部大于2.300,均报告为布鲁氏菌,表明鉴定结果的可信度很高。 聚类分型结果表明,在蛋白质水平,MALDI-TOF-MS将测试的布鲁氏菌分成3大类。结论MALDI-TOF-MS对布鲁氏菌进行鉴定,具有快速、准确、灵敏等优点,可以实现对布鲁氏菌属的准确鉴定,对布鲁氏菌病的临床诊断具有较高的使用价值。
布鲁氏菌;MALDI-TOF-MS; 鉴定
布鲁氏菌病(brucellosis、简称布病)是由布鲁氏菌(Brucella)感染引起的人畜共患的传染—变态反应性疾病[1-2]。布病在《中华人民共和国传染病防治法》中被列为乙类传染病,由于其较高的感染率,它会给流行地区和国家造成巨大的经济损失并且严重危害人类健康[3]。人布病也称马耳他热,主要通过皮肤黏膜、消化道和呼吸道感染,主要临床表现是长期发热、伴有多汗、关节痛及肝脾肿大等,但往往又缺乏特异性[4-5],一般在临床易被误诊为其他疾病,如果治疗不及时可导致机体多个系统发生病变。布鲁氏菌是经典的生物战剂,被列入《国际禁止生物武器公约》核查清单,也是美国反生物恐怖袭击中的生物剂之一[6]。
布鲁氏菌属于兼性胞内寄生的革兰阴性菌,无芽胞、无鞭毛;营养条件要求高,生长缓慢,生长最适温度为37 ℃,个别菌种初代生长需一定浓度的CO2[7-8]。布鲁氏菌在布氏琼脂培养基上生长的菌落呈无色透明、圆形、表面光滑、稍隆起、均质样,在菌落中央常带有很小的颗粒,最初为透明状,以后逐渐浑浊。布鲁氏菌传统的分类方法是根据宿主倾向性,将其分为6个经典的种。根据培养和生物学特性的不同,又将6个经典种分为19个生物型,包括羊种布鲁氏菌(Brucellamelitensis)3个生物型,牛种布鲁氏菌(Brucellaabortus)8个生物型,猪种布鲁氏菌(Brucellasuis)5个生物型,沙林鼠种布鲁氏菌(Brucellaneotomae),绵羊附睾种布鲁氏菌(Brucellaovis)和犬种布鲁氏菌(BrucellaCanis)各有1个生物型。后来又分离到3个新的种型,分别被称为田鼠型(B.microti),鳍型(B.pinnipediae)和鲸型(B.cetacea或B.ceti)布鲁氏菌[9-11]。引起我国目前布鲁氏菌病疫情的是羊种、牛种和猪种布鲁氏菌,而在实验动物或宠物犬中经常能分离到犬种布鲁氏菌。传统的表型鉴定分类方法主要从菌株的形态学、生理生化反应特征以及免疫学特性加以鉴定分型的,这些传统经典分型方法是国际公认的,并且经过多年的实践证明其对流行病学和布鲁氏菌病的防治具有重要的意义。但是,在实际工作中约有10%~30%是用传统方法无法鉴定的非典型菌株[12],而且这些传统的鉴定方法检测周期长、判读结果繁琐、不能提供同一血清型中不同菌株之间的亲缘关系,而且还存在实验室生物安全隐患[13]。因此,寻找更加安全、可靠、快速的鉴定方法对布病的溯源工作、流行病学调查和预防控制都具有重要的意义。
基质辅助激光解吸电离飞行时间质谱(matrix assisted laser desorption ionization time of flight mass spectrometry,MALDI-TOF-MS)是近几年发展起来的一种全新的用于微生物鉴定和分型的技术,已越来越多地用于微生物的检测与鉴定。其基本原理为:样品与基质在靶盘上形成共结晶,利用激光作为能量来源辐射共结晶体,基质分子吸收能量与样品解吸附并使其电离,经过飞行时间检测器,将不同质荷比(m/z) 的离子分开,形成细菌特异性的质谱图[14-15]。用许多种已知标准菌株或来源分型明确的菌株建立参考细菌质谱图库,将待测细菌的质谱图与已有的质谱图进行比较,即可确定细菌种属[16]。由于其具有快速、灵敏、准确、高通量等优点,MALDI-TOF-MS 已经逐渐成为临床诊断、食品生产、环境监测以及军事领域研究的一种有力手段。本研究通过MALDI-TOF-MS采集布鲁氏菌特征性指纹图谱,创建布鲁氏菌鉴定质谱图数据库,实现对布鲁氏菌的快速准确鉴定,从而可以为布鲁氏菌病疫情的暴发和溯源提供技术支持。
1 材 料
1.1实验菌株选取20株(1-20)布鲁氏菌菌株用于建立MALDI-TOF-MS鉴定数据库。使用39株(21-58)布鲁氏菌分离菌株(见表1)用于评价所建立数据库的鉴定可信度。所用实验菌株均由中国疾病预防控制中心传染病预防控制所布鲁氏菌病室保存和提供。
1.2菌株鉴定59株受试菌株均按照传统方法进行生化反应鉴定。
2 布鲁氏菌MALDI-TOF-MS鉴定数据库的建立
2.1样品处理与点样
2.1.1取适量(5~10 mg)布鲁氏菌新鲜培养物,加入300 μL去离子水,仔细混匀,再加入900 μL无水乙醇混匀。如不马上测定可放置在-20 ℃保存。微生物中的核糖体蛋白在该环境下很稳定可以保存几个星期甚至几个月。
2.1.2高速(10 000-16 000 r/min)离心2 min,弃去上清(必要时,再离心一次尽量去除乙醇和水)。
2.1.3加入50 μL 70%甲酸,混匀,再加入50 μL乙腈,混匀,高速离心2 min,吸出上清置于一新的离心管中。
2.1.4取1 μL上清液置于MALDI-TOF-MS样品靶盘上,晾干后用2 μL基质溶液覆盖,在空气中晾干。
2.1.5分别取等体积的标准肽溶液与基质溶液混合后,取2 μL混合液置于MALDI-TOF-MS样品靶盘上,在空气中晾干,待完全干燥后进行MALDI-TOF-MS检测。每个样品和标准肽各做3个平行孔。
表1用于建立和评价布鲁氏菌MALDI-TOF-MS数据库的菌株信息
Tab.1Strain information of Brucella for MALDI-TOF-MS database establishment and evaluation
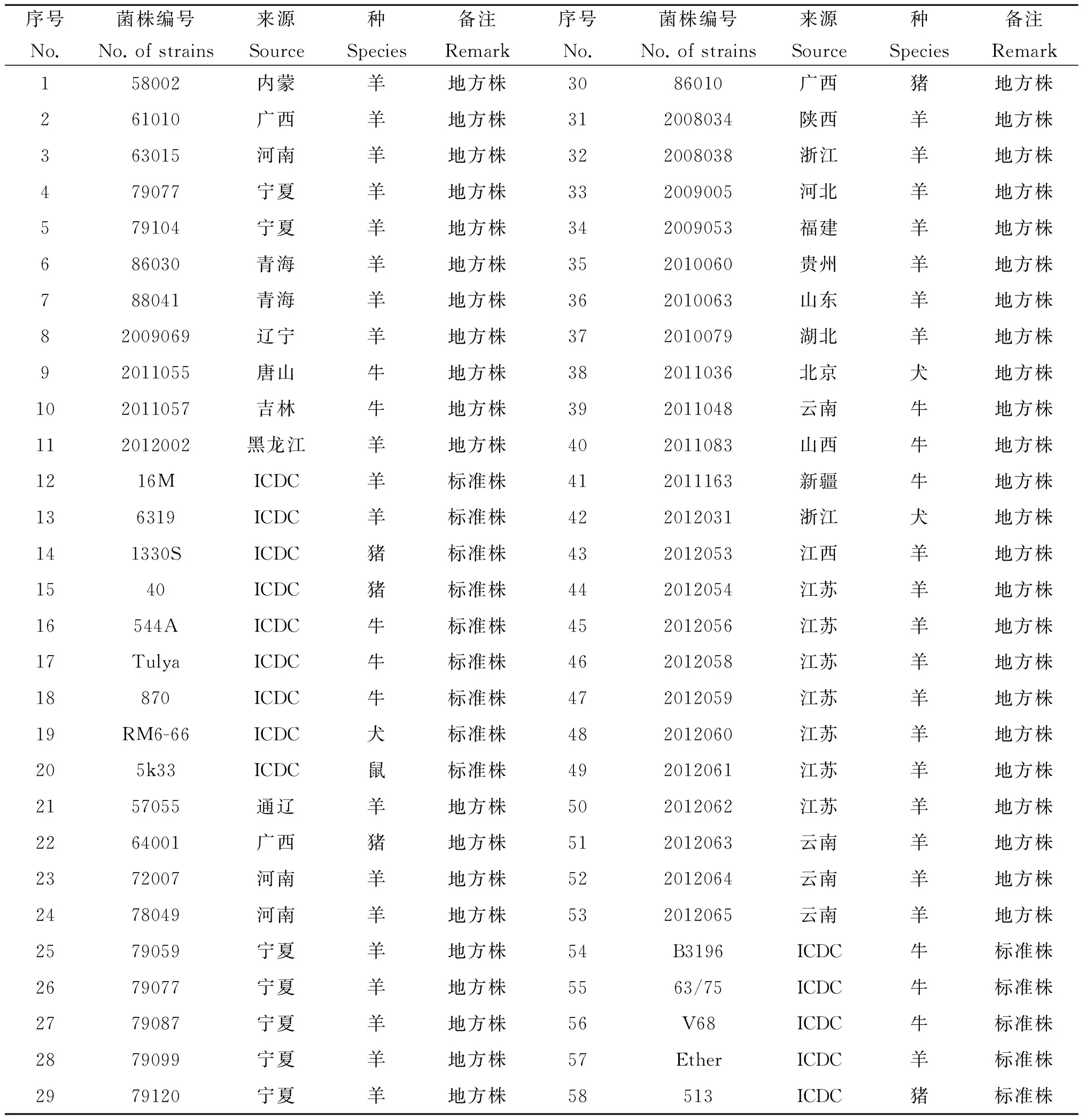
序号No.菌株编号No.ofstrains来源Source种Species备注Remark序号No.菌株编号No.ofstrains来源Source种Species备注Remark158002内蒙羊地方株3086010广西猪地方株261010广西羊地方株312008034陕西羊地方株363015河南羊地方株322008038浙江羊地方株479077宁夏羊地方株332009005河北羊地方株579104宁夏羊地方株342009053福建羊地方株686030青海羊地方株352010060贵州羊地方株788041青海羊地方株362010063山东羊地方株82009069辽宁羊地方株372010079湖北羊地方株92011055唐山牛地方株382011036北京犬地方株102011057吉林牛地方株392011048云南牛地方株112012002黑龙江羊地方株402011083山西牛地方株1216MICDC羊标准株412011163新疆牛地方株136319ICDC羊标准株422012031浙江犬地方株141330SICDC猪标准株432012053江西羊地方株1540ICDC猪标准株442012054江苏羊地方株16544AICDC牛标准株452012056江苏羊地方株17TulyaICDC牛标准株462012058江苏羊地方株18870ICDC牛标准株472012059江苏羊地方株19RM6-66ICDC犬标准株482012060江苏羊地方株205k33ICDC鼠标准株492012061江苏羊地方株2157055通辽羊地方株502012062江苏羊地方株2264001广西猪地方株512012063云南羊地方株2372007河南羊地方株522012064云南羊地方株2478049河南羊地方株532012065云南羊地方株2579059宁夏羊地方株54B3196ICDC牛标准株2679077宁夏羊地方株5563/75ICDC牛标准株2779087宁夏羊地方株56V68ICDC牛标准株2879099宁夏羊地方株57EtherICDC羊标准株2979120宁夏羊地方株58513ICDC猪标准株
2.2数据采集将上样的样品板小心置于板孔中,加有样品一面朝上,盖上盖子,抽真空。打开仪器控制软件FlexControl,调好仪器参数,校准仪器。采集标准品及样品的质谱图。对采集的数据进行保存,每次试验前都要在采集数据的质量范围内使用标准肽蛋白进行校准,校准后进行质谱数据采集,通过Biotyper软件进行分析鉴定。
2.3建立数据库的方法按2.2.1的方法进行样品处理与点样,每个平行样品重复点2个样品孔。每株菌共点l0个样品孔。按2.2.2的方法校准仪器后,点击样品孔采集数据,每个样品孔点击10次,采集2个质谱图。使用BioTyper软件,分别调入所采集的每株布鲁氏菌的质谱图,将此质谱图汇总生成整合图谱,对所得的图谱进行分析统一化,该图谱便作为布鲁氏菌质谱图数据库的标准图谱。
2.4对布鲁氏菌质谱数据库的验证采用39株常规和PCR方法已经鉴定为布鲁氏菌菌株对所建立的数据库进行验证。
2.5结果判断应用MALDI-TOF-MS BioTyper分析软件进行细菌检测时,将待测细菌的检测结果通过软件在细菌库中检索,判定标准是待检样品与细菌库里标准菌株的图谱的符合度,这种符合度用分数表示,0.000~1.699之间,颜色显示为红色,则无法判定,标记为(-);在1.700~1.999之间,颜色显示为黄色,判定为该种细菌的概率极低,标记为(+);得分在2.000~2.299之间,颜色显示为绿色,判定为该种细菌的概率很高,标记为(++);得分在2.300~3.000之间,颜色显示为绿色,判定为该菌种,标记为(+++)。
3 结 果
3.1布鲁氏菌MALDI-TOF-MS鉴定数据库的建立对20株布鲁氏菌菌株全部进行了图谱的采集与汇总,建立标准图谱,最终建成包含20株布鲁氏菌信息的独立的鉴定数据库。这20株布鲁氏菌菌株的主要离子峰一致,峰谱稳定。以羊种布鲁氏菌标准菌株(16M)为例,如图1所示,经MALDI-TOF-MS分析,羊种布鲁氏菌(16M)主要离子峰为:m/z3675.991、4536.143、5167.895、6672.614、7356.699、9075.223、9789.003。
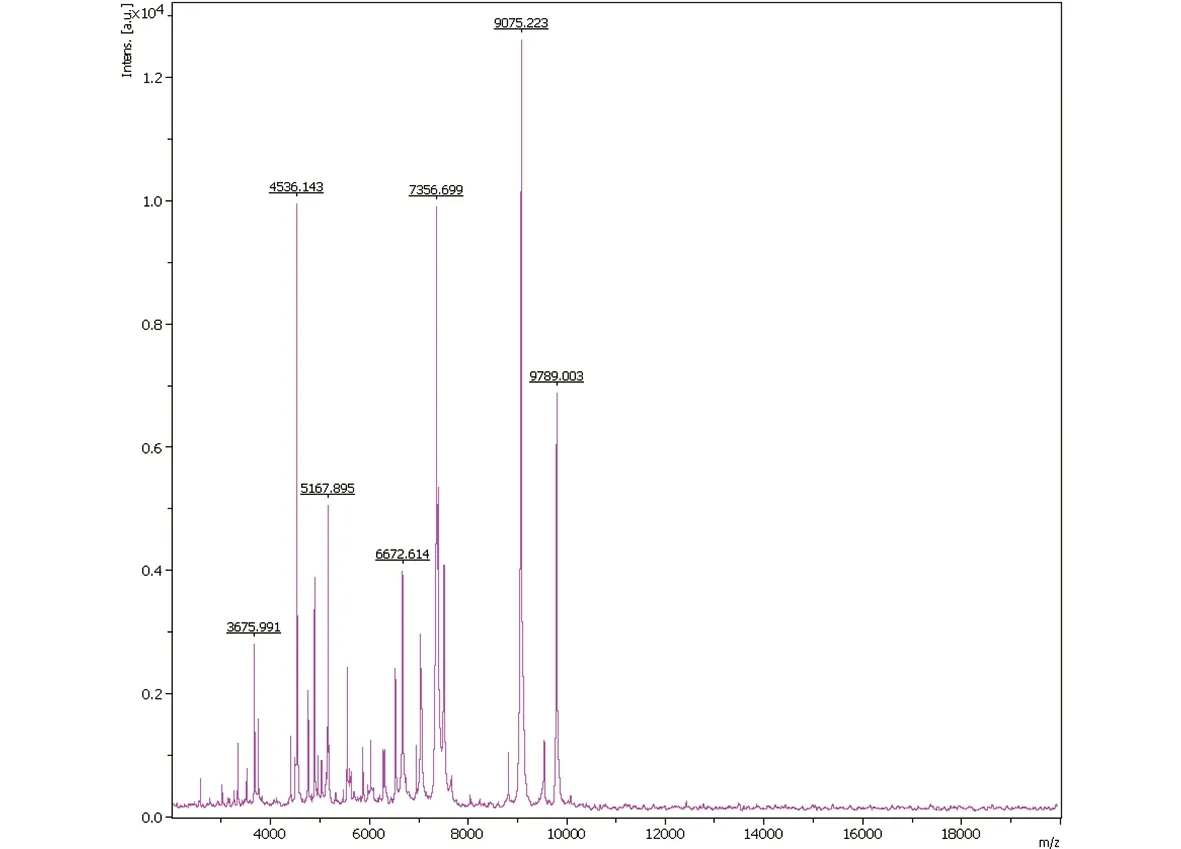
图1 MALDI-TOF-MS分析布鲁氏菌(16M)的主要离子峰图谱Fig.1 The main spectra of Brucella (16M) obtained from MALDI-TOF-MS
3.2对布鲁氏菌MALDI-TOF-MS鉴定数据库的验证
3.2.1通过Biotyper分析软件对样品进行判定打开Biotyper分析软件,调入选中样品数据进行分析,图2左侧是编号为2008034菌株的指纹图谱和细菌库中编号为2009069菌株的对照图,横线以上是样品的指纹图,由多条竖线组成,若样品指纹图与标准图成镜像关系,说明样品在此处的图谱与标准菌株完全一样,以绿色表示。若样品在某处的指纹图与标准图不同,则以红色表示。绿色竖线越多说明判定为该菌株的概率越大。图2右侧是鉴定结果,得分由高到低,第一行是得分最高的,细菌也由得分最高的进行判定。图2-2中最高得分为2.748,在2.3~3之间,判定为布鲁氏菌。
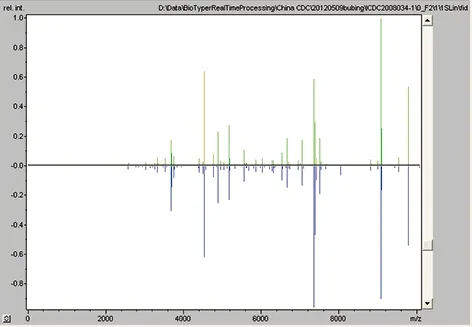
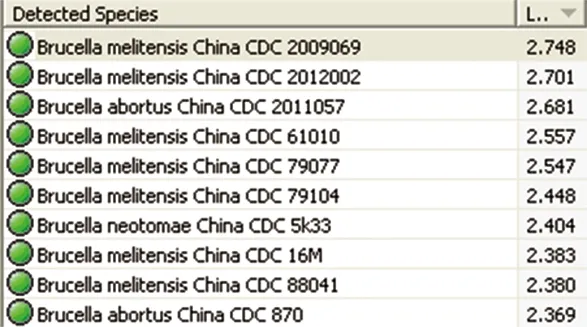
图2 编号为2008034的菌株,得分为2.748的质谱鉴定结果
3.2.2MALDI-TOF-MS对39株布鲁氏菌菌株的鉴定结果将39株布鲁氏菌菌株(见表2)分别进行MALDI-TOF-MS鉴定,分析在布鲁氏菌数据库中的匹配结果。经鉴定,39株布鲁氏菌菌株的匹配分数全部大于2.300,均报告为布鲁氏菌,表明鉴定结果的可信度很高。
表239株布鲁氏菌菌株MALDI-TOF-MS的鉴定结果
Tab.2Identification results of 39 Brucella strains with MALDI-TOF-MS
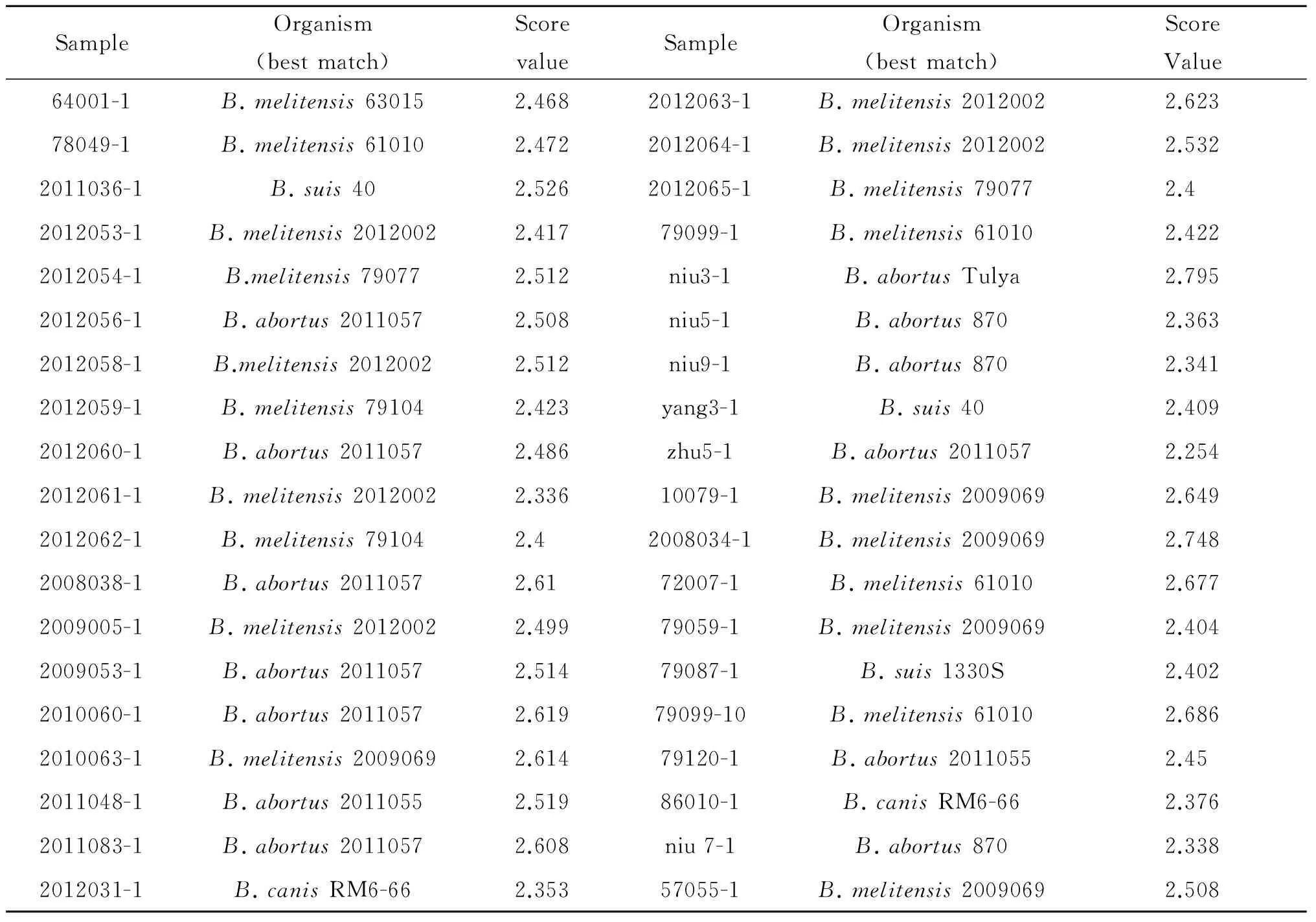
SampleOrganism(bestmatch)ScorevalueSampleOrganism(bestmatch)ScoreValue64001-1B.melitensis630152.4682012063-1B.melitensis20120022.62378049-1B.melitensis610102.4722012064-1B.melitensis20120022.5322011036-1B.suis402.5262012065-1B.melitensis790772.42012053-1B.melitensis20120022.41779099-1B.melitensis610102.4222012054-1B.melitensis790772.512niu3-1B.abortusTulya2.7952012056-1B.abortus20110572.508niu5-1B.abortus8702.3632012058-1B.melitensis20120022.512niu9-1B.abortus8702.3412012059-1B.melitensis791042.423yang3-1B.suis402.4092012060-1B.abortus20110572.486zhu5-1B.abortus20110572.2542012061-1B.melitensis20120022.33610079-1B.melitensis20090692.6492012062-1B.melitensis791042.42008034-1B.melitensis20090692.7482008038-1B.abortus20110572.6172007-1B.melitensis610102.6772009005-1B.melitensis20120022.49979059-1B.melitensis20090692.4042009053-1B.abortus20110572.51479087-1B.suis1330S2.4022010060-1B.abortus20110572.61979099-10B.melitensis610102.6862010063-1B.melitensis20090692.61479120-1B.abortus20110552.452011048-1B.abortus20110552.51986010-1B.canisRM6-662.3762011083-1B.abortus20110572.608niu7-1B.abortus8702.3382012031-1B.canisRM6-662.35357055-1B.melitensis20090692.508
3.359株布鲁氏菌MALDI-TOF-MS聚类分型结果本研究通过模式匹配计算59株布鲁氏菌主要谱图的相似性,采用相似分值构建系统树,对59株菌株进行了聚类分型分析。59株布鲁氏菌聚类分型树状图如图3所示。参与分析的菌株被分成3个主要的类别,并用不同的颜色标示,其中绿色的类别中以羊种布鲁氏菌为主,蓝色的类别中以牛种布鲁氏菌为主,红色的类别中以猪种布鲁氏菌为主。
4 讨 论
MALDI-TOF-MS是近几年发展起来的一种全新的用于微生物鉴定和分型的生物质谱技术,它利用不同细菌间特异的质荷比作为生物标志分子鉴定细菌,具有快速、准确、高通量等优点。虽然传统的布鲁氏菌鉴定方法准确、可靠,是目前公认的鉴定方法,但缺点是步骤繁琐,检测周期长,而且对有些生化特性不典型的布鲁氏菌难以鉴定。基于细菌全蛋白分析的MALDI-TOF-MS方法对布鲁氏菌进行鉴定与分型丰富或弥补了现有方法的不足,其原理、操作程序均与现在惯用方法不同,无需提取DNA,仅在样品处理时滴加少量基质,即可上机测定,大容量样品板一次可测定100多份样品,1 h内即可获得结果,可以连续测定,结果以直观的图谱形式表达,数据便于统计学分析,实现了目前任何一种检测仪器或方法所无法比拟的高通量和高效率。但在应用MALDI-TOF-MS分析全细菌时,应当考虑到可能影响全细菌图谱质量和重复性的实验因素。目前对仪器参数和样品制备的调查结果显示,细菌的培养和样品的制备是两个重要影响因素[17]。培养条件(培养基种类和培养时间)能影响生物标记分子的出现,从而引起图谱的变化[18]。其次,所用的基质、酸的种类和浓度的不同也导致不同实验室对同一菌株的分析结果出现偏差[19],但通过将分析程序标准化即可解决上述问题[20]。
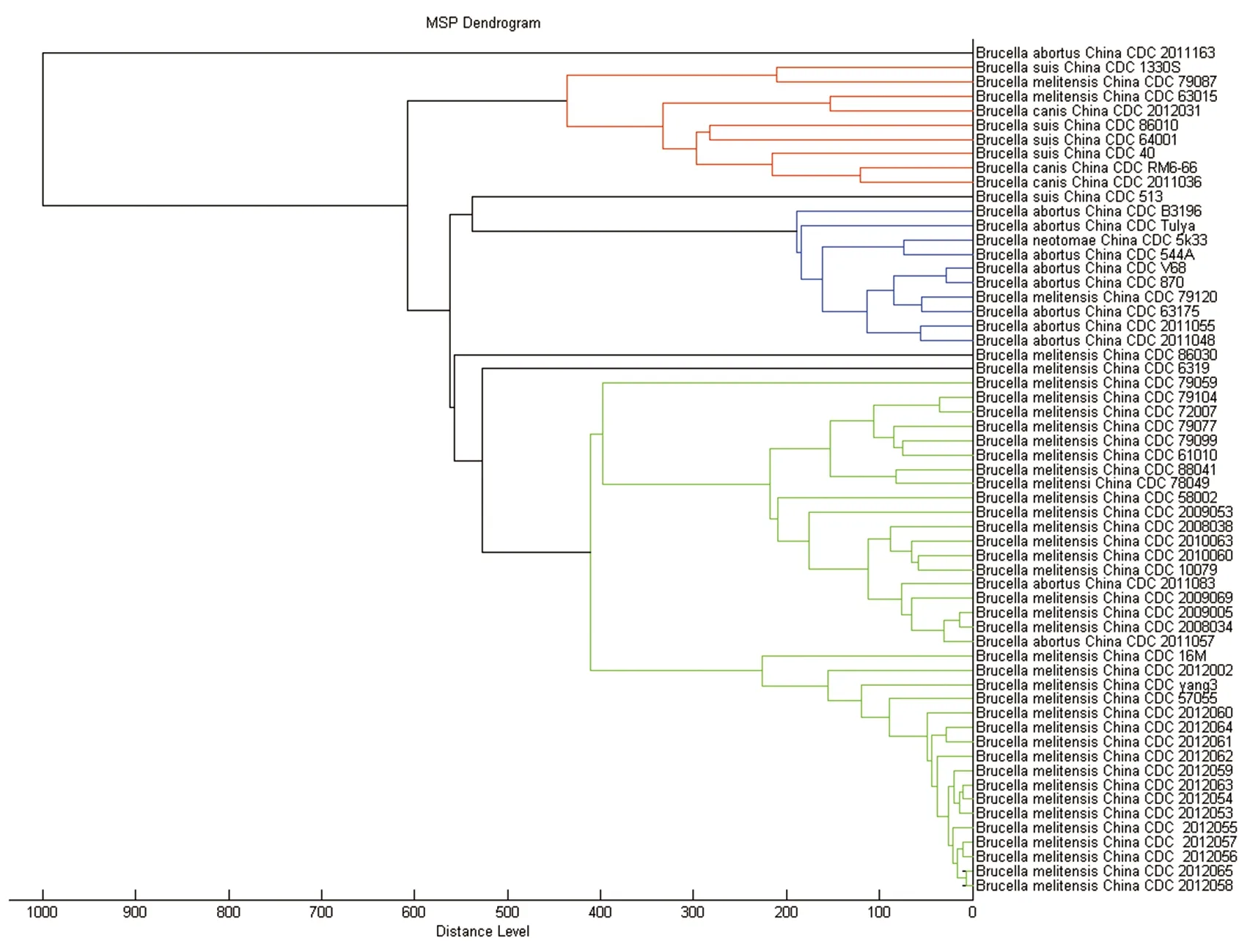
图3 59株布鲁氏菌MALDI-TOF-MS聚类分型树状图Fig.3 Cluster analysis of 59 Brucella strains according to the results of MALDI-TOF-MS
本研究通过采集不同时间不同地点分离的20株布鲁氏菌菌株的数据,获得了特征指纹图谱,创建了鉴定布鲁氏菌的质谱图数据库。并通过39株布鲁氏菌菌株对建立的数据库进行验证,结果显示,布鲁氏菌菌株的匹配分数均大于2.300,MALDI-TOF-MS能够实现对布鲁氏菌的准确鉴定。本研究在建立布鲁氏菌质谱鉴定数据库的同时,还根据不同菌株的谱图特点对所研究的59株布鲁氏菌进行了聚类分型。结果表明,质谱聚类分型的结果一方面与传统分型的类别比较吻合,另一方面也说明传统的经验分型方法存在不足,而质谱分型基于不同菌株的蛋白图谱特征,更精确地体现了不同菌株的生物学特点,是一种更为科学可靠的分型方法。从而可以为布鲁氏菌疫情的暴发和流行的预防和溯源提供技术支撑。
MALDI-TOF-MS对未知细菌分型鉴定必须建立在含有足够多的已知菌株的谱库中进行检索,方可实现最匹配且真实可靠的菌株鉴定结果,本实验所用仪器软件支持用户自定义数据库。因此,后续研究可按统一的建库标准扩大检测数据库,提高对细菌的分型鉴定能力。相信随着数据库的不断补充与应用软件的完善,MALDI-TOF-MS不仅在微生物的鉴定中体现其快速、准确、灵敏的特点,还可能成为直接在分子层次上揭示菌体活动的有力工具,用于病原菌溯源性研究、生物武器鉴别、微生物的致病性和毒力特征描述等方面。
[1] Cui BY. Surveillance and control of brucellosis in China[J]. Dis Surveill,2007,22(10): 649-651. (in Chinese)
崔步云. 中国布鲁菌病疫情监测与控制[J]. 疾病监测,2007,22(10): 649-651.
[2] Liu BY,Sun X,Shang DQ,et al. Brucellosis[M]. Beijing: People’s Medical Publishing House, 1981:1. (in Chinese)
刘秉阳,孙玺,尚德秋,等. 布鲁氏菌病学[M]. 北京: 人民卫生出版社,1989:1.
[3] Corbel MJ. Brucellosis: an overview[J]. Emerg Infect Dis,1997,3(2): 213-221. DOI: 10.3201/eid0302.970219
[4] Qi JW. Summary of brucellosis[J]. Chin J Vet Med,2009,20(30): 50-53. (in Chinese)
齐景文. 布鲁菌病概述[J]. 中国兽医杂志,2009,20(3): 50-53.
[5] Xiao DL,Jang SL,Cui BY,et al. Brucellosis prevention manual[M]. Beijing: People’s Medical Publishing House,2008: 38-52. (in Chinese)
肖东楼,江森林,崔步云,等. 布鲁菌病防治手册[M]. 北京: 人民卫生出版社,2008: 38-52.
[6] Zhang XF. A retrospective study of 1988-1990 years of brucellosis in Almeria[J]. Local Dis,1994,15(6): 58. (in Chinese)
张雪峰. 阿尔梅里亚省布氏菌病1988-1990年的回顾性研究[J]. 地方病丛,1994,15(6): 58.
[7] Zhao ZX,Cui BY,Hao SZ. The research progress of typing ofBrucellaspecies[J]. Chin J Ctrl Endem Dis,2007,18(2): 172-175. (in Chinese)
赵振祥,崔步云,郝素珍. 布鲁菌分种分型研究进展[J]. 中国地方病防治杂志,2007,18(2): 172-175.
[8] Wang YC,Wang ZL. Experimental report of oxidative metabolism test forBrucellatyping[J]. Chin J Ctrl Endem Dis,1991,6(4): 209-210. (in Chinese)
王永成,王芝兰. 氧化代谢试验用于布鲁菌分型的实验报告[J]. 中国地方病防治杂志,1991,6(4): 209-210.
[9] Bricker BJ,Ewalt DR,MacMillan AP,et al. Molecular characterization ofBrucellastrains isolated from marine mammals[J]. J Clin Microbiol,2000,38(3): 1258-1262.
[10] Nielsen O,Stewart RE,Nielsen K,et al. Serologic survey ofBrucellaspp. antibodies in some marine mammals of North America[J]. J Wildl Dis,2001,37(1): 89-100.
[11] Scholz HC,Hubalek Z,Sedlacek I,et al.Brucellamicroti sp. nov.,isolated from the common voleMicrotusarvalis[J]. Int J Syst Evol Microbiol,2008,58(2): 375-382. DOI: 10.1099/ijs.0.65356-0
[12] Cui BY,Yin JM,Li LY et al. Study on the typing ofBrucellaRep-PCR[J]. Dis Surveill,2005,20(8): 397-400. (in Chinese)
崔步云,尹继明,李兰玉,等. 布鲁氏菌的Rep-PCR分型研究[J]. 疾病监测,2005,20(8): 397-400.
[13] Gee JE,De BK,Levett PN,et al. Use of 16S rRNA gene sequencing for rapid confirmatory identification ofBrucellaisolates[J]. J Clin Microbiol,2004,42(8): 3649-3659. DOI: 10.1128/JCM.42.8.3649-3654.2004
[14] Liu HH. Application of MALDL-TOF-MS in the detection and identification of bacteria[J]. Progr Microbiol Immunol,2003,31: 47-53. (in Chinese)
刘海洪. MALDL TOF MS 在细菌检测和鉴定中的应用[J]. 微生物学免疫学进展,2003,31: 47-53.
[15] Yi P,Zhang HM. Technology and application of MALDL-TOF-MS[J]. Mod Instruments,2002,1: 118-119. (in Chinese)
怡萍,张红明. 激光解析电离飞行时间质谱技术及应用[J]. 现代仪器,2002,1: 118-119.
[16] Xu J,Broun R,Winograd N. Applicability of imaging time-of-flight secondary ion ms to the characterization of solid phase synthesized combinatorial libraries[J]. Anal Chem,2003,75: 6155-6162.
[17] Williams TL,Andrzejewski D,Lay JO,et al. Experimental factors affecting the quality and reproducibility of MALDI TOF mass spectra obtained from whole bacteria cells[J]. J Am Soc Mass Spectromet,2003,14(4): 342-351. DOI: 10.1016/S1044.0305(03)00065-5
[18] Conway GC,Smole SC,Sarracino DA,et al. Phyloproteomics: species identification ofEnterobacteriaceaeusing matrix-assisted laser desorption/ionization time of flight mass spectrometry[J]. J Mol Microbiol Biotechnol,2001,3(1): 103-112.
[19] Sharon CW,Kristin HJ,Catherine EP,et al. Bacterial analysis by MALDI-TOF mass spectrometry: an interlaboratory comparison[J]. J Am Soc Mass Spectromet,2005,16(4): 456-462. DOI: 10.1016/j.jasms.2004.12.004
[20] Vargha M,Takats Z,Konopka A,et al. Optimization of MALDI-TOF MS for strain level differentiation ofArthrobacterisolates[J]. J Microbiologic Methods,2006,66(3): 399-409. DOI: 10.1016/j.mimet.2006.01.006
Establishment and evaluation of identification ofBrucellaby MALDI-TOF-MS
TANG Xu1,XIAO Di2,ZHANG Hui-fang2,CHEN Jing-diao3,MAO Ling-ling4,CUI Bu-yun2,ZHANG Jian-zhong2,JIANG Hai2
(1.HaidianCenterforDiseaseControlandPrevention,Beijing100094,China;2.StateKeyLaboratoryforInfectiousDiseasePreventionandControl,CollaborativeInnovationCenterforDiagnosisandTreatmentofInfectiousDiseases,NationalInstituteforCommunicableDiseaseControlandPrevention,ChineseCenterforDiseaseControlandPrevention,Beijing102206,China;3.GuangdongProvincialCenterforDiseaseControlandPrevention,Guangzhou511430,China;4.LiaoningProvincialCenterforDiseaseControlandPrevention,Shenyang110005,China)
Fast,accurate and convenient detection and identification ofBrucella,MALDI-TOF-MS was established. The sensibility and specificity of the method was evaluated too. MALDI-TOF-MS database was established by collecting the unique protein fingerprint of 20Brucella
trains and endemic strains and was evaluated by 39Brucellaisolates. On the basis of database information,59Brucellastrains were grouped into 3 clusters. Mass spectra database ofBrucellaidentification was created through the acquisition of theBrucellacharacteristic fingerprint.Brucellaisolates were selected to verify established database. Match scores between 39 strains and reference strains in database were all greater than 2.300 and results are reported asBrucella. This revealed that degree of confidence was very high. Clustering results indicated thatBrucellawas divided into three categories at the protein level by MALDI-TOF-MS. MALDI-TOF-MS was used to identifyBrucellaat protein levels. The method is fast,accurate,sensitive and has higher value on clinical diagnosis of brucellosis.
Brucella; MALDI-TOF-MS; identification
Jiang Hai,Email: jianghai@icdc.cn
10.3969/j.issn.1002-2694.2016.09.002
姜海,Email:jianghai@icdc.cn
1. 北京市海淀区疾病预防控制中心,北京100094;
2. 中国疾病预防控制中心传染病预防控制所,传染病预防控制围家重点实验室,感染性疾病协同诊治协同创新中心,北京102206;
R378.5
A
1002-2694(2016)09-0772-07
2016-06-01;
2016-07-25
国家自然科学基金 (No.81271900)
3. 广东省疾病预防控制中心,广州511430;
4. 辽宁省疾病预防控制中心,沈阳110005
Supported by the National Natural Science Foundation of China (No. 81271900)
