陕棉抗病种质及其衍生品种的遗传多样性与群体结构研究
2016-10-14邢泽农贺道华邢宏宜雷忠萍
邢泽农, 蒋 雷, 贺道华*, 邢宏宜, 雷忠萍
(1 宝鸡市农业科学研究所,陕西岐山 722400;2 西北农林科技大学 农学院,陕西杨陵 712100;3 西北农林科技大学 生命科学学院,陕西杨陵 712100)
陕棉抗病种质及其衍生品种的遗传多样性与群体结构研究
邢泽农1,2, 蒋 雷2, 贺道华2*, 邢宏宜2, 雷忠萍3
(1 宝鸡市农业科学研究所,陕西岐山 722400;2 西北农林科技大学 农学院,陕西杨陵 712100;3 西北农林科技大学 生命科学学院,陕西杨陵 712100)
通过72个分布于棉花全基因组的SSR标记,对54份陕棉抗病种质及其衍生品种的遗传多样性与群体结构进行了分析。结果表明:(1)54份陕棉种质间的遗传相似系数在0.733 3~0.987 2之间,其中材料间相似系数≤0.90的占11.1%,相似系数≥0.95的占55.6%,相似系数在0.90~0.95的占33.3%。(2)72个SSR标记等位基因变异的多态性信息含量(PIC值)在0.04~0.68,平均为0.33。(3)基于遗传距离的UPGMA聚类分析显示,在遗传相似系数为0.877时将54个品种分为5类,第Ⅰ类44个品种,第Ⅲ类7个品种,第Ⅱ、Ⅳ、Ⅴ类各1个品种。(4)基于数学模型的聚类和群体结构分析显示,54份种质归属于4个组群。研究认为,54份材料间遗传相似系数较大,遗传基础比较狭窄,多样性很低。
棉花;抗病种质;SSR;遗传多样性;群体结构
枯萎病和黄萎病是危害棉花最为严重的两种病害,严重影响了棉花生产。为了经济有效地控制棉花枯、黄萎病,西北农林科技大学(原陕西省棉花研究所)采用多种杂交方法,通过在发重病且均匀的枯、黄萎病混生病圃连续选择,创造出高抗枯、黄萎病的材料,先后育成一批抗病能力强且综合性状优良的种质,如‘陕棉4号’、‘陕棉5号’、‘陕401’、‘陕1155’等[1]。这些陕棉抗病种质被全国棉花育种单位用作亲本或系统育种的原始材料,育成广为种植的品种60余个,如‘86-1’、‘晋棉7号’、‘中棉所17’、‘中棉所19’、‘中棉所23’(被导入抗虫基因育成‘中棉所41’)、‘中棉所35’等[2-3]。据统计,陕棉抗病种质及其衍生品种在中国累计种植0.2×109hm2,取得巨大的经济、社会、生态效益[1]。
国内外利用DNA分子标记技术对棉花抗病品种的遗传多样性已经做了大量的研究[4-8],结果表明:中国供试抗病品种群体的遗传基础较为狭窄,基于分子标记的类群划分与品种系谱来源有较强的相关性。徐秋华等[6]研究表明中国抗枯萎病棉花品种之间的相似性较高,认为其原因在于中国陆地棉品种资源整体上遗传多样性水平低下,以及抗病品种资源狭窄的遗传基础。刘文欣等[9]研究显示:陆地棉品种的多样性低于棉属的其他品种,陆地棉栽培品种的遗传基础较为狭窄,且中国自育陆地棉品种的遗传基础窄于国外引进品种。诸多的研究一方面说明了分子标记在遗传多样性研究上的重要价值,另一方面也显示了陆地棉品种间很低的多样性,尤其是在同一个国家或地区内。
在棉花种质研究方面,利用分子标记研究抗病种质的遗传多样性虽然已屡见不鲜[10],但利用均匀来自全基因组的DNA分子标记同时研究陕棉血统的抗病种质的遗传多样性及群体遗传结构,在分子水平上明确该群体的遗传基础,在国内外还鲜见报道。本研究选用已被定位且广泛分布于棉花基因组的各染色体框中SSR标记,系统地分析陕棉抗病种质及其衍生品种的群体遗传结构及遗传多样性水平,了解陕棉血统抗病种质的遗传分化,将有助于拓宽棉花抗病遗传基础、发掘抗病基因和改良棉花抗病品种。
1 材料和方法
1.1 供试材料
本实验取20世纪60年代以来在生产上被大面积种植的陕棉抗病种质,以及应用陕棉抗病种质育成的衍生抗病品种,衍生品种只取至第三轮。其中陕棉抗病种质9个,衍生品种45个,共54份材料(表1),均由中国农业科学院棉花研究所提供。
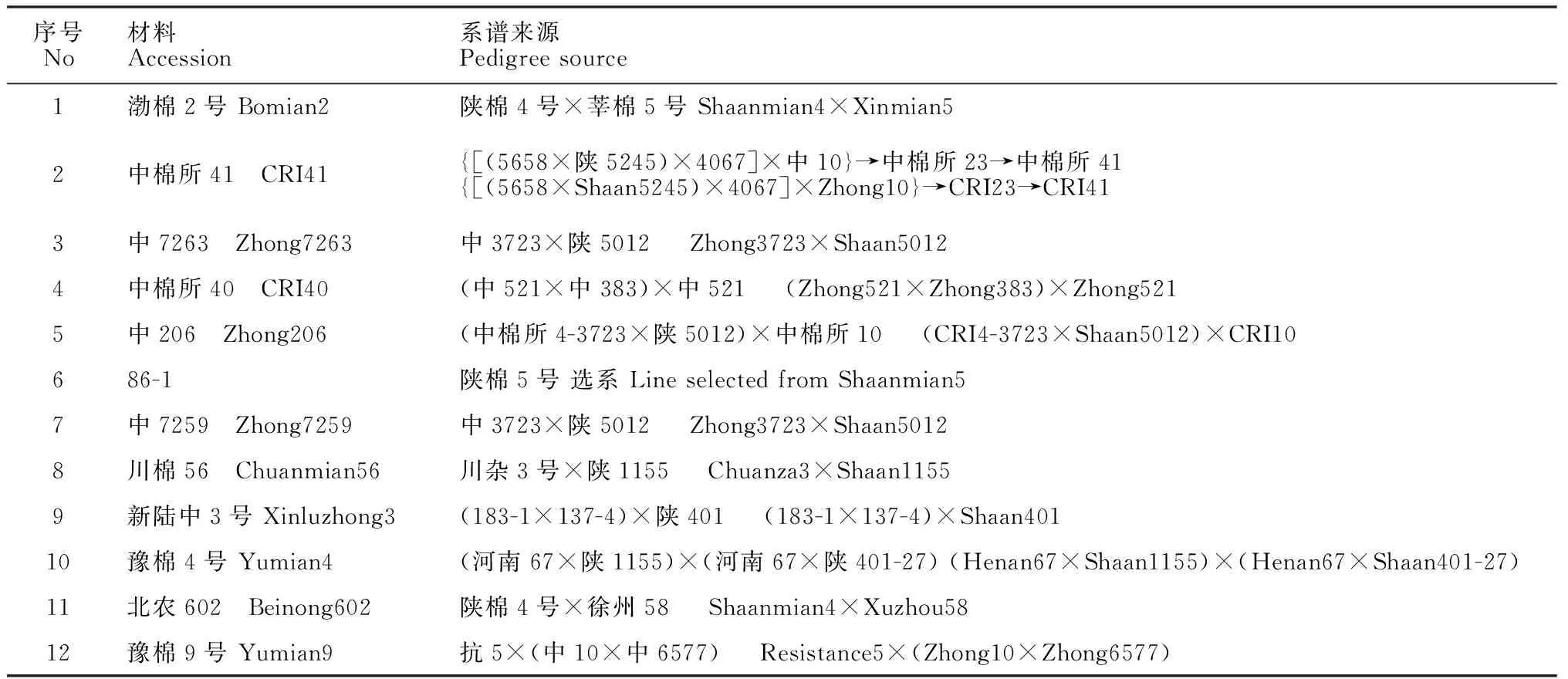
表1 54份陆地棉品种系谱来源
续表1 Continued Table 1

序号No材料Accessions系谱来源Pedigreesource13川抗414Chuankang41川364×陕401Chuan364×Shaan40114辽棉10Liaomian10陕32-15选系LineselectedfromShaan32-1515中6331Zhong6331(中7-198×陕32-15)×冀棉1号→5691(Zhong7-198×Shaan32-15)×Jimian1→569116冀合321Jihe321陕棉4号×陕3765→冀75-7×75-23Shaanmian4×Shaan3765→Ji75-7×75-2317陕3619Shaan3619(55-90×岱字棉)×(徐州209×岱福棉)→F1×陕棉3号(55-90×Deltapine)×(Xuzhou209×DeltapineFoster)→F1×Shaanmian318盐棉48Yanmian48陕棉5号→86-1×鲁棉1号Shaanmian5→86-1×Lumian119晋棉13号Jinmian13[(晋棉4号×陕棉5号→79-105)×岱字棉SR-1]×冀1724[(Jinmian4×Shaanmian5→79-105)×DeltapineSR-1]×Ji172420冀棉11号Jimian11(冀邯4号×珂字4104)→F2×74育102(Jihan4×Coker4104)→F2×74Yu10221冀棉3号Jimian3陕棉10号×中长1号→陕2319Shanmian10×Zhongchang1→Shaan231922江苏棉1号Jiangsumian1陕棉5号→86-1Shaanmian5→86-123鲁1024Lu1024山东129×陕5245Shandong129×Shaan524524陕5012Shaan5012陕401选系LineselectedfromShaan40125川73-27Chuan73-27陕棉4号选系LineselectedfromShaanmian426豫棉1号Yumian1陕棉4号×刘庄1号Shaanmian4×Liuzhuang127鲁棉研16Lumianyan16中棉所12×(泗棉1号×陕棉9号→盐抗76-75×791→泗棉3号)CRI12×(Simian1×Shaanmian9→Yankang76-75×791→Simian3)28绵阳83-21Mianyang83-21洞庭1号×陕3563Dongting1×Shaan356329陕3215Shaan3215陕401选系LineselectedfromShaan40130盐抗7305Yankang7305新棉4号×陕棉4号Xinmian4×Shaanmian431江苏棉3号Jiangsumian3(陕棉4号×刘庄1号→新291)×77-369(Shaanmian4×Liuzhuang1→Xin291)×77-36932李台8号Litai8中棉所7×陕棉9号CRI7×Shaanmian93386-3陕1155×632-124Shaan1155×632-12434陕954Shaan954陕棉6号选系LineselectedfromShaanmian635湘棉10号Xiangmian10(岱红岱×陕棉4号)×岱红岱(DeltRedDelt×Shaanmian4)×DeltRedDelt3686-4岱字棉16×(陕棉5号→86-1)Deltapine16×(Shaanmian5→86-1)37石无16Shiwu16[豫无19×(河南69×陕5245)]→豫棉2号[Yuwu19×(Henan69×Shaan5245)]→Yumian238冀植17Jizhi17辽632-125×(陕棉10号×冀棉1号)Liao632-125×(Shaanmian10×Jimian1)39陕8092Shaan8092辽642×陕棉10号Liao642×Shaanmian1040运1729Yun1729岱4067×[(86-1×贝无)×(中棉所3号×斯字棉7A)Deltapine4067×[(86-1×Beiwu)×(CRI3×Stoneville7A)]41苏棉12Sumian128004×(陕棉4号×陕棉6号→冀75-7×75-23→冀合328)8004×(Shaanmian4×Shaanmian6→Ji75-7×75-23→Jihe328)42豫79-10Yu79-10(岱字棉×中棉所7号)×(陕棉5号→86-1)(Deltapine×CRI7)×(Shaanmian5→86-1)43豫棉16Yumian16[(中棉所3号×陕棉4号→豫抗1号)×商丘66]×冀棉366[(CRI3×Shaanmian4→Yukang1)×Shangqiu66]×Jimian36644陕81-1Shaan81-1辽6913×陕1155Liao6913×Shaan115545陕401Shaan401陕棉3号Shaanmian346豫棉8号Yumian8豫3478×陕1155→豫3009×混合花粉Yu3478×Shaan1155→Yu3009×Manymales47陕3563Shaan3563陕2021选系LineselectedfromShaan202148泗棉3号Simian3泗棉1号×陕棉9号→盐抗76-75×泗阳791Simian1×Shaanmian9→Yankang76-75×Siyang79149陕5245Shaan5245陕棉4号×陕棉6号Shaanmian4×Shaanmian650豫抗1号Yukang1中棉所3号×陕棉4号CRI3×Shaanmian451冀合232Jihe232(冀无303×3016→F2)×3016(Jiwu303×3016→F2)×301652豫棉2号Yumian2豫无19×(河南69×陕5245)Yuwu19×(Henan69×Shaan5245)53中棉所15CRI156429×(陕棉5号→86-1)6429×(Shaanmian5→86-1)54晋棉14Jinmian14汾无195×陕1155Fenwu195×Shaan1155
1.2 方 法
1.2.1 棉花基因组DNA提取 摘取幼嫩叶片,采用修改的CTAB法提取基因组DNA[11],利用Beckman DU800紫外光分光光度计对DNA纯度和浓度进行检测,以λDNA作对照,通过0.8%琼脂糖凝胶电泳进一步确定其浓度和质量。
1.2.2 SSR分析 根据已公开发表的遗传图谱[12-13],从每个连锁群上不同的标记框[13]中选择SSR标记。从26个连锁群上选择出72个标记,由上海捷瑞生物工程有限公司进行引物合成,SSR分析所用试剂由北京全式金公司提供。PCR反应总体系为15 μL,包含基因组DNA(20 ng/μL)5 μL,10× Buffer (含Mg2+) 1.5 μL,10 mmol/L的dNTPs 0.6 μL,Taq DNA聚合酶(5U/μL) 0.21 μL,ddH2O 6.49 μL,正反引物(25 μmol/L)各0.6 μL。SSR反应程序为:95 ℃预变性3 min;94 ℃ 45 s, 55 ℃ 45s, 72 ℃ 1 min, 35个循环;72 ℃延伸10 min;4 ℃保存。扩增后的产物与10 μL的上样缓冲液混合,95 ℃变性6 min,立即转移至冰水浴冷却。采用变性聚丙烯酰胺凝胶(6%)电泳对SSR扩增产物进行分离,75 W恒功率电泳1 h。电泳后银染显带、晾干照相并记读带型。
1.2.3 数据处理与分析 根据电泳结果记录清晰可重复的条带,采取0/1赋值计带,观察扩增条带的有无,有则记为“1”,无则记为“0”,并根据DNA Marker推测并记录该条带的长度(bp)。SSR标记的多态性信息含量(PIC)根据所有材料中等位基因出现的频率计算,具体公式为:
PIC= 1 -∑(Pi)2
其中,Pi表示第i个等位基因出现的频率。利用NTSYS-pc (Ver 2.1)软件[14]处理SSR分型数据,计算两两品种间的遗传相似系数(GS),采用离差平方和方法进行系统聚类分析。
为了估测该群体的遗传结构,应用Structure 2.3.1软件[15],进行基于数学模型的类群划分,并计算54份材料相应的Q值(第i材料其基因组变异源于第k群体的概率)。先设定群体数目(K)为1~10,并假定位点都是独立的,对72个SSR位点的分型数据进行分析,将MCMC开始时的不作数迭代设为10 000次, 再将不作数迭代后的MCMC设为100 000次, 然后依据似然值LnP(D)最大的原则选取一个合适的K值,并绘出基于模型的群体遗传结构图[16]。
2 结果与分析
2.1 SSR标记的多态性分析
供试的72对SSR引物共扩增出176条多态性条带,平均每对SSR引物扩增2.44条多态性条带(表2),其中以CGR6905、CGR5396与BNL3860最少,为1条多态性条带;以CGR6185与BNL3442最多,为4条多态性条带。这说明,在CGR6185和BNL3442的扩增区域上,品种间遗传多态性比较丰富。SSR位点的多态性信息含量(PIC)范围为0.04~0.68之间,平均多态性信息含量为0.33,其中PIC≥ 0.6的标记有8个,占11%。总的来看,所选用的SSR引物在本试验群体中多态性信息含量不高,品种间在被检测SSR位点上,变异的频率不高,遗传多样性比较低。另外,在研究的过程中,通过扩增条带与Marker的比较分析发现,大多数SSR扩增片段的长度较小,且都集中在100~450 bp之间。
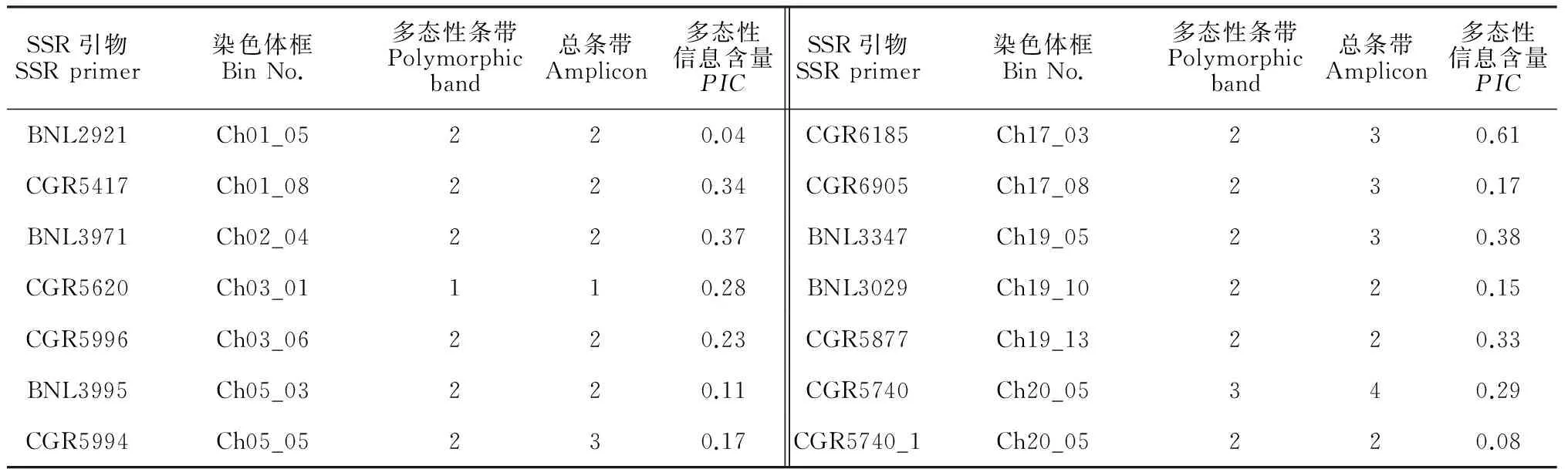
表2 SSR标记在染色体框中的分布、扩增情况和多态性信息含量
续表2 Continued Table 2
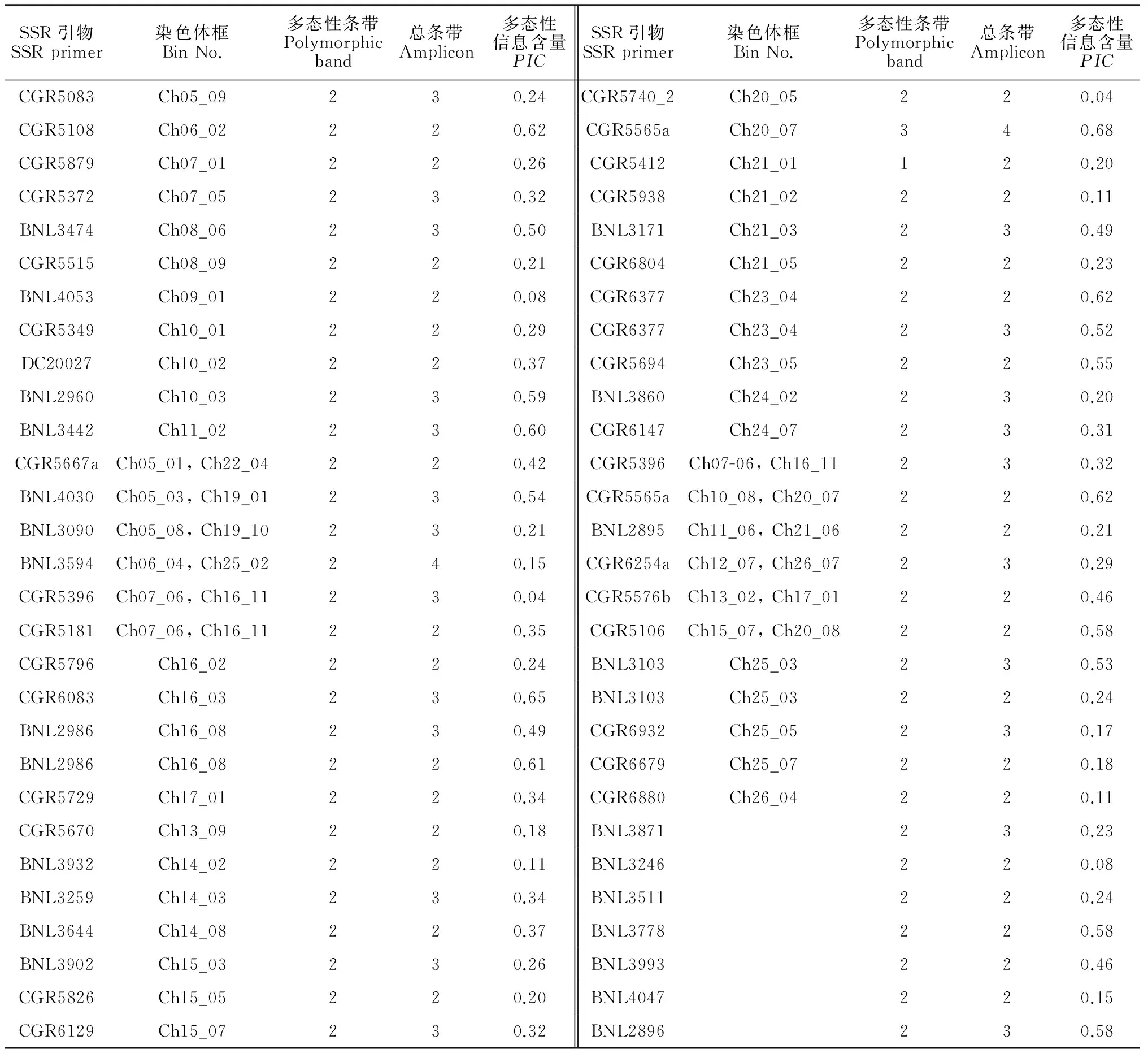
SSR引物SSRprimer染色体框BinNo.多态性条带Polymorphicband总条带Amplicon多态性信息含量PICSSR引物SSRprimer染色体框BinNo.多态性条带Polymorphicband总条带Amplicon多态性信息含量PICCGR5083Ch05_09230.24CGR5740_2Ch20_05220.04CGR5108Ch06_02220.62CGR5565aCh20_07340.68CGR5879Ch07_01220.26CGR5412Ch21_01120.20CGR5372Ch07_05230.32CGR5938Ch21_02220.11BNL3474Ch08_06230.50BNL3171Ch21_03230.49CGR5515Ch08_09220.21CGR6804Ch21_05220.23BNL4053Ch09_01220.08CGR6377Ch23_04220.62CGR5349Ch10_01220.29CGR6377Ch23_04230.52DC20027Ch10_02220.37CGR5694Ch23_05220.55BNL2960Ch10_03230.59BNL3860Ch24_02230.20BNL3442Ch11_02230.60CGR6147Ch24_07230.31CGR5667aCh05_01,Ch22_04220.42CGR5396Ch07-06,Ch16_11230.32BNL4030Ch05_03,Ch19_01230.54CGR5565aCh10_08,Ch20_07220.62BNL3090Ch05_08,Ch19_10230.21BNL2895Ch11_06,Ch21_06220.21BNL3594Ch06_04,Ch25_02240.15CGR6254aCh12_07,Ch26_07230.29CGR5396Ch07_06,Ch16_11230.04CGR5576bCh13_02,Ch17_01220.46CGR5181Ch07_06,Ch16_11220.35CGR5106Ch15_07,Ch20_08220.58CGR5796Ch16_02220.24BNL3103Ch25_03230.53CGR6083Ch16_03230.65BNL3103Ch25_03220.24BNL2986Ch16_08230.49CGR6932Ch25_05230.17BNL2986Ch16_08220.61CGR6679Ch25_07220.18CGR5729Ch17_01220.34CGR6880Ch26_04220.11CGR5670Ch13_09220.18BNL3871230.23BNL3932Ch14_02220.11BNL3246220.08BNL3259Ch14_03230.34BNL3511220.24BNL3644Ch14_08220.37BNL3778220.58BNL3902Ch15_03230.26BNL3993220.46CGR5826Ch15_05220.20BNL4047220.15CGR6129Ch15_07230.32BNL2896230.58
2.2 基于遗传距离的聚类
利用软件NTSYS-pc2.1计算材料两两间的相似系数,54个陆地棉品种之间成对相似系数在0.733 3~0.987 2之间,其平均遗传相似系数为0.898。其中,相似系数小于0.90的为11.1%,相似系数大于0.95的占55.6%,相似系数在0.90~0.95之间的占33.3%,这说明这批品种间遗传相似系数较大,其亲缘关系比较近,遗传基础比较狭窄。
由聚类结果(图1,A)可看出,在相似系数为0.877时,可将该群体分为5大类,第Ⅰ类包括44个品种,第Ⅱ类只有‘陕401’ 1个品种,第Ⅲ类包括7个品种,第Ⅳ类包括‘中棉所15’ 1个品种,最后,由‘晋棉14’独聚一类,为第V类。在相似系数为0.887处又可将第Ⅰ类分为3个亚类,第Ⅰ-1亚类包括了37个品种,第Ⅰ-2亚类包含6个品种,第Ⅰ-3类只有1个品种(陕81-1)。图1,A显示,近67%陆地棉品种间遗传相似系数大于0.90,这说明绝大多数以陕棉抗病种质及其衍生品种间遗传多样性非常低,其亲缘关系很近。
根据聚类分析结果,54个品种中晋棉14单独聚为一类,与其它品种的亲缘关系较远。但是查阅系谱图可知,‘晋棉14’和‘陕81-1’的直接父本都是‘陕1155’,导致这种结果的原因可能是由于在长期育种和种植的过程中‘晋棉14’发生了某些变异,使得‘晋棉14’在聚类时单独成为一类。

A. 54个棉花材料根据Jaccard’s相似系数所得的UPGMA聚类图; B. 同UPGMA聚类图的排列顺序所绘群体遗传结构;C. 根据Q值来排列的群体遗传结构图1 54个陕棉抗病种质及衍生品系的遗传距离聚类(A)和模型聚类(B, C)结果比较A. UPGMA dendrogram based on Jaccard’s similarity coefficients of the 54 cotton accessions; B. Bar plot of the genetic composition of individual lines, based on SSR data by using admixture model in Structure 2.3.1, sorted by accession No in the left UPGMA dendrogram; C. Sort by QFig. 1 Dendrogram of the 54 ShaanResistance cotton collection and derived accession based on distance-based cluster (A) and model-based cluster method (B, C)
2.3 基于数学模型的聚类和遗传结构分析
通过基于数学模型的类群划分可以看出:当组群数(K)在1~10之间时,LnP(D)值持续增大,但当K=4及K>4时,α值趋向稳定,说明马尔可夫链(Markov chain)在K=4时开始收敛(图2)。根据Δk最大值,可以推测K值为4,根据Evanno等[16]描述的方法推断出54份陕棉及衍生品系中存在4大组群,组群A包含11份资源,组群B包含14份资源,组群C包含18份资源,组群D包含11份资源(表3)。
根据群体结构检测结果,利用Structure 2.3.1软件,每份资源以竖直细线条表示(图1,B和C),红(组群A)、绿(组群B)、蓝(组群C)、黄(组群D)4色分别代表组群趋向。组群A以‘陕棉3号’、‘陕棉4号’同‘中棉所3号’杂交衍生的系统为主,也包含‘陕3619’、‘陕3719’、‘陕2021’等血统;组群B以‘陕棉3号’、‘陕棉4号’、‘陕1155’同‘徐州1818’、‘中长1号’杂交衍生的系统为主,组群C以‘陕棉3号’、‘陕棉4号’、‘陕401’同‘徐州209’、‘彭泽1’杂交衍生的系统为主,组群D以‘陕棉6号’、‘陕1155’同‘中棉所3号’杂交衍生的系统为主。图中显示,红色线条中几乎全部含有‘陕棉3号’、‘陕棉4号’和‘中棉所3号’的血统;绿色线条在所有的品种中都有大量分布,但在‘陕1155’、‘陕棉3号’、‘陕棉4号’同‘徐州1818’的衍生品系中占比例最大;‘陕棉3号’、‘陕棉4号’、‘陕401’同‘徐州209’、‘彭泽1’杂交衍生的系统中蓝色占绝对优势。图中信息表明,54份陕棉抗病种质资源间分界十分明显,属于截然不同的组群,其组群划分取决于育成亲本以及地域来源;以中棉所品种或以其为材料与陕棉品种所育成的衍生品种另形成一个组群。
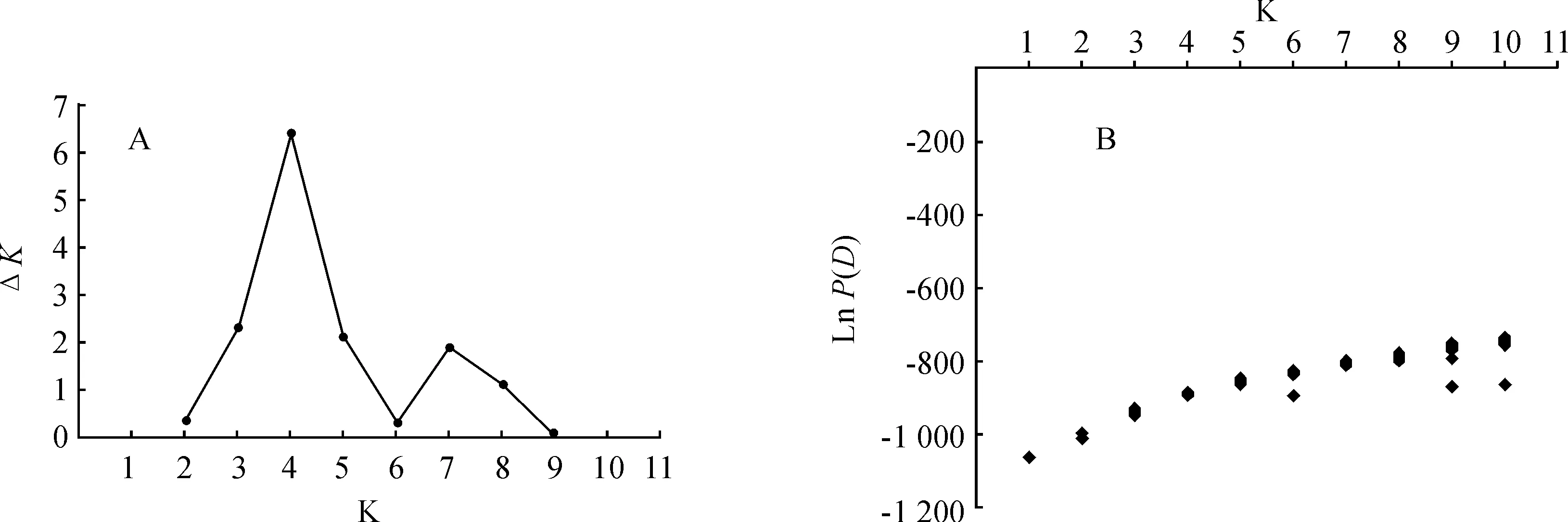
A.ΔK的变化曲线; B. 20次重复的Ln P(D)变化图图2 陕棉抗病种质及衍生品系的最适群体结构(K值)估测A. Change curve in the log probability of data between successive K-value (ΔK); B. Mean Ln P(D) (±SD) over 20 runs for each K valueFig. 2 Calculation of true K of ShaanResistance cotton collection and derived accessions
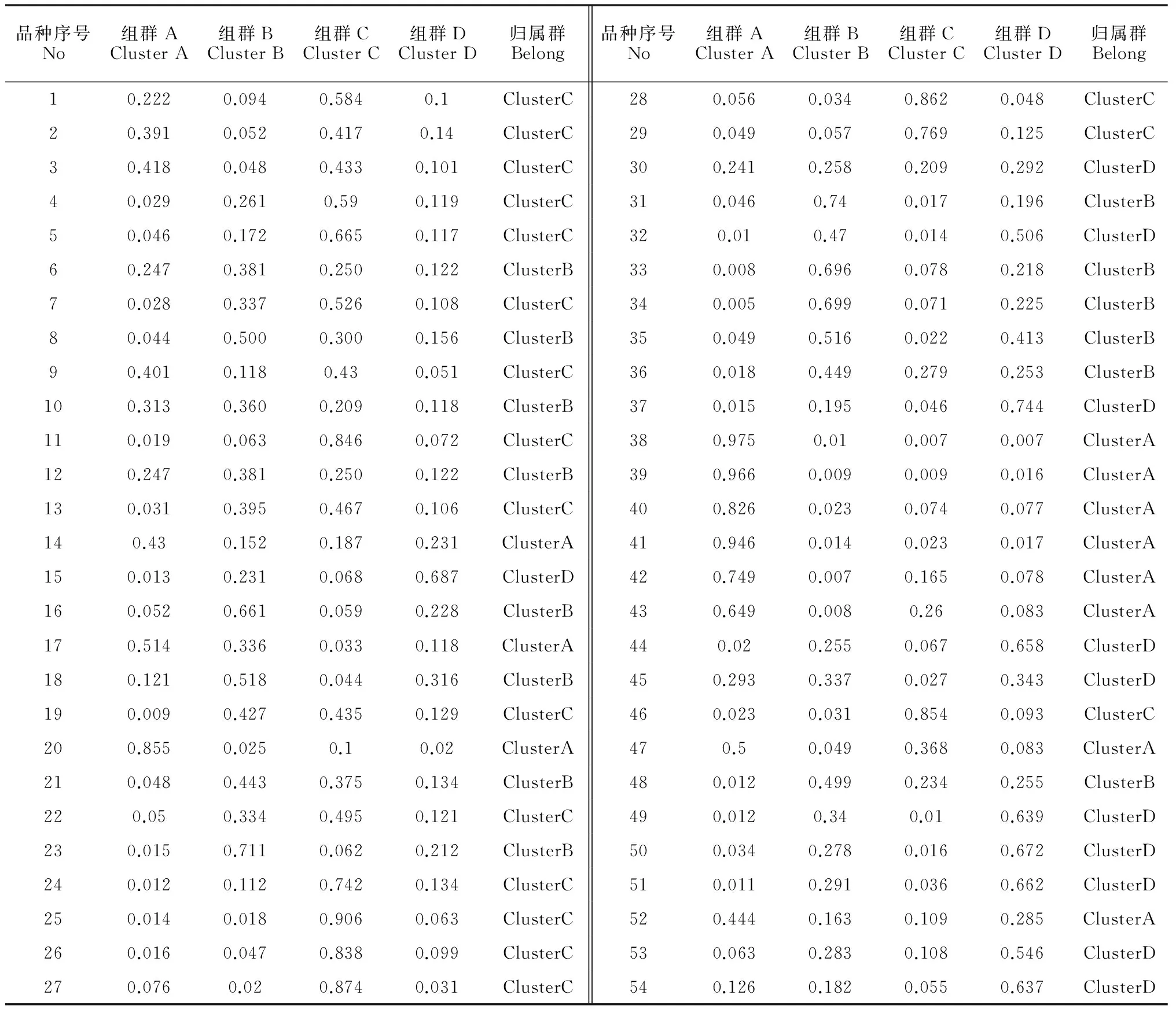
品种序号No组群AClusterA组群BClusterB组群CClusterC组群DClusterD归属群Belong品种序号No组群AClusterA组群BClusterB组群CClusterC组群DClusterD归属群Belong10.2220.0940.5840.1ClusterC280.0560.0340.8620.048ClusterC20.3910.0520.4170.14ClusterC290.0490.0570.7690.125ClusterC30.4180.0480.4330.101ClusterC300.2410.2580.2090.292ClusterD40.0290.2610.590.119ClusterC310.0460.740.0170.196ClusterB50.0460.1720.6650.117ClusterC320.010.470.0140.506ClusterD60.2470.3810.2500.122ClusterB330.0080.6960.0780.218ClusterB70.0280.3370.5260.108ClusterC340.0050.6990.0710.225ClusterB80.0440.5000.3000.156ClusterB350.0490.5160.0220.413ClusterB90.4010.1180.430.051ClusterC360.0180.4490.2790.253ClusterB100.3130.3600.2090.118ClusterB370.0150.1950.0460.744ClusterD110.0190.0630.8460.072ClusterC380.9750.010.0070.007ClusterA120.2470.3810.2500.122ClusterB390.9660.0090.0090.016ClusterA130.0310.3950.4670.106ClusterC400.8260.0230.0740.077ClusterA140.430.1520.1870.231ClusterA410.9460.0140.0230.017ClusterA150.0130.2310.0680.687ClusterD420.7490.0070.1650.078ClusterA160.0520.6610.0590.228ClusterB430.6490.0080.260.083ClusterA170.5140.3360.0330.118ClusterA440.020.2550.0670.658ClusterD180.1210.5180.0440.316ClusterB450.2930.3370.0270.343ClusterD190.0090.4270.4350.129ClusterC460.0230.0310.8540.093ClusterC200.8550.0250.10.02ClusterA470.50.0490.3680.083ClusterA210.0480.4430.3750.134ClusterB480.0120.4990.2340.255ClusterB220.050.3340.4950.121ClusterC490.0120.340.010.639ClusterD230.0150.7110.0620.212ClusterB500.0340.2780.0160.672ClusterD240.0120.1120.7420.134ClusterC510.0110.2910.0360.662ClusterD250.0140.0180.9060.063ClusterC520.4440.1630.1090.285ClusterA260.0160.0470.8380.099ClusterC530.0630.2830.1080.546ClusterD270.0760.020.8740.031ClusterC540.1260.1820.0550.637ClusterD
2.4 遗传距离聚类和模型聚类结果差异比较
对比图1,A、B和C发现, 2种聚类方法划分的类群存在一定差异, 但模型聚类法能将遗传距离聚类中类群归属不明确的5个品种(‘豫棉2号’、‘中棉所15’、‘晋棉14’、‘陕81-1’和‘陕401’)明确划分到相应类群。2种聚类方法中有14个品种的类群归属不一致,其中,7个品种在距离聚类中都属于第I类,而在模型聚类中根据最大Q值则分别属于组群A(‘辽棉10’和‘冀棉11号’)、组群B(‘冀合321’、‘盐棉48’、‘冀棉3号’和‘鲁1024’)和组群D(‘中6331’)。4个品种(‘豫棉8号’、‘陕3563’、‘泗棉3号’和‘豫棉2号’)在距离聚类中都属于第Ⅱ类,而在模型聚类中根据最大Q值则分别属于组群C、组群A、组群B、组群A群。可见,第Ⅱ类群的品种易在2种聚类结果中发生类群归属变动。
3 讨 论
陕棉抗病种质因其抗性强,综合性状优良,在中国枯、黄萎病流行的棉区广为种植,直接控制了病害的爆发。另外,因抗病性遗传力高,陕棉抗病种质被国内各育种单位用作抗源亲本,培育出一系列的突破性品种,如‘中棉所17’、‘中棉所19’、‘86-1’等。为挖掘陕棉抗病种质及其衍生品种的特点,为今后更充分地利用陕棉抗病种质以及新抗源的创制,本研究选择了陕棉抗病种质及其衍生品种作为研究对象,利用SSR标记研究其遗传多样性及其群体结构。从已发表的遗传图谱上选择了72对均匀分布于全基因组上的SSR标记,可以初步探析54份棉花品种间在全基因组水平上的差异和多样性。通过SSR分析,共扩增出276条SSR条带,其中多态性条带176条,多态性位点占63.8%。PIC为0.04~0.68,遗传相似系数为0.733 3~0.987 2。这些均说明了带有陕西棉花血统的陕棉抗病种质及其衍生品种间遗传多样性很低,遗传基础比较狭窄。
国内外利用分子标记对棉花的遗传多样性进行了诸多研究,均显示遗传基础狭窄。这主要是因为在育种过程中,仅少数材料被用作杂交育种的亲本,其他材料因为某些性状不佳,从而被育种者所舍弃,从而导致遗传基础越来越狭窄。本研究所选的54份材料,一部分是在陕西的生态条件和病圃压力下培育而成(陕棉抗病种质),另一部分是以这些陕棉抗病种质为育种亲本在其他生态区衍生(培育)而来。这些材料在抗病性、产量、品质或其他性状上均有优势且各不相同,在相应的生态区曾为主栽品种,大幅度提高了棉花产业的经济效益。但回溯这些材料的亲本来源,它们均含有自美国引进的斯字棉、岱字棉、德字棉等品种的血统,同时又经历了陕西的生态条件的筛选,所以遗传基础越来越低。
总的来看,54个棉花品种DNA标记聚类分析与系谱分析结果有一定的吻合性。例如系谱显示‘豫棉1号’和‘川73-21’都是以‘陕棉4号’为亲本选育而成,这也与聚类结果中‘豫棉1号’与‘川73-21’聚为一类相吻合;同时,‘江苏棉1号’与‘盐棉48’的聚类结果也与系谱记录相一致。这表明:这类品种聚类的主要决定因素是其亲缘关系的远近,而非其它生态环境或外部条件,亲缘关系近,其遗传相似系数也比较大,则会较早地聚为一类。但是,也有很大一部分品种的聚类结果与系谱显示不一致,例如‘鲁棉研16’与‘绵阳83-21’在聚类中很早就聚为一类,但是查阅系谱可知‘鲁棉研16’与‘绵阳83-21’的亲缘关系较远,而同是来源于‘中棉所3号’的‘豫抗1号’和‘渤棉2号’在聚类上却并没有聚为一类,‘渤棉2号’属于第I类,而‘豫抗1号’则聚在第Ⅲ类。造成这种差异可能是由于广泛而频繁引种和杂交育种过程中的生物学混杂,导致各地种质资源类似并相互融合,克服了地域的限制性,使得各地品种群体的遗传变异变小,亲缘关系越来越近,遗传基础也变得越来越狭窄。因此在今后的育种过程中亲本选择不能只依靠系谱,而应该在系谱的基础上再通过分子鉴定,选择出遗传多样性高,遗传关系较远的品种作为杂交亲本。
要扩宽枯、黄萎病抗性种质的遗传基础,丰富陕西棉花枯、黄萎病抗源种质基因库,就需在今后育种过程中引入更多的亲缘关系较远的种质资源,特别是对野生棉种质资源的引入,使得遗传基因库更加丰富,以克服抗病品种间遗传关系较近的不足,进一步为棉花育种提供新的抗性资源。
[1] 邢宏宜, 贺道华, 易永华, 等.陕西棉花抗枯黄萎病种质资源评价[J].中国农学通报, 2008, 24(10): 222-227.
XING H Y, HE D H, YI Y H,etal. The evaluation of germplasms with resistance toFusariumwilt andVerticilliumwilt from Shaanxi cotton section [J].ChineseAgriculturalScienceBulletin, 2008, 24(10): 222-227.
[2] 黄滋康.中国棉花品种及其系谱[M]. 北京:中国农业出版社,1994: 215-286.
[3] 杜雄明,刘国强. 中国棉花品种志(1978~2007)[M]. 北京:中国农业科学技术出版社,2009: 273-277.
[4] 王省芬, 张桂寅, 李喜焕, 等. 黄河、长江流域棉区棉花抗病品种的AFLP分析[J]. 遗传学报, 2004, 31(12): 1 426-1 433.
WANG X F, ZHANG G Y, LI X H,etal. AFLP analysis of cotton withFusariumandVerticilliumwilts from the Huanghe and Changjiang valleys [J].ActaGeneticaSinica, 2004, 31(12): 1 426-1 433.
[5] 王省芬, 马峙英, 张桂寅, 等. 我国棉花抗枯、黄萎病骨干品种(系)基于AFLP的遗传多样性[J]. 棉花学报, 2005, 17(1): 23-28.
WANG X F, MA Z Y, ZHANG G Y,etal. Genetic diversity of Chinese key cottons withFusariumandVerticilliumwilts resistance based upon AFLPs [J].CottonScience, 2005, 17(1): 23-28.
[6] 徐秋华, 张献龙, 聂以春, 等. 我国棉花抗枯萎病品种的遗传多样性分析[J]. 中国农业科学, 2002, 35(3): 272-276.
XU Q H, ZHANG X L, NIE Y C,etal. Genetic diversity evaluation of cultivars (G.hirsumtumL.) resistant toFusariumwilt by RAPD markers [J].ScientiaAgriculturaSinica, 2002, 35(3): 272-276.
[7] 郭旺珍, 周兆华, 张天真, 等. RAPD鉴定棉花抗耐黄萎病品种系的遗传变异研究[J]. 江苏农业学报, 1999, 15(1): 1-6.
GUO W Z, ZHOU Z H, ZHANG T Z,etal. Detection of genetic variation of Upland cotton cultivars (lines) resistant (tolerant) toVerticilliumdahliae with RAPD technique[J].JiangsuJournalofAgriculturalSciences, 1999, 15(1): 1-6.
[8] 温小杰,马峙英,王省芬,等. 中国抗枯、黄萎病陆地棉材料分子水平的遗传差异评价[J]. 中国农业科学, 2005, 38(5): 936-943.
WEN X J, MA Z Y, WANG X F,etal. Molecular divergence estimation of Chinese Upland cottons withFusariumandVerticilliumwilt resistance [J].ScientiaAgriculturaSinica, 2005, 38(5): 936-943.
[9] 刘文欣,孔繁玲,郭志丽,等.建国以来我国棉花品种遗传基础的分子标记分析[J]. 遗传学报,2003,30(6): 560-570.
LIU W X, KONG F L, GUO Z L,etal. An analysis about genetic basis of cotton cultivars in China since 1949 with molecular markers [J].ActaGeneticaSinica, 2003, 30(6): 560-570.
[10] 贺道华,邢宏宜,李婷婷,等. 92份棉花资源遗传多样性的SSR分析[J]. 西北植物学报, 2010, 30(8): 1 557-1 564.
HE D H, XING H Y, LI T T,etal. Genetic diversity of 92 cotton accessions evaluated with SSR markers [J].ActaBotanicaBoreali-OccidentaliaSinica, 2010, 30(8): 1 557-1 564.
[11] PATERSON A H. A rapid method for extraction of cotton (Gossypiumspp.) genomic DNA suitable for RFLP or PCR analysis [J].PlantMolecularBiologyReport, 1993, 11(2): 122-127.
[12] HE D H, LIN Z X, ZHANG X L,etal. QTL mapping for economic traits based on a dense genetic map of cotton with PCR-based markers using the interspecific cross ofGossypiumhirsutum×G.barbadens[J].Euphytica, 2007, 153(1-2): 181-197.
[13] XIAO J, WU K, FANG D D,etal. New SSR markers for use in cotton (Gossypiumssp.) improvement [J].TheJournalofCottonScience, 2009, 13(2): 75-175.
[14] ROHLF F J. In: NTSYS-pc numerical taxonomy and multivariate analysis system [M]. Version 1.8 Exter publication, New York: 1990.
[15] PRITCHARD J K, WEN X, FALUSH D. Documentation for structure software: version 2.3 [EB/OL]. (2009-01-21) [2010-01-25]. http://pritch.bsd.uchicago.edu/structure.html.
[16] EVANNO G, REGNAUT S, GOUDET J. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study [J].MolecularEcology, 2005, 14: 2 611-2 620.
(编辑:宋亚珍)
Genetic Diversity and Population Structure of Disease-Resistance Cotton Cultivars Developed in Shaanxi and Their Descendants by Mapped SSRs
XING Zenong1, 2, JIANG Lei2, HE Daohua2*, XING Hongyi2, LEI Zhongping3
(1 Baoji Institute of Agricultural Sciences,Qishan,Shaanxi 722400,China; 2 College of Agronomy,Northwest A&F University,Yangling,Shaanxi 712100,China; 3 College of Life Sciences,Northwest A&F University,Yangling,Shaanxi 712100,China)
Fifty-four accessions, including disease-resistance cotton lines developed in Shaanxi and their descendants, were genotyped to evaluate their genetic diversity and population structure by 72 SSR markers spanning the whole genome of cotton. (1) The genetic similarity coefficient of 54 accessions ranged from 0.733 3 to 0.987 2. Among 1 431 similarity coefficient of pairwise accessions, 11.1% of the similarity coefficient was less than 0.90, 55.6% of the similarity coefficient was more than 0.95. (2) The polymorphic information content (PIC) varied from 0.04 to 0.68 with an average of 0.33. (3) A genetic-distance-based UPGMA cluster analysis revealed that all the accessions could be divided into five groups at cut-off value of 0.877 in the genetic similarity coefficient. Group I included 44 accessions, Group Ⅲ included 7 accessions, and each of the remained three groups included one accession, respectively. (4) A model-based clustering method (implemented by the program STRUCTURE) showed that all the accessions originated from four clusters. This study indicated that the cotton population, which was comprised of disease-resistance accessions developed in Shaanxi and their descendants, was characterized by high genetic similarity coefficient, narrow genetic base (genomic ancestry) and poor genetic diversity.
cotton; disease-resistance germplasm; SSR; genetic diversity; population structure
1000-4025(2016)08-1551-09
10.7606/j.issn.1000-4025.2016.08.1551
2016-04-05;修改稿收到日期:2016-07-08
现代农业产业技术体系建设专项资金(CARS-18-45);转基因生物新品种培育科技重大专项(2013ZX08005-002)
邢泽农(1988-),男,助理农艺师,硕士,宝鸡市农业科学研究所工作。E-mail: xznong@126.com
*通信作者:贺道华,讲师,主要从事棉花生物技术与育种研究。E-mail: daohuahe@nwafu.edu.cn
Q346+.5; Q789
A
