五个湖泊中华刺鳅(Mastacembelus sinensis)种群线粒体细胞色素b遗传多样性分析*
2016-10-12赵子明叶建生刘美剑沈冬冬黄世熠
赵子明,叶建生,刘美剑,沈冬冬,黄世熠
(江苏农牧科技职业学院,泰州 225300)
五个湖泊中华刺鳅(Mastacembelus sinensis)种群线粒体细胞色素b遗传多样性分析*
赵子明,叶建生,刘美剑,沈冬冬,黄世熠
(江苏农牧科技职业学院,泰州 225300)
采用特异性引物对中华刺鳅基因组DNA进行扩增和测序,获得了江苏省5个湖泊的中华刺鳅种群线粒体cyt b基因767bp的序列. 分析表明,cyt b基因中A+T含量略高于G+C含量,144个中华刺鳅共获得cyt b基因单倍型28个,变异位点38个,其中单倍型Hap1分布最为广泛,可能为祖先型单倍型. 平均单倍型多样性和平均核苷酸多样性指数分别为0.58751和0.00214,遗传多样性较低. 群体间遗传分化系数为 0.0356,绝大多数的遗传变异都来自群体内部,而群体间的遗传分化极小. 构建的系统进化树未出现明显的以单个湖泊群体的聚簇,5个湖泊的种群间基因交流极其强烈. 研究结果表明,当前5个湖泊的中华刺鳅遗传多样性水平比较低,亟待加强保护.
中华刺鳅;线粒体DNA;cyt b;遗传多样性;淡水湖
中华刺鳅(Mastacembelus sinensis)属于鲈形目(Perciformes)、刺鳅科(Mastacembelidae)、刺楸属(Mastacembelus),俗称石铲、刀鳅、钢鳅等,是广泛分布我国辽河、黄河、长江、钱塘江、珠江等水系的中小型淡水鱼类[1]. 改革开放以来,由于人为活动和江河环境污染,特别是农作物秸秆向水体中抛置,导致水体污染严重,中华刺鳅野生资受到严重破坏. 目前,中华刺鳅资源量不断下降.
cyt b基因作为常用线粒体DNA分子标记,具有较强的中间解析和鉴别能力,广泛应用于动物的群体遗传结构、物种鉴别和系统进化的研究中[2-5]. 近年来,cyt b基因也越来越多地应用于鱼类遗传多样性研究. 赵凯等[6]分析了黄河上游和柴达木盆地托索湖的裸裂尻鱼cyt b基因序列变异,初步认为黄河和柴达木水系托索湖的裸裂尻鱼未显著分化,两种群表现出较低水平的遗传多样性. 阎雪岚等[7]采用cyt b序列分析了采自新疆克孜河3个群体的塔里木裂腹鱼及1个斑重唇鱼群体的种群遗传多样性和遗传结构,结果显示克孜河的塔里木裂腹鱼和斑重唇鱼均处于很低的遗传多样性水平,物种维持力较弱. 赵峰等[8]采用cyt b基因片段为遗传标记,对黄海南部和东海的6个银鲳群体遗传结构进行了分析,结果表明6个群体均呈现出高单倍型多样性和低核苷酸多样性的特点,群体间具有高度的基因交流,是一个随机交配群体. 田辉伍等[9]则利用cyt b基因研究了长江上游的长江干流、岷江、赤水河和嘉陵江的8个群体长薄鳅的遗传结构,表明长薄鳅种群内的变异大于种群间的变异,种群基因交流十分频繁. 目前,有关刺鳅的研究很少,仅有的几篇报道主要集中于基础生物学[10-15]、病害研究[16]以及分类地位讨论[17-22]. 尤其是其分类地位一直存在争议,存在刺鳅(Mastacembelus aculeatus)和中华光盖刺鳅(Pararhynchobdlla sinensis)等多种学名,倪勇与伍汉霖先生[1]根据其形态特征比对,最终确定了其分类地位,学名为中华刺鳅(Mastacembelus sinensis). 本研究对采自江苏省5个湖泊中华刺鳅种群cyt b片段进行了测序与比较分析,研究5个湖泊中华刺鳅群体间的遗传多样性和遗传结构,以深入了解和掌握5个湖泊中华刺鳅种群当前的种质资源现状,为其今后的保护与开发提供基础分子生物学依据.
1 材料和方法
1.1 研究材料
实验用中华刺鳅于2015年5-7月采集自江苏省苏中和苏北地区的5个湖泊水域,分别为江苏淮安的白马湖(B)、高邮湖(G)、洪泽湖(H)、骆马湖(L)以及江苏泰州的溱潼湖(Q),剪取尾鳍,无水乙醇固定后,-20℃ 冷冻保存备用.
1.2 DNA提取
基因组DNA采用试剂盒提取(SangonEzupColumnAnimalGenomicDNAPurificationKit). 总DNA经检测后稀释至100ng/μl,-20℃冷冻保存备用.
1.3 PCR扩增及测序
参照GenBank数据库中大刺鳅(Mastacembelus armatus)的线粒体全序列(登入号:KJ184553),设计引物扩增cyt b 基因,引物序列为F:5’-ATCCCTCCTGGGACTCTGTC-3’;R:5’-CGTGTGGAGGATTGGGACAA-3’,引物由上海生工生物公司合成.PCR反应体系为50μl:模板DNA50ng;2×PCRMix25μl(包含Taq酶 2.5U,dNTPs10μmol,MgCl20.1mmol),上、下游引物(10μmol/L)各1μl,其余体积用水补足. 反应程序为:94℃预变性2min,94℃变性45s,62℃退火1min,72℃延伸1min,经35个循环后再72℃延伸10min.PCR产物经割胶纯化后,直接送上海生工生物公司利用正反引物进行双向测序.
1.4 数据分析
根据测序峰图对双向测序序列进行人工拼接,之后用BioEdit7.0.5[23]软件比对获得同源序列,MEGA6.0软件[24]分析碱基的组成,并构建系统进化树.DnaSP软件[25]统计变异位点类型与数目,计算单倍型种类、平均核苷酸差异数、单倍型多样性指数和核苷酸多态性等,用Arlequin3.01 软件[26]中的Kimura2-Parameters方法构建5个种群间的遗传距离,分子生物学方差分析(AMOVA)方法计算遗传分化指数(Fst)及遗传变异组成.
2 结果
2.1 序列分析
5个湖泊中华刺鳅种群144个个体的cyt b基因,经拼接比对后,获得长度为 767bp的同源序列.MEGA6软件分析表明,T、C、A、G碱基组成平均为29.2%、30.1%、26.1%和14.6%,A+T含量为 55.3%,略高于G+C含量. 序列共发现多态性位点38个,占核苷酸总数的4.95%,其中包含18个单态突变位点和20个简约信息位点,未检测到碱基缺失. 144个个体中共检出28个单倍型序列,所有单倍型序列已提交至GeneBank(登入号:KT448786~KT448813),种群间共享单倍型共8个,其中单倍型Hap1 5个群体间都有分布且数量也大,可能是原始单倍型(表1). 5个种群的单倍型平均多样性指数为0.58751,平均核苷酸差异数为1.63947,平均核苷酸多样性指数为0.00214(表2). 5个湖泊的中华刺鳅群体中,高邮湖群体的单倍型多样性指数最高,溱潼湖群体的核苷酸多样性指数最高,而骆马湖群体的单倍型多样性指数和核苷酸多样性指数均为最低.

表2 5个湖泊的中华刺鳅种群的单倍型及遗传多样性参数Tab.2 Haplotypes and genetic diversity of spiny eel populations from five lakes
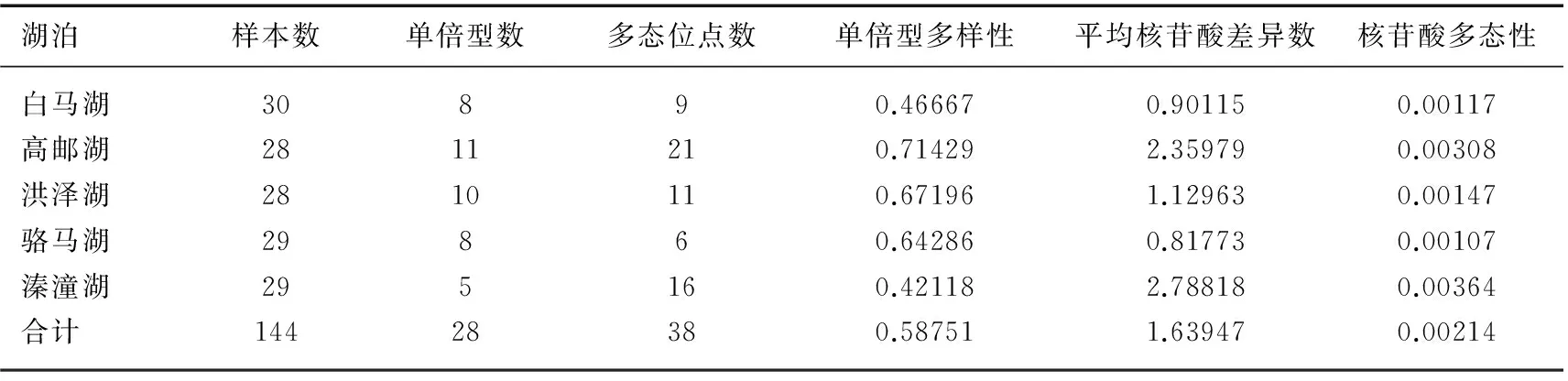
湖泊样本数单倍型数多态位点数单倍型多样性平均核苷酸差异数核苷酸多态性白马湖30890.466670.901150.00117高邮湖2811210.714292.359790.00308洪泽湖2810110.671961.129630.00147骆马湖29860.642860.817730.00107溱潼湖295160.421182.788180.00364合计14428380.587511.639470.00214
2.2 群体变异和遗传结构
将测序获得的144个个体的cyt b基因序列输入Arlequin软件,使用Kimura2-Parameters方法构建5个湖泊种群间的相对遗传距离和Fst(表3). 5个种群的平均遗传距离为0.00216,高邮湖种群与溱潼湖种群间的遗传距离最远为0.00338,遗传距离最近的是白马湖和骆马湖种群之间为0.00116. 而5个湖泊刺鳅种群内部遗传距离从大到小依次为:溱潼湖(0.00368)>高邮湖(0.00311)>洪泽湖(0.00148)>白马湖(0.00118)>骆马湖 (0.00107). 5个种群的平均Fst为0.03556,5个湖泊的中华刺鳅种群间存在一定的遗传分化,但是种群间的Fst值均较低,种群间Fst显著性检验有近一半(P>0.05),表明5个湖泊中华刺鳅种群间存在较大的基因流,遗传分化差异较低(表4).
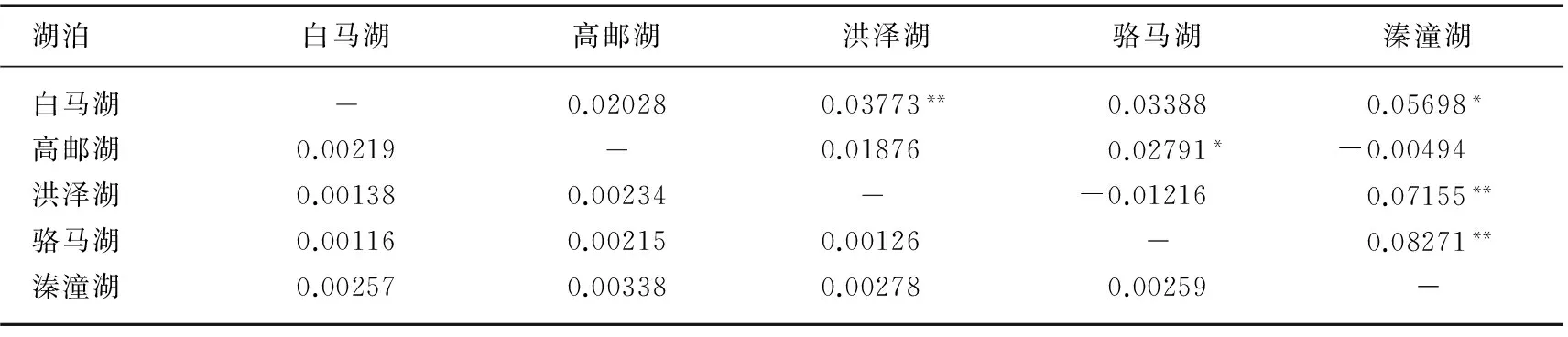
表3 5个湖泊的中华刺鳅种群间K 2-P遗传距离和遗传分化系数Tab.3 Average K 2-P distance and fixation indice between spiny eel populations from five lakes
*表示差异显著(P<0.05);**表示差异极显著(P<0.01);对角线上方数值表示种内分化系数;对角线下方数值表示群体间遗传距离.
表4 5个湖泊中华刺鳅种群间遗传差异的AMOVA分析Tab.4 Analysis of AMOVA of spiny eelpopulations from five lakes
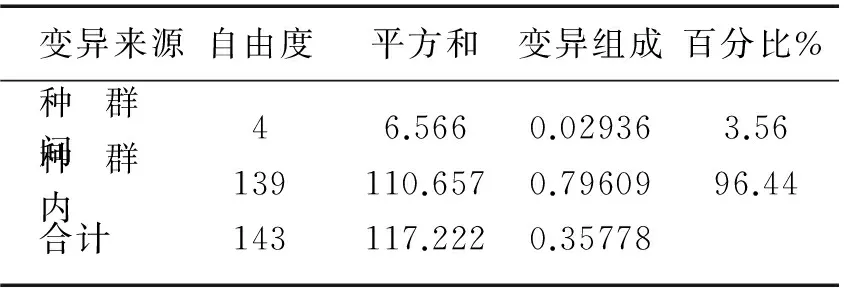
变异来源自由度平方和变异组成百分比%种群间46.5660.029363.56种群内139110.6570.7960996.44合计143117.2220.35778
AMOVA法估算5个群体间遗传变异结构和来源的结果表明,种群间的变异占总变异的3.56%,而种群内的变异对总变异的贡献率达到96.44%(表4),种群内未出现遗传分化. 另外,用DnaSP软件分析单倍型数据所得遗传分化系数(Gst)为0.01092,基因流(Nm)为22.65;而144个个体的Gst为0.05601,Nm为4.21,也表明种群间的基因交流很频繁.
2.3 系统进化分析
将144个中华刺鳅个体的cyt b基因序列输入MEGA6软件,对5个湖泊群体进行系统进化分析,得到UPGMA系统进化树(图1A). 5个中华刺鳅群体没有明显聚类,形成了单系类群,京杭大运河水系连接的白马湖、骆马湖、洪泽湖和高邮湖群体首先聚类,最后与溱潼湖聚合. 而144个个体的构建的UPGMA系统进化树则表明,5个群体的个体都没有独立聚类,相互散布在不同的地理群体中(图1B).
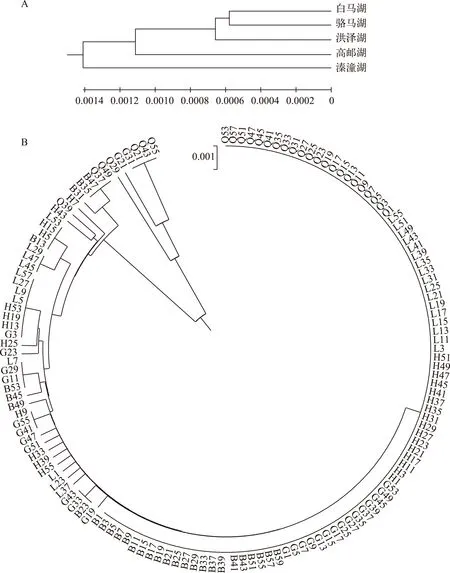
图1 5个湖泊中华刺鳅群体间(A)及个体间(B)的UPGMA系统进化树Fig.1 Dendrogram of spiny eel populations (A) and 144 individuals (B) from five lakes by UPGMA method based genetic distance
2.4 群体历史动态
用Fu’Fs检验方法检验5个湖泊中华刺鳅群体的中性突变,结果表明种群的整体Fs=-23.887(P<0.05),显著偏离中性突变,表明5个中华刺鳅群体整体上有一定的扩张趋势,但白马湖、骆马湖、高邮湖和洪泽湖群体的Fs值都小于0,而溱潼湖群体的Fs值大于0. 所有个体的线粒体cyt b基因序列进行碱基歧点分布分析,个体间碱基歧点分布的观测曲线与期望曲线大体一致,中间只有一个较低的凸起(图2),表明5个湖泊的中华刺鳅历史上种群相对比较稳定,经历过一个比较小的种群扩张事件.
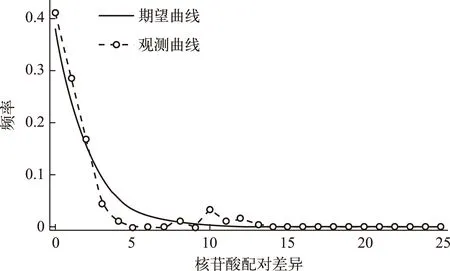
图2 5个湖泊的中华刺鳅群体线粒体cyt b基因错配碱基分布Fig.2 The mismatch distribution of the Mitochondrial DNA cyt b of spiny eel populations from five lakes
3 讨论
3.1 群体遗传多样性
本研究获得了5个湖泊中华刺鳅群体的线粒体 cyt b 基因片段,序列碱基组分析表明,中华刺鳅线粒体cyt b 基因的A+T含量为55.3%,略高于G+C平均含量,而G含量显著低于其他碱基,表现出反G偏倚,与其他动物线粒体DNA的研究结果一致[27].
遗传多样性是一个物种长期生存和进化的前提,物种的遗传多样性越高,该物种的进化潜力也就越大,对环境变化的适应能力也就越强[5]. 本研究获得5个湖泊144 尾中华刺鳅cyt b基因767bp的序列,共获得基因单倍型28个,变异位点38个,单倍型多样性和核苷酸多样性分别为0.58751 和0.00214,说明江苏省5个湖泊的中华刺鳅单倍型多样性和核苷酸多样性都很低,远低于黄颡鱼[4](0.945、0.00419)和福建近海竹荚鱼[28](0.937、0.336)等经济鱼类的cyt b基因单倍型多样性和核苷酸多样性,甚至低于野生濒危物种鱇浪白鱼[5](0.996、0.00434) cyt b基因的单倍型多样性和核苷酸多样性,仅与濒危物种青海湖裸鲤[29](0.783、0.00205)、黄河裸体裂尻[6](0.700、0.0026)和长薄鳅[9](0.60852、0.00089) cyt b基因的单倍型多样性和核苷酸多样性相当,高于怒江角鱼[30](0.579、0.00070) cyt b基因单倍型多样性和核苷酸多样性. 如此低的遗传多样性,可能意味着中华刺鳅种群曾经历过“瓶颈效应”的打击[31],说明当前该物种的进化潜力比较低,对环境变化的适应能力很弱,存在着较大的灭绝风险.
3.2 种群遗传结构
AMOVA分析表明,种群内的遗传变异显著超过各种群间的遗传变异,占全部遗传变异的96.44%,这表明中华刺鳅群体间差异不显著,群体间未出现遗传分化.
Fst值可以反映群体之间的遗传分化程度,当Fst值介于 0~0.05时表示分化较弱. 本文中5个湖泊中华刺鳅群体的Fst值为0.0356,说明5个中华刺鳅种群间存遗传分化较弱,不存在显著的系统地理分化.Shaklee等[32]根据已发表的文献认为鱼类在种群、种和属三级水平上的遗传距离D值分别为0.05、0.30和0.9. 中华刺鳅5个湖泊群体间遗传距离为0.00216,说明中华刺鳅群体间的遗传分化极低,远没有达到种群的分化水平. 另外,与其他鱼类群体间的遗传距离相比较(0.035~0.019)[33],中华刺鳅群体间遗传距离(0.00116~0.00338)可以看出,中华刺鳅种群无论是群体间还是群体内部序列差异都极小,说明其遗传多样性水平极低.Wright提出种群Nm>4时,群体就是一个随机的单位[34]. 而本研究中单倍型间的Nm高达22.65,个体间的Nm为4.21,表明5个群体间基因流动的相当强烈,也进一步说明中华刺鳅种群间未出现分化,应作为一个整体进行资源保护. 系统进化分析表明5个湖泊的中华刺鳅群体没有明显聚类,形成了单系类群. 以上结果都表明中华刺鳅5个湖泊群体在cyt b水平上未出现群体遗传分化.Grant等依据单倍型多样度h 和核苷酸多样度π推测了鱼类群体的4 种进化情景[35],本研究中5个湖泊的中华刺鳅种群遗传多样性低,遗传没有分化,接近于低h、低π情形,可能因为湖泊间地理距离近,水系连通,使得基因交流的可能性很大;同时,也可能存在建立者效应,即5个湖泊的中华刺鳅群体都源于单一建立者;此外,江河等水体环境污染和人为活动导致栖息地减少,致使中华刺鳅群体数量急剧下降,而群体变小后,瓶颈效应和遗传漂变可能在某种程度上导致其遗传多样性的降低,损失的遗传变异也会增多.
遗传多样性是生命进化的基础,遗传多样性的缺失会威胁到种群的生存. 本研究以cyt b基因为工具,研究了5个湖泊中华刺鳅群体间的遗传多样性和遗传结构,以深入了解和掌握当前中华刺鳅种质资源现状,为其今后的保护与开发提供基础分子生物学依据. 5个湖泊的中华刺鳅种群处于较低的遗传多样性水平,且未形成遗传分化,物种维持力较弱. 本研究结果表明当前5个湖泊的中华刺鳅资源现状不容乐观,亟待加强保护. 种群一旦遭受破坏,就很难以复,这个宝贵物种也将面临灭绝的危险. 同时,应尽快对其他水域中华刺鳅遗传多样性现状进行研究,全面了解该物种种质资源现状.
[1]NiYong,WuHanlineds.FishesofJiangsuProvince.Beijing:ChinaAgriculturePress, 2006: 740-742(inChinese).[倪勇, 伍汉霖. 江苏鱼类志. 北京: 中国农业出版社, 2006: 740-742.]
[2]BrownWM.EvolutionofanimalmitochondrialDNA.In:NeiM,KoehnRKeds.Evolutionofgenesandproteins.SunderlandMA:Sinauer, 1983: 62-88.
[3]SacconeC,PesoleG,SbisaE.ThemainregulatoryregionofmammalianmitochondrialDNA:structure-functionmodelandevolutionarypattern. J Mol Evol, 1991, 33(1): 83-91.
[4]ZhongLiqiang,LiuPengpeng,PanJianlinet al.Geneticvariationanalysisofyellowcatfish(Pelteobagrus fulvidraco)fromfivelakesinthemiddleandlowerreachesoftheYangtzeRiverbasedonMitochondrialDNAcytb. J Lake Sci, 2013, 25(2): 302-308(inChinesewithEnglishabstract).DOI::10.18307/2013.0219. [钟立强, 刘朋朋, 潘建林等. 长江中下游5个湖泊黄颡鱼种群线粒体细胞色素b基因的遗传变异分析. 湖泊科学, 2013, 25(2): 302-308.]
[5]LiuHongyan,XiongFei,YangDonget al.MitochondrialcytochromebgenesequencediversityinwildandculturedpopulationsofAnabarilius grahami. J Huazhong Agric Univ, 2011, 30(1): 94-98(inChinesewithEnglishabstract).[刘红艳, 熊飞, 杨东等. 基于细胞色素b基因的鱇浪白鱼野生群体和养殖群体遗传多样性分析. 华中农业大学学报, 2011, 30(1): 94-98.]
[6]ZhaoKai,YangGongshe,LiJunbinget al.PhylogeneticstructureofSchizopygopsis pylzovipopulationsfromMitochondrialcytochromebgenesequencevariations. Acta Hydrob Sinica, 2006, 30(2): 129-133(inChinesewithEnglishabstract).[赵凯, 杨公社, 李俊兵等. 黄河裸裂尻鱼群体遗传结构和cytb 序列变异. 水生生物学报, 2006, 30(2): 129-133.]
[7]YanXuelan,YangJinquan,TangWenqiaoet al.GeneticdiversityoftwoCyprinidfishes: Schizothorax biddulphiandDiptychus maculatesinKezilesuRiver,XinjiangUygurAutonomousRegionbasedonCytbgenesequencesvariation. Chinese J Zool, 2009, 44(5): 8-13(inChinesewithEnglishabstract).[阎雪岚, 杨金权, 唐文乔等. 基于线粒体Cytb基因序列变异的克孜河塔里木裂腹鱼和斑重唇鱼遗传多样性. 动物学杂志, 2009, 44(5): 8-13.]
[8]ZhaoFeng,ZhuangPing,ZhangLongzhenet al.PopulationgeneticstructureofPampus argenteusinthesouthYellowSeaandEastChinaSeabasedonthemitochondrialCytbsequence. Acta Hydrob Sinica, 2011, 35(5): 745-752(inChinesewithEnglishabstract).[赵峰, 庄平, 章龙珍等. 基于线粒体Cytb基因的黄海南部和东海银鲳群体遗传结构分析. 水生生物学报, 2011, 35(5): 745-752.]
[9]TianHuiwu,DuanXinbin,WangDengqianget al.SequencevariabilityofCytochromebandgeneticstructureofLeptobotia elongataintheupperYangtzeRiver. Freshw Fish, 2013, 43(6): 13-18(inChinesewithEnglishabstract).[田辉伍, 段辛斌, 汪登强等. 长江上游长薄鳅Cytb基因的序列变异与遗传结构分析. 淡水渔业, 2013, 43(6): 13-18.]
[10]WuLv.CellularcompositionoftheisletsinMastacembelus. Science, 1950, 32(3): 91(inChinese).[伍律. 刺鳅胰岛之细胞组成. 科学, 1950, 32(3): 91.]
[11]WuLv.Thepancreasandthepancreaticisletsofateleost, Mastacembelus aculeatus. Acta Hydrob Sinica, 1956, 2(2): 272-278(inChinesewithEnglishabstract).[伍律. 刺鳅的胰脏和胰岛. 水生生物学集刊, 1956, 2(2): 272-278.]
[12]ZhouRongjia,YuQixing,ChengHanhuaet al.PCRamplificationofSRYboxgeneinMonopterus albusandMastacembelus aculeatus. Chinese Sci Bull, 1996, 41(7): 640-642(inChinesewithEnglishabstract).[周荣家, 余其兴, 程汉华等.PCR扩增黄鳝和刺鳅SRY盒基因. 科学通报, 1996, 41(7): 640-642.]
[13]LiuJiangdong,HuangXiao,YuQixinget al.ThecytogeneticevidencesforconfirmingtheexistenceofsexchromosomesinMastacembelus aculeatus (Osteichthyes,Perciformes). J Wuhan Univ (Nat Sci Ed), 1999, 45(2): 185-190(inChinesewithEnglishabstract).[刘江东, 黄晓, 余其兴等. 刺鳅性染色体的细胞遗传学确定证据. 武汉大学学报: 自然科学版, 1999, 45(2): 185-190.]
[14]ChenJi,ZhaoGang,ZangYatinget al.TheconstructionofXchromosomelibraryofspinyeel(Mastacembelus aculeatus). Acta Hydrob Sinica, 2009, 33(4): 571-576(inChinesewithEnglishabstract).[陈戟, 赵刚, 臧亚婷等. 刺鳅X染色体DNA文库的构建. 水生生物学报, 2009, 33(4): 571-576.]
[15]WuYuanan,LiangZhiqiang,LiChuanwuet al.AnalysisandevaluationofnutritionalcomponentsinthemuscleoftwokindsofMastacembelus. Acta Nutrimenta Sinica, 2010, 32(5): 499-502(inChinesewithEnglishabstract).[伍远安, 梁志强, 李传武等. 两种刺鳅肌肉营养成分分析及评价. 营养学报, 2010, 32(5): 499-502.]
[16]HanJiugao.InfectionandcontrolofIchthyophthiriasisspp.onMastacembelus aculeatus. J Anhui Agri Sci, 2007, 35(16): 4826-4834(inChinesewithEnglishabstract).[韩九皋. 刺鳅感染小瓜虫病及防治研究. 安徽农业科学, 2007, 35(16): 4826-4834.]
[17]TraversRA.AreviewoftheMastacembeloidei,asuborderofsynbranchiformteleostfishes.PartII:hylogeneticanalysis. Bull Br Mus Nat Hist Zool, 1984, 47(2): 83-150.
[18]KottelatM.NotesonthetaxonomyanddistributionofsomewesternIndonesianfreshwaterfishes,withdiagnosesofanewgenusandsixnewspecies(Pisces:Cyprinidae,Belonidae,andChaudhuriidae). Ichthyol Explor Fres, 1991, 2(3): 273-287.
[19]JohnsonGD,PattersonC.Percomorphphylogeny:AsurveyofAcanthomorphsandanewproposal. Bull Mar Sci, 1993, 52(1): 554-626.
[20]KottelatM,LimKP.DiagnosisoftwonewgeneraandthreenewspeciesofearthwormeelsfromtheMalayPeninsulaBorneo(Teleostei:Chaudhuriidae). Ichthyol Explor Fres, 1994, 5(2):181-190.
[21]BritzR.OntogenyofethmoidalregionandhyopalatinearchinMacrognathus pancalus(Percomorpha,Mastacembeloidei),withcriticalremarksonmastacembeloidinter-andintrarelationships. Am Mus Novit, 1996, 3181: 1-18.
[22]VrevenEJ.Mastacembelidae(Teleostei;Synbranchiformes)subfamilydivisionandAfricangenericdivision:Anevaluation. J Nat Hist, 2005, 39(4):351-370.
[23]HallTA.BioEdit:auser-friendlybiologicalsequencealignmenteditorandanalysisprogramforWindows95/98/NT. Nucl Acids Symp Ser, 1999, 41: 95-98.
[24]TamuraK,StecherG,PetersonDet al.MEGA6:Molecularevolutionarygeneticsanalysisversion6.0. Mol Biol Evol, 2013, 30(4): 2725-2729.
[25]RozasJ,Anchez-DelBarrioJC,EsseguerXMet al.DnaSP,DNApolymorphismanalysesbythecoalescentandothermethods. Bioinformatics, 2003, 19(18):2496-2497.
[26]ExcoffierL,LavalG,SchneiderS.Arlequinver3.01:Anintegratedsoftwarepackageforpopulationgeneticsdataanalysis.Berne:ComputationalandMolecularPopulationGeneticsLaboratory(CMPG),Switzerland:UniversityofBerne, 2006.
[27]WolstenholmeDR.AnimalmitochondrialDNA:Structureandevolutionin:Mitochondrialgenomes.SanDiego:AcademicPress, 1992:173-372.
[28]NiuSufang,SuYongquan,WangJunet al.GeneticpolymorphismofmitochondrialcontrolregionandcytbinTrachurus japonicusfromFujiancoastalwaters. J Fish Sci China, 2011, 18(1): 66-74(inChinesewithEnglishabstract).[牛素芳, 苏永全, 王军等. 福建近海竹荚鱼线粒体DNA控制区和细胞色素b遗传多态性. 中国水产科学, 2011, 18(1): 66-74.]
[29]ZhaoKai,HeShunping,PengZuoganget al.MitochondrialdiversityandpopulationstructureofGymnocypris przewalskii przewalskii (Teleostei:Cyprinidae). J Qinghai Univ (Nat Sci), 2006, 24(4): 1-4(inChinesewithEnglishabstract).[赵凯, 何舜平, 彭作刚等. 青海湖裸鲤的种群结构和线粒体DNA变异. 青海大学学报, 2006, 24(4): 1-4.]
[30]ZhangDongya,ChenYong,LiuShaopinget al.PopulationgeneticstructureoftheendangeredspeciesEpalzeorhynchus bicornisintheNujiangRiver. Freshw Fish, 2009, 39(2): 3-7(inChinesewithEnglishabstract).[张东亚, 陈勇, 刘绍平等. 怒江濒危鱼类角鱼种群遗传结构研究. 淡水渔业, 2009, 39(2): 3-7.]
[31]BillingtonN,HebertPD.MitochondrialDNAdiversityinfishesanditsimplicationsforintroductions. Canadian Journal of Fisheries and Aquatic Sciences, 1991, 48(suppl.1): 8-94.
[32]ShakleeJB,TamaruCS,WaplesRS.Speciationandevolutionofmarinefishesstudiedbytheelectrophoreticanalysisofproteins. Pac Sci, 1982, 36(2): 141-157.
[33]ChenDi.AnalysisofgeneticstructureinmitochondrialcytochromebofCoilia ectenesinYangtzeRiver[Dissertation].Guangzhou:JinanUniversity, 2006 (inChinesewithEnglishabstract).[陈迪. 长江刀鲚遗传多样性的细胞色素b基因序列分析[学位论文]. 广州: 暨南大学, 2006.]
[34]LüBaozhong,ZhongYang,GaoLipinget al.Molecularevolutionandphylogenetics.Beijing:HighEducationPress, 2002.[吕宝忠, 钟扬, 高莉萍等译. 分子进化与系统发育. 北京: 高等教育出版社, 2002.]
[35]GrantWS,BowenBW.Shallowpopulationhistoriesindeepevolutionarylineagesofmarinefishes:insightsfromsardinesandanchoviesandlessonsforconservation. Journal of Heredity, 1998, 89: 415-426.
Genetic diversity of spiny eel (Mastacembelus sinensis) from five lakes based on Mitochondrial DNA cyt b gene
ZHAO Ziming, YE Jiansheng, LIU Meijian, SHEN Dongdong & HUANG Shiyi
(Jiangsu Agri-animal Husbandry Vocational College, Taizhou 225300, P.R.China)
ThemitochondrialDNAcyt bgene(767bp)ofspinyeelfromfivelakesinJiangsuProvincewasamplifiedandsequenced.TheaveragecontentsofA+TwerealittlehigherthanthoseofG+Cwhen38variablesitesweredetected,and28haplotypeswererecovered.Theaveragehaplotypediversity(0.58751)andnucleotidediversity(0.00214)demonstratedverylowgeneticdiversityofthespinyeelpopulationsinthefivelakes.Thefixationindiceofanalysisofmolecularvariancewas0.0356,whichshowednosignificantgeneticdifferenceamongpopulations.TheUPGMAphylogenetictreeofcyt bgenebuiltwithMEGA6.0showedthatfivepopulationswerenotclusteredintocladesbasedongeneticdistance.TherewasanextensivegeneralflowamongspinyeelpopulationsfromthefivelakesinJiangsuProvince.Theresultshowedthatspinyeelfromthefivelakeshadlowgeneticdiversityandgeneticallyhomogeneouspopulationstructure,indicatingtheirweakviabilitytomaintainpopulation.Thespinyeelneedtobewellprotected.
Mastacembelus sinensis;MitochondrialDNA; cyt b;geneticdiversity;freshwaterlakes
*江苏农牧科技职业学院课题(NSFRC1304)资助. 2015-08-29收稿; 2015-11-13收修改稿. 赵子明(1962~), 男, 副教授;E-mail:zzm0282678@163.com.
