以玉米淀粉废水为反硝化碳源的污染物降解特征与微生物群落结构研究
2016-09-21郭晓娅年跃刚闫海红殷勤高鹏陈光伟
郭晓娅,年跃刚*,闫海红,2,殷勤,2,高鹏,陈光伟
1.中国环境科学研究院水污染控制技术研究中心,北京 100012 2.北京师范大学水科学研究院,北京 100875 3.中蓝连海设计研究院,上海 201204 4.中粮生化能源(公主岭)有限公司,吉林 四平 136100
以玉米淀粉废水为反硝化碳源的污染物降解特征与微生物群落结构研究
郭晓娅1,年跃刚1*,闫海红1,2,殷勤1,2,高鹏3,陈光伟4
1.中国环境科学研究院水污染控制技术研究中心,北京100012 2.北京师范大学水科学研究院,北京100875 3.中蓝连海设计研究院,上海201204 4.中粮生化能源(公主岭)有限公司,吉林 四平136100
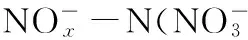
反硝化;碳源;玉米淀粉废水;三维荧光光谱;平行因子分析;微生物群落分析
废水的脱氮效果很大程度上取决于废水中反硝化碳源的含量及性能[1]。利用废物作为有机碳源,不仅可以减少处理废物的压力,相对于目前开发的其他类碳源,如天然缓释碳源[2-3]、人工降解材料[4-5]等,更能进一步降低处理成本,在污水处理中更具有实际意义。淀粉废水有机物浓度高、毒性小[6],且废水中的有机物以易降解有机物为主,是培养微生物的理想介质[7-10]。
目前针对反硝化过程中有机物的降解特征主要采用CODCr和TOC浓度进行表征,较少涉及溶解性有机物(dissolved organic matter,DOM)的降解特征。对反硝化过程中的DOM进行分析,不仅能够了解进出水的DOM信息,也能反映反硝化过程中DOM随时间的变化规律。三维荧光光谱(excitation-emission matrix spectrum, EEMs)技术近年来被广泛用于研究DOM荧光性质,其不仅能够反映有机物的浓度,同时还可以提供有机物组成的信息[11]。但由于荧光峰之间的相互叠加,传统的寻峰法以及区域积分法对重叠峰无法识别,2种方法均有一定的局限性。平行因子分析方法(parallel factor analysis,PARAFAC)是基于三线性数据分解理论,采用交替最小二乘法实现的数学模型[12],可客观识别出样品中荧光峰的个数、种类及各荧光峰的荧光强度等信息,具有广泛的应用前景。利用三维荧光光谱结合平行因子方法分析反硝化过程中DOM的降解特征,可以为反硝化反应器的底物投加提供理论和试验基础。
碳源种类是影响污泥菌群结构的重要因素[13],然而淀粉废水对反硝化脱氮的菌群结构影响却鲜有报道。由于污泥中微生物群落组成复杂,试图充分了解群落中微生物的结构往往比较困难,近年来高通量测序技术的快速发展为解决该问题提供了新的方法。借助高通量技术深度研究淀粉废水作为碳源的微生物多样性,有助于进一步研究反硝化过程的脱氮机理,对碳源的选择和利用均有重要的指导作用。笔者分析了以玉米淀粉废水处理过程中初沉池出水作为碳源时硝态氮和CODCr的降解过程,利用三维荧光光谱结合平行因子方法分析DOM的降解特征,并通过Illumina Hiseq 2500高通量测序技术分析废水驯化污泥中的微生物多样性,以期为淀粉废水作为碳源的过程控制与资源化利用提供依据。
1 材料与方法
1.1废水来源
废水取自吉林省某大型玉米深加工企业,初沉池废水水质指标:CODCr为12 234 mgL;TN浓度为631 mgL;TP浓度为161 mgL;浓度为24 mgL;浓度为2.3 mgL;浓度为4.9 mgL;pH为4.0。
1.2试验方法

1.3分析方法
1.3.1常规指标分析方法

1.3.2三维荧光光谱分析方法
水样经0.45 μm滤膜过滤后稀释10倍,使用F-7000型三维荧光分光光度计采集样品的荧光信号。发射波长为250~440 nm,激发波长为200~310 nm,激发与发射波长的狭缝宽度均为5 nm,扫描速度为12 000 nmmin。
在Matlab软件上应用drEEM工具箱[14]对样品的三维荧光数据进行平行因子分析。在应用模型之前,采用Delaunnay三角插值法去除拉曼和瑞利散射。结合核一致诊断法初步确定模型组分数,通过对半检验法来验证模型的可靠性,确定组分数。
1.3.3微生物多样性的测定
基因DNA的提取和聚合酶链式反应(PCR)扩增:采用CTAB方法对样本的基因组DNA进行提取,利用琼脂糖凝胶电泳检测DNA的纯度和浓度,取适量的样品于离心管中,用无菌水稀释样品至1 ngμL。以稀释后的基因组DNA为模板,选择16S V4区,根据测序区域的选择,使用带Barcode的特异引物(515F和806R),New England Biolabs公司的Phusion® High-Fidelity PCR Master Mix with GC Buffer和高效高保真酶进行PCR扩增。
文库构建和上机测序:使用TruSeq® DNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建,构建好的文库经过Qubit和Q-PCR定量,文库合格后,使用Hiseq 2500 PE250进行上机测序。
生物多样性与分类学分析:利用Uparse 软件[15]对所有样品的全部有效序列条数(Effective Tags)进行聚类,默认以97%的一致性(identity)将序列聚类成为OTUs(operational taxonomic units),筛选OTUs中出现频数最高的序列作为OTUs的代表序列。对OTUs代表序列进行物种注释,用RDP Classifier方法[16]与GreenGene数据库[17]进行物种注释分析,并分别在界(kingdom)、门(phylum)、纲(class)、目(order)、科(family)、属(genus)、种(species)分类水平下统计样本的群落组成。
2 结果与讨论
2.1反硝化过程中硝态氮浓度和CODCr的变化
污泥培养稳定后,反硝化系统中硝态氮浓度和CODCr的变化如图1所示。

图1 反硝化过程中硝态氮浓度和CODCr变化Fig.1 Changes of the concentrations of nitrogen and CODCr in denitrification process
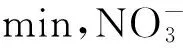
CODCr校准=CODCr检测-8
由图1可知,反硝化过程中CODCr的降解速率较稳定,在270 min内,从864 mgL快速下降至102 mgL,说明废水中含有丰富的易降解有机物,可以被微生物快速利用。由于试验完全以废水作为碳源,实际工程废水中含有一定量碳源,废水仅作为补充碳源进行添加,废水添加量可以进一步减少,并且缺氧反硝化工艺后通常设有好氧工艺,有机物可以被进一步降解,因此出水CODCr可以达到GB 8978—1996《污水综合排放标准》一级标准(100 mgL)限值,进一步证明玉米淀粉废水作为碳源的可行性。
2.2平行因子法解析反硝化过程
采用三维荧光光谱结合平行因子法分析反硝化过程中DOM的降解特征。利用核一致诊断法初步判断模型组分,一般认为核一致值大于60%时组分数的选择较合适。图2为以初沉池出水作为反硝化碳源时组分数分别为2和3的核一致诊断法结果。组分数为2和3时,核一致值分别为99.87%和17.20%,因此初步拟定模型的组分数为2。
对半分析法是将数据库分为几个部分,通过检验各部分获得的模型是否一致来判断组分数的合理性,以排除单独一个数据库造成误差的可能。将数据库分解为A、B、C、D 4组,两两组合为1个半数据库,验证AB、CD、AC、BD、AD以及BC 6个半数据库与总数据库的组分在激发和发射波长的载荷同总数据库的一致性。根据文献[20]设定Matlab程序,即各组分的Tucker一致性系数大于0.95时可运行成功,否则运行错误。图3为组分数为2时的对半分析结果。由图3可见,当模型组分数为2时,各半数据库与总数据库的组分在激发和发射波长的载荷基本重合。当模型组分数为3时,程序无法运行成功,即数据库各组分的Tucker一致性系数小于0.95,因此可以确定模型的组分数为2。
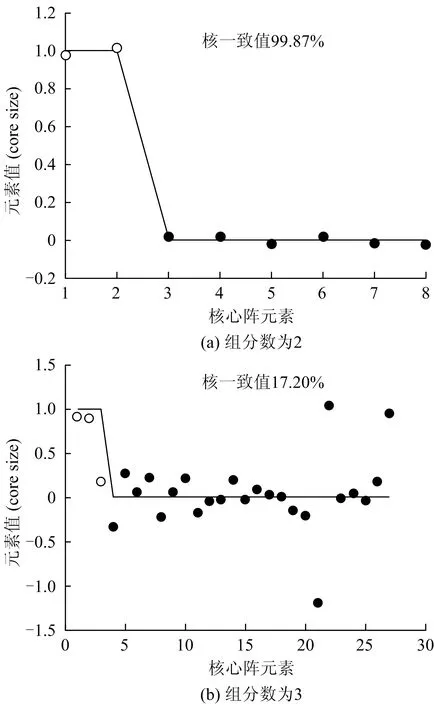
图2 核一致法判断模型组分数Fig.2 Model component estimation by the core consistency diagnostic
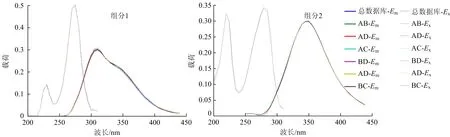
图3 组分数为2时的对半分析结果Fig.3 The results of split-half analysis when there were two components
根据平行因子模型识别出初沉池出水作为碳源的反硝化系统中DOM的2个荧光组分及各组分的最大激发和发射波长,根据文献[21-22]获得对应的荧光物质类型,如表1所示。2个荧光组分的三维荧光光谱如图4所示。

表1 反硝化过程中2个荧光组分特征
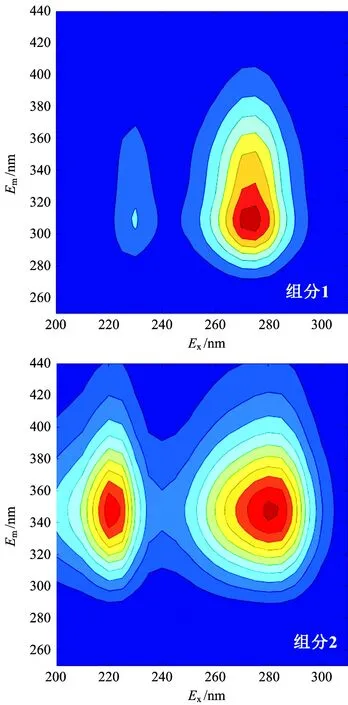
图4 平行因子模型鉴别出的2个荧光组分Fig.4 Two fluorescence components identified by the PARAFAC model
以初沉池出水为碳源的反硝化过程中各组分荧光强度和总荧光强度随时间的变化如图5所示。
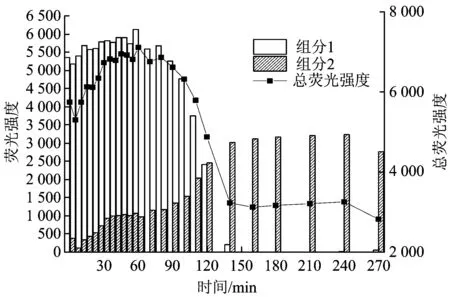
图5 平行因子模型解析反硝化过程荧光强度变化Fig.5 Variation of fluorescence intensity against time identified by the PARAFAC model
由图5可见,废水中的荧光组分以组分1为主,随着反硝化反应的进行,组分1的荧光强度以及总荧光强度在60 min之前呈小幅上升趋势,这可能是由于加入废水后,处于内源呼吸期的微生物为应对外界环境变化产生的代谢副产物所致。60 min之后,组分1的荧光强度迅速下降,直至消失,说明微生物可以快速利用废水中的类络氨酸。系统中组分2在初始时刻荧光强度较弱,随着时间的推移,其荧光强度逐渐加强,且组分1与组分2荧光强度不显著相关,因此可以认为组分2是反硝化过程中微生物代谢产生的类色氨酸副产物。
2.3微生物群落结构和多样性
2.3.1污泥驯化前后纲水平下微生物群落结构
经初沉池废水驯化过的二沉池活性污泥中,微生物群落在纲水平下的组成和相对丰度如图6所示(仅列出相对丰度大于0.5%的纲类,相对丰度低于0.5%的纲类归为其他)。经初沉池废水驯化后,活性污泥中优势种群种类基本不变,但微生物丰度明显发生变化。其中,β-变形菌纲(Betaproteobacteria)丰度显著增加,由原二沉污泥中38.03%增加至59.34%。而β-变形菌纲的许多种类都已证明具有良好的反硝化能力[23]。
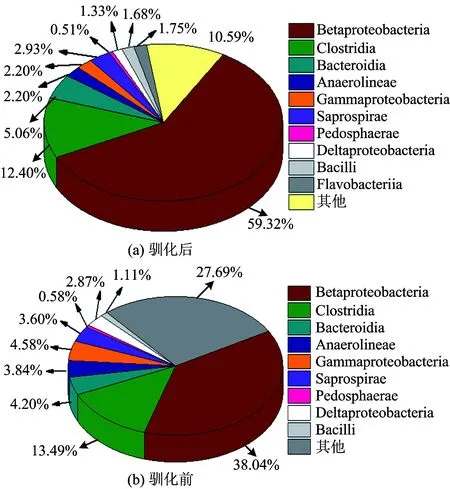
图6 微生物群落在纲水平的组成和相对丰度Fig.6 Composition and relative abundance of microbial communities at the class level
2.3.2驯化污泥目水平下微生物群落结构
以初沉池废水为碳源的反应器内有9种优势菌群,其中,第一优势菌群为未经培养菌种(uncultured bacterium),相对丰度为18.54%。其次为伯克氏菌目(Burkholderiales)、红环菌目(Rhodocyclales)和嗜氢菌目(Hydrogenophilales),相对丰度分别为14.65%、14.13%和9.63%。这3类微生物均属于β-变形菌纲。之后依次为梭菌目(Clostridiales)、拟杆菌目(Bacteroidales)、Saprospirales、厌氧绳菌目(Anaerolineales)和假单孢菌目(Pseudomonadales),相对丰度分别为8.14%、5.06%、2.93%、1.57%和1.23%。
3 结论
(2)利用三维荧光光谱结合平行因子方法分析反硝化过程中DOM的降解特征,识别出反硝化过程中的2个溶解性有机物的荧光组分,分别为类络氨酸荧光组分〔组分1(230 nm310 nm、275 nm310 nm)〕和类色氨酸荧光组分〔组分2(220 nm350 nm、280 nm350 nm)〕。组分1的荧光强度随时间先升高后降低,直至荧光峰消失;组分2荧光强度逐渐升高,推测组分2为微生物代谢过程中产生的副产物;总荧光强度随时间呈先升高后逐渐降低的趋势。
(3)经初沉池出水驯化后,污泥中的β-变形菌纲丰度明显增加。目水平下三大优势菌群(相对丰度在10%以上)为未经培养菌种(uncultured bacterium)、伯克氏菌目(Burkholderiales)和红环菌目(Rhodocyclales),相对丰度分别为18.54%、14.65%和14.13%。
[1]HENZE M.Capabilities of biological nitrogen removal processes from wastewater[J].Water Science & Technology,1991,23(456):669-679.
[2]OVEZ B,OZGEN S,YUKSEL M.Biological denitrification in drinking water usingGlycyrrhizaglabraandArundadonaxas the carbon source[J].Process Biochemistry,2006,41(7):1539-1544.
[3]金赞芳,陈英旭,小仓纪雄.以棉花为碳源去除地下水硝酸盐的研究[J].农业环境科学学报,2004,23(3):512-515.
JIN Z F,CHEN Y X,OGURA N.Denitrification of groundwater using cotton as energy source[J].Journal of Agro-Environment Science,2004,23(3):512-515.
[4]WU W Z,YANG L H,WANG J L.Denitrification using PBS as carbon source and biofilm support in a packed-bed bioreactor[J].Environmental Science & Pollution Research,2012,20(1):333-339.
[5]CHU L,WANG J.Denitrification performance and biofilm characteristics using biodegradable polymers PCL as carriers and carbon source[J].Chemosphere,2013,91(9):1310-1316.
[6]LOSS E,ROYER A R,BARRETO-RODRIGUES M,et al.Use of maize wastewater for the cultivation of thePleurotusspp.mushroom and optimization of its biological efficiency[J].Journal of Hazardous Materials,2009,166(2):1522-1525.
[7]BRAR S K,VERMA M,TYAGI R D,et al.Efficient centrifugal recovery ofBacillusthuringiensisbiopesticides from fermented wastewater and wastewater sludge[J].Water Research,2006,40(6):1310-1320.
[8]HUANG L P,JIN B,LANT P,et al.Simultaneous saccharification and fermentation of potato starch wastewater to lactic acid byRhizopusoryzaeandRhizopusarrhizus[J].Biochemical Engineering Journal,2005,23(3):265-276.
[9]O-THONG S,HNIMAN A,PRASERTSAN P,et al.Biohydrogen production from cassava starch processing wastewater by thermophilic mixed cultures[J].International Journal of Hydrogen Energy,2011,36(5):3409-3416.
[10]MUNIRAJ I K,XIAO L W,HU Z H,et al.Microbial lipid production from potato processing wastewater using oleaginous filamentous fungiAspergillusoryzae[J].Water Research,2013,47(10):3477-3483.
[11]杨长明,汪盟盟,马锐,等.城镇污水厂尾水人工湿地深度处理过程中DOM三维荧光光谱特征[J].光谱学与光谱分析,2012,32(3):708-713.
YANG C M,WANG M M,MA R,et al.Excitation-emission matrix fluorescence spectra characteristics of DOM in a subsurface constructed wetland for advanced treatment of municipal sewage plant effluent[J].Spectroscopy and Spectral Analysis,2012,32(3):708-713.
[12]BRO R.PARAFAC:tutorial and applications[J].Chemometrics and Intelligent Laboratory Systems,1997,38(2):149-171.
[13]刘秀红,甘一萍,杨庆,等.碳源对反硝化生物滤池系统运行及微生物种群影响[J].水处理技术,2013,39(11):36-40.
LIU X H,GAN Y P,YANG Q,et al.Effect of carbon source types on the operation and nitrifying microbial community of denitrifying biofilter for advanced nitrogen removal[J].Technology of Water Treatment,2013,39(11):36-40.
[14]MURPHY K R,STEDMON C A,GRAEBER D,et al.Fluorescence spectroscopy and multi-way techniques:PARAFAC[J].Analytical Methods,2013,5(23):6557-6566.
[15]EDGAR R C.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods,2013,10(10):996-998.
[16]WANG Q,GARRITY G M,TIEDJE J M,et al.Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J].Applied & Environmental Microbiology,2007,73(16):5261-5267.
[17]DESANTIS T Z,HUGENHOLTZ P,LARSEN N,et al.Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Applied & Environmental Microbiology,2006,72(7):5069-5072.
[18]KUJAWA K,KLAPWIJK B.A method to estimate denitrification potential for predenitrification systems using NUR batch test[J].Water Research,1999,33(10):2291-2300.
[19]付昆明,曹相生,孟雪征,等.污水反硝化过程中亚硝酸盐的积累规律[J].环境科学,2011,32(6):1660-1664.
FU K M,CAO X S,MENG X Z,et al.Characteristics of nitrite accumulation during wastewater denitrification[J].Environmental Science,2011,32(6):1660-1664.
[20]LORENZO-SEVA U,TEN B J M F.Tucker′s congruence coefficient as a meaningful index of factor similarity[J].Methodology,2006,2(2):57-64.
[21]CHEN W,WESTERHOFF P,LEENHEER J A,et al.Fluorescence excitation-emission matrix regional integration to quantify spectra for dissolved organic matter[J].Environmental Science & Technology,2003,37(24):5701-5710.
[22]LEENHEER J A,CROUÉ J P.Peer reviewed:characterizing aquatic dissolved organic matter[J].Environmental Science & Technology,2003,37(1):18A-26A.
[23]LEE H W,PARK Y K,CHOI E,et al.Bacterial community and biological nitrate removal:comparisons of autotrophic and heterotrophic reactors for denitrification with raw sewage[J].Journal of Microbiology & Biotechnology,2008,18(11):1826-1835. ▷
汪燕南,李文文,孙运飞,等.改性甘蔗渣去除模拟废水中孔雀石绿的研究[J].环境工程技术学报,2016,6(5):434-439.
WANG Y N, LI W W, SUN Y F, et al.Removal of malachite green in simulated wastewater by modified bagasse[J].Journal of Environmental Engineering Technology,2016,6(5):434-439.
Pollutants Degradation Characteristics and Microbial Community Structure Using Cornstarch Wastewater as Denitrification Carbon Source
GUO Xiaoya1, NIAN Yuegang1, YAN Haihong1,2, YIN Qin1,2, GAO Peng3, CHEN Guangwei4
1.Research Center of Water Pollution Control Technology, Chinese Research Academy of Environmental Sciences, Beijing 100012, China 2.College of Water Science, Beijing Normal University, Beijing 100875,China 3.China Bluestar Lehigh Engineering Corporation, Shanghai 201204, China 4.COFCO Bio-chemical Energy(Gongzhuling) Company Limited, Siping 136100, China
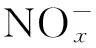
denitrification; carbon source; cornstarch wastewater; excitation-emission matrix spectroscopy; parallel factor analysis; community structure
2016-03-07
国家水体污染控制与治理科技重大专项(2012ZX07202-009-01)
郭晓娅(1990—),女,硕士研究生,主要研究方向为水污染控制与资源化,xiaoyaguo1990@163.com
*责任作者:年跃刚(1963—),男,研究员,博士,主要从事生态修复、中水回用研究,nianyg@craes.org.cn
X703
1674-991X(2016)05-0427-07
10.3969j.issn.1674-991X.2016.05.063
