酵母耐盐相关基因HAL1在棉花中的功能表达
2016-09-19郭丽雪樊伟丽阴祖军王俊娟王德龙叶武威
穆 敏,舒 娜,王 帅,郭丽雪,樊伟丽,阴祖军,王俊娟,王德龙,叶武威
(中国农业科学院棉花研究所/棉花生物学国家重点实验室/农业部棉花遗传改良重点开放实验室,河南安阳 455000)
酵母耐盐相关基因HAL1在棉花中的功能表达
穆 敏,舒 娜,王 帅,郭丽雪,樊伟丽,阴祖军,王俊娟,王德龙,叶武威
(中国农业科学院棉花研究所/棉花生物学国家重点实验室/农业部棉花遗传改良重点开放实验室,河南安阳 455000)
【目的】克隆酿酒酵母(Saccharomyces cerevisiae)耐盐基因HAL1(halotolerance)并转化棉花,探究该基因在棉花中的功能,进一步探究酵母耐盐相关基因在高等植物中的具体功能。【方法】根据NCBI核酸数据库中酵母耐盐基因HAL1的mRNA全长序列信息,利用RT-PCR技术从酿酒酵母As2.375中克隆该基因。选择酶切位点XbaI和SmaI对表达载体pBI121∶∶GFP进行双酶切,采用In-Fusion技术构建pBI121-ScHAL1∶∶GFP融合表达载体。以自发荧光较弱的陆地棉品种Y-2067、ZA-23、GZ-2的花粉为材料,用基因枪轰击棉花花粉进行瞬时表达研究。采用基因枪活体转化技术将外源基因表达载体 pBI121-HAL1∶∶GFP转化棉花盐敏感材料中s9612,获得T0棉花转基因种子。用100 mmol·L-1NaCl盐溶液对转基因T0种子进行耐盐性发芽试验,并进行分子检测,对鉴定出的转基因植株进行叶盘耐盐性分析。【结果】从酿酒酵母As2.375中克隆耐盐基因ScHAL1,基因全长885 bp,共编码294个氨基酸。对其序列进行分析,发现HAL1蛋白中丝氨酸所占比例最大,整个蛋白呈碱性且带正电,属于亲水性蛋白。根据蛋白二级结构预测结果,推测该蛋白的结构功能域可能主要由无规则卷曲和β-折叠片构成。棉花花粉瞬时表达结果表明,转化HAL1后,3种陆地棉花粉的绿色荧光现象都明显增强,说明该基因可以在这3种陆地棉的花粉中表达。用100 mmol·L-1NaCl盐溶液对转基因棉花T0种子和受体材料中s9612自交种进行胁迫,发现转基因种子萌发能力明显强于受体材料,表明HAL1可以提高种子耐盐性。根据基因核苷酸序列设计2对引物对T0转基因棉花幼苗进行分子检测,将纯化后的PCR产物进行直接测序,测序结果证明转基因成功。对鉴定出的转基因植株进行叶盘耐盐性分析发现,在600 mmol·L-1NaCl和400 mmol·L-1NaCl盐胁迫下转基因植株叶盘叶绿素含量均高于对照植株,且在600 mmol·L-1NaCl溶液胁迫后,转HAL1植株叶绿素含量反而高于400 mmol·L-1NaCl溶液处理后的叶盘。【结论】成功从酿酒酵母中克隆基因HAL1,酵母HAL1对提高棉花耐盐性具有重要作用。
酵母;HAL1;陆地棉;花粉瞬时表达;分子检测
0 引言
【研究意义】土壤盐渍化已成为影响世界农业发展的一个主要因素。据报道,盐渍化影响着全世界约1/5的耕地,并导致其以每年1 000万公顷的速度流失[1-3]。棉花是盐碱地的先锋作物,是开发利用盐渍化土地的理想作物之一,通过基因工程和转基因技术提高棉花耐盐性已经成为当前棉花育种的一个迫切需求和重要方向。【前人研究进展】目前,对棉花的耐盐机制、内源基因克隆和功能分析方面做了大量的研究,已从棉花中分离克隆了许多抗逆相关基因。而在外源基因转化棉花方面也有大量报道。郭三堆等[4]将来源于苏云金芽孢杆菌具有高杀虫活性的 Bt杀虫基因GFM Cry1A导入棉花,成功培育出国产单价转基因抗虫棉品种,使中国成为继美国之后世界上第二个研制成功转基因抗虫棉的国家;拟南芥抗旱基因AtHDG11可以提高烟草、水稻等转基因植株的抗旱性,将其转化陆地棉后发现该基因不仅可以提高转基因棉花的耐旱性,同时提高棉花产量[5];利用农杆菌介导法将山菠菜(Atriplex hortensis)AhCMO转化泗棉3号,0.5% NaCl胁迫转基因棉花和对照,发现转基因棉花所受盐害程度明显小于非转基因棉株,证明AhCMO可以提高转基因棉花的耐盐性[6];PASAPULA等[7]将拟南芥 AVP1转化棉花,转基因棉花在 200 mmol·L-1NaCl条件下依然可以旺盛生长,同时在干旱条件下,转基因植株的纤维产量至少提高了20%。酿酒酵母(Saccharomyces cerevisiae)中,HAL1 (halotolerance)是重要的耐盐因子,其参与细胞内离子的浓度调节[8]。1992年,GAXIOLA等[9]和RIOS等[10]通过在高盐条件下对转化酵母菌株进行筛选,首次分离出HAL1并在酵母中超表达,证明其可以减少细胞内Na+积累,同时促进K+吸收。HAL1编码一种可以调节单离子通道的水溶性蛋白(32 kD),它通过提高反馈调节中K+吸收的量来促进K+的吸收[9]。相关研究表明,在高等植物中可能存在酵母HAL1的同源基因,这意味着酵母HAL1转化高等植物后可能与植物耐盐性相关[11-14]。已有报道证明,ScHAL1转化甜瓜[15]、番茄[16]、拟南芥[17]、西瓜[18]、苜蓿[19]后,转基因植株在耐盐性方面比野生型植株均有不同程度的提高;转化番茄后同时提高其产量[20];转化水稻后提高其耐碱性[21]。【本研究切入点】ScHAL1转化植物后可提高转基因植物的耐盐性,但转化棉花的功能作用未见报道。【拟解决的关键问题】本研究以酿酒酵母As2.375为材料,克隆ScHAL1并构建表达载体,运用基因枪活体转化技术转化陆地棉(Gossypium hirsutum L.)中s9612,旨在进一步探索该基因在高等植物中的确切功能,同时为获得棉花耐盐性新材料奠定基础。
1 材料与方法
1.1试验材料
酿酒酵母(Saccharomyces cerevisiae)As2.375购自上海工业微生物研究所。
1.2RNA的提取和基因全长序列克隆
使用UltraClean Microbial RNA Isolation Kit(MO BIO)试剂盒提取酿酒酵母 As2.375的 RNA,用Nanodrop 2000核酸分析仪测定总 RNA的浓度和纯度,用ReverTra Ace qPCR RT Master Mix with gDNA Remover试剂盒将RNA反转录成第一链cDNA。用Primer Premier 5软件设计基因全长引物,分别命名为HAL1-F和HAL1-R(表1)。以反转录获得的cDNA为模板,用2×TransTaq-T PCR SuperMix扩增ScHAL1全长mRNA序列。PCR程序为94℃ 5min;94℃ 45 s,55℃ 45 s,72℃ 1 min,40个循环;72℃ 10 min。连接pMD-18 T载体转化至大肠杆菌感受态细胞DH5α,挑取阳性克隆,测序验证。
1.3序列分析
Protparam(http://web.expasy.org/protparam/)在线软件分析蛋白质的理化性质。ProtScale(http://web. expasy.org/protscale/)预测亲疏水性。SOPMA(http:// npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/MPSA /npsa_sopma.html)分析蛋白的二级结构。
1.4构建pBI121-ScHAL1::GFP荧光表达载体
在线(http://bioinfo.clontech.com/infusion)设计In-Fusion引物,命名为InHAL1-F和InHAL1-R(表1)。该引物分别在基因克隆引物的上下游5′端加上载体正向酶切位点和反向酶切位点连接处15 bp的重叠碱基。以ScHAL1质粒为模板,用In-Fusion引物进行扩增。选择的酶切位点为 XbaI和 SmaI,对表达载体pBI121::GFP进行双酶切。采用In-Fusion连接技术进行 pBI121-ScHAL1::GFP表达载体的构建,其构建原理基本流程如图1所示,验证重组克隆并测序。
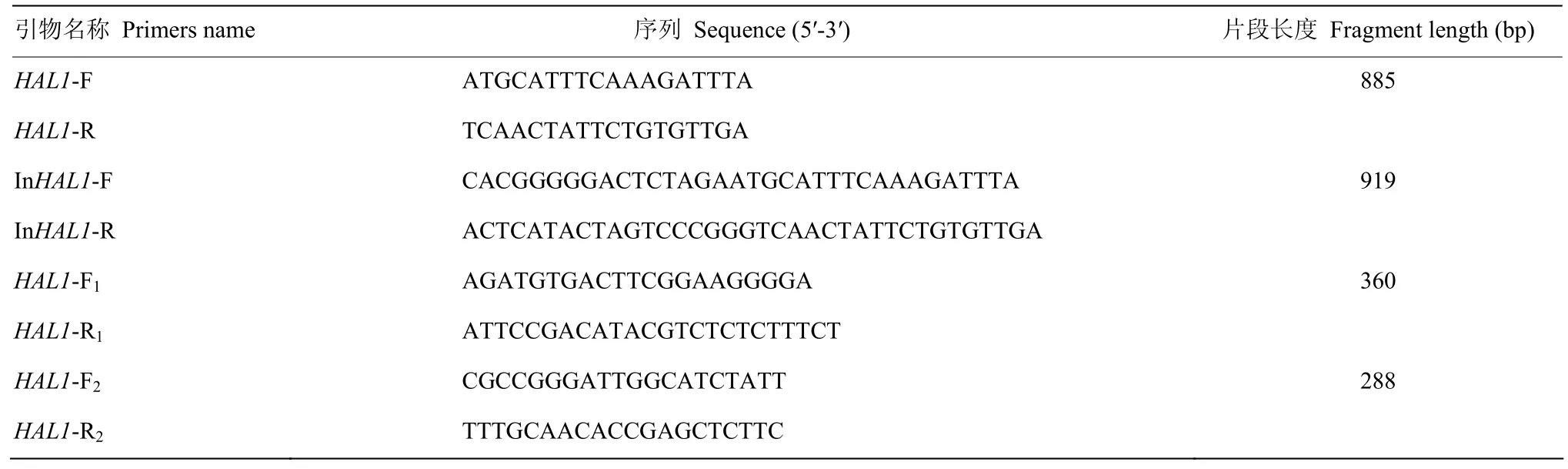
表1 试验所用引物Table 1 Primers used in the experiments
1.5ScHAL1在棉花花粉中的瞬时表达分析
收集陆地棉Y-2067、ZA-23和GZ-2 3种材料的花粉,便携式基因枪 GDS-80轰击花粉,在激光共聚焦显微镜FV1000(日本Olympus)下观察花粉荧光。
1.6基因枪活体转化棉花
提取pBI121-ScHAL1::GFP载体质粒(1 mg·mL-1)备用,金粉和质粒的处理参照孔静静[22]的试验步骤;参考周凯[23]的操作方法进行基因枪活体转化工作,即:(1)转化的第一天下午采集花朵并计数,已去掉雄蕊的花用蜡管套住柱头;(2)第二天上午收集花粉,将质粒和金粉的悬浮液加入基因枪中并轰击花粉;(3)将轰击后的花粉人工涂抹在前一天用蜡管套住的雄蕊的柱头,并将蜡管重新套好,以免发生杂交。(4)待种子成熟,将收获的种子做好标记,晒干后保存。
1.7转基因棉花T0种子耐盐性发芽试验
采用双夹层滤纸法[22]对收获的 T0种子进行发芽试验,用100 mmol·L-1NaCl溶液处理为试验组,清水处理为对照组,放入光照培养箱中进行培养(培养条件为28℃,光照14 h;25℃,黑暗10 h),试验组和对照组各重复3次,7 d后统计发芽结果。
1.8转基因棉花分子检测
根据ScHAL1在棉花基因组中的比对情况,对基因序列设计2对引物,分别命名为HAL1-F1、HAL1-R1和HAL1-F2、HAL1-R2(表1)。采用CTAB法提取转基因植株DNA进行PCR扩增,所用程序为94℃,5 min;94℃ 45 s,58℃ 45 s,72℃ 30 s,40 cycle;72℃ 10 min。
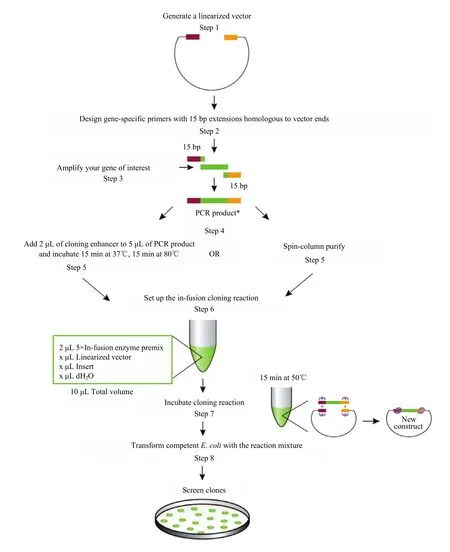
图1 表达载体构建流程图(摘自TaKaRa Clontech 技术)Fig.1 The flowing chart of construction expression vector (from TaKaRa Clontech technology)
1.9转基因棉花叶盘耐盐性分析
对鉴定出的转基因植株随机选择6棵进行叶盘耐盐性分析试验。具体操作步骤参照张莹[24]的方法,分别取6棵植株同一叶位的叶片进行打孔,灭菌水、400 和600 mmol·L-1NaCl盐水处理,同一时期中s9612植株同叶位的叶片为对照,重复3组;28℃,光照条件培养96 h,观察叶盘颜色和形态变化并测定每组叶绿素含量。
2 结果
2.1ScHAL1的克隆
在 NCBI数据库中进行检索得到 ScHAL1 (GenBank:EF015596.1)的全长序列信息,设计基因特异性引物,以cDNA为模板,扩增ScHAL1完整序列。得到一条长度为885 bp编码294个氨基酸的核苷酸序列(图2),测序结果正确。
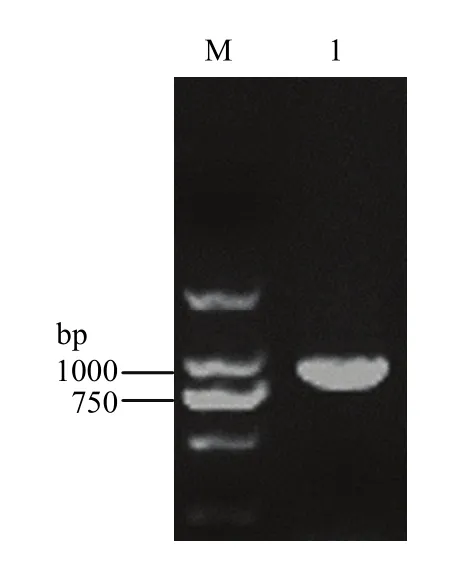
图2 ScHAL1的克隆Fig.2 Cloning of ScHAL1
2.2生物信息学分析
在线分析软件 Protparam (http://web.expasy.org/ protparam/)的结果表明,HAL1蛋白的分子式为C1443H2269N409O460S7,分子量为32.9 kD,等电点为9.01,基因定位在细胞质中。其氨基酸组成中,丝氨酸(Ser)所占比例最大,为13%;其次是赖氨酸(Lys)9%(图3-A)。带正电荷的碱性氨基酸(Arg+Lys)有38个,带负电荷的酸性氨基酸(Asp+Glu)有31个,该蛋白呈碱性且带正电。分析其不稳定指数为50.08,半衰期大于20 h,属于不稳定蛋白。分析HAL1蛋白的亲疏水性发现,蛋白中亲水性氨基酸明显多于疏水性氨基酸(图3-B),其亲水性平均数(GRAVY)为-0.787,即整个蛋白为亲水性。蛋白二级结构预测结果(图3-C)表明,该蛋白中无规则卷曲的氨基酸有150个,占51.02%,组成该蛋白的主体结构;β-折叠片为73个,占24.83%;α螺旋区为39个氨基酸,占13.27% ;β-转角占10.88%,包含32个氨基酸;推测该蛋白的结构功能域可能主要由无规则卷曲和β-折叠片构成。
2.3pBI121-ScHAL1::GFP荧光表达载体的构建
将ScHAL1插入植物表达载体pBI121::GFP中,构建融合蛋白表达载体,转化至大肠杆菌DH5α感受态细胞,挑选阳性克隆进行测序,序列比对结果正确。选用限制性内切酶BglⅡ酶切验证插入正确(图4),表明表达载体构建成功,命名为 pBI121-ScHAL1:: GFP。
2.4ScHAL1在棉花花粉中的瞬时表达分析
棉花花粉具有自发荧光现象。在波长为488 nm的激发光下,棉花花粉的自发荧光为绿色。根据实验室前期研究结果[22],选择花粉自发荧光较弱的3种棉花材料GZ-2、Y-2067、ZA-23收集花粉,采用基因枪活体转化技术对棉花花粉进行轰击,室温下过夜培养,然后在激光共聚焦显微镜下观察(图5),发现3种材料的花粉绿色荧光都明显增强,证明该基因在棉花花粉中得到了表达,同时证明该外源基因转化棉花的可行性,为使用基因枪活体转化技术获得转基因耐盐材料奠定基础。
2.5转基因棉花T0种子耐盐性检测
将酿酒酵母耐盐相关基因ScHAL1转入盐敏感材料中s9612,共收获T0种子187粒,同时选取材料中s9612自交种子作为对照,各取籽粒饱满,均匀一致的种子20粒,采用双夹层滤纸培养法,用100 mmol·L-1的NaCl溶液处理为试验组,0 mmol·L-1处理为对照组,然后放入光照培养箱中进行培养,7 d后统计发芽结果(图6),在NaCl溶液胁迫下,转基因种子萌发能力明显高于对照,表明HAL1可以明显提高种子耐盐性。
2.6转基因植株的分子检测
将正常发芽的种子置于温室中出苗,取幼苗叶片提取DNA进行分子检测。以载体质粒为阳性对照,中s9612自交种DNA为阴性对照(图7)。转基因植株和质粒中均扩出条带,而阴性对照中无带。将PCR产物纯化后进行测序,测序结果使用DNAMAN软件与ScHAL1的mRNA序列进行比对(图8),认为酵母HAL1已成功转入棉花中。
2.7叶盘耐盐性分析结果
对分子检测正确的9棵转基因植株中随机选择6棵,进行叶盘耐盐性试验(图9-A),400和600 mmol·L-1NaCl盐水胁迫96 h后,棉花叶盘颜色逐渐变黄,叶边缘变白,而0 mmol·L-1NaCl溶液处理下叶盘依然保持绿色;且在相同浓度盐溶液处理下,转HAL1植株叶盘颜色形态强于对照叶盘。对处理后的叶盘测定其叶绿素含量(图9-B),结果发现与对照中s9612相比,0 mmol·L-1盐溶液处理后转基因植株叶盘的叶绿素含量较高,但差异并不明显;400 mmol·L-1NaCl处理后,二者叶绿素含量都有所降低,转基因植株叶绿素含量明显高于对照;600 mmol·L-1NaCl处理后,转基因棉花叶盘叶绿素含量高于对照一倍多。同时发现,转基因植株叶盘在600 mmol·L-1NaCl溶液胁迫后,叶绿素含量反而高于400 mmol·L-1NaCl溶液处理后的叶盘,推测酿酒酵母HAL1在棉花耐受高盐胁迫方面发挥巨大作用。
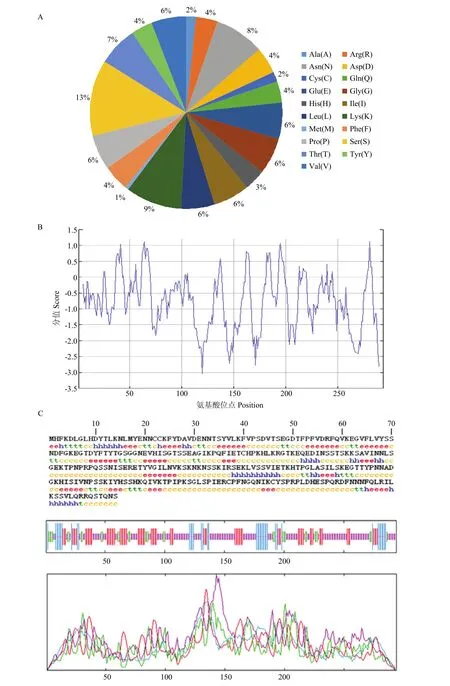
图3 ScHAL1蛋白生物信息学分析Fig.3 Bioinformatics analysis of ScHAL1 protein
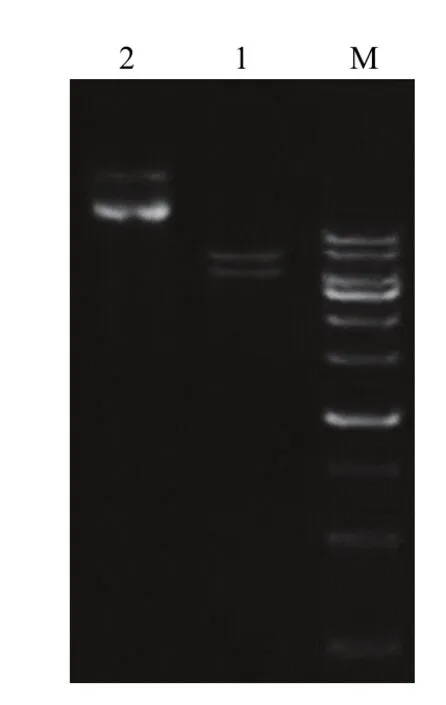
图4 pBI121-ScHAL1::GFP表达载体酶切验证Fig.4 Enzyme digestion of expression vector pBI121-ScHAL1::GFP

图5 棉花花粉的瞬时表达分析Fig.5 The transient expression analysis of cotton pollen
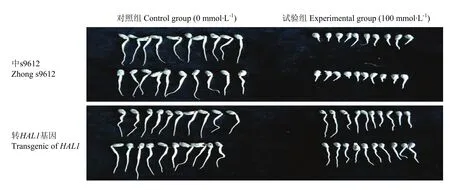
图6 转ScHAL1棉花种子发芽试验Fig.6 Seed germination test of ScHAL1 transgenic cotton

图7 转基因植株分子检测Fig.7 The transgenic plants molecular detection

图8 测序结果Fig.8 Sequencing result
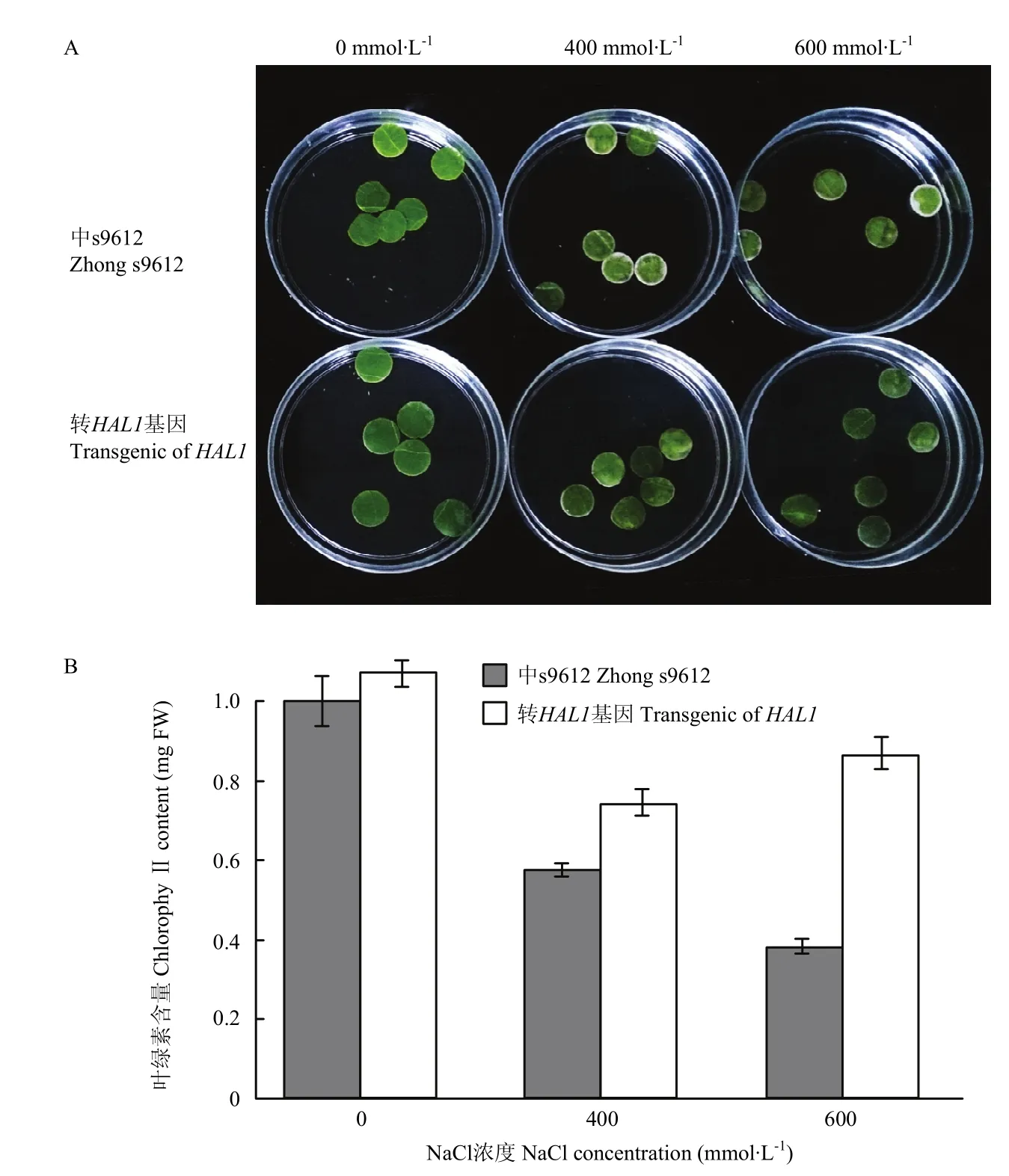
图9 转基因棉花叶盘耐盐性分析Fig.9 Transgenic cotton semal salt resistance analysis
3 讨论
大量的研究表明,耐盐真菌的相关耐盐基因能提高微生物和植物的耐盐能力[25-26]。ZHAO等[27]将酵母超氧化物歧化酶基因SOD转入水稻中,转基因植株耐盐性具有一定的提高;张丽青[28]克隆来自嗜热毛壳菌SOD转化烟草,除了提高转基因植株的耐盐能力,对烟草赤星病和炭疽病有一定的抵抗能力;谢丽霞[29]将曲霉中分离出的耐盐基因RPS3ae和RPL44转化拟南芥和烟草,结果表明,转基因植株对盐渍化的耐受型明显高于对照野生型。
目前用于植物转基因的方法较多,相比于农杆菌侵染法、花粉管注射和台式基因枪轰击法,本研究使用的基因枪活体转化技术操作步骤简单,减少材料污染,大大缩短试验周期。
结合HAL1序列与GFP报告基因构建融合表达载体,通过基因枪活体转化技术在棉花花粉中进行瞬时表达研究,结果表明,HAL1在棉花花粉中可以表达,为下一步转化棉花获得转基因新材料奠定基础。利用PCR扩增产物直接测序,缩短转基因种子检测的时间。而对于PCR扩增无法获得基因全长的结果,认为可能是由于ScHAL1整合到棉花基因组后基因编码链被打断导致的,还需进一步检测鉴定。
张荃等[30]从啤酒酵母中克隆了HAL1并利用叶圆盘法转化番茄,对T1转基因番茄和对照番茄进行盐处理,15 d后观察发现,在任何盐浓度下,转基因植株和对照植株的生长情况均有所缓滞,但转基因植株的生长情况明显优于对照。在175 mmol·L-1NaCl处理10 d后,对照番茄叶片变黄,基部叶片脱落并伴有坏死,而转基因植株的叶片呈深绿色,生长状况明显强于对照。盐胁迫下,转基因番茄地上部分的鲜重、干重也明显高于对照番茄。HAL1的转入提高了转基因番茄的耐盐性。100 mmol·L-1NaCl溶液对转基因种子进行耐盐性发芽试验,结果表明,转基因种子在盐胁迫下的芽长度明显比0 mmol·L-1对照组短,但比盐胁迫后的非转基因种子芽长度长。对转基因植株的叶盘耐盐性分析表明,酵母HAL1转化棉花后提高转基因植株叶盘的耐盐性,且在高盐浓度下对其耐受性提高具有重要作用,后续试验将对转基因植株进行进一步研究。
4 结论
克隆酿酒酵母As2.375耐盐基因HAL1并转化陆地棉中s9612,在100 mmol·L-1NaCl溶液胁迫下,转HAL1的棉花种子耐盐性明显强于受体材料中 s9612自交种;不同浓度盐胁迫条件下,转基因植株叶盘叶绿素含量均高于对照植株,且在 600 mmol·L-1NaCl溶液胁迫后,转HAL1植株叶绿素含量反而高于400 mmol·L-1NaCl溶液的处理。证明ScHAL1可以提高棉花耐盐性。
References
[1] BOYER J S. Plant productivity and environment. American Association for the Advancement of Science,1982,218: 443-448.
[2] FLOWERS T J,YEO A R. Breeding for salinity resistance in crop plants: Where next Australian. Australian Journal Plant Physiology,1995,22: 875-884.
[3] NELSON D E,SHEN B,BOHNERT H J. Salinity tolerancemechanisms,models and the metabolic engineering of complex traits. Genetic Engineering,1998,20: 153-176.
[4] 郭三堆,倪万潮,徐琼芳. 编码杀虫蛋白融合基因和表达载体及其应用: 中国,ZL95119563.8. GUO S D,NI W C,XU Q F. Encoding insecticidal protein fusion gene and expression vectors and its application: China,ZL95119563.8. (in Chinese)
[5] YU L H,WU S J,PENG Y S,PENG Y S,LIU R N,CHEN X,ZHAO P,XU P,ZHU J B,JIAO G L,PEI Y,XIANG C B. Arabidopsis EDT1/HDG11 improves drought and salt tolerance in cotton and poplar and increases cotton yield in the field. Plant Biotechnology Journal,2016,14: 72-84.
[6] 张慧军,董合忠,石跃进,陈受宜,朱永红. 山菠菜胆碱单加氧酶基因对棉花的遗传转化和耐盐性表达. 作物学报,2007,33(7): 1073-1078. ZHANG H J,DONG H Z,SHI Y J,CHEN S Y,ZHU Y H. Transformation of cotton (Gossypium hirsutum L.) with AhCMO gene and the expression of salinity tolerance. Acta Agronomica Sinica,2007,33(7): 1073-1078. (in Chinese)
[7] PASAPULA V,SHEN G X,KUPPU S,VALENCIA J P,MENDOZA M,HOU P,CHEN J,QIU X Y,ZHU L F,ZHANG X L,AULD D,BLUMWALD M,ZHANG H,GAXIOLA R,PAYTON P. Expression of an Arabidopsis vacuolar H+-pyrophosphatase gene (AVP1) in cotton improves drought- and salt tolerance and increases fibre yield in the field conditions. Plant Biotechnology Journal,2011,9: 88-99.
[8] SERRANO R,GAXIOLA R. Microbial models and salt stress tolerance in plants. Critical Reviews in Plant Sciences,1994,13: 121-138.
[9] GAXIOLA R,DE LARRINOA I F,VILLALBA J M,SERRANO R. A novel and conserved salt-induced protein is an important determinant of salt tolerance in yeast. The EMBO Journal,1992,11: 3157-3164.
[10] RIOS G,FERRANDO A,SERRANO R. Mechanisms of salt tolerance conferred by overexpression of the HAL1 gene in Saccharomyces cerevisiae. Yeast,1994,13: 515-528.
[11] PARK J M,PARK C J,LEE S B,HAM B K,SHIN R,PAEK K H. Overexpression of the tobacco Tsi1 gene encoding an EREBP/AP2-type transcription factor enhances resistance against pathogen attack and osmotic stress in tobacco. The Plant Cell,2001,13: 1035-1046.
[12] WINICOV I. Alfin1 transcription factor overexpression enhances plant root growth under normal and saline conditions and improves salt tolerance in alfalfa. Planta,2000,210: 416-422.
[13] WINICOV I,BASTOLA D R. Transgenic overexpression of the transcription factor Alfin1 enhances expression of the endogenous MsPRP2 gene in alfalfa and improves salinity tolerance of the plants. Plant Physiology,1999,120: 473-480.
[14] XU D P,DUAN X L,WANG B Y,HO T H,WU R. Expression of a late embryogenesis abundant protein gene,HVA1,from barley confers tolerance to water deficit and salt stress in transgenic rice. Plant Physiology,1996,110: 249-257.
[15] BORDAS M,MONTESINOS C,DABAUZA M,SALVADOR A,ROIG L A,SERRANO R,MORENO V. Transfer of the yeast salt tolerance gene HAL1 to Cucumis melo L. cultivars and in vitro evaluation of salt tolerance. Transgenic Research,1997,6: 41-50.
[16] GISBERT C,RUS A M,BOLARIN M C,CORONADO J.M L,ARRILLAGA I,MONTESINOS C,CARO M,SERRANO R,MORENO V. The yeast HAL1 gene improves salt tolerance of transgenic tomato. Plant Physiology,2000,123: 393-402.
[17] Yang S X,Zhao Y X,Zhang Q,HE Y K,ZHANG H,LUO D. HAL1 mediate salt adaptation in Arabidopsis thaliana. Cell Research,2001,11: 142-148.
[18] Ellul P,RIOS G,ATARES A,ROIG L A,SERRANO R,MORENO V. The expression of the Saccharomyces cerevisiae HAL1 gene increases salt tolerance in transgenic watermelon [Citrullus lanatus (Thunb.)Matsun & Nakai]. Theoretical and Applied Genetics,2003,107: 462-469.
[19] 刘艳芝,韦正乙,邢少辰,谭化. 高明,董英山. HAL1基因转化苜蓿再生植株及其耐盐性. 吉林农业科学,2008,33: 21-24. LIU Y Z,WEI Z Y,XING S C,TAN H,GAO M,DONG Y S. Transgenetic alfalfa with HAL1 gene and its salt tolerance. Journal of Jilin Agricultural Sciences,2008,33: 21-24. (in Chinese)
[20] RUS A M,ESTAN M T,GISBERT C,SOGO B G,SERRANO R,CARO M,MORENO M,BOLARIN M C. Expressing the yeast HAL1 gene in tomato increases fruit yield and enhances K+/Na+selectivity under salt stress. Plant Cell and Environment,2001,24: 875-880.
[21] 于志晶,王岭,金永梅,林秀峰,马瑞. 酵母 HAL1基因转化水稻及其耐碱性研究. 吉林农业科学,2014,39: 17-20. YU Z J,WANG L,JIN Y M,LIN X F,MA R. Studies on transgenic rice with HAL1 gene and its tolerance to alkali. Journal of Jilin Agricultural Sciences,2014,39: 17-20. (in Chinese)
[22] 孔静静. 藻类耐盐相关基因的克隆及转化棉花的初步研究[D]. 开封: 河南大学,2013. KONG J J.Gene cloning of salt tolerance on algae and its preliminary research of transformation on cotton [D]. Kaifeng: Henan University,2013. (in Chinese)
[23] 周凯. 陆地棉耐盐相关基因(GhPTAC、GhGnT)克隆及其表达分析[D]. 北京: 中国农业科学院,2011. ZHOU K. Cloning and expression analysis of salt-tolerance related genes (GhPTAC、GhGnT) on Gossypium hirsutum L.[D]. Beijing: Chinese Academy of Agrecultural Sciences,2011. (in Chinese)
[24] 张莹. 转 TsVP-AtNHX1基因棉花的耐盐性研究[D]. 济南: 山东大学,2014. ZHANG Y. The study of salt tolerance in cotton co-expressing TsVP and AtNHX1[D]. Jinan: Shandong University,2014. (in Chinese)
[25] YAN J,SONG W N,EVIATAR N. A MAPK gene from Dead Sea fungus confers stress tolerance to lithium salt and freezing-thawing: Prospects for saline agriculture. Proceedings of the National Academy of Sciences of the USA,2005,102: 18992-18997.
[26] DIETMAR K. Evolution of the cellular stress proteome: From monophyletic origin to ubiquitous function. The Journal of Experimental Biology,2003,206: 3119-3124.
[27] ZHAO F Y,GUO S L,ZHANG H,ZHAO Y X. Expression of yeast SOD2 in transgenic rice results in increased salt tolerance. Plant Science,2006,170: 216-224.
[28] 张丽青. 嗜热毛壳菌超氧化物歧化酶基因的克隆、表达及转基因烟草耐盐性研究[D]. 泰安: 山东农业大学,2011. ZHANG L Q. Cloning,expression of Superoxide Dismutase (SOD)gene on Chaetomium thermophilum CT2 and salt resistance of transgenic tobacco[D]. Taian: Shandong Agricultural University,2011. (in Chinese)
[29] 谢丽霞. 极端嗜盐曲霉核糖体蛋白基因SpRPS3ae和SpRPL44的克隆及其抗逆功能研究[D]. 长春: 吉林农业大学,2013. XIE L X. Cloning and stress resistance analys of ribosomal protein genes (SpRPS3ae and SpRPL44 ) in extreme halotolerant Aspergillus sp[D]. Changchun: Jilin Agricultural University,2013. (in Chinese)
[30] 张荃,王淑芳,赵彦修,赵可夫,张慧. HAL1基因转化番茄及耐盐转基因番茄的鉴定. 生物工程学报,2001,17(6): 659-662. ZHANG Q,WANG S F,ZHAO Y X,ZHAO K F,ZHANG H. HAL1 gene transform into tomatoe and salt-tolerance identification of transgenic tomato. Chinese Journal of Biotechnology,2001,17(6): 659-662. (in Chinese)
(责任编辑 李莉)
The Function Expression of Salt-Tolerant Yeast Gene Halotolerance ( HAL1 ) in Cotton
MU Min,SHU Na,WANG Shuai,GUO Li-xue,FAN Wei-li,YIN Zu-jun,WANG Jun-juan,WANG De-long,YE Wu-wei
(Institute of Cotton Research,Chinese Academy of Agricultural Sciences/State Key Laboratory of Cotton Biology/Key Laboratory for Cotton Genetic Improvement,Ministry of Agriculture,Anyang 455000,Henan)
【Objective】 The objective of this study is to clone Saccharomyces cerevisiae halotolerance (ScHAL1) gene and transformed into cotton ,explore the function of the gene in cotton,to further explore function of the salt-tolerant yeast genes in higher plants. 【Method】According to total length of mRNA sequence information of ScHAL1 in NCBI,the gene was cloned using RT-PCR technology from Saccharomyces cerevisiae As2.375,double enzyme digestion was made with XbaIand SmaIforpBI121::GFP,and pBI121-ScHAL1::GFP fusion expression vector was constructed by In-Fusion technique. With weak auto-fluorescence upland cotton varieties,Y-2067,ZA-23 and GZ-2 pollen as materials,using the gene bombarding technique to study transient expression of cotton pollen. The expression vector pBI121-ScHAL1::GFP was transformed into cotton salt-sensitive material Zhong s9612 with gene gun in vivo conversion technology,T0generation cotton genetically seeds were modified. Solution with 100 mmol·L-1NaCl was used to test the salt resistance of seeds in the transgenic T0generation seed germination experiment,molecular detection was carried out,and semal salt resistance of transgenic plants was analysis. 【Result】 ScHAL1 cloned from Saccharomyces cerevisiae As2.375 and ScHAL1 is 885 bp in length,which encoding 294 amino acids. After its sequence analysis,it was found that the largest proportion of the whole HAL1 protein is serine,and it is alkaline and positively charged and it is a hydrophilic protein. According to the results of protein secondary structure prediction,it is speculated that the structure of the protein function domain may mainly made up of random curl and beta sheet. Cotton pollen instantaneous expression results showed that after conversion of HAL1 gene,three land cotton powder green fluorescence were obviously enhanced,suggesting that the gene expressed in these three upland cotton pollen. With 100 mmol·L-1NaCl,transgenic T0generation seed germination ability obviously stronger than receptor s9612 selfing seed material,which preliminary showed that HAL1 could also improve the seed salt resistance. According to the gene nucleotide sequences,two pairs of primers were designed for molecular detection of T0generation. Direct sequencing of PCR product was conducted and the sequencing results preliminarily evidence that the transgene is successful. With the semal salt resistance analysis,it was found that the chlorophyll contents of transgenic plants in 600 mmol·L-1NaCl and 400 mmol·L-1NaCl were higher than the control,and the chlorophyll contents of transgenic plants in 600 mmol·L-1NaCl were higher than that in 400 mmol·L-1NaCl. 【Conclusion】HAL1 gene was Successfully cloned from Saccharomyces cerevisiae,yeast HAL1 gene plays an important role in improving cotton salt resistance.
yeast; HAL1; upland cotton; pollen instantaneous expression; molecular detection
2016-02-15;接受日期:2016-05-23
国家转基因生物新品种培育科技重大专项(2014ZX0800504B)
联系方式:穆敏,Tel:15890348192;E-mail:15890348192@163.com。通信作者叶武威,Tel:0372-2562283;E-mail:yew158@163.com
