黄酒酿造过程中真菌群落组成及挥发性风味分析
2016-09-07牟穰毛健孟祥勇刘芸雅粮食发酵工艺与技术国家工程实验室江南大学江苏无锡214122江南大学食品学院江苏无锡214122国家黄酒工程技术研究中心浙江绍兴312000
牟穰,毛健*,3,孟祥勇,3,刘芸雅(1.粮食发酵工艺与技术国家工程实验室,江南大学,江苏无锡214122;2.江南大学食品学院,江苏无锡214122;3.国家黄酒工程技术研究中心,浙江绍兴312000)
黄酒酿造过程中真菌群落组成及挥发性风味分析
牟穰1,2,毛健*1,2,3,孟祥勇1,2,3,刘芸雅1,2
(1.粮食发酵工艺与技术国家工程实验室,江南大学,江苏无锡214122;2.江南大学食品学院,江苏无锡214122;3.国家黄酒工程技术研究中心,浙江绍兴312000)
利用宏基因组学技术对不同发酵时期黄酒醪液中的真菌进行鉴定,并利用GC-MS分析黄酒中的主要高级醇、醛类和酯类等挥发性风味物质。结果表明:黄酒前酵阶段真菌有着较高的群落多样性,在发酵排料当天鉴定出22种真核属类;其中曲霉属(Aspergillus)在整个发酵过程相对丰度均达到90%以上,远远高于其它真菌,是黄酒发酵过程中的特征微生物;随着黄酒发酵的进行,优势真菌呈现先降低后增加趋势。挥发性风味物质的测定结果表明,在发酵过程中挥发性物质的生成受发酵液性质影响较大,大部分在第4天有着较高的含量,随着发酵的进行,发酵液的较高酸度和酒精度以及低pH降低了微生物的多样性,使得挥发性风味物质含量有所降低。关键词:麦曲;发酵醪液;真菌;多样性;挥发性风味物质
黄酒是我国特有的民族特产,在中国已有数千年的历史,它主要以谷物(稻米、小米、黍米、玉米等)为原料,加入麦曲、酵母等进行发酵而成。由于原料、地理环境和酿造工艺的不同,不同区域的黄酒均呈现出独特的风味和口感,按照黄酒风格分为传统型黄酒、清爽型黄酒和特型黄酒[1]。黄酒发酵过程中存在着复杂的微生物群落,包括细菌以及霉菌、酵母菌等真菌。它们不仅能够产各种酶类,降解大分子物质,还能够促进黄酒风味物质的代谢合成,包括有机酸、游离氨基酸、酯类等,赋予了黄酒丰富的营养成分和独特的风味特征[2-3]。
微生物是黄酒酿造过程中必不可少的组成成分,对微生物群落的解析有助于了解黄酒发酵机理。目前对黄酒微生物的分析大多采用传统培养方法,但这种方法存在弊端,如采用分离、纯化、分子鉴定步骤,需经过一系列繁杂的形态特征和生理生化试验[4-5]。同时利用培养基方法仅能够筛选出少量微生物,大部分微生物因不能培养而遗漏,造成检测的微生物群落结构不准确。
随着分子生物学技术的发展,越来越多的研究人员使用非纯培养的方法和手段对环境微生物进行全面分析,如16SrRNA克隆建库[6]、条形码测序[7]以及基因芯片[8]等技术已被广泛应用于环境微生物多样性的研究。高通量测序(High throughput sequencing,HTS)是一种全新的测序技术,它能一次进行多个样品测定,还能一次并行对几十万到几百万条DNA分子进行序列测定,给研究提供了方便[9]。通量高、快速、低成本等优点,使得该方法一经出现就被广泛应用于各种生态系统中,比如土壤[10]、草鱼肠道[11]、海洋沉积物[12]等生态环境中微生物多样性的检测和研究中。近年来,HTS也逐渐应用到传统发酵食品中微生物的多样性研究,如泡菜[13]、干酪[14]、白酒[15]、豆瓣酱[16]等发酵食品,而应用于黄酒中的真菌研究还未见报道。
已发表的有关黄酒微生物的研究主要针对细菌,对于发酵液中的真菌研究较少。然而,真菌在黄酒酿造过程中能产生大量酶类,对黄酒发酵起到十分重要的作用。为探明发酵过程中发酵液风味物质及理化指标的代谢规律,解析黄酒麦曲及发酵醪液中真菌的群落结构,采用宏基因组学对海派清爽型黄酒(上海金枫黄酒)酿造用麦曲和发酵过程中的真菌群落结构进行分析,同时对发酵液风味物质及理化指标进行测定,旨在为解析黄酒酿造过程提供一定的理论依据。
1 材料与方法
1.1实验样品
黄酒发酵醪液,取自上海金枫酒业股份有限公司,样品发酵天数分别为0、2、4、6、9、12、18 d和24 d,0 d表示投料当天;黄酒生麦曲,取自企业麦曲仓库,为2014年生产。所有样品当天采集完后放入-20℃冰箱保存备用。
1.2样品宏基因组DNA提取
称取不同天数发酵醪液和麦曲样各1 g,采用玻璃珠破碎法对样品进行破碎处理,用Soil DNA kit试剂盒(OMEGA,Bio-Tek,USA)对样品进行宏基因组DNA提取,提取方法按说明书进行。
1.3目的区段扩增及高通量测序
扩增选取真菌18S rRNA基因中的ITS1F-ITS2区段作为扩增对象,采用引物ITS1F:CTTGGTCATT TAGAGGAAGTAA和2043R:GCTGCGTTCTTCATC GATGC进行扩增[17-18]。此外,用于PCR扩增的正向引物前加入一段由7个碱基构成的barcode序列,所有样品分别用不同barcode序列进行区分,具体扩增体系及程序如下。
1.3.1PCR反应体系PCR试验采用Trans Gen AP221-02:TransStartFastpfuDNAPolymerase (Trans Gen Biotech,China);共20 μL的反应体系:5×FastPfu Buffer 4 μL,2.5 mmol/L dNTPs 2 μL,正反向引物各(5 μmol/L)0.8 μL,FastPfu Polymerase 0.4 μL,模板DNA 10 ng,剩余的用ddH2O补足至20 μL。
1.3.2PCR扩增程序PCR仪采用ABI Gene Amp○R 9700(Applied Biosystems,USA),其反应参数为:1)1×(95℃3 min);2)30×(95℃30 s,55℃30 s,72℃45 s);3)72℃延伸10 min。得到的PCR产物经2 g/dL的琼脂糖凝胶电泳检测后,送至上海美吉生物医药科技有限公司进行Illumina双末端测序分析。
1.4黄酒发酵醪液挥发性风味物质测定
采用顶空固相微萃取技术(HS-SPME)与GCMS相结合测定黄酒发酵醪液中的挥发性物质。
1.4.1HS-SPME条件取4 mL稀释后的黄酒发酵液,加入到20 mL的顶空瓶中,加1.0 g NaCl和20 μL内标(2-辛醇,质量浓度为8 800 μg/L),使用50/30 μm DVB/CAR/PDMS萃取头,50℃预热15 min,萃取吸附40 min,250℃解吸7 min,用于GC-MS分析。
1.4.2 GC条件色谱柱TG-WAXMS(30 m×0.25 μm× 0.25 mm);载气为高纯氦气(纯度>99.999%),不分流,体积流量为1.0 mL/min;进样口温度250℃;程序升温,起始温度40℃,保持3 min,以6℃/min的速率升温至100℃,然后以10℃/min升温至230℃,保持7 min。
1.4.3MS条件离子化方式EI;发射电流50 μA;电子能量70 eV;离子源温度230℃;传输线温度250℃;扫描质量范围33~400 u。
1.4.4物质的定性分析未知化合物的质谱通过与NIST05a.L Database(Agilent Technologies Inc)进行比对,并通过计算保留指数(RI)和查阅已经报道文献中相应物质RI确认。物质的定量分析采用半定量分析。
1.5黄酒醪液理化指标测定
黄酒醪液中氨基酸态氮、总酸及pH测定方法参照与黄酒相关的国家标准[1]。
1.6数据分析
用Mothur软件包以97%为划定阈值对序列划分操作分类单元(OTU);利用得出的OTU结果计算样品中微生物的α-多样性,包括Chao 1指数、ACE指数、Shannon指数、Simpson指数等。此外,为尽量减少错误测序,确保测序质量,在测序前需要将各序列的引物和barcode去掉,并对序列长度进行筛选[12]。删掉长度小于50 bp的序列,以及含有其他不明确的碱基(如N)和含有同聚体的区段(重复次数大于6)。试验结果均采用均值±标准差的形式。
2 结果与分析
2.1宏基因组DNA提取
采用玻璃珠破碎法对样品进行破碎处理后,提取样品中微生物的宏基因组,用1 g/dL的琼脂糖凝胶电泳进行分析,结果如图1所示。

图1 黄酒发酵醪液及麦曲微生物宏基因组DNA琼脂糖凝胶电泳图谱Fig.1 Agarose gel electrophoresis of metagenomic DNA extracted from ferment mash and wheat koji
从图1可以看出,玻璃珠破碎细胞提取宏基因组DNA时产生了很多不同大小的片段,造成电泳时的弥散拖尾现象。但在靠近图谱上方有着较为清晰的目的条带,几乎能保持在同一水平,说明样品中目的DNA被提取出,可以进行下一步的切胶回收实验。从图谱中还可以看出,随着发酵的进行,亮度有所降低,这说明微生物多样性随着发酵的进行而降低。
2.2序列及α-多样性分析
对不同发酵期的麦曲(9个样品)进行测序后共获得179 330条序列,平均每个样品获得19 926条序列。样品的多样性指数结果见表1。
从表1可以看出,麦曲的Shannon指数最高,达到1.42,是发酵投料当时的4.5倍,是发酵第24天时的35.5倍;从Simpson指数来看,发酵液样品均达到0.9以上,而麦曲仅为0.4,表明麦曲中的微生物多样性要高于发酵期间醪液中的微生物多样性。同时从Coverage指数来看,每个样品的数值均达到0.999以上,说明利用HTS技术对麦曲及发酵液进行高通量测序得到的数据准确可靠。
从表1中OTU数量可以看出,黄酒麦曲及投料当天的发酵液中真菌多样性较为复杂,分别达到39种和33种,而随着发酵的进行,发酵醪液中微生物种类呈递减趋势,微生物多样性越来越低,发酵醪液中真核群落变得更加简单。这是由于真菌大部分都是好氧菌,贮存期间麦曲与空气充分接触,同时麦曲中含有充足的营养物质,使麦曲中的微生物能够充分生长,因此显示出较高的微生物多样性。

表1 样品多样性指数Table 1 Alpha-diversity of samples
在黄酒发酵启动后,由于营养物质的消耗,以及发酵产生的CO2使得微生物处于缺氧的环境,同时发酵液中微生物产生的代谢产物改变了发酵醪液的理化特性,如pH降低、酒精度升高等,这些都对真菌生长产生抑制作用,从而降低了真菌的多样性。
2.3发酵过程中真菌群落结构
对黄酒麦曲、前酵开始阶段(0、2 d)和后酵开始阶段(6 d)发酵液中的真菌群落组成进行鉴定分析(表2)。结果表明,在黄酒麦曲中一共鉴定出16种不同的真核属类,其中相对丰度大于1.00%的有5种,分别为曲霉属(Aspergillus,72.55%),链格孢属(Alternaria,6.00%),嗜热真菌属(Thermomyces,2.37%),镰刀菌属(Fusarium,1.69%)和附球菌属(Epicoccum,1.43%)。而前酵开始阶段鉴定出22种不同的真菌属类,数量上相对于麦曲增加了6种。这是由于黄酒投料时真菌不仅由麦曲带入,加入的米浆水、酒母和酿造器具等也会带入其他微生物,同时由于发酵初期氧含量比较充足,微生物所处的环境较为适宜,醪液的理化特性还没有发生很大改变,对真菌生长的抑制作用并不明显,此时发酵醪液中的微生物多样性相对复杂。
2.4优势真菌属变化情况分析
随着发酵的进行,优势真菌呈现先减少后稍有上升的变化趋势,特别是第2天的真核群落多样性在整个发酵阶段是最低的,真核数量急剧下降到4种(图2所示)。当黄酒发酵启动后,微生物开始发酵并释放代谢产物,其中有些物质是对微生物生长有害的,如酵母菌发酵产生的酒精对自身和其他真菌有着很大的毒害作用,抑制了真菌的生长[19];同时一些微生物代谢产生的有机酸也使得发酵液酸度增大,像曲霉菌代谢出柠檬酸,细菌中醋酸菌及乳酸菌分别产生的醋酸、乳酸等[20]。微生物在较高酸和酒精度环境中生长受到抑制,使其群落多样性降低。

表2 黄酒发酵过程中真菌群落组成分布Table2Distributionofeukaryoticmicroorganism communities during rice wine brewing

图2 不同发酵时期真菌在属水平上的数目变化情况Fig.2 Changes of the eukaryotic microorganism in different fermentation periods at genus level
到了发酵后期真核种类稍有上升,在第12天时优势微生物从6种上升到10种;发酵液中特征微生物曲霉属相对丰度增大,从第9天的98.54%上升到第12天时的99.27%。其他一些糖化真菌也在后酵阶段出现,如根霉属、毛霉属等。这是由于到了后酵时期整个发酵液温度下降,酵母和一些细菌代谢活动变缓,而此时的氧气量相对前酵阶段比较充足,一些霉菌能够在低温和低酸下存活而不受抑制[21]。像根霉、毛霉等,它们能够产生淀粉酶、蛋白酶等物质,降解发酵液中残余物质产生可供代谢的营养物质,如糖类、蛋白质等,微生物利用这些营养物质而生长,重新使得发酵醪液的真菌多样性增大[22-23]。
2.5发酵液挥发性物质变化情况
利用GC-MS对黄酒发酵过程中的挥发性风味物质进行鉴定,一共得到60种化合物,其中高级醇类12种,酯类化合物34种,醛类化合物7种,其他类如呋喃、吡嗪等共7种,其中主要挥发性物质变化情况如图3所示。

图3 发酵液主要挥发性风味物质变化情况Fig.3 Changes of the main volatile flavor compounds in ferment mash
从图3可以看出,主要高级醇变化趋势较为一致,在前酵阶段均随着发酵时间进行,含量不断增加;到了后酵阶段,3种高级醇增长变缓,都在第12天时达到最大值,随后含量稍有降低并保持稳定。主要醛类物质中,苯甲醛含量在整个发酵过程中一直呈现增长趋势,苯乙醛和壬醛呈现出先增加后降低的趋势,其中苯乙醛在第2天达到最大值,壬醛含量在第9天达到最大值,随后随着发酵的进行,化合物含量维持在较为稳定的状态。主要挥发性酯类中,己酸乙酯和乙酸乙酯呈现出先增加后稳定的趋势,而乙酸异戊酯含量在前4 d上升到最大值,在第6天下降后又上升,之后其含量呈降低趋势。
高级醇是黄酒发酵过程中酵母合成细胞蛋白质的副产物,主要由酵母产生[24]。在黄酒的前酵阶段,由于发酵醪液中具有较多的营养物质,适宜的环境使得酵母产生酒精和大量的高级醇;醇被氧化成醛,使得醛类物质相应增加;有机酸和高级醇发生酯化脱水产生酯,使得发酵液中酯类含量增加。到了后期,由于温度降低,微生物产酸也使得发酵液变成了较高酸度、高酒精度环境,大量微生物生长受到抑制,酵母产酒精能力也受到影响,因此在后酵时产高级醇能力降低,相应的醛类和酯类化合物生成量也就降低了。
2.6发酵液理化指标变化情况
从图4可以看出,在前酵阶段,总酸增加明显,第4天达到最大值,随后保持不变;氨基酸态氮呈现出缓慢的增长趋势;发酵液pH在第4天最低,总体上呈现先降低后稳定的变化趋势。
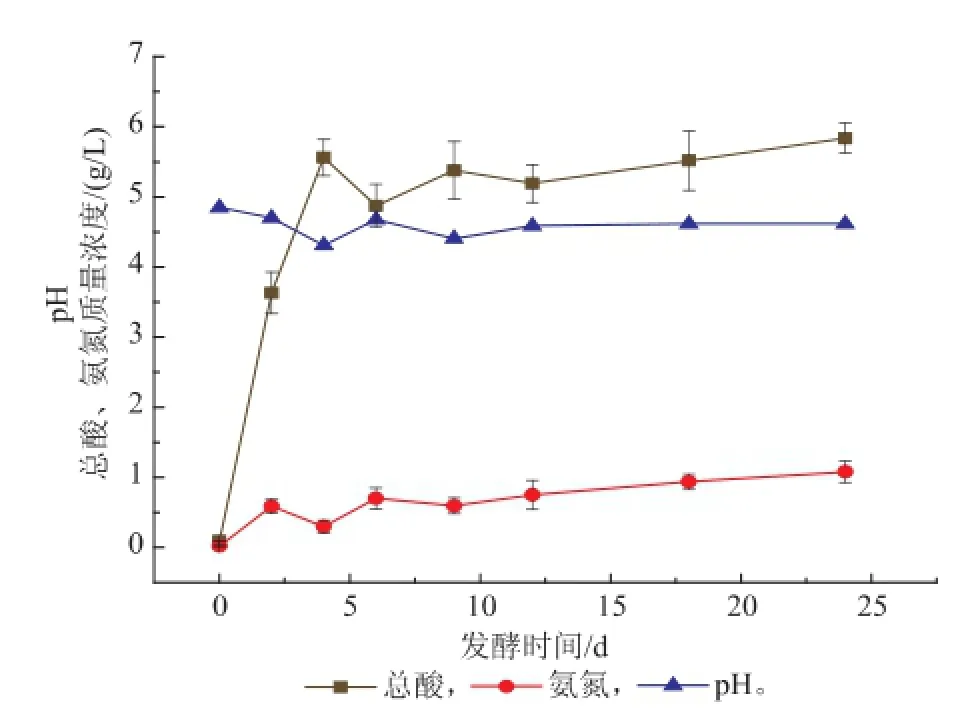
图4 发酵液理化指标变化情况Fig.4 Changes of the physical and chemicalproperties in ferment mash
黄酒中的有机酸主要是乳酸、醋酸和琥珀酸,其中非挥发性的乳酸和琥珀酸是导致黄酒回味的主要物质[25]。由于在发酵前期,充足的养分和空气,使得产酸微生物大量生长,微生物代谢出大量有机酸,使得发酵液总酸增大,这时候的pH也随之降低;到了发酵后期,由于发酵液不适合微生物生长,一些产酸微生物生长受到抑制,因而总酸和pH能够保持较为稳定的状态。氨基酸主要是由生物合成和转氨基作用产生,这个过程需要利用微生物产生的酶来合成或转化。由于大部分合成酶及转氨酶都是从麦曲中带入,并且在发酵过程中含量保持较为稳定,因而酶催化产生的氨氮能够稳定增长。
3 结语
采用宏基因组学的方法分析了清爽型黄酒发酵过程中真菌群落组成,发现在整个黄酒发酵过程中曲霉属具有较高的相对丰度,并一直以优势真菌存在于整个黄酒发酵过程中。采用GC-MS对发酵液挥发性风味物质进行测定,结果表明高级醇呈现出先升高后保持稳定的趋势,醛类和酯类物质大多呈现出先增加后降低的变化规律。在发酵前期,发酵液中含有大量的营养物质,此时的环境适合微生物的繁殖,因而发酵液中微生物不断生长并保持较高的多样性和活性,微生物代谢合成的风味物质含量也随着升高。随着发酵的进行,由于一些产酸微生物产生大量有机酸,同时酵母发酵产生了酒精,使得发酵醪液变成高酸、高酒精度环境,造成大量的微生物不适应环境而消失或丰度降低。利用宏基因组学技术能够快速准确地解析黄酒发酵过程中微生物的群落结构,同时跟踪主要微生物的变化情况,这对于解析黄酒发酵过程中微生物的动态变化以及指导黄酒生产,具有重要的理论意义和实践价值。
本文主要从属水平对各种微生物种类及相对丰度进行了研讨,主要是对黄酒发酵醪液中的真菌进行鉴定。后续研究中,可对发酵液的理化性质进一步研究,如营养物质分布、溶氧等,为解释微生物群落结构变化奠定理论基础。
[1]GB/T 13662-2008黄酒[S].北京:中国标准出版社,2008.
[2]罗涛,范文来,徐岩.中国黄酒中挥发性和不挥发性物质的研究现状与展望[J].酿酒,2007(1):44-48. LUO Tao,FAN Wenlai,XU Yan.The review of volatile and non-volatile compounds in Chinese Rice Wine[J].Liquor Making,2007(1):44-48.(in Chinese)
[3]寿虹志,凌志勇,杨旭,等.浅析黄酒麦曲中的微生物与黄酒风味的关系[J].中国酿造,2007(8):55-57. SHOU Hongzhi,LING Zhiyong,YANG Xu,et al.Relationship between metabolites of wheat Qu microorganisms and flavor of rice wine[J].China Brewing,2007(8):55-57.(in Chinese)
[4]曹钰,陆健,方华,等.绍兴黄酒麦曲中真菌多样性的研究[J].食品科学,2008(3):277-282. CAO Yu,LU Jian,FANG Hua,et al.Fungal diversity of wheat Qu of Shaoxing Rice Wine[J].Food Science,2008(3):277-282.(in Chinese)
[5]Shang Y,Chen L,Zhang Z,et al.A comparative study on the fungal communities of wheat Qu for Qingshuang-type Chinese Rice Wine[J].Journal of the Institute of Brewing,2012,118(2):243-248.
[6]Han Y,Li L,Liu J.Characterization of the airborne bacteria community at different distances from the rotating brushes in a wastewater treatment plant by 16S rRNA gene clone libraries[J].Journal of Environmental Sciences,2013,25(1):5-15.
[7]Cheng C,Zaichao Z,Aizhong D,et al.Bar-Coded pyrosequencing reveals the bacterial community during microcystis water bloom in guanting reservoir,Beijing[J].Procedia Engineering,2011,18:341-346.
[8]Hazen T C.The super chip for microbial community structure,and function from all environments[J].Microbial Biotechnology,2013,6(5):450-452.
[9]Metzger J,Tonda R,Beltran S,et al.Next generation sequencing gives an insight into the characteristics of highly selected breeds versus non-breed horses in the course of domestication[J].Bmcgenomics,2014,15(1):1-13.
[10]Zhao J,Zhang R,Xue C,et al.Pyrosequencing reveals contrasting soil bacterial diversity and community structure of two main winter wheat cropping systems in China[J].Microbial Ecology,2014,67(2):443-453.
[11]Wu S,Tian J,Gatesoupe F,et al.Intestinal microbiota of gibel carp(Carassius auratus gibelio)and its origin as revealed by 454 pyrosequencing[J].World Journal of Microbiology&Biotechnology,2013,29(9):1585-1595.
[12]Zhu D,Tanabe S,Yang C,et al.Bacterial community composition of South China Sea sediments through Pyrosequencing-Based analysis of 16S rRNA genes[J].PLoS ONE,2013,8(10):1-9.
[13]Park E,Chun J,Cha C,et al.Bacterial community analysis during fermentation of ten representative kinds of kimchi with barcoded pyrosequencing.[J].Food Microbiology,2012,30(1):197-204.
[14]Aldrete-Tapia A,Escobar-Ramírez M C,Tamplin M L,et al.High-throughput sequencing of microbial communities in Poro cheese,an artisanal Mexican cheese[J].Food Microbiology,2014,44:136-141.
[15]Wu Q,Chen L,Xu Y.Yeast community associated with the solid state fermentation of traditional Chinese Maotai-flavor liquor[J]. International Journal of Food Microbiology,2013,166(2):323-330.
[16]Nam Y,Lee S,Lim S.Microbial community analysis of Korean soybean pastes by next-generation sequencing[J].International Journal of Food Microbiology,2012,155(1-2):36-42.
[17]Orgiazzi A,Lumini E,Nilsson R H,et al.Unravelling soil fungal communities from different mediterranean Land-Use backgrounds[J].PLoS ONE,2012,7(4):1-9.
[18]Bruns T D,Gardes M.ITS primers with enhanced specificity for basidiomycetes-application to the identification of mycorrhizae and rusts[J].Molecular Ecology,1993,2(2):113-118.
[19]杨鲁君.优良黄酒酵母菌株的筛选、鉴定和发酵特性研究[D].杭州.浙江工商大学,2013.
[20]赵佳,雷振河,吕利华,等.汾酒产酸细菌的分离鉴定及其产酸条件研究[J].食品与发酵工业,2009(12):43-46. ZHAO Jia,LEI Zhenghe,LV Lihua,et al.Identify acidogenic bacterium and study on the acidogenic condition of the fen liquor [J].Food and Fermentation Industries,2009(12):43-46.(in Chinese)
[21]毛青钟,鲁瑞刚,陈宝良,等.黄酒发酵醪抑制霉菌生长的初步探讨[J].中国酿造,2006(9):56-59. MAO Qingzhong,LU Ruigang,CHEN Baoliang,et al.Study on fermented mash of rice wine on inhibition to mold[J].China Brewing,2006(9):56-59.(in Chinese)
[22]刘芳,曹新志,任晓华.甜酒曲华根霉淀粉酶菌株紫外线诱变育种[J].中国酿造,2009(7):78-80. LIU Fang,CAO Xinzhi,REN Xiaohua.Brewing of rhizopus chinensis amylase strain from sweet-liqueur Daqu by ultraviolet radiation[J].China Brewing,2009(7):78-80.(in Chinese)
[23]李理,罗泽民,卢向阳.毛霉产蛋白酶的特性及其应用研究[J].湖南农业大学学报.1999(3):216-219. LI Li,LUO Zemin,LU Xiangyang.Studies on the properties of mucor producing proteinase and mucor protease application[J]. Journal of Hunan Agricultural University,1999(3):216-219.(in Chinese)
[24]谭檑华.黄酒固定化发酵及其高级醇含量的控制研究[D].杭州:浙江工商大学,2013.
[25]冯德明,张洋,赵惠明,等.反相高效液相色谱法测定黄酒中的有机酸[J].中国酿造,2009(3):157-161. FENG Deming,ZHANG Yang,ZHAO Huiming,et al.Determination of organic acids in rice wine by reverse phase high performance liquid chromatography[J].China Brewing,2009(3):157-161.(in Chinese)
Analysis of Fungi Diversity and Volatile Flavor Compounds in Chinese Rice Wine Fermentation Process
MOU Rang1,2,MAO Jian*1,2,3,MENG Xiangyong1,2,3,LIU Yunya1,2
(1.National Engineering Laboratory for Cereal Fermentation Technology,Jiangnan University,Wuxi 214122,China;2.School of Food Science and Technology,Jiangnan University,Wuxi 214122,China;3.National Engineering Research Center of Chinese Rice Wine,Shaoxing 312000,China)
As the Chinese national specialty,Chinese rice wine has a long history and unique production and fermentation technology,the microbial community and metabolites contribute to the unique flavor during the rice wine brewing.In this study,the metagenome technology was applied to explore the fungi diversity and the GC-MS was used to analyze the volatile flavor compounds in ferment mash of different fermentation periods.The results showed that the ferment mash had higher fungi diversity at the initial stage,22 genera were identified in 0 day.The relative abundance of Aspergillus were all above 90%,which was much higher as compared with other eukaryotic microorganisms during the fermentation process.The advantageous fungi changed obviously,the genera number decreased at first four days and increased later.The analyzed of volatile flavor compounds showed that the generation of volatile substances were greatly influenced by physical and chemical properties of fermented mash.High acidity,alcohol and low pH reduced the fungidiversity,and lowered the contents of volatile flavor compounds.
wheat koji,ferment mash,fungi,diversity,volatile flavor compounds
TS 262.4
A
1673—1689(2016)03—0303—07
2014-11-25
国家863计划项目(2013AA102203-06)。
毛健(1970—),男,安徽宿州人,工学博士,教授,主要从事食品生物技术研究。E-mail:biaomao@263.net
