基于ISSR的硬叶兜兰居群遗传多样性研究
2016-09-02李宗艳管名媛李名扬
李宗艳,管名媛,李 静,李名扬
(1 西南林业大学 园林学院, 昆明 650224;2 西南大学 园艺园林学院,重庆 400715)
基于ISSR的硬叶兜兰居群遗传多样性研究
李宗艳1,管名媛1,李静1,李名扬2*
(1 西南林业大学 园林学院, 昆明 650224;2 西南大学 园艺园林学院,重庆 400715)
利用ISSR标记对7个硬叶兜兰居群160个体扩增,10对引物共扩增出101个条带,其中多态性条带97个,种水平上的多态性条带比率(PPB)达96.02%,香侬指数(I)为0.488 9,Nei’s基因多样性指数(H)为0.332 4。居群水平的遗传参数,多态性条带比率(PPB)为 33.51%,香侬指数(I)为0.194 3,Nei’s基因多样性指数(H)为0.133 2。总的遗传多样性(HT)为0.323 9,个体遗传多样性(Hs)达到0.133 2。 AMOVA结果揭示居群间遗传分化大,居群间基因流Nm为0.349 2。居群间的Nei’s遗传距离0.148 1~0.4385。基于UPGMA聚类,在遗传距离为0.327时,7个居群聚为两支, 第一支由古林箐和马固、田坝、夹寒箐和杨柳井居群组成,第二支由斗咀和小坝子居群组成,聚类关系反映居群间遗传分化与地理距离无显著相关性。
硬叶兜兰;遗传多样性;遗传分化;遗传距离;亲缘关系
硬叶兜兰(Paphiopedilummicranthum)分布于云南东南、贵州西南和北部、广西西部的亚热带石灰岩山区[1-2],因其具有较高的观赏价值,自20世纪80年代发现以来,种质资源大量被采挖,被CITES列为一级保护植物,禁止贸易。
作为珍稀花卉,当前主要开展了硬叶兜兰繁殖、传粉生物学、育种和遗传多样性等研究[3-9]。硬叶兜兰主要依靠蜂类和食蚜蝇传粉[3],自然状态下,硬叶兜兰结实率和萌发率较低,人工授粉后种子萌发率最大可达32.4%[4];因其具有较高的观赏特性,硬叶兜兰参与许多园艺栽培品种的杂交,如著名的魔鬼灯笼P.magicLantern(硬叶兜兰×德氏兜兰)[5];硬叶兜兰植株形态多样,花器官变异较大,如花色、花瓣、中萼和合萼的分化系数较其它器官高[6];李昂等[7]分析4个硬叶兜兰居群遗传多样性,结果表明:其物种水平的遗传多样性高于居群水平,有20.3% 的遗传多样性来自于居群间的分化,居群间遗传分化水平高于7个云南居群的SRAP数据的结果[8];进一步的空间自相关研究表明:硬叶兜兰群体中遗传变异在空间上的分布呈斑块状,在短距离内表现出显著的正相关,在较大距离内表现出显著的负相关[9]。
ISSR(inter simple sequence repeat) 称为简单重复序列区间扩增多态DNA ,实验操作简单快速[10],自创建以来,在居群遗传多样性和遗传结构分析中显示了突出的优势[11-13]。滇东南的石灰岩地区极可能是兜兰的起源中心和演化中心,亦是国内硬叶兜兰采集压力最大的区域,采用ISSR技术对该地区居群遗传多样性进行分析,为其保护提供理论依据具有现实意义。
1 材料和方法
1.1材料收集
本研究材料采集选择文山、马关和麻栗坡3县7个居群,每居群按随机采样原则,为避免无性克隆个体,每个单株取样距离在2 m以上,7个居群共160个样本(表2),置于保温箱中带回实验室于-70 ℃超低温冰箱保存。
1. 2引物筛选和PCR扩增
采用改良的1.5×CTAB法提取硬叶兜兰基因组总DNA,DNA经0.8%琼脂糖凝胶电泳检测后,稀释为30 ng/μL在-20 ℃保存备用。
实验选用的ISSR引物是由加拿大哥伦比亚大学(UBC)开发设计的77个引物,从中选取10对能稳定扩增、产生条带清晰丰富、多态性较好的引物组合进行遗传多样性的分析。优化的PCR扩增反应体系为:2.0 mmol/L MgCl2、dNTPs 0.40 mmol/L、模板质量浓度为5.0 ng/μL、1.25 U/20μLTaqDNA聚合酶、引物质量浓度0.35 μmol/L。ISSR-PCR扩增程序为:94 ℃预变性5 min,94 ℃变性45 s,48~52 ℃退火45 s,72 ℃延伸90 s,35个循环,72 ℃延伸10 min,4 ℃保存。扩增产物经2.0%琼脂糖凝胶电泳检测。
1.3数据处理
条带统计时将具有相同迁移率的扩增片段,按照0/1编码,应用POPGENE 1.32软件[14,15]进行多态位点百分率(PPB)、观测等位基因数(Na)、有效等位基因数(Ne)、Nei’s基因多样性指数(H)、Shannon信息指数(I)、群体总的遗传多样性(HT)、群体内基因多样性(Hs)、群体间的遗传分化系数(Gst)、Nei’s遗传距离(D)和基因流(Nm)计算。根据Nei’s遗传距离,利用NTSYS-pc1.21[16]软件对居群进行UPGMA居群间亲缘关系分析。分子遗传变异的方差分析(AMOVA)用GenALEX软件计算[17]。
2 结果与分析
2.1引物筛选结果
采用优化的ISSR-PCR扩增反应体系,对77个ISSR引物扩增有产物的有42个,从中筛选出10个扩增稳定性较高、多态性较好和条带清晰的引物进行居群遗传多样性分析(表1)。引物扩增条带最多为12条,为引物855、856和840,多数引物扩增条带为8~10条,有6个引物扩增多态百分率达100%。

表1 不同引物的扩增效果

表2 基于ISSR数据的硬叶兜兰7个居群的遗传多样性指数
注:Na.观测等位基因数;Ne.有效等位基因数;H.Nei’s基因多度;I.Shannon’s 指数;PPB. 多态性百分率
Note:Na. Observed number of alleles;Ne. Effective number of alleles;H. Nei gene diversity;I. Shannon’s information index;PPB. The percentage of polymorphic bands
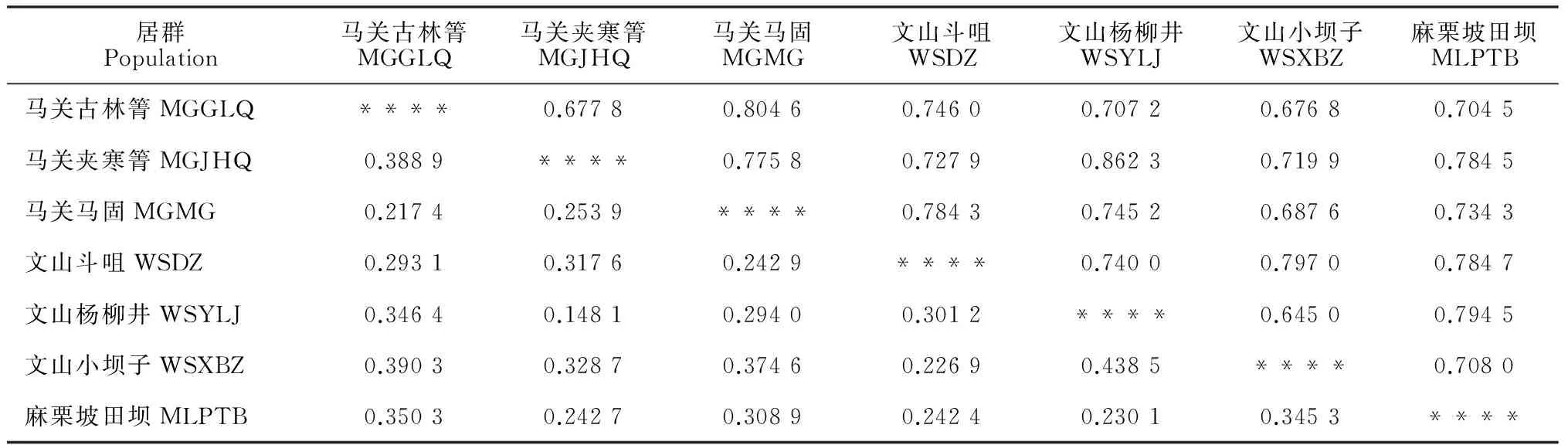
表3 硬叶兜兰7个居群的Nei’s遗传一致度(对角线上方)和遗传距离(对角线下方)
2.2硬叶兜兰的遗传多样性
10个引物共扩增出101个条带(表2),其中多态性条带97个,多态条带比率达96.04%,每个引物平均扩增产生10.1个条带。古林箐和马固居群多态性百分率较高,分别为40.59%和36.63%,夹寒箐和杨柳井居群的多态性百分率最小,仅有28.71%。7个居群的有效等位基因数(Ne)在1.180 1~1.268 2之间;Nei’s基因多度(H)变幅为0.102 0~0.151 9,Shannon指数(I)值在0.151 6~0.224 1。小坝子和田坝居群的遗传多样性相差不大,夹寒箐居群内的遗传多样性较低(PPB=28.71%;H=0.102 0;I=0.151 6)。古林箐居群的遗传多样性水平最高,Shannon指数(I)为0.224 1。在居群水平的Shannon指数和Nei’s基因多度为0.194 3和0.133 2,在物种水平的分别为0.488 9和0.324 4。总的遗传多样性(HT)为0.323 9, 居群内的遗传多样性(Hs)为0.133 2。
2.3居群间遗传距离与亲缘关系
依据显示的居群间的Nei’s遗传距离(表3),近距离居群的小坝子和杨柳井间的遗传距离最大,为0.438 5, 而夹寒箐和杨柳井居群间有最小遗传距离, 为0.148 1,7个居群间平均遗传距离为0.299 2。基于UPGMA聚类(图1),在遗传距离达0.327时,7个居群聚为两支,第一支由两个分支组成,第一分支由古林箐和马固居群组成,第二分支由田坝、夹寒箐和杨柳井居群组成,第二支由斗咀和小坝子居群组成,结果证实了居群间遗传距离与地理距离无显著相关性。

图1 硬叶兜兰7个居群的亲缘关系聚类Fig. 1 Dendrogram analysis showing relationships among seven populations
2.4硬叶兜兰居群间的遗传分化
群体间遗传分化系数是是衡量遗传多样性的变异来源的指标[18],遗传分化的高低表明居群间的变异和分化水平[19-20]。Gst、PhiPT是不同软件计算的遗传分化指数。
首先,7个居群间的遗传分化系数(Gst)达0.588 8,揭示了居群间存在较高的遗传分化水平。AMOVA揭示有39.60%的分子遗传变异来源于居群间,这一结果稍高于黄家林等报道的结果[21],小于与Gst的分化水平,与PhiPT值近相同。而居群间平均基因流(Nm)仅为0.349 2。
其次,同一地区间居群的遗传分化系数(PhiPT)相差较大,文山小坝子与斗咀间的PhiPT值为0.304,斗咀与杨柳井的为0.430, 小坝子与杨柳井间的遗传分化系数达0.492,平均遗传分化系数达0.409;而马关马固和古林箐仅为0.319,古林箐与夹寒箐的PhiPT为0.472,马固与夹寒箐之间为0.381。田坝居群与其余6个居群间PhiPT值在0.338~0.413之间, 与斗咀分化最小,与古林箐最大。7个居群两两间遗传分化最小的为杨柳井和夹寒箐,达0.278;最大的为杨柳井和小坝子,达0.492。7个居群间平均PhiPT值为0.392。居群间PhiPT值反映出的遗传分化程度与Nei’s距离关系是一致的。尽管杨柳井和斗咀居群为近缘居群,但居群两两间的平均遗传分化系数却较高,说明相同地区近距离居群间亦存在较高的遗传分化,反映了居群间遗传分化水平与地理距离无明显相关性。
3 讨 论
3.1硬叶兜兰居群的遗传多样性
一般认为,广布种都比窄布种具有较高的遗传多样性,濒危植物有较低的遗传多样性,遗传多样性越低的植物越濒危[22-23]。但越来越多的研究表明,濒危植物也有很高的遗传多样性。采用ISSR技术对珊瑚菜(Glehnialittoralis)、毛瓣金花茶(Camelliapubipetala) 濒危种遗传多样性水平的检测显示,在种和居群水平都保持较高的遗传多样性,居群间遗传分化也不高[24-25]。
采用ISSR技术检测兰科濒危种的研究表明,流苏石斛(Dendrobiumfimbriatum)在种水平的遗传多样性不低,但居群水平的遗传多样性较低[26]。本项目对7个硬叶兜兰居群的ISSR检测结果表现出物种水平的遗传多样性较高(PPB=96.04%,H=0.324 4,I=0.488 9),居群水平的遗传多样性均高于上述种(PPB=33.51%,H=0.133 2,I=0.194 3)。物种总的遗传多样性(HT)为0.323 9,居群内的遗传多样性仅为0.133 2。此结论与SRAP数据和李昂的初步检测结论是一致的[8-9],即硬叶兜兰种水平遗传多样性远高于居群水平,但与黄家林等报道的较高水平的居群遗传多样性(PPB=80.28%,H=0.284 7,I=0.423 6)结果有较大出入[21]。
珍稀濒危物的遗传多样性与很多因素有关,如从祖先广布种的物种中分化而来、分布范围、生境条件、繁育系统等。Hamrick[22]认为居群遗传变异大小的主要影响因素依次为繁育系统、分布范围和生活型,广布、异交和靠动物传播种子的物种往往具有较高的遗传多样性。硬叶兜兰能保持种较高水平的遗传多样性与其异交、长命草本、种子传播特性、分布生境的多样密切相关。
3.2硬叶兜兰居群的遗传分化
一般认为,PhiPT分化指数若在0~0.05之间说明居群间分化很弱,若在0.05~0.15之间说明居群间存在中等程度分化,若在0.15~0.25之间说明居群间分化大,若PhiPT大于0.25表明居群间分化极大[27]。7个硬叶兜兰居群的分化指数(PhiPT)平均水平达0.392,充分说明硬叶兜兰居群间分化大,与AMOVA结果近相同,低于基于Nei’s指数的Gst(0.588 8),接近于相同地区铁皮石斛居群间Gst(0.563 7),远高于硬叶兜兰在云贵桂交界区的Gst水平[21]。基于硬叶兜兰的ISSR分子标记,利用不同的分析方法(PhiPT,AMOVA)所得的遗传分化值基本一致。研究表明:异交类群的平均基因遗传分化系数Gst为0.23,混交类群Gst为0.19,自交类群Gst为0.59[28]。硬叶兜兰属于异花传粉植物,居群间的遗传分化显著。
物种的繁育系统是影响居群水平上的遗传分化水平的重要因素,Avise[29]认为由弱飞行能力的动物授粉和靠种子迁移的植物居群之间的遗传分化显著。硬叶兜兰主要由蜂类传粉[3],其飞行距离有限,主要是进行居群内开花植株之间的授粉,造成遗传变异主要来源于居群内,而居群间的花粉流是非常有限的;再者,由于硬叶兜兰种子萌发特性具有强烈的生境选择特点,易导致物种地理间断性分布,造成居群间的地理距离较远。一般认为,地理隔离会引起近交,而近交繁殖方式将引起近交衰退和降低群体间的遗传多样性,同样也会降低群体间基因交流,加剧群体间的分化[30-32]。居群遗传学研究中,基因流根据Nm的大小划分为高(≥1.0)、中(0.250~0.99)、低(0.0~0.249)三个等级水平[33]。若按此标准划分,此次研究的硬叶兜兰居群间基因流处于低水平,仅为0.349 2,相同的ISSR技术揭示的云南、贵州和广西交界区域的硬叶兜兰的基因流也仅有0.7201[21]。一般而言,在选择作用不明显时,只要有很少量的基因流便可有效地阻止群体分化[34],可判断基因流不是造成硬叶兜兰居群间遗传分化显著的主要因素。
有越来越多的研究证明,遗传变异主要是由选择、突变、基因流和遗传漂变等产生,而空间距离并不一定产生遗传变异[35]。彼此相距很近的群体即使存在频繁的基因流动,强有力的选择作用也能使得群体间产生遗传上的变异和分化。
有学者[35]认为遗传距离与地理距离之间缺乏显著相关性意味着遗传漂变在种群间的分化中起着不可忽视的作用。在人为干扰严重的硬叶兜兰居群中,居群个体主要依赖无性克隆产生,导致居群中有效个体数量下降形成小居群,则更易加大发生遗传漂变的机率;此次供试斗咀、小坝子和杨柳井居群为小居群,大概也增加了居群间的遗传分化。再者,在人为干扰小的居群中,开花植株少和个体数量减少,易造成居群内花粉流水平下降和结实率降低,居群内遗传多样性水平下降和遗传变异变小,而居群间的种子流有限则也加剧居群间的遗传分化。
[1]陈心启,吉占和. 中国兰花全书[M]. 北京: 中国林业出版社, 2000: 123-124.
[2]CRIBB P, LUO Y B, SIU G. .PaphiopedilummicranthuminNorth-east Guizhou, a significant range extension[J].OrchidReview, 2002, 43: 40-43.
[3]BANZIGER H. Studies on the natural pollination of three species of wild lady slipper orchids (Paphiopedilum) in Southeast Asia[C]// PRIDGEON A. Proceedings of the 14th World Orchid Conference. HMSO, Edinburgh, UK, 1994: 201-202.
[4]陈之林, 叶秀粦,梁承邺, 等. 杏黄兜兰和硬叶兜兰的种子试管培养[J]. 园艺学报, 2004,31(4): 540-542.
CHEN Z L, YE X L, LIANG C Y,etal. Seed germinationinvitroofPaphiopedilumarmeniacumandP.micranthum[J].ActaHorticulturaeSinica,2004,31(4): 540-542.
[5]曾宋君, 田瑞雪, 陈之林, 等. 兜兰属植物杂交育种研究进展[J]. 热带亚热带植物学报, 2010, 18(2): 459-468.
ZENG S J, TIAN R X, CHEN Z L,etal. Research progress on cross breeding ofPaphipedilum[J].JournalofTropicalandSubtropicalBotany, 2010,18(2):459-468.
[6]李宗艳,李名扬. 硬叶兜兰居群表型变异研究[J]. 植物遗传资源学报,2015, 16(4): 765-771.
LI Z Y, LI M Y. Study on phenotypic variation ofPaphiopedilummicranthumpopulation[J].JournalofPlantGeneticResources,2015,16(4):765-771.
[7]LI A, LUO Y B, GE S. A preliminary study on conservation genetics of an endangered orchid (Paphiopedilummicranthus) from southwestern China[J].BiochemicalGenetics,2002,40: 195-201.
[8]李宗艳,李静,曾万标,等. 滇东南硬叶兜兰核心种质区群体遗传结构研究[J]. 植物遗传资源学报, 2013, 14(3): 407-413.
LI Z Y, LI J, ZENG W B,etal. Genetic structure ofPaphiopedilummicranthumpopulations from core distribution in Southeast Yunnan[J].JournalofPlantGeneticResources, 2013, 14(3): 407-413.
[9]李昂,罗毅波, 葛颂. 采用空间自相关分析研究两种兰科植物的群体遗传结构[J]. 生物多样性, 2002,10(3):249-254.
LI A, LUO Y B, GE S. Spatial autocorrelation study of population genetic structure of two orchid species[J].BiodiversityScience,2002,10(3):249-254.
[10]ZIETKIEWICZ E, FAFALSKI A, LABUDA D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification[J].Genomics,1994,20(2):176-183.
[11]钱韦, 葛颂, 洪德元. 采用RAPD和ISSR标记探讨中国疣粒野生稻的遗传多样性[J]. 植物学报, 2000,42(7):741-750.
QIAN W, GE S, HONG D Y. Assessment of genetic variation ofOryzagranulatedetected by RAPDs and ISSRs[J].ActaBotanicaSinica,2000,42(7):741-750.
[12]杨淑达, 施苏华, 龚洵, 等. 滇牡丹遗传多样性的ISSR分析[J]. 生物多样性, 2005,13(2): 105-111.
YANG S D, SHI S H, GONG X.etal. Genetic diversity ofPaeoniadelavayiasrevealed by ISSRs[J].BiodiversityScience,2005,13(2):105-111.
[13]金则新, 李钧敏, 李增鸿. 浙江仙居长叶榧自然居群遗传多样性的ISSR分析[J]. 北京林业大学学报, 2007, 29(1):53-59.
JIN Z X, LI J M, LI Z H. ISSR analysis of the genetic diversity ofTorreyajactiiChun natural populations in Xianju County,Zhejiang Province[J].JournalofBeijingForestryUniversity,2007,29(1):53-59.
[14]NEI M. Genetic distance between populations[J].AmericanNaturalist, 1972,106(949):283-292.
[15]NEI M. Analysis of gene diversity in subdivided populations[J].ProceedingsoftheNationalAcademyofSciences, 1973,70(12):3 321-3 323.
[16]PEAKALL R, SMOUSE P E. GENALEX 6: Genetic analysis in Excel. Population genetic software for teaching and research[J].MolecularEcologyNotes, 2006,6(1):288-295.
[17]ROHLF F J. NTSYS-pc: Numerical Taxonomy and Multivariate Analysis System[M]. New York: Applied Biostatistics Incorporated,1988:24-32.
[18]FISCHER M, HUSI R, PRATI D,etal. RAPD variation among and within small and large populations of the rare clonal plantRammculusreptans(Ranunculaceae)[J].AmericanJournalofBotany,2000, 87(8):1 128-1 137.
[19]GROOM M J, MEFFE G K, CARROLL C R. Principles of Conservation Biology[M]. Sunderland, Massachusetts: Sinauer Associates Incorporated,2005:85-97.
[20]HARTL D L, CLARK A G. Principles of Population Genetics[M]. Sunderland, Massachusetts: Sinauer associates Incorporated,1997:67-69.
[21]黄家林,李树云,胡虹.珍稀濒危植物硬叶兜兰的遗传多样性及遗传结构研究[J].植物分类与资源学报, 2014, 36(2):209-218.
HUANG J L, LI S Y, HU H. ISSR and SRAP markers reveal genetic diversity and population structure of an endangered slipper orchid,Paphiopedilummicranthum(Orchidaceae)[J].PlantDiversityandResources,2014,36(2):209-218.
[22]HAMRICK J L. Plant population genetics and evolution[J].AmericanJournalofBotany,1982, 69(10):1 685-1 693.
[23]洪德元,葛颂,张大明,等. 植物濒危机制研究的原理和方法[M]. 北京:中国科学技术出版社, 1995:125-133.
[24]王爱兰,王贵琳,李维卫. 濒危物种珊瑚菜遗传多样性的ISSR分析[J]. 西北植物学报,2015, 35(5): 1 541-1 546.
WANG A L, WANG G L,LI W W. Genetic diversity ofGlehnialittoralispopulations revealed by ISSR molecular markers[J].ActaBotanicaBoreali-OccidentaliaSinica,2015,35(5):1 541-1 546.
[25]柴胜丰,庄雪影,邹蓉,等. 濒危植物毛瓣金花茶遗传多样性的ISSR分析[J]. 西北植物学报, 2014,34(1): 93-98.
CHAI S F,ZHUANG X Y,ZOU R,etal. Genetic diversity analysis of endangered plantCamelliapubipetaladetected by ISSR[J].ActaBotanicaBoreali-OccidentaliaSinica,2014,34(1): 93-98.
[26]马佳梅, 殷寿华. 西双版纳地区流苏石斛遗传多样性的ISSR分析[J]. 云南植物研究, 2009,31(1): 35-41.
MA J M, YIN S H. Genetic diversity ofDendrobiumfimbriatum(Orchidaceae), an endangered species, detected by inter-simple sequence repeat (ISSR)[J].ActaBotanicaYunnanica,2009,31(1): 35-41.
[27]BUSO G S, RANGEL P H, FERREIRA M E. Analysis of genetic variability of South American wild rice populations(Oryzaglumaepatula)with isozymes and RAPD markers[J].MolecularEcology,1998,7(1):107-117.
[28]NYBOM H, BARTISH I V. Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants[J].PerspectivesinPlantEcology,EvolutionandSystematic,2000,3(2):93-114.
[29]AVISE J C. Molecular Markers, Natural History and Evolution[M]. New York: Chapman & Hall, 1994:125.
[30]HAMRICK J L. Gene Flow and Distribution of Genetic Variation in Plant Populations[M]. New York: Academes Press,1987:87-103.
[31]BROWN A. Genetic Characterization of Plant Mating Systems. Plant Population Genetics, Breeding, and Genetic Resources[M]. Sunderland, Massachusetts: Sinauer Associates Incorporated,1990:145-162.
[32]URBANSKA K M. Differentiation Patterns in Higher Plants[M]. New York: Academes Press,1987:57-76.
[33]GOVINDARAJU D R. Relationship between dispersal ability and levels of gene flow in plants[J].Oikos,1988, 52(1):31-35.
[34]FISCHER M. Species loss after habitat fragmentation[J].TrendsinEcologyandEvolution,2000,15(10):396-404.
[35]FISCHER M, MATTHIES D. RAPD variation in relation to population size and plant fitness in the rareGentianellagermanica(Gentianaceae)[J].AmericanJournalofBotany,1998,85(6): 811-819.
(编辑:宋亚珍)
Genetic Diversity ofPaphiopedilummicranthumDetected by ISSR Data
LI Zongyan1, GUAN Mingyuan1, LI Jing1, LI Mingyang2*
(1 Landscape and Architecture Faculty, Southwest Forestry University, Kunming 650224, China; 2 Horticulture and Landscape College, Southwest University, Chongqing 400715, China )
Paphiopedilummicranthumhas the high ornamental value known as slipper orchids, which was restrictedly distributed in karst limestone areas of southeast Yunnan, southwest Guizhou, northern and western Guanxi Province. This study focused on the genetic diversity of 7 populations from Southeast Yunnan detected by ISSR markers. 101 bands were generated from 160 individuals by 10 primers, among which 97 bands belonged to polymorphic and each primer amplified 10.1 bands. There was a higher genetic diversity index at species level (PPB=96.02%;I=0.488 9;H=0.332 4), but a lower genetic diversity index at population level (PPB=33.51%;I=0.194 3;H= 0.133 2). Total gene diversity reached 0.323 9 and gene diversity within populations attained to 0.133 2. Nei’s genetic differetation among populations (Gst)was 0.148 1-0.438 5. Gene flow (Nm) was 0.349 2. These indicated that there was a higher population differentiation among populations. Based on the UPGMA cluster dendrogram, the seven populations were classfied into two groups at genetic distance of 0.327. First group included Gulinqing, Magu, Tianba, Jiahanqing and Yangliujing; Second group was made up by Douzui and Xiaobazi populations. The cluster result indicated that there was no significant correlation between genetic distance and geographical distance.
Paphiopedilummicranthum; genetic diversity; genetic differentiation; genetic distance; blood relationship
1000-4025(2016)07-1351-06
10.7606/j.issn.1000-4025.2016.07.1351
2016-04-21;修改稿收到日期:2016-06-13
云南省高校优势特色生态学重点学科建设项目(05000511311);云南省优势特色重点学科生物学一级学科建设项目(50097501)
李宗艳(1974- ), 女,博士, 副教授,从事濒危植物保护研究。E-mail: lizyan74@sina.com
李名扬,男,博士,教授,从事植物分子生物学研究。E-mail: limy@swu.edu.cn
Q746+.5
A
