基于宏基因组比较分析石斑鱼育苗两种模式水体中的菌群多样性和分布特点
2016-08-24孙敏秋林克冰黄种持郑乐云
孙敏秋,林克冰,葛 辉,黄种持,郑乐云,周 宸
(福建省水产研究所,福建省海洋生物增养殖与高值化利用重点实验室,福建 厦门 361013)
基于宏基因组比较分析石斑鱼育苗两种模式水体中的菌群多样性和分布特点
孙敏秋,林克冰,葛辉,黄种持,郑乐云,周宸
(福建省水产研究所,福建省海洋生物增养殖与高值化利用重点实验室,福建 厦门 361013)
为比较分析石斑鱼育苗新旧模式水体中菌群的分布差异性,研究应用高通量测序技术测定六批次(A1、A2、B1、B2、C1和C2)水样的菌群16S rDNA V4变异区序列,并使用Qiime和Mothur等软件整理和统计样品序列数目和操作分类单元(OTUs)数量,分析物种的丰度、分布和Alpha多样性以及物种丰度的差异性。结果显示,获得用于分析的序列和OTU数为667 27/3 724(A1)、66 418/3 391(A2)、87 403/3 980(B1)、57 519/3 438(B2)、62 349/3 530(C1)和66 673/3 004(C2);稀疏曲线表明测序深度充分,OTU的数量接近实际情况。A1、A2、B1、B2、C1和C2 六个样品的丰富度指数分别为6 535.21/8 872.16、6 583.85/8 855.40、8 062.60/10 640.49、6 878.85/9 352.25、6 859.85/9 295.45和5 291.87/6 868.28,多样性指数分别为0.05/4.72、0.04/4.77、0.04/4.69、0.04/4.74、0.05/4.45和0.08/4.06。传统模式育苗场暴发石斑鱼病毒性神经坏死病的同一时期,对两种模式育苗水体中的菌群在门、属水平分别进行单样品物种丰度、多样品物种分布和物种丰度差异性分析。在门水平,两种育苗模式育苗水体的优势菌群分布相似,但在属水平,传统模式育苗水体的优势菌群中易致病菌假单胞菌属含量显著高于新模式育苗水体。获得了暴发病毒性神经坏死病前后,石斑鱼育苗新旧两种模式共6批次育苗水样的细菌均匀度、丰富度和菌群结构,推测石斑鱼“生态优化及病害防控人工育苗创新模式”可能有利于抑制致病菌,富集益生菌。
斜带石斑鱼;育苗创新模式;细菌菌群;宏基因组学
石斑鱼(Epinephelussp.)是一种重要的名贵海水鱼类,其肉质鲜美、营养丰富、深受人们喜爱。21世纪初,石斑鱼苗种的人工繁育进入蓬勃发展时期,然而病毒性神经坏死病(Viral nervous necrosis,VNN)的出现和肆虐严重阻碍了石斑鱼养殖产业的发展,神经坏死病毒(Nervous necrosis virus,NNV)已成为现今石斑鱼育苗过程中危害最大的病原[1]。由于至今,仍未能找到有效治疗病毒性神经坏死病的方法,因此,现阶段病毒防控成为苗种培育的关键[2]。福建省水产研究所石斑鱼课题组多年来致力于新型石斑鱼育苗养殖模式研发,总结各方经验,在传统育苗模式基础上趋利避害,建立“石斑鱼生态优化及病害防控人工育苗”创新模式,旨在由受精卵发育到大规格苗种的过程中,从养殖水环境、饵料、日常用具、管理等各个环节对神经坏死病毒病进行有效的防控[3]。
研究表明,细菌环境与疾病的产生存在相关性。Boissiere等[4]揭示了按蚊肠道共生菌可影响疟疾病原体(疟原虫)的发育。Menno R.等[5]发现,婴幼儿患上呼吸道疾病与同时存在于上呼吸道的多种细菌和病毒的相互作用有关。因此,研究推测石斑鱼患神经坏死病毒病并不是一个单一的过程,该病毒病的暴发也许跟水体及周围细菌的分布存在着一定的联系,水体中细菌的结构种类可能促进或抑制神经坏死病毒病的发生。为证实这一点,研究比较分析了石斑鱼“生态优化及病害防控人工育苗创新模式”(以下简称新模式)与传统育苗模式下育苗水体中的菌群分布特点。
长久以来,微生物研究都依靠实验室培养,这一局限性被宏基因组学(Metagenomics)的出现打破。宏基因组学诞生于20世纪90年代[6-8],通过直接从环境样品中提取全部微生物的总DNA,构建宏基因组文库,利用高通量测序技术等策略研究环境样品所包含的全部微生物的遗传组成及其群落功能,使对占微生物总体99%以上不可培养微生物的研究成为现实[9]。研究者可通过对宏基因组文库进行分析,探讨在各种环境下微生物间相互作用和微生物与周围环境间相互影响的规律[10]。南春燕等[11]就曾采用宏基因组方法研究了中华按蚊幼虫肠道细菌的组成;也有研究者利用宏基因组高通量测序技术分析污水及饮用水中的细菌群落结构[12-13]。但目前,采用宏基因组研究水产生物的报道还不多见。邢孟欣[14]采用高通量Solexa测序技术分析了养殖大菱鲆(Scophthalmusmaximus)肠道微生物的多样性及其功能;杨章武等[15]利用宏基因组测序技术分析凡纳滨对虾(Litopenaeusvannamei)育苗中生物絮团的细菌群落结构。本文是首次于易发病毒性神经坏死病的石斑鱼育苗过程中,采用基于细菌16S rDNA[16-17]的宏基因组学高通量测序技术对比分析石斑鱼育苗新模式与传统模式间育苗水体的菌群多样性和分布特点,了解石斑鱼育苗新旧模式间细菌的物种丰富度和均匀度,以期为进一步探讨石斑鱼病毒性神经坏死病的暴发与育苗水体中细菌组成分布的相互关系,以及验证“石斑鱼生态优化及病害防控人工育苗”创新模式的确实可行性和高效性积累研究数据。
1 材料和方法
1.1样品采集与处理
水样分别取自漳州市诏安县的两个斜带石斑鱼(Epinepheluscoioides)育苗场——诏安大华水产有限公司(“石斑鱼种业创新与产业化工程建设”项目育苗新模式示范基地)和另一传统模式育苗场。依次于2013年3月27日(A)、4月11日(B)、4月27日(C)分三批次分别采集两个场的育苗水样,每次每场采五个育苗池,每池等量采集五个点的水样混合成一个样,每个水样的采集量为5 L(其中4月11日,传统育苗场暴发神经坏死病毒病,大华场正常。)。采集的水样先依次经由2 μm、0.8 μm滤膜除去杂质,再经0.22 μm滤膜富集水样中的细菌,将附有细菌的滤膜匀浆,以备提取细菌的基因组DNA。
1.2主要试剂、材料和仪器
细菌基因组快速提取试剂盒购自上海生物工程技术公司,操作步骤参照试剂盒说明书。采用超微量分光光度计(Nano drop 2000,美国Thermo Scientific公司)和琼脂糖凝胶电泳对提取的细菌基因组DNA进行质量检测。MisSeq宏基因组测序和生物信息学部分由上海派森诺生物科技有限公司利用测序仪(MisSeq System SY-410-1003,美国Illumina公司)完成。
1.3细菌MiSeq宏基因组测序和生物信息学分析
扩增细菌的16S rDNA-V4区片段引物如下:正向引物5-AYTGGGYDTAAAGNG-3′;反向引物 5′- TACNVGGGTATCTAATCC-3′。PCR产物胶回收后进行定量,通过3′→5′核酸外切酶和聚合酶的共同作用,修复带有突出末端的DNA片段,在3′端引入单碱基“A-”,与3′端含有单碱基“T-”的接头连接,利用PCR选择性富集连有接头的DNA片段,同时扩增DNA文库,定量并质检后,逐步稀释定量上机测序,按照美国Illumina公司标准流程进行。对测得的原始数据进行质量控制,舍弃低质量序列,应用Flash(V1.0.3)软件连接序列,过滤连接的序列(连续相同碱基<6,模糊碱基N<1),获得最终用于分析的序列。用软件Qiime,将相似度大于0.97的序列归为1个操作分类单元(Operational taxonomic unit,OTU),统计各样品所含OTU的数量,去除无法分类的OTU后进行后续分析,绘制稀疏曲线。基于物种丰度的分析,包括应用Mothur软件计算Alpha多样性指数中的丰富度(Chao/Ace)、覆盖率(coverage)和多样性(Simpson/Shannon)。基于群落结构分析,在门和属两个分类水平上统计单样品的物种丰度;在门和属水平统计多样品的物种分布;分析物种丰度差异性时在门和属水平,先将原始丰度归一到同数量级,以log2(样品1/样品2)计算倍数差异,差异倍数>1和<-1者,均被认为差异具有显著性。
2 结果
2.1水样细菌宏基因组DNA质量
分别提取新模式石斑鱼育苗场水样(按时间依次编号为A1、B1、C1)和传统模式育苗场水样[按时间依次编号为A2、B2(暴发神经坏死病毒病)、C2]共六批次的细菌基因组DNA,其质量均符合宏基因组测序的基本要求(表1、图1)。
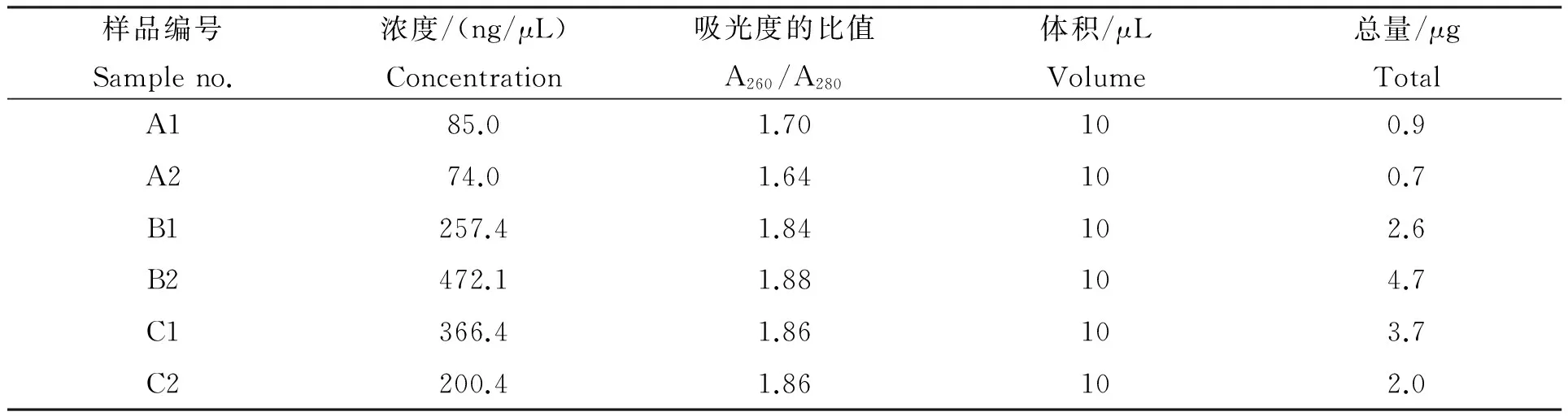
表1 水样细菌宏基因组DNA浓度质量检测结果
2.2水样细菌物种丰度分析
2.2.1样品序列数目和稀疏曲线
六个水样获得最终用于分析的序列条数分别为66 727(A1)、66 418(A2)、87 403(B1)、57 519(B2)、62 349(C1)和66 673(C2)。对比分析各序列,分别得到的OTU数目为3 724(A1)、3 391(A2)、3 980(B1)、3 438(B2)、3 530(C1)和3 004(C2)。
从稀疏曲线看,总体的趋势为:在<10 000条序列时,OTU数量随着样品的序列数增加而迅速增加;在10 000~30 000条序列时,OTU数目增加缓慢;之后则趋于平台期。本研究中的六个水样均趋于平台期(图2)。
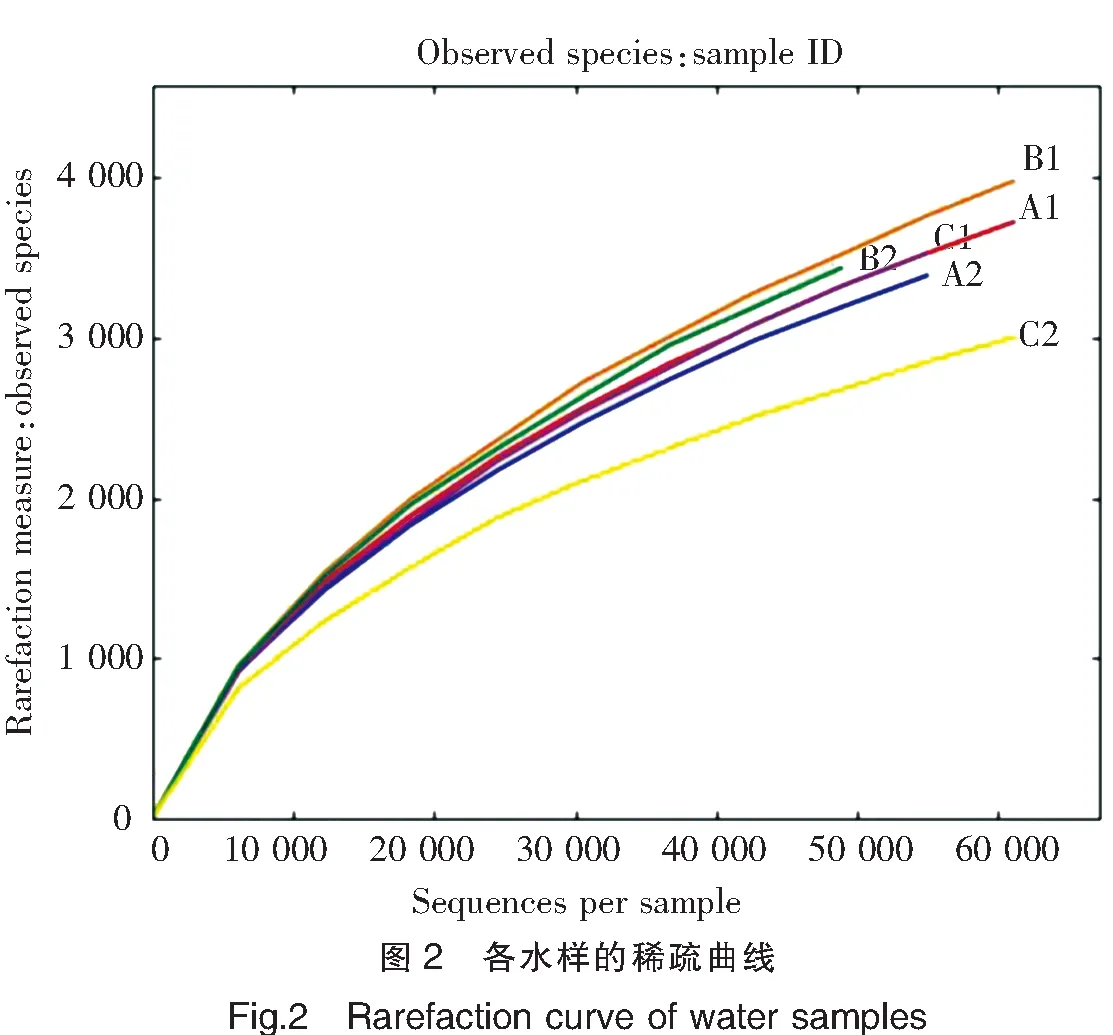
注:横坐标:各水样序列数;纵坐标:OTU数量。
Notes:X-axis:No.sequences per water sample;Y-axis:No.OTUs.
2.2.2Alpha多样性
Alpha多样性是指一个特定区域或生态系统内的多样性,经常用物种丰富度来度量。在OTU水平,A1、A2、B1、B2、C1和C2等六个水样的丰富度指数(Chao/Ace)分别为6 535.21/8 872.16、6 583.85/8 855.40、8 062.60/10 640.49、6 878.85/9 352.25、6 859.85/9 295.45和5 291.87/6 868.28,覆盖率指数(Coverage)分别为97%、97%、97%、96%、97%和98%,多样性指数(Simpson/Shannon)分别为0.05/4.72、0.04/4.77、0.04/4.69、0.04/4.74、0.05/4.45和0.08/4.06。
2.3水样细菌群落结构分析
2.3.1单样品物种丰度
对单个水样中OTU在门和属水平进行归类和整理。分析了4月11日(旧模式育苗场暴发病毒性神经坏死病)新旧模式两育苗场水样的情况,即水样B1和水样B2(暴发病毒性神经坏死病水样)。
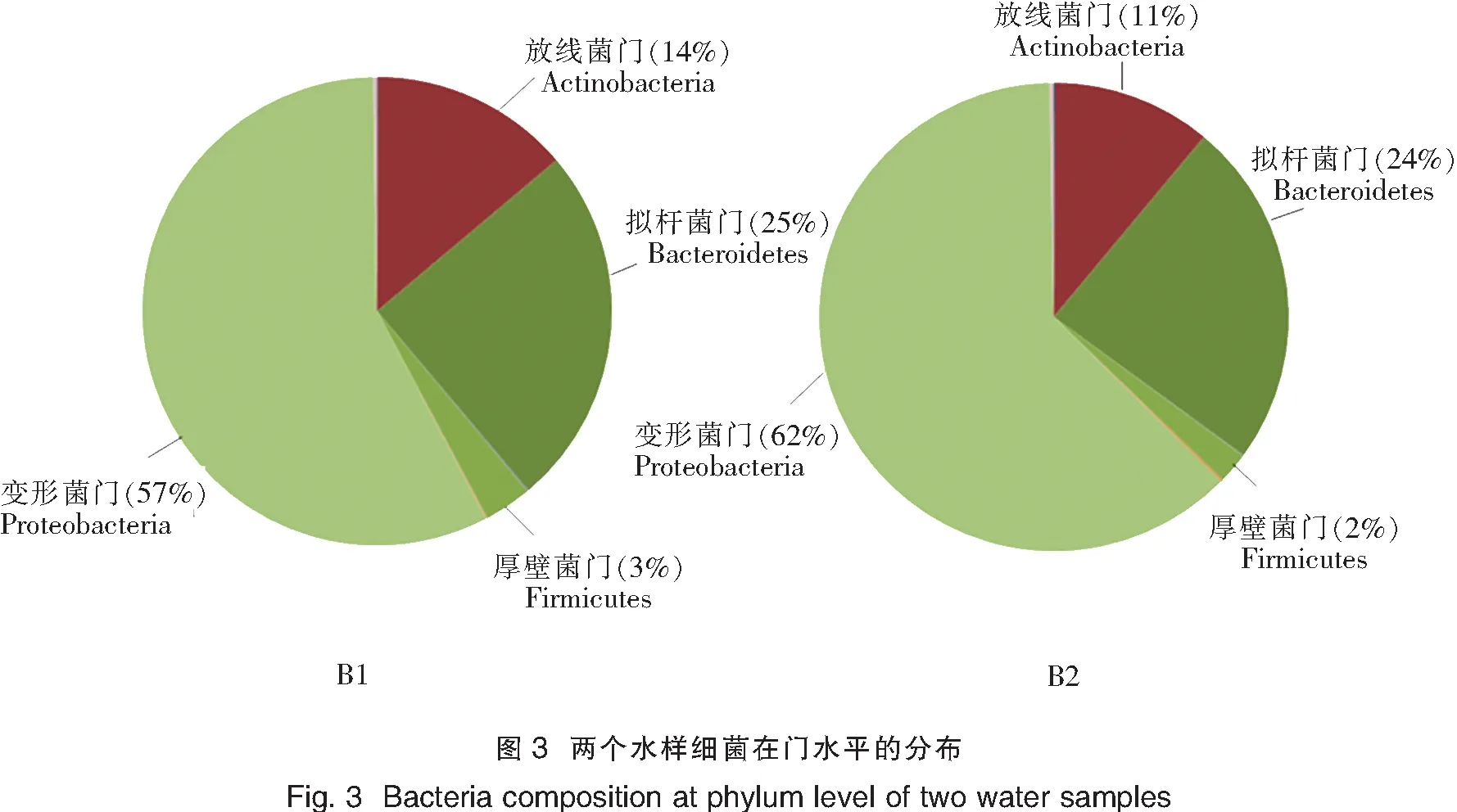
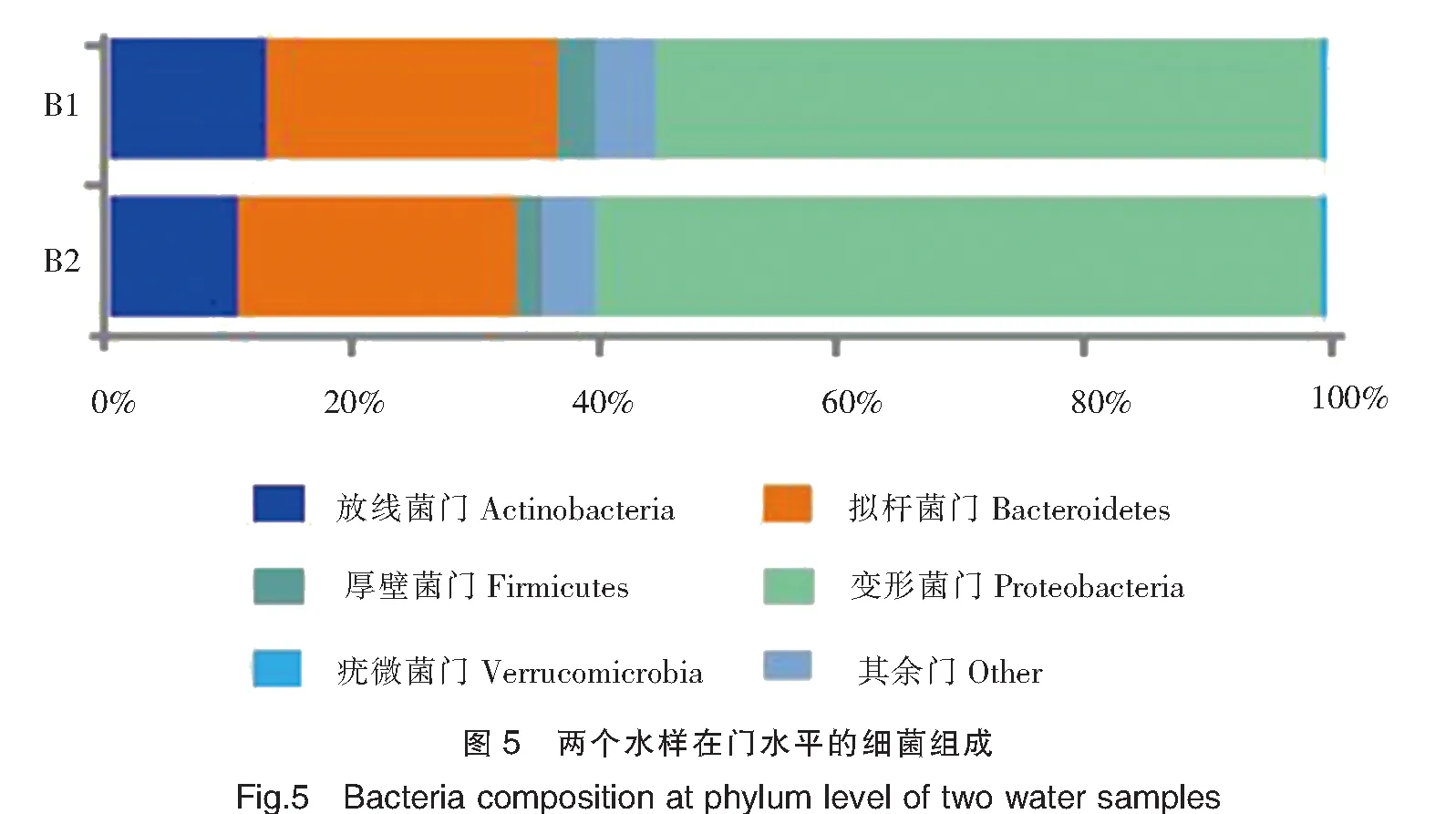
在门水平,B1水样细菌隶属18个门,其中变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)和厚壁菌门(Firmicutes)共占99%,变形菌门所占比例最大(占57%),其余14个门仅占1%(图3)。B2水样细菌隶属20个门,变形菌门、拟杆菌门、放线菌门和厚壁菌门共占99%,变形菌门所占比例仍最大(占62%),而其余16个门仅占1%(图3)。B2中有18个门与B1的重叠,其它2个为独有的门——梭杆菌门(Fusobacteria)和OP10。
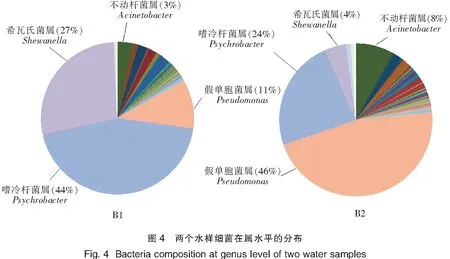
在属水平,B1水样细菌隶属154个属,丰度≥3%的属有嗜冷杆菌属(Psychrobacter,44%)、希瓦氏菌属(Shewanella,27%)、假单胞菌属(Pseudomonas,11%)和不动杆菌属(Acinetobacter,3%),其余150个属占15%(图4)。B2水样细菌隶属159个属,丰度≥3%的属有假单胞菌属(46%)、嗜冷杆菌属(24%)、不动杆菌属(8%)和希瓦氏菌属(4%),其余155个属占18%(图4)。
2.3.2多样品物种分布
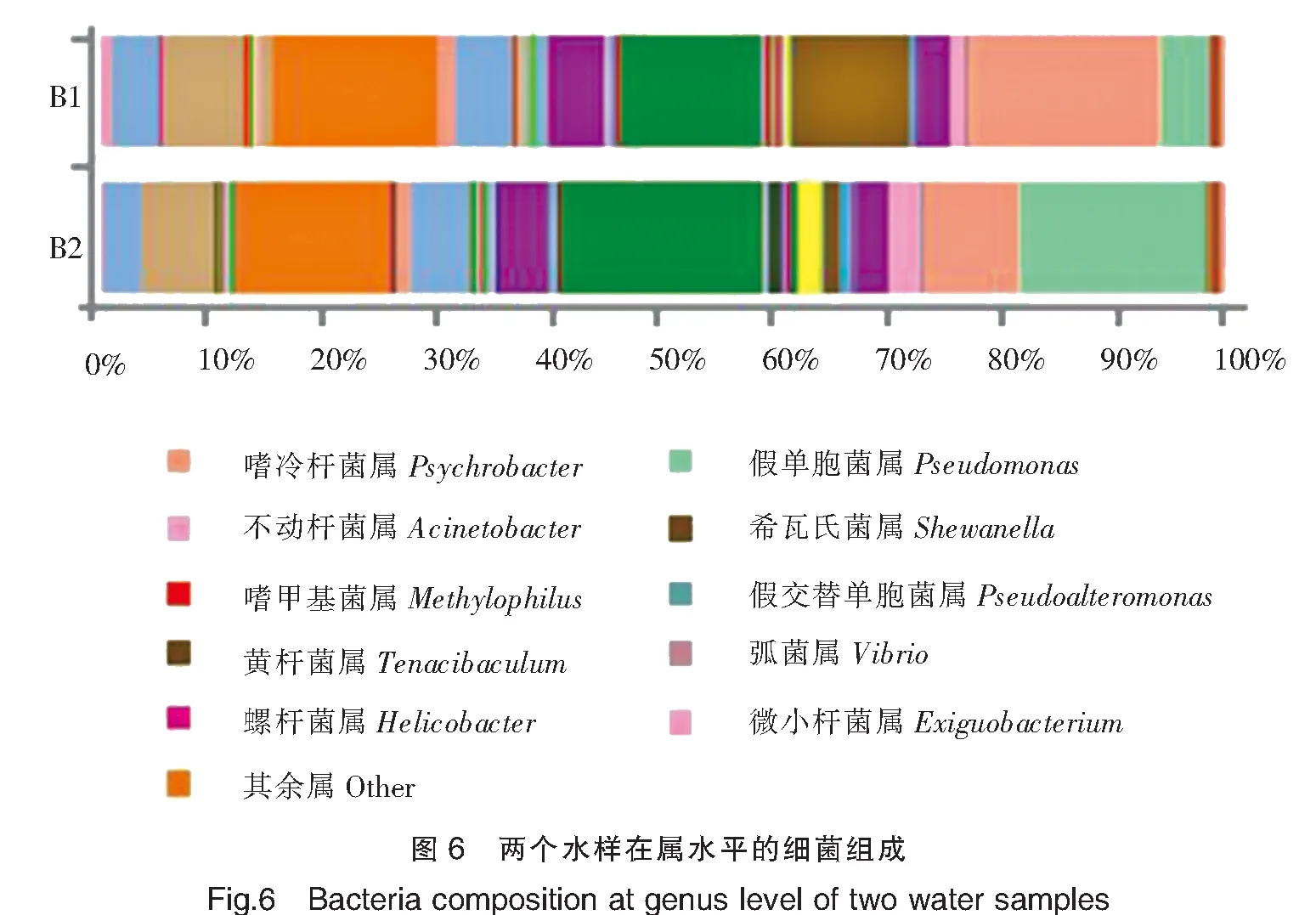
在门和属水平对各水样的菌种分布进行了统计(图5,图6)。在门水平,B1水样和B2水样中细菌隶属的各门所占比例相似。在属水平,两个水样共检测到76个属,B1水样检测到59个属,B2水样检测到64个属,其中47个属为两者共有,12个属为B1水样独有,17个属为B2水样独有。且B2水样中假单胞菌属(占16.33%)所占比例明显高于B1水样(占4.09%) ,相反B2水样中嗜冷杆菌属(占8.29%)所占比例明显低于B1水样(占17.08%)。
2.3.3物种丰度差异性
在门和属水平分析两个水样间的细菌丰度差异性。水样B1和B2相比,有7个门存在显著差异,差异最大的为梭杆菌门(Fusobacteria)(差异倍数 -11.35)(表2)。
水样B1和B2有差异的属有169个,由于数量较大,在此仅列出两者差异倍数超过10倍以上的属。其中,水样B1独有的属25个,B2独有的属47个。假单胞菌属虽不在列,但两者具有显著性差异(差异倍数-2.00,<-1)。
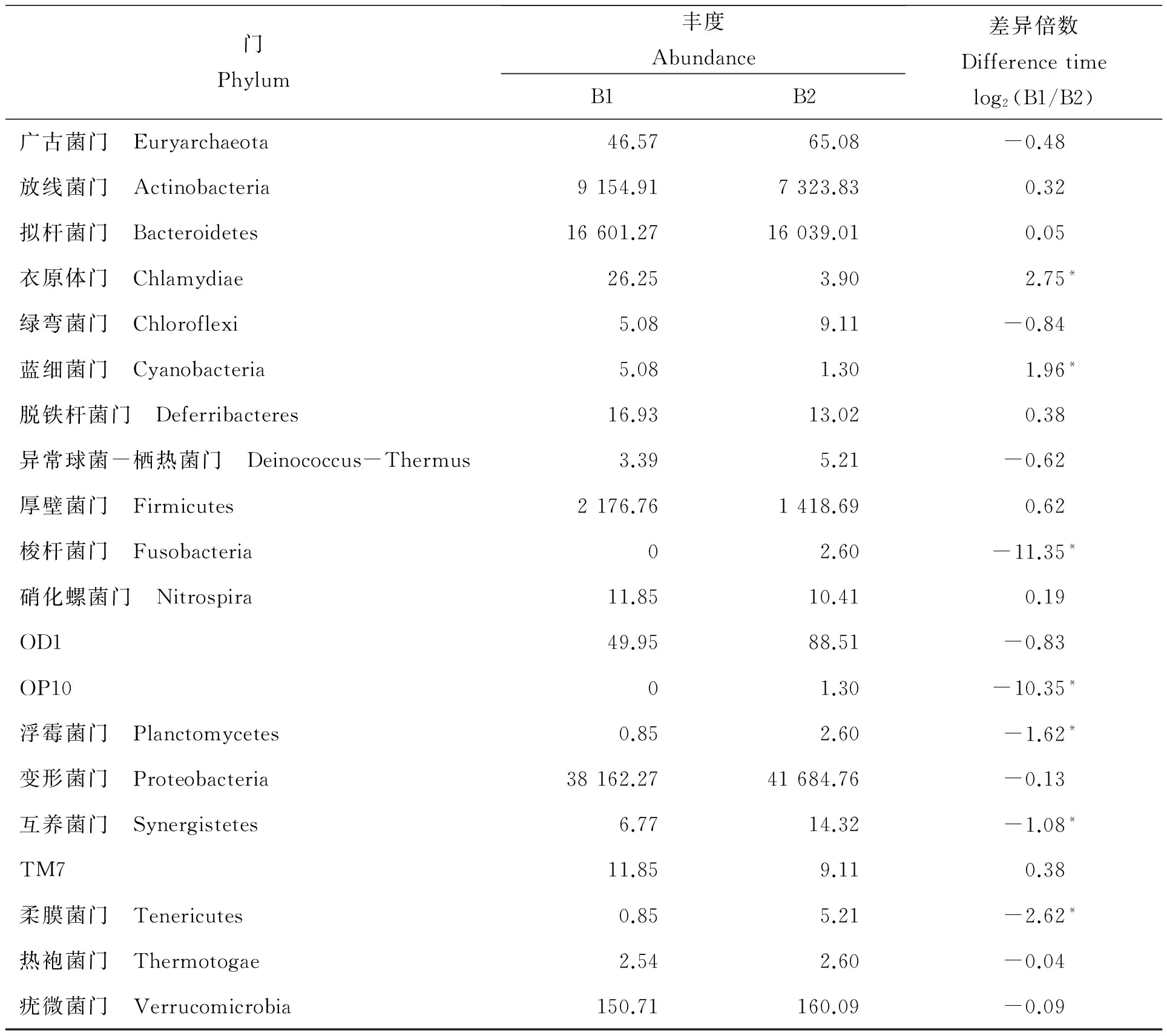
表2 两个水样在门水平的丰度及差异倍数
注:*差异显著。
Note:*indicated significant difference.
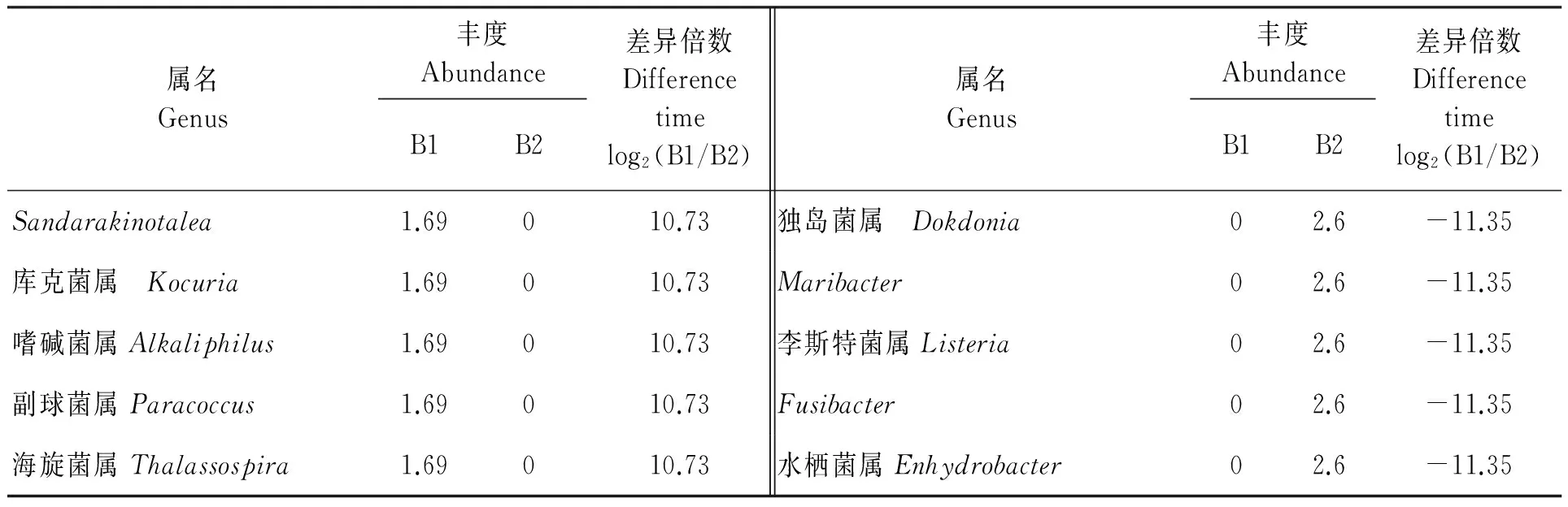
表3 两个水样在属水平的丰度及差异倍数(大于10)
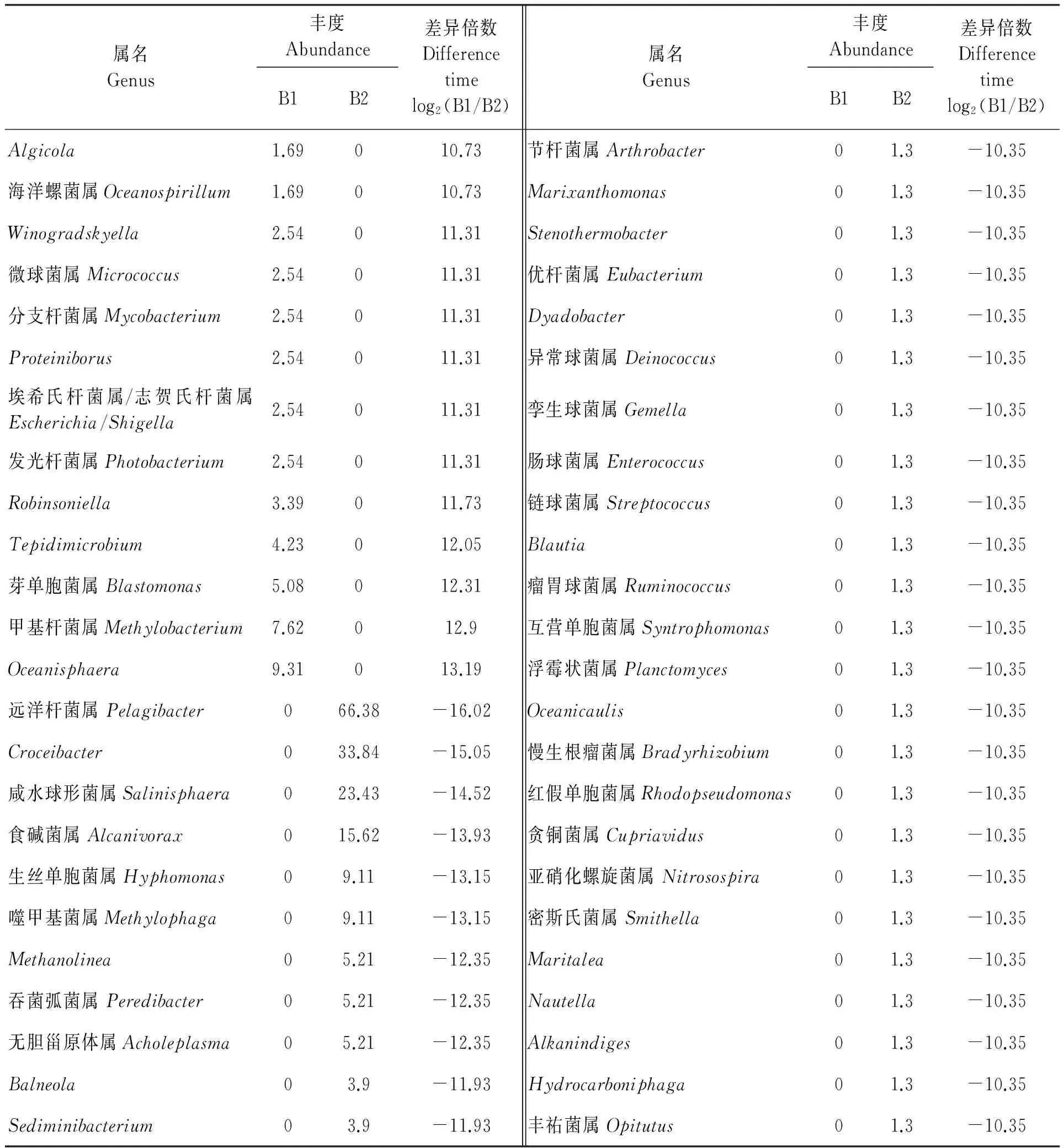
续表3
3 讨论
细菌的16S rDNA由于突变率小、分子大小适中、具有物种特异性,因此常被用作分子标志,鉴定实验中分离出的细菌种类[13,18]。本研究利用细菌16S rDNA V4可变区序列的扩增与宏基因组高通量测序技术相结合,分析新旧两种石斑鱼育苗模式间育苗水体中细菌群落分布的特点和差异。
结果显示,抽提的宏基因组DNA质量较高,稀疏曲线表明测序深度充分,评估OTU的数量应接近实际情况。当传统模式育苗场暴发病毒性神经坏死病时,采用高通量测序获得新旧两种模式在同一时期育苗水体细菌丰度。在门水平,丰度较高的有变形菌门、拟杆菌门、放线菌门和厚壁菌门等;在属水平,丰度较高的有假单胞菌属、嗜冷杆菌属、不动杆菌属和希瓦氏菌属等。在门水平,新旧两种模式育苗场水体中细菌隶属的各门所占比例相差不大,但在属水平,假单胞菌属在传统模式育苗场(当时暴发病毒性神经坏死病)水体中所占比例明显高于新模式育苗场水体。同时,在物种丰度差异性分析中,虽然假单胞菌属、嗜冷杆菌属等分别在两水体间的丰度差异倍数小于10,但在各自水体中都属于较优势菌群,更具有宏观比较的意义,且丰度差异数据显示,两水体间假单胞菌属的丰度差异是具有显著性的。
在哺乳动物疾病研究中,Pettigrew等[19]发现特定细菌与病毒的相互作用会提高儿童患上呼吸道感染并发症——急性中耳炎的风险;同一科研团队的Ruohola等[20]比较分析了患急性中耳炎的儿童和正常儿童的鼻咽中细菌和呼吸道病毒的关系,得出儿童患急性中耳炎的风险与其鼻咽中细菌和病毒间的相互作用存在相关性的结论。由此说明,同一区域范围内的细菌和病毒并非各自孤立,而是密切联系的。在石斑鱼育苗过程中,育苗水体所含的微生物种类繁多,其中细菌含量尤为丰富。众所周知,石斑鱼病毒性神经坏死病常规暴发于每年的特定时节,推测除了受宏观的气温等因素影响外,与水体中微观的细菌群体也应存在一定联系。上述研究结果显示,当传统模式育苗场暴发病毒性神经坏死病时,大多为致病菌的假单胞菌属成为育苗水体中的绝对优势菌群,在水体菌群中所占比例显著高于同一时期未暴发病毒性神经坏死病的新模式育苗场。
因此,由研究结果推测,传统育苗模式,育苗水体中有害菌群含量较石斑鱼育苗新模式下的要高。同时,传统育苗模式下石斑鱼病毒性神经坏死病的暴发很可能与其育苗水体中假单胞菌属等有害菌群含量增加有关。至于这之间具体存在着怎样的相关性,以及它们相互间是如何作用的还需在今后的研究中进一步探索。然而,可以初步确定的是,石斑鱼育苗新模式下的育苗水体中致病菌含量显著低于传统育苗模式,表明石斑鱼育苗新模式水体小生态调控方法较为可行,育苗前期水体菌群结构较为均衡,有益菌占优势,有害菌群含量较低,体现出“石斑鱼生态优化及病害防控人工育苗”创新模式的明显优势。
[1]赵平平.石斑鱼病毒性神经性坏死病研究现状[J].黑龙江水产,2011,5:44-46.
[2]林克冰,方琼珊,吴建绍,等.石斑鱼神经坏死病毒传播途径阻断的初步研究[J].福建水产,2011,33(5):15-19.
[3]林克冰,方琼珊,王涵生,等.斜带石斑鱼生态优化与病害防控育苗模式的研究[J].福建水产,2013,35(5):368-374.
[4]Boissiere A,Tchioffo M T,Bachar D,et al.Midgut microbiota of the malaria mosquito vectorAnophelesgambiaeand interaction withPlasmodiumfalciparuminfection[J].PLoS Pathog,2012,8(5):901-923.
[5]Menno R van den Bergh,Giske Biesbroek,John W Rossen,et al.Associations between Pathogens in the Upper Respiratory Tract of Young Children:Interplay between Viruses and Bacteria[J].PLoS One,2012,7(10):2476-2481.
[6]Schmidt M T,DeLong E F,Pace N R.Analysis of a marine picoplankton community by 16S rRNA gene cloning and sequencing[J].J Bacteriol,1991,173(14):4371-4378.
[7]Handelsman J,Rondon M R,Brady S F,et al.Molecular biological access to the chemistry of unknown soil microbes a new frontier for natural products[J].Chem Biol,1998,5(10):R245-R249.
[8]贺纪正,张丽梅,沈菊培,等.宏基因组学(Metagenomics)的研究现状和发展趋势[J].环境科学学报,2008,28(2):209-218.
[9]Hugenholtz P,Tyson G W.Microbiology:Metagenomics[J].Nature,2008,455(7212):481-483.
[10]孟飞,俞春娜,王秋岩,等.宏基因组与宏基因组学[J].中国生物化学与分子生物学报,2010,26(2):116-120.
[11]南春燕,马雅军,徐建农,等.中华按蚊幼虫肠道细菌宏基因组的组成研究[J].中国寄生虫学与寄生虫病杂志,2013,31(2):114-119.
[12]Ye L,Zhang T.Bacterial communities in different sections of a municipal wastewater treatment plant revealed by 16S rDNA 454 pyrosequencing[J].Applied Microbiology and Biotechnology,2013,97:2681-2690.
[13]Shi P,Jia S,Zhang T,et al.Metagenomic insights into chlorination effects on microbial antibiotic resistance in drinking water[J].Water Research,2013,47:111-120.
[14]邢孟欣.养殖大菱鲆肠道微生物多样性及功能分析[D].北京:中国科学院大学,2013.
[15]杨章武,杨铿,张哲,等.基于宏基因组测序技术分析凡纳滨对虾育苗中生物絮团细菌群落结构[J].福建水产,2015,37(2):91-97.
[16]Zwolinski M D.DNA sequencing:Strategies for soil microbiology[J].Soil Science Society of America Journal,2007,71(2):592-600.
[17]He J Z,Xu Z H,Hughes J.Molecular bacterial diversity of a forest soil under residue management regimes in subtropical Australia[J].FEMS Microbiology Ecology,2006,55(1):38-47.
[18]He J Z,Xu Z H,Hughes J.Analyses of soil fungal communities in adjacent natural forest and hoop pine plantation ecosystems of subtropical Australia using molecular approaches based on 18S rRNA genes[J].FEMS Microbiology Letters,2005,247(1):91-100.
[19]Pettigrew M M,Gent J F,Pyles R B,et al.Viral-bacterial interactions and risk of acute otitis media complicating upper respiratorytract infection[J].J Clin Microbiol,2011,49(11):3750-3755.
[20]Ruohola A,Pettigrew M M,Lindholm L,et al.Bacterial and viral interactions within the nasopharynx contribute to the risk of acute otitis media[J].J Infect,2013,66(3):247-254.
Comparative analysis of bacterial diversity and distribution characteristics in water collected from two Epinephelus coioides seedling models with Metagenomics
SUN Minqiu,LIN Kebing,GE Hui,HUANG Zhongchi,ZHENG Leyun,ZHOU Chen
(Key Laboratory of Cultivation and High-value Utilization of Marine Organisms in Fujian Province,Fisheries Research Institute of Fujian,Xiamen 361013,China)
To compare the difference of bacterial distribution in water of new or traditional grouper seedling model,the 16S rDNA V4 region of six water samples(A1,A2,B1,B2,C1 and C2)collected from one farm using “the new breeding model of ecological optimization and disease control and prevention” and another traditional model farm on Zhao’an county of Zhangzhou City were sequenced by high-throughput pyrosequencing.Using Qiime and Mothur software,the number of sequences and operational taxonomic units(OTUs)for each sample were sorted and calculated,the species abundance and distribution,Alpha diversity index and difference times of species abundance among samples were analyzed.The number of sequences and OTUs for each sample were 66 727/3 724(A1),66 418/3 391(A2),87 403/3 980(B1),57 519/3 438(B2),62 349/3 530(C1)and 66 673/3 004(C2).The rarefaction curves showed that adequate sampling was achieved.The number of OTUs was close to actual situation.The value of richness index was 6 535.21/8 872.16(A1),6 583.85/8 855.40(A2),8 062.60/10 640.49(B1),6 878.85/9 352.25(B2),6 859.85/9 295.45(C1)and 5 291.87/6 868.28(C2);diversity index was 0.05/4.72(A1),0.04/4.77(A2),0.04/4.69(B1),0.04/4.74(B2),0.05/4.45(C1)and 0.08/4.06(C2).At the time NNV breaking in the traditional model farm,the single sample species abundance,multi-samples species abundance and different times of species abundance at phylum and genus levels between two model samples were analyzed.At phylum level,the distribution of dominant bacterial communities in seedling water between two models were similar.At genus level,the content of pathogenicPseudomonasof dominant bacterial community in traditional model sample was significantly higher than the new model.Evenness and richness of bacteria flora in the six water samples collected from new and traditional grouper seedling model were obtained before and after the onset of NNV breaking.It was suggested that “the new breeding model of ecological optimization and disease control and prevention” for grouper seedling might good for inhibition of pathogenic bacteria and enrichment of probiotics.
Epinepheluscoioides;new seedling model;bacterial flora;Metagenomics
2016-05-09
厦门南方海洋研究中心项目(14GZP75NF39);闽海洋高新项目[2014]21号;省属公益类科研院所基本科研专项(2014R1003-15);省属公益类科研院所基本科研专项(2014R1003-11);闽台重要海洋生物资源高值化开发技术公共服务平台(2014FJPT01);福建重要海洋经济生物种质库与资源高效开发技术公共服务平台(14PZY017NF17).
孙敏秋(1986-),女,实习研究员,硕士,研究方向:微生物及水产动物免疫学.E-mail:minqiusun@sina.com
S917.1
A
1006-5601(2016)03-0181-11
孙敏秋,林克冰,葛辉,等.基于宏基因组比较分析石斑鱼育苗两种模式水体中的菌群多样性和分布特点[J].渔业研究,2016,38(3):181-191.
