CRISPR/Cas9基因编辑系统研究进展及其在动物基因编辑研究中的应用
2016-08-05王玮玮刘瑞琪吴勇延杨严格王勇胜卿素珠
王玮玮,刘瑞琪,吴勇延,杨严格,王勇胜,卿素珠
(西北农林科技大学动物医学院,杨凌 712100)
CRISPR/Cas9基因编辑系统研究进展及其在动物基因编辑研究中的应用
王玮玮,刘瑞琪,吴勇延,杨严格,王勇胜,卿素珠*
(西北农林科技大学动物医学院,杨凌 712100)
摘要:CRISPR/Cas(Clustered regularly interspaced short palindromic repeats(CRISPR)/CRISPR-associated (Cas)系统是由一种短小RNA调控的对DNA修饰的基因编辑工具,是一种较锌指核酸酶(Zinc-fingers nuclease,ZFN)和转录激活因子样效应物核酸酶(Transcription activator-like (TAL) effector nucleases,TALEN)打靶更加快速、高效、精准的新型基因组编辑工具,它易于设计,且具有特异性。本文回顾了CRISPR/Cas系统的结构与功能,概述了Cas9的设计策略、影响Cas9基因编辑效率的因素、脱靶检测分析方法及其在动物基因编辑研究中的应用。基于CRISPR/Cas9已经在多种动物上成功实现了基因编辑,它有望成为建立动物模型和研究疾病防治的一种新型可行性途径。
关键词:CRISPR/Cas9系统;基因编辑;sgRNA;基因敲除;基因敲入;动物模型
1CRISPR/Cas的兴起
1987年,日本科研人员[1]在大肠杆菌的基因组中发现了一系列相互间隔的短回文序列(Clustered regularly interspaced short palindromic repeats,CRISPR),这种短回文序列可以给细菌与古细菌提供获得性免疫[2],当时有研究者预测了 CRISPR/Cas可能是一种新的DNA修复系统[3-4]。CRISPR/Cas系统广泛存在于原核生物中,87%的古细菌和48%的细菌中都含有此种蛋白免疫系统[5]。最初,CRISPR/Cas系统无法应用在人和动物上,经过改造,它作为一种以核酸酶为核心的基因编辑技术逐渐被广泛应用。
2CRISPR/Cas9系统的基因结构和作用机制
2.1CRISPR/Cas9系统的基因结构
目前有3种CRISPR/Cas系统(Ⅰ、Ⅱ、Ⅲ),Cas9核酸蛋白酶主要构成和表达Ⅱ型CRISPR/Cas系统。每个系统包含一群与CRISPR相关的基因、非编码RNA和一个独特阵列的正向重复元件,其中最常见是的肺炎链球菌S.pneumoniaeCas9(spCas9)系统,即Ⅱ型CRISPR/Cas9系统。CRISPR/Cas9系统至少需要3个组件:一个CRISPR相关核酸酶、一个特异性CRISPR RNA(CRISPR RNAs,crRNA)和一个反式激活CRISPR RNA(Transactivating crRNA,tracrRNA)[6]。有研究人员为了精简这一技术,设计出了代替crRNA- tracrRNA复合物的单链向导RNA(Singleguide RNA,gRNA)[7],它可以将Cas9核酸酶引导到目标打靶位点对双链DNA进行切割。此外,tracrRNA上还有一个结构叫做tracrRNA尾巴(TracrRNA tail),该结构有利于增强Cas9核酸酶的表达[8]。
CRISPR是一些相互间隔的短回文重复序列,序列长度为21~48 bp,这些重复序列常常产生发卡结构,而且发卡结构的重复次数可达200次以上[5]。每个重复序列都被外源性DNA靶点中与之结构相似的短重复序列所隔开,叫做典型间隔区域(Protospacer),这些间隔区域序列决定了CRISPR系统的种类以及靶基因的识别位点[9]。在DNA靶点之内,每个典型间隔区域始终与典型间隔相邻基序(Protospacer adjacent motif,PAM)相邻,PAM可以根据特异性CRISPR系统而变化[10]。常用的PAM有3种类型,分别为NGG、NAG和NNGG[11]。Cas9蛋白含有两个核酸酶结构域,一个是HNH结构域,另一个是RuvC样结构域。HNH核酸酶结构域和RuvC样核酸酶结构域分别切割目标DNA链的一条单链[12]。这样的单链切割容易产生突变,可能是由于HNH和RuvC的存在[13]。
2.2CRISPR/Cas9系统的作用机制
如图1所示,PAM序列引起Cas9蛋白识别,使得与tracrRNA相连的单链向导RNA(Single guide RNA,sgRNA)与目标位点(Target sequence)识别,保证了Cas9蛋白和基因组稳定地结合,引起目标位点的切割,从而产生DNA双链断裂(Double-strand breaks,DSB)[14]。DNA双链断裂后引起非同源末端连接(Non-homologous end joining,NHEJ)或者同源性定向修复(Homology-directed repair,HDR),各个单链通过高精确度的碱基切除修复机制(Base-excision repair,BER)进行修复[15-16]。CRISPR/Cas9系统可以同时对靶基因进行高效的基因敲除(Knockout)和基因敲入(Knock-in)编辑,如图2所示,只需对目的片段进行切割,通过NHEJ机制修复而引入插入或缺失的突变,就可以达到Knockout的效果。Knock-in则是将外源有功能的基因转入基因的同源序列中,通过HDR修复,进行Knock-in或者点突变,使插入基因组得以在细胞内表达[17],高效地对特定位点同时进行Knockout和Knock-in是Cas9系统的一种特色,只需要改变供体载体(Donor vector)上的外源基因即可。
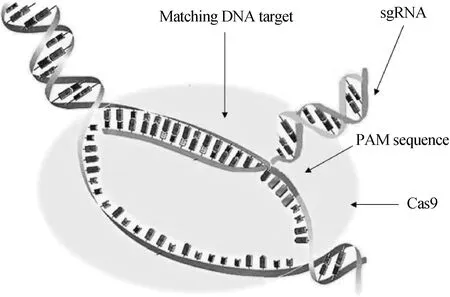
图1 CRISPR/Cas9系统的作用机制Fig.1 Mechanism of CRISPR/Cas9 system
2.3Cas9与传统技术的对比
早期的基因编辑技术主要有锌指核酸酶(ZFNs)和转录激活因子样效应物核酸酶(TALENs)。ZFNs是由锌指序列组成,FokI核酸酶结构域通过二聚体作用产生核酸内切酶活性从而切开DNA双链。转录激活因子样效应物核酸酶(TALENs)与 ZFNs的构建原理相似,设计更加优化,靶向特异性更加明确。CRISPR/Cas9系统作为第3代的人工核酸酶技术,与ZFNs和TALENs相比,其优势在于打靶位点多,平均8个碱基就有一个打靶位点,ZFNs几乎到500个碱基才有一个合适的打靶位点[18]。其次是Cas9载体构建简单,只需根据特异性打靶位点设计sgRNA,而ZFNs 和TALENs则需要对每个打靶位点设计和组装一对核酸酶。再者就是Cas9可以高效地进行多位点编辑,而ZFNs和TALENs的一对核酸酶一次只能切割一个位点,很难提高突变率。但是Cas9的脱靶率要高于ZFNs和TALENs[19]。
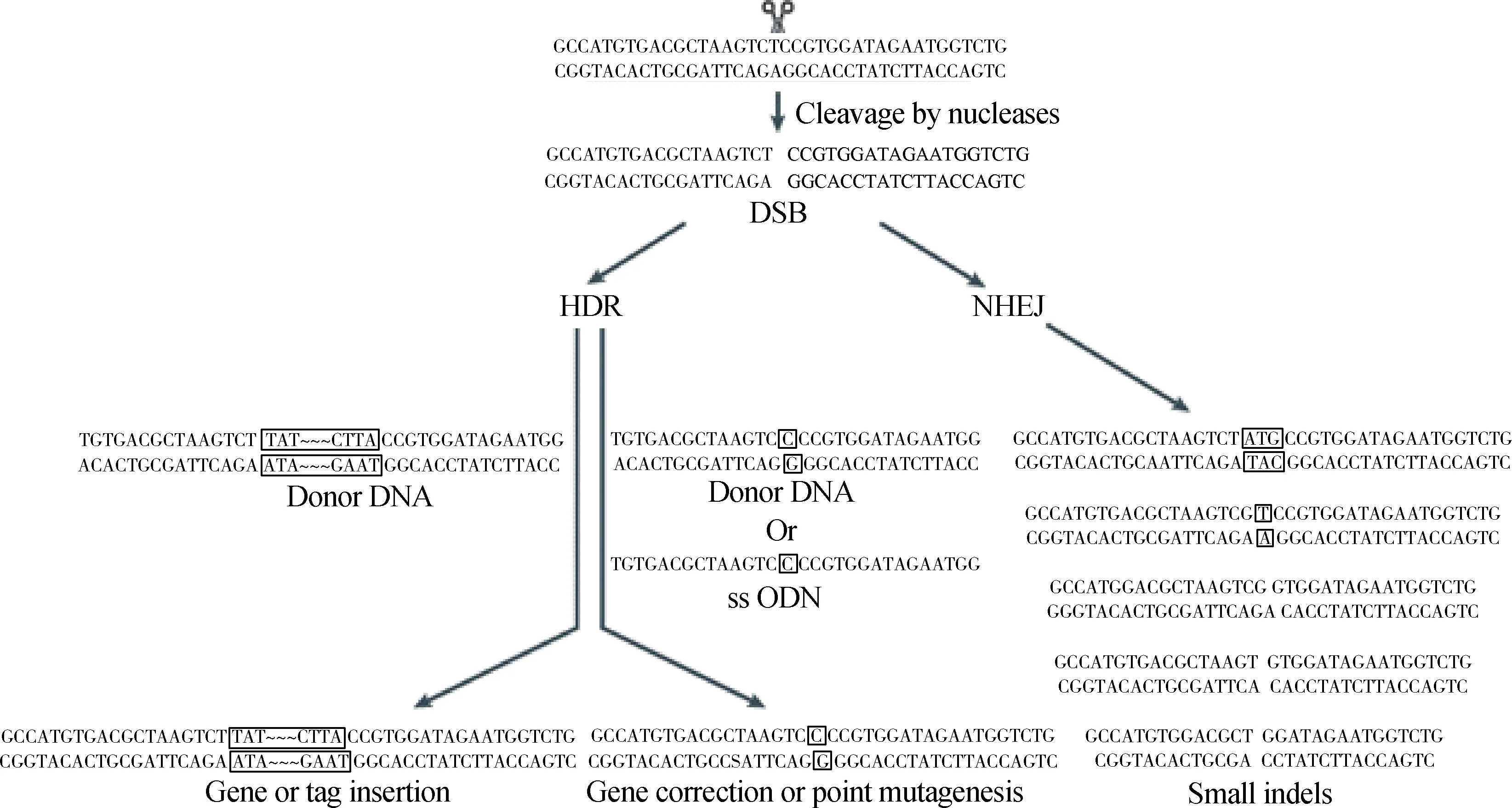
图2 Cas9核酸酶的切割和修复机制[17]Fig.2 Cleavage by nucleases and repair mechanism of Cas9 nucleases
3Cas9的设计策略
3.1单链向导RNA的设计
sgRNA从5′开始到3′依次为DNA互补区、crRNA和tracrRNA,其中DNA互补区的设计对打靶效率有至关重要的影响[20]。sgRNA中有一个12个碱基组成的毗邻PAM上游的独特序列,被称作“种子区”[21],“种子区”发生错配对靶位点的识别影响更大[22]。sgRNA和目的片段之间一定数量的错配是可以被容忍的,特别是当这些错配离PAM较远的情况下[23],因此在设计sgRNA时需要注意发生错配的序列位置。针对基因组设计sgRNA的一般标准是:紧邻着PAM序列(5′-NGG-3′)的12个碱基的种子区的3′端最多允许出现1个错配;在单链向导RNA(sgRNA)5′末端8个碱基的区域允许出现5个错配[21]。总的来说就是要严格遵守碱基互补原则来设计靠近PAM的12个碱基序列,尽量保证该序列内没有突变或者错配。但是相对于前人的研究,第7到第12个碱基也没有一些特别的序列[24-25]。也有研究表明,增加茎环结构和3′尾部结构可以加强向导RNA的稳定性,有利于Cas9形成正确构象[26]。
3.2关于DNA打靶序列
目前仍然不能确定设计打靶序列的精确标准[27],只是根据一些常规标准,在众多打靶位点中选择出较好的打靶位点。目前设计打靶序列的在线软件:http://zifit.partners.org/ZiFiT/ChoiceMenu.aspx、http://www.e-crisp.org/E-CRISP/designcrispr.html、http://www.rgenome.net/,http://crispr.mit.edu/、https://chopchop.rc.fas.harvard.edu/。这几款在线软件中,前两个的主要功能是寻找打靶序列,之后需要进行人工筛选,结果较为可靠。http://www.rgenome.net/ 这个软件可用来寻找打靶序列并且设计向导RNA,而且还可以设定所需的碱基错配数。http://crispr.mit.edu/和https://chopchop.rc.fas.harvard.edu/可以对生成的sgRNA进行排名以供选择,唯一不足是其数据库内的物种有限[28]。
3.3影响Cas9打靶效率的因素
设计向导RNA时,GC含量一般为45%~60%为宜,超过这个范围都会影响Cas9的靶向效率以及C碱基富集[29]。C碱基富集使得容易打靶在DNA甲基化的位置,尽量避开甲基化位点有利于减少表观遗传突变。但是与之矛盾的是有研究指出甲基化只影响ZFNs和TLAENs,并不影响Cas9的切割效率[8]。DNA聚合酶I超敏感位点(DHS)属于染色质结构的一种状态,可以显著提高转录因子结合位点的细胞特异性预测,将靶向序列设计在DHS内可提高基因编辑效率[30]。此外,选择靶向位点时,尽量选择外显子区域而非内含子区域,这是因为内含子对翻译产物的意义不大且比外显子更易产生突变。
4切割效率和脱靶效应的分析及检测
人工设计的核酸酶经常会产生脱靶切割,在基因序列中引起突变(插入、缺失、倒置或易位),从而使基因异常表达、细胞死亡或发生肿瘤。20 nt 的sgRNA与目的片段之间一定数目的错配数导致大量的脱靶突变产生[31],因此很难彻底和有效地确认潜在的脱靶位点。体外试验表明,不同类型的PAM可能会影响CRISPR/Cas9的切割活性,按照PAM的要求,在基因组上可以产生很大的打靶范围,打靶位点过多会导致基因片段在多个位点被切断,这也是Cas9容易脱靶的主要原因。
4.1检测切割效率和脱靶效应的主要手段
检测切割效率和脱靶效应的主要手段:1.SURVEYOR法或T7EN1酶切法。两种方法都属于利用核酸内切酶检测脱靶情况。SURVEYOR酶是CEL-I错配特异性核酸酶的一种,能准确切割异源双链DNA错配位点。T7EN1酶能识别并切割不配对的双链DNA,将基因编辑后序列与基因编辑前序列相比较,看一定范围内碱基序列是否发生变化。2.SSA报告载体检测法。该方法是建立SSA报告载体,报告载体上Luciferase蛋白失去活性后,在目标位点发生打靶后通过比对Luciferase的活性,评价核酸内切酶的切割效率和脱靶情况[32]。该法是较为精密的测试脱靶效应的方法。3.Sanger测序法。直接性地对PCR产物进行测序或者间接性地转入TA克隆载体中,挑选单克隆进行测序。4.Digenome-Seq技术。提取基因组后进行高通量测序,它属于全基因测序的一种方法,可以全面有效地检测脱靶效应[33]。
4.2降低脱靶效率的方法
目前,可通过3个方面来降低Cas9基因编辑中的脱靶效应[8]:第一,提高sgRNA的特异性:(1)尽量降低预测的脱靶位点与sgRNA之间的配对碱基数;(2)设计的sgRNA与预测的脱靶位点在“种子区”至少有2个碱基的错配;(3)尽量避免出现sgRNA与预测的脱靶位点序列有连续的或间隔的4个碱基配对。S.W.Cho等[34]的研究证明修饰sgRNA可以降低Cas9的脱靶效应。(4)缩短sgRNA的长度。将20 nt的sgRNA改造成17~18 nt的缩短型sgRNA,有效地降低了脱靶率[35]。第二,改造Cas9蛋白。F.A.Ran等致力于改造更高特异性的Cas9蛋白,将Cas9蛋白改造为双切口酶(Double-nicking),将Cas9切口酶突变体与成对的sgRNA相结合产生一种新的复合蛋白以完成特异性双链断裂[31]。第三,还可以将Cas9-D10A切口酶与一对sgRNA配合使用产生SSB(Single-strand breaks,SSB),以减少因细胞核内不存在DNA同源修复模板而引起的脱靶突变[36]。
5Cas9在动物基因编辑研究上的应用
5.1基因敲除
2013年,美国科研工作者基于CRISPR/Cas9技术在细胞系上进行基因敲除,利用靶位点特异性RNA,将Cas9核酸酶引导到基因组的目的位点上进行切割并引起突变。以小鼠为动物模型的Cas9基因打靶技术已经革新为“一步法”,该法可实现多重基因组编辑[37]。H.Wang等[38]选择对Tet家族基因(Ten-eleven translocation family members)进行敲除,Tet基因的突变可以引起多种肿瘤尤其是造血系统肿瘤。研究人员用显微共注射方法,将Cas9 mRNA和sgRNA共同注射到小鼠受精卵中同时敲除Tet1和Tet2,经验证敲除效率在80%左右,该试验成功地产生双等位基因突变的小鼠,并且是首次在动物体内同时敲除2个内源性基因。
Y.Niu等选择Ppar-γ(Peroxisome proliferator-activated receptor gamma)和Rag1(Recombination activating gene 1)两个靶基因,将Cas9 mRNA和单链向导RNA共同注入食蟹猴受精卵中,在一个步骤中同时打靶这2个基因,在全基因分析检测中没有发现脱靶现象,最后成功获得了经过基因修饰的转基因食蟹猴[39]。灵长类动物在遗传特性和生理功能方面与人类接近,可以作为研究模型来进行人类某些疾病致病机制及治疗策略的研发。
F.Chen等[40]利用CRISPR/Cas9系统敲除猪免疫球蛋白M(IgM)重链基因的JH区域(Joining chain),此区域对免疫系统的发育和分化有至关重要的作用。在体细胞核移植技术的支持下,将IgM抗体的Cas9质粒转染仔猪胎儿成纤维细胞后,基因敲除的效率高达53.3%,其中25%阳性克隆有双等位基因修饰,这比传统的同源重组效率高很多[41]。X.Zhou等人采用Cas9/sgRNAs的方式,敲除了猪胎儿成纤维细胞上PARK2基因(Parkin2)和PINK1基因(PTEN-induced putative kinase 1),以突变型细胞克隆为体细胞移植的供体,产生纯合子的转基因猪[42]。MSTN基因(Myostatin)编码的氯化筒箭毒碱会抑制肌肉的分化和生长,研究人员利用CRISPR/Cas9技术将特异性MSTN基因的CRISPR/Cas9 mRNA显微共注射到绵羊受精卵的胞质中,结果显示胚胎发育突变率达50%[43]。在山羊上,在成纤维细胞敲除MSTN和FGF5基因的效率都是60%,而在98只试验动物中分别敲除了MSTN和FGF5基因后存活下来的动物只有15%和21%,双基因修饰后存活下来的动物为10%[44],这些研究提示CRISPR/Cas9系统可作为动物优质性状新品种培育和抗病育种有效的基因编辑工具。通过注射IL2RG和Rag1基因的Cas9 mRNA和sgRNA到原核期胚胎的细胞质中,可获得高达100%效率的双等位基因敲除兔,同时敲除3个基因(IL2RG,RAG1和RAG2)和5个基因(IL2RG,RAG1,RAG2,TIKI1和ALB)的效率均可达到33.3%[45]。N.Véron等运用活体点穿孔技术,有效敲除鸡胚胎干细胞中的转录因子PAX7(Paired box 7),使野生型多细胞动物产生镶嵌式基因突变[46],使鸡胚胎干性基因的相关功能丢失。Cas9技术在斑马鱼上也实现了高效率的多基因修饰,crRNA-tracrRNA-Cas9蛋白质复合物使内源基因表达可视化,这是在冷水动物模型上的首次突破[47]。
5.2基因敲入
有研究发现如果提供同源DNA,可将外源性序列定点敲入到斑马鱼胚胎的特定靶点,插入效率为3.5%~15.6%[48]。潜在脱靶位点的突变率只有1.1%~2.5%,体现了Cas9系统的特异性。截至目前,利用Cas9系统编辑牛基因组的相关文章屈指可数,可能是因为牛较长的妊娠周期和Cas9易于脱靶的性质[49]。2016年,研究者利用NHEJ途径有效地将一个4.6 kb的无启动子载体整合到GADPH基因位点上,在人体细胞和胚胎干细胞上的敲入率分别为20%和1.7%,并且证明NHEJ途径比HDR途径的敲入效率要高[50],但并没有评估NHEJ途径可能引起的细胞毒性问题。
6结语
Cas9系统是微生物自我保护的一种独特机制,这种保护机制是为了防止外来微生物的侵入[51]。它是继ZFN和TALEN之后出现的更为快捷和有效的基因编辑工具,该技术旨在提高对特异性序列的识别与结合的能力以及酶切效率,越来越多的研究利用Cas9系统提高了基因编辑在不同物种上的可能性。由于Cas9核酸酶的PAM(5′-NGG-3′)很短,几乎在任何物种上都可以找到,这就解决了基因编辑工具在跨物种方面的难题,其他基因编辑工具并不能直接做到。高效、时间成本低、一个基因组上可进行多个基因位点编辑以及基本不受物种限制都是Cas9基因编辑技术异军突起的优势。当然,CRISPR/Cas9基因编辑技术也存在着一些不足,如容易脱靶且潜在脱靶位点多、基因突变以及影响打靶效率的因素过多等。脱靶和影响打靶效率的因素尚可预测,但基因突变是难以预测的。基因编辑过程中产生的基因突变是CRISPR/Cas9打靶系统的副作用,Cas9核酸酶可能会结合到意想不到的位点,导致某些位点发生基因突变。这些都是Cas9系统需要改进的方面。
研究者们也在致力于不断探索和优化CRISPR系统的研究,以使其更加快捷、简便、完善。I.M.Slaymaker等在金色葡萄球菌中找到了比Cas9核酸酶更小的一种蛋白,它的强大在于更易进入成熟细胞以及更容易切割产生黏性末端以增加DNA序列与载体间的连接[52],随后该团队又在2016年改造出新型eSpCas9(“Enhanced specificity”,SpCas9)基因编辑系统,eSpCas9既降低了脱靶效率又延续了高效、高准确度打靶效率[53]。相信随着CRISPR/Cas9基因编辑技术的不断优化和完善,其在动物基因编辑研究中的应用将更为广泛,在动物新品种培育、抗病育种及生物医学领域的疾病研发方面发挥重要作用。
参考文献(References):
[1]ISHINO Y,SHINAGAWA H,MAKINO K,et al.Nucleotide sequence of the iap gene,responsible for alkaline phosphatase isozyme conversion inEscherichiacoli,and identification of the gene product[J].JBacteriol,1987,169:5429-5433.
[2]GAJ T,GERSBACH C A,BARBAS C F,et al.ZFN,TALEN,and CRISPR/Cas-based methods for genome engineering[J].Cell,2013,31:397-405.
[3]MAKAROVA K S,ARAVIND L,GRISHIN N V,et al.A DNA repair system specific for thermophilic Archaea and bacteria predicted by genomic context analysis[J].NucleicAcidsRes,2002,30(2):482-496.
[5]GRISSA I,VERGNAUD G,POURCEL C,et al.The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J].BMCBioinformatics,2007,8:172.
[6]HSU P D,LANDER E S,ZHANG F,et al.Development and Applications of CRISPR-Cas9 for Genome engineering[J].Cell,2014,157:1262-1278.
[7]PELLETIER S,GINGRAS S,GREEN D R,et al.Mouse genome engineering via CRISPR-Cas9 for study of immune function[J].Immunity,2015,42:18-27.
[8]HSU P D,SCOTT D A,WEINSTEIN J A,et al.DNA targeting specificity of RNA-guided Cas9 nucleases[J].NatBiotechnol, 2013,31(9):827-832.
[9]DOUDNA J A,CHARPENTIER E.The new frontier of genome engineering with CRISPR-Cas9[J].Science,2014,246:1258096(1-9).
[10]RAN F A,HSU P D,WRIGHT J,et al.Genome engineering using the CRISPR-Cas9 system[J].NatProtocols,2013,8:2281-2308.
[11]XIAO A,CHENG Z,KONG L,et al.CasOT:a genome-wide Cas9/gRNA off-target searching tool[J].Bioinformatics,2014,30(8):1180-1182.
[12]HOCKEMEYER D,WANG H,KIANI S,et al.Genetic engineering of human pluripotent cells using TALEnucleases[J].NatBiotechnol,2011,29(8):731-734.
[13]CONG L,RAN F A,COX D,et al.Multiplex genome engineering using CRISPR/Cas systems[J].Science,2013,339:819-823.
[14]SHALEM O,SANJANA N E,HARTENIAN E,et al.Genome-scale CRISPR-Cas9 knockout screening in human cells[J].Science,2014,343:84-87.
[15]MALI P,YANG L,ESVELT K M,et al.RNA-guided human genome engineering via Cas9[J].Science,2013,339(6121):823-826.
[16]DIANOV G L,HÜBSCHER U.Mammalian base excision repair:the forgotten archangel[J].NucleicAcidsRes, 2013,41(6):3483-3490.
[17]KIM H,KIM J S.A guide to genome engineering with programmable nucleases[J].NatRevGenet,2014,15(5):321-334.
[18]方锐,畅飞,孙照霖,等.Crispr/Cas9介导的基因组定点编辑技术[J].生物化学与生物物理进展,2013,40(8):691-702.
FANG R,CHANG F,SUN Z L,et al.New method of genome editing derived from CRISPR/Cas9[J].ProgressinBiochemistryandBiophysics,2013,40(8):691-702.(in Chinese)
[19]GAJ T,GERSBACH C A,BARBAS C F.ZFN,TALEN,and CRISPR/Cas-based methods for genome engineering[J].TrendsBiotechnol,2013,31(7):397-405.
[20]SAPRANAUSKAS R,GASIUNAS G,FREMAUX C,et al.The Streptococcus thermophilus CRISPR/Cas system provides immunity inEscherichiacoli[J].NucleicAcidsRes,2011,39(21):9275-9282.
[21]DOW L E,FISHER J,O’ROURKE K P,et al.Inducible in vivo genome editing with CRISPR-Cas9[J].NatBiotechnol, 2015,33(4):390-394.
[22]KLEINSTIVER B P,PREW M S,TSAI S Q,et al.Engineered CRISPR-Cas9 nucleases with altered PAM specificities[J].Nature,2015,523(7561):481-485.
[23]JIANG W,BIKARD D,COX D,et al.RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J].NatBiotechnol,2013,31(3):233-239.
[24]CHO S W,KIM S,KIM J M,etal.Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease[J].NatBiotechnol,2013,31(3):230-232.
[25]JINEK M,EAST A,CHENG A,et al.RNA-programmed genome editing in human cells[J].Elife,2013,2(1):e00471.
[26]NISHIMASU H,RAN F A,HSU P D,et al.Crystal structure of Cas9 in complex with guide RNA and target DNA[J].Cell,2014,156(5):935-949.
[27]CARROLL D.Staying on target with CRISPR-Cas[J].NatureAmerica,2013,31:807-809.
[28]MONTAGUE T G,CRUZ J M,GAGNON J A,et al.CHOPCHOP:a CRISPR/Cas9 and TALEN web tool for genome editing[J].NucleicAcidsRes,2014,25:1-7.[29]WILES M V,QIN W,CHENG A W,et al.CRISPR-Cas9-mediated genome editing and guide RNA design[J].MammGenome,2015,26(9-10):501-510.
[30]GUSMAO E G,DIETERICH C,ZENKE M,et al.Detection of active transcription factor binding sites with the combination of DNase hypersensitivity and histone modifications[J].Bioinformatics,2014,30(22):3143-3151.
[31]RAN F A,HSU P D,LIN C Y,et al.Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J].Cell,2013,154(6):1380-1389.
[32]BHAKTA M S,SEGAL D J.The generation of zinc finger proteins by modular assembly[J].MethodsMolBiol,2010,649:3-30.
[33]KIM D,BAE S,PARK J,et al.Digenome-seq:genome-wide profiling of CRISPR-Cas9 off-target effects in human cells[J].NatMethods,2015,12(3):237-243.
[34]CHO S W,KIM S,KIM Y,et al.Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases[J].GenomeRes,2014,24(1):132-141.
[35]FU Y F,SANDER J D,REYON D,et al.Improving CRISPR-Cas nuclease specificity using truncated guide RNAs[J].NatBiotechnol,2014,32(3):279-284.
[36]TREVINO A E,ZHANG F.Chapter eight-genome editing using Cas9 nickases[J].MethodsEnzymol,2014,546:161-174.
[37]YANG H,WANG H,SHIVALILA C S,et al.One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering[J].Cell,2013,153:1370-1379.
[38]WANG H,YANG H,SHIVALILA C S,et al.One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering[J].Cell,2013,153:910-918.
[39]NIU Y,SHEN B,CUI Y,et al.Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos[J].Cell,2014,156:836-843.
[40]CHEN F,WANG Y,YUAN Y,et al.Generation of B cell-deficient pigs by highly efficient CRISPR/Cas9-mediated gene targeting[J].JGenetGenomics,2015,42(8):437-444.
[41]TU Z,YANG W,YAN S,et al.CRISPR/Cas9:a powerful genetic engineering tool for establishing large animal models of neurodegenerative diseases[J].MolNeurodegener,2015,10:35-37.
[42]ZHOU X,XIN J,FAN N,et al.Generation of CRISPR/Cas9-mediated gene-targeted pigs via somatic cell nuclear transfer[J].CellMolLifeSci,2015,72(6):1175-1184.
[43]CRISPO M,MULET A P,TESSON L,et al.Efficient generation of myostatin knock-out sheep using CRISPR/Cas9 technology and microinjection into zygotes[J].PLoSOne,2015,10(8):366-375.
[44]WANG X,YU H,LEI A,et al.Generation of gene-modified goats targeting MSTN and FGF5 via zygote injection of CRISPR/Cas9 system[J].SciRep,2015,5:13878.
[45]YAN Q,ZHANG Q,YANG H,et al.Generation of multi-gene knockout rabbits using the Cas9/gRNA system[J].CellRegen(Lond),2014,3(1):12.
[46]VÉRON N,QU Z,KIPEN P A,et al.CRISPR mediated somatic cell genome engineering in the chicken[J].DevBiol,2015,407(1):68-74.
[47]KOTANI H,TAIMATSU K,OHGA R,et al.Efficient multiple genome modifications induced by the crRNAs,tracrRNA and Cas9 protein complex in zebrafish[J].PLoSOne,2015,10(5):128-134.
[48]HRUSCHA A,KRAWITZ P,RECHENBERG A,et al.Efficient CRISPR/Cas9 genome editing with low off-target effects in zebrafish[J].Development,2013,140(24):4982-4987.
[49]WANG Z.Genome engineering in cattle:recent technological advancements[J].ChromosomeRes,2015,23(1):17-29.
[50]HE X,TAN C,WANG F,et al.Knock-in of large reporter genes in human cells via CRISPR/Cas9-induced homology-dependent and independent DNA repair[J].NucleicAcidsRes,2016,[Epub ahead of print].
[51]ZETSCHE B,GOOTENBERG J S,ABUDAYYEH O O,et al.Cpfl is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system[J].Cell,2015,163(3):759-771.
[52]SLAYMAKER I M,GAO L,ZETSCHE B,et al.Rationally engineered Cas9 nucleases with improved specificity[J].Science,2016,351(6268):84-88.
[53]JIANG W,ZHOU H,BI H,et al.Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis,tobacco,sorghum and rice[J].NucleicAcidsRes, 2013,41(20):188-193.
(编辑郭云雁)
doi:10.11843/j.issn.0366-6964.2016.07.001
收稿日期:2016-01-08
基金项目:陕西省农业科技创新与攻关项目(2014K02-05-01);2015年西北农林科技大学第二批基本科研业务费科技创新专项(Z109021566)
作者简介:王玮玮(1990-),女,甘肃兰州人,硕士生,主要从事动物组织胚胎学方面的研究,E-mail:weiweiwang1990@163.com *通信作者:卿素珠,主要从事动物组织胚胎学及生殖内分泌调控研究,E-mail:suzhuqing@163.com
中图分类号:S813.3
文献标志码:A
文章编号:0366-6964(2016)07-1299-07
The Progress of CRISPR/Cas9 System and Its Application in Animal Genetic Engineering
WANG Wei-wei,LIU Rui-qi,WU Yong-yan,YANG Yan-ge,WANG Yong-sheng,QING Su-zhu*
(CollegeofVeterinaryMedicine,NorthwestA&FUniversity,Yangling712100,China)
Abstract:CRISPR/Cas (Clustered regularly interspaced short palindromic repeats (CRISPR)/CRISPR-associated(Cas)) system is a DNA modified gene editing tools regulated by a kind of short RNA.This technology is a novel tool,which is more rapid,efficient and accurate than ZFN (zinc-fingers nuclease) and TALEN (transcription activator-like (TAL) effector nucleases) in genetic engineering.It is easy to be designed and has specificity.This paper reviews the structure and function of CRISPR/Cas9 system and the design strategy of Cas9 and its application in animal genetic engineering.CRISPR/Cas9 is successfully used in a variety of animal genetic editing,it is likely to become a new way to establish animal models and to develop a new feasible approach to prevent disease.
Key words:CRISPR/Cas9 system;genetic engineering;sgRNA;gene knockout;gene knock-in;animal models
