桃PME基因家族的鉴定与分析
2016-07-23霍如雪刘振宁杨青
霍如雪++刘振宁++杨青
摘要:果胶甲酯酶属于碳水化合物酯酶家族CE-8,是调节果胶甲酯化程度的一种普遍存在的酶,是植物细胞壁的主要成分。为深入研究桃PME基因家族的功能,利用生物信息学方法对桃基因组中PME基因家族成员进行鉴定,并对其基因组信息、蛋白生理生化特征、基因结构、保守结构域和系统进化树等方面进行了研究。结果表明:在桃基因组中共鉴定出69个PME基因,可以分为Group Ⅰ、Group Ⅱ两大类,Group Ⅱ又可以进一步分成3个分支;PME基因在桃的8条染色体上呈现不均匀分布,并存在明显的串联重复现象,共发现18个串联重复基因簇,包含47个基因,串联重复是桃PME基因扩增的主要方式。对桃PME基因结构的分析表明,PME基因在进化上存在着一定的保守性;对桃PME蛋白氨基酸序列的多重比对发现了PME蛋白5个典型的保守结构区域;通过MEME软件对桃PME蛋白保守基序的分析发现了12个比较保守的基序。
关键词:桃;果胶甲酯酶;基因家族;生物信息学
中图分类号: S662.101文献标志码: A文章编号:1002-1302(2016)05-0024-07
E-mail:wgq@lcu.edu.cn。果胶是植物细胞初生壁和胞间层的主要成分[1],果胶的去酯化能够引起植物细胞结构和功能特性的改变,在果实成熟软化、器官脱落和衰老、花粉发育、花粉管生长和抗逆性等方面具有重要作用[2-9]。果胶的去酯化是由果胶甲酯酶(pectin methylesterase,PME,EC 3.1.1.11)催化完成的,果胶甲酯酶广泛存在于所有高等植物以及一些细胞壁降解的微生物如细菌、真菌中[10]。在高等植物中,果胶甲酯酶通常以多种异构体的形式存在,表现出对特定细胞类型的特异性[11]。
果胶甲酯酶基因(PMEs)属于一类比较大的基因家族,在高等植物中,根据其基因和蛋白的结构特点可以分为2类:Type Ⅰ、Type Ⅱ。Type Ⅱ类PME蛋白具有1个比较长的N-端PRO区域,而Type Ⅰ类PME蛋白的PRO区域很短甚至缺失。对PRO区域功能的研究表明,该区域具有将PME蛋白定位到细胞壁上、蛋白的正确折叠及对PME活性的抑制等作用[12]。对PME基因功能的研究表明,PME基因参与了形成层细胞的分化[13]、下胚轴的伸长[14]、花粉发育[7,15]、花粉管生长[5-6]和果实成熟[2,16]等一系列生理生化过程。另外,在食品加工和造纸等生产工艺中已广泛利用果胶甲酯酶活性来改善产品品质[17]。目前已经对拟南芥[6]、水稻[18]、杨树[19]、小立碗藓[20]等物种中的PME基因进行了全基因组鉴定和分析,而对桃PME基因的研究仅限于对桃果实成熟过程中PME蛋白活性变化[21-22]和PME基因表达量变化[23]的分析,缺乏从全基因组水平上对桃PME基因家族的相关研究。
桃(Prunus persica)是蔷薇科(Rosaceae)李属(Prunus)植物,是世界上广泛栽培的落叶果树之一。与蔷薇科木本植物如李、酸樱桃等多倍体果树相比,桃是二倍体(n=8 ),具有相对较小的基因组(230 Mbp),而且具有相对短的童期( 2~3年),这些独特的优点使得桃成为李属及其他蔷薇科植物的模式基因组物种[24]。桃基因组测序的完成和序列释放[25-26]使得在全基因组水平上对桃重要功能基因的发掘比较和功能研究成为可能。本研究利用生物信息学方法对桃PME基因家族成员进行鉴定和基因组注释,并分析了其基因结构和保守域,以期为进一步对桃PME家族基因的功能研究提供基础信息并最终在分子水平上对桃品种的改良提供明确候选基因。
1材料与方法
1.1桃PME基因家族的鉴定
首先,利用拟南芥PME蛋白的氨基酸序列作为种子序列在桃基因组数据库(http://www.rosaceae.org/species/prunus_persica/genome_ version 2.0)[26]中进行BLASTp比对搜索。其次,为保证候选基因没有被遗漏,我们又利用搜索到的PME蛋白的氨基酸序列对桃基因组数据库进行2次BLASTp比对搜索。最后,利用Pfam数据库(http://pfam.janelia.org/)[27]、SMART数据库(http://smart.embl-heidelberg.de/)[28]、NCBI的保守域数据库(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi) [29]分析候选蛋白的结构域,根据候选蛋白的氨基酸序列是否具有PME的保守结构域进行候选PME基因的筛选和鉴定。
1.2桃PME基因的基因组信息和染色体定位
桃PME基因的序列和基因组信息通过桃基因组数据库获得,并根据每个PME基因在染色体上的精确位置和染色体长度使用Photoshop软件将其人工定位到对应染色体上。
1.3桃PME蛋白的生理生化分析
PME蛋白的分子量和等电点通过Compute pI/Mwsoftware (http://www.expasy.ch/tools/pi_tool.html) [30]来预测。信号肽序列通过SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP/)[31]进行分析。糖基化位点通过NetNGly (http://www.cbs.dtu.dk/services/NetNGlyc/)[32]进行预测。
1.4桃PME基因的结构分析、保守域分析和进化树分析
利用GSDS网站(http://gsds1.cbi.pku.edu.cn/)[33]来分析PME基因的外显子-内含子结构。用在线的MEME软件(http://meme.sdsc.edu/meme/meme.html) [34]鉴定PME蛋白氨基酸序列中的基序。用Clustal X软件[35]对PME蛋白的氨基酸序列进行比对。在进化树的构建方面,首先利用MEGA5.0软件(http://www.megasoftware.net/)[36]自带的Clustal W应用对蛋白的氨基酸序列进行比对分析,空格罚分设置为10,空格扩展罚分设置为0.2。然后利用MEGA5.0软件将比对好的序列构建进化树,进化树使用邻接法构建,采用泊松校正、成对删除、1 000次重复等建树参数[37]。
2结果与分析
2.1桃PME基因家族的鉴定与注释
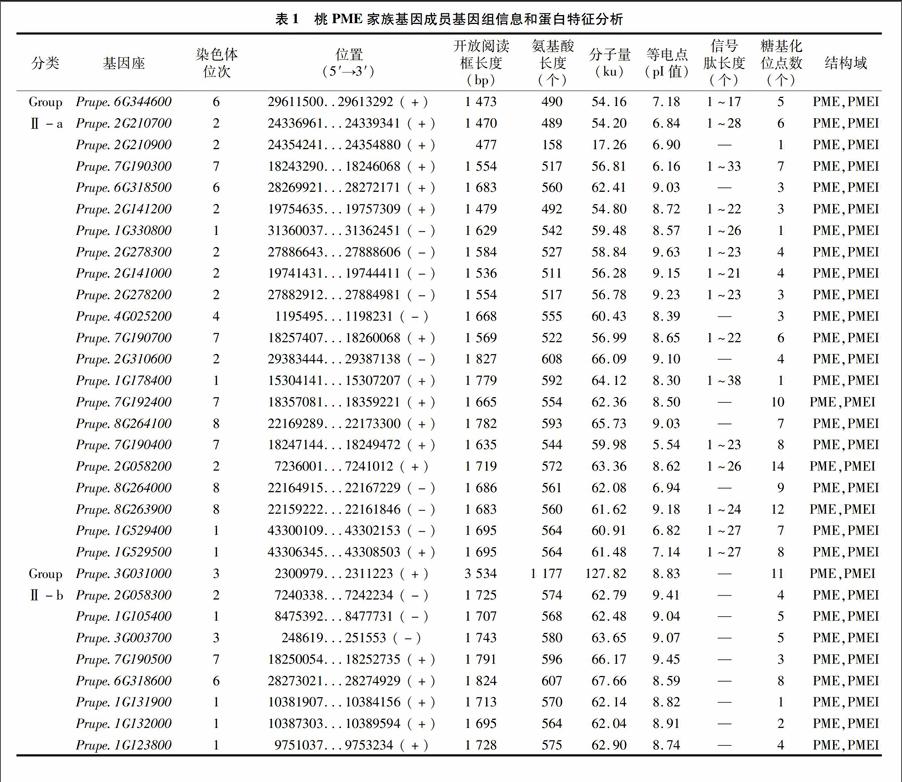
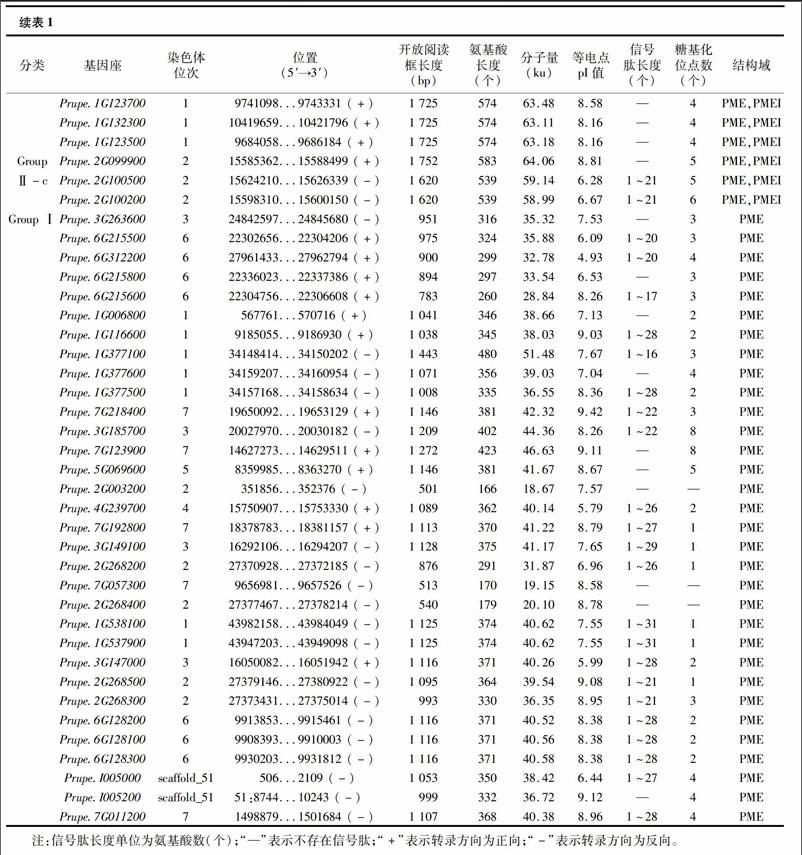
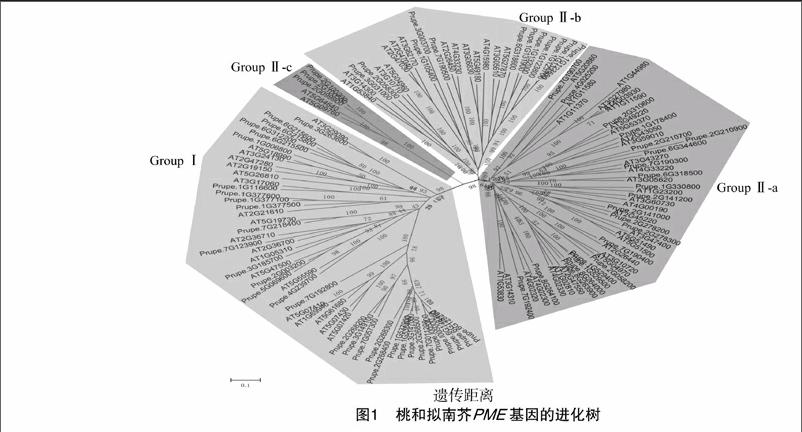
根据拟南芥PME蛋白的氨基酸序列,通过对桃基因组数据库的BLASTp比对搜索,在桃基因组中共鉴定出69个PME蛋白(表1)。根据PME蛋白中有无PME、PMEI结构域可以将这69个PME蛋白分成2大类:Group Ⅰ、Group Ⅱ。利用MEGA 5.0软件对桃和模式植物拟南芥中PME蛋白进化树构建和聚类分析的研究,又可以将Group Ⅱ细分成Group Ⅱ-a、Group Ⅱ-b、Group Ⅱ-c 3类(图1)。Group Ⅰ包括32个PME蛋白成员,只具有1个PME结构域;基因的开放阅读框长度相对较短,长度为501~1 443 bp,编码166~480位氨基酸,相应的蛋白分子量为18.67~51.48 ku,等电点范围为 4.93~9.42。Group Ⅱ包括37个PME蛋白成员,同时具有PME、PMEI结构域;基因的开放阅读框长度为477~3 534 bp,编码158~1 177位氨基酸,相应的蛋白分子量为17.26~127.82 ku,等电点为5.54~9.63。对桃PME蛋白信号肽的预测表明,其中39个PME蛋白存在1段N末端信号肽,信号肽长度范围在1~16到1~33个氨基酸之间。值得注意的是,Group Ⅱ-b中的12个蛋白成员均没有预测到信号肽的存在。前人研究发现,PME蛋白的糖基化影响其酶活性质,如热稳定性、对果胶的活性等[38-39]。笔者对桃PME蛋白糖基化位点的预测表明,PME蛋白中广泛存在着1~14个不等的糖基化位点。2.2桃PME基因的染色体定位分析
为对桃PME基因在染色体上的分布有更加明确的认识,根据桃PME基因的基因组信息,分别将其定位到桃的8条染色体上,其中Prupe.I005000、Prupe.I005200这2个PME基因位于scaffold_51上而未能正确定位到染色体上(图2)。结果表明,PME基因在桃的8条染色体上呈现不均匀分布,其中第1、2、6、7条染色体上具有较多的PME基因,而第3、4、5、8条染色体上的PME基因比较少。此外,PME基因在染色体上的分布存在明显的串联重复现象,共发现18个串联重复的基因簇,共包含47个基因,占PME基因总数的68.1%。
2.3桃PME基因的基因结构分析
为研究桃PME基因的基因结构,根据桃基因组信息获取了每个PME基因的DNA、CDS序列信息并构建了其基因结构(图3)。结果表明,大部分PME基因都有内含子结构。通过对基因结构和基因进化树的比较分析发现:每个分支中
2.4桃PME蛋白的氨基酸序列比对和保守域分析
为研究桃PME蛋白的保守结构域,利用CLUSTAL X对桃PME蛋白的氨基酸序列进行多重比对。图4的比对结果表明,PME蛋白一般具有5个典型的保守结构区域:Region Ⅰ(GxYxE)、Region Ⅱ(QAVAxR)、Region Ⅲ(QDTL)、Region Ⅳ(GxxDFIFG)、Region Ⅴ(YLGRxWx)。另外,利用MEME软件对桃PME蛋白保守基序的分析发现了12个比较保守的基序(motif 1~motif 12)(图5),其中motif 7、motif 6、motif 4、motif 1、motif 2分别对应Ⅰ—Ⅴ 5个保守结构域。
3结论与讨论
桃、拟南芥、水稻、杨树基因组大小分别为230、125、466、480 Mbp,这4个物种基因组中PME基因的数量分别为69、66、35、89个,说明桃基因组中PME基因出现了一定的数目扩增。基因扩增是基因组进化最主要的驱动力之一,是产生具有新功能的基因和进化出新物种的主要原因之一。基因可以通过多种方式进行扩增,包括全基因组复制、串连复制、片
段复制和逆转座复制等。对桃PME基因染色体定位的研究发现,PME基因在染色体上的分布存在明显的串联重复现象,共有18个串联重复基因簇,包含47个基因,占PME基因总数的68.1%,说明串联重复是桃PME基因扩增的主要方式。
对桃、拟南芥、杨树、水稻和小立碗藓等5个代表性物种中PME基因家族成员分类比较的研究发现,桃、拟南芥、杨树、水稻这4个高等植物中均同时具有Group Ⅰ、Group Ⅱ 2类PME基因,但是小立碗藓中只具有Group Ⅰ类的PME基因。笔者也分析了6种真菌、10种细菌中的PME基因,发现它们也都属于Group Ⅰ类,这说明Group Ⅱ类的PME基因是在苔藓植物、维管植物分化完成后才出现的,这与Markovi等对细菌、真菌和高等植物中PME基因进化树的分析结果[10]是一致的。但是关于PME基因到底是在哪一类物种中首先出现的,还需要进一步研究。
桃的使用主要是鲜食、加工2种方式,而这2种方式对果实的硬度、成熟度要求不同[40-41]。果实的软化主要是多聚半乳糖醛酸酶(PG)、PME相互作用的结果。目前一般认为,PME的作用是去除果胶分子链上半乳糖醛酸羧基上的酯化基团(主要是羟甲基或羟乙基),增加果胶在水中的溶解度,从而造成适于PG作用的条件[42]。Tieman等应用反义RNA技术在番茄中高表达反义PME mRNA,抑制了果实中果胶甲酯酶的活性,其成熟过程虽然没有受到明显干扰,但是果胶的高度甲酯化明显影响了果胶代谢,改善了果实品质[43]。另外,果胶甲酯酶在果汁生产中广泛用于澄清果汁、提高果浆出汁率、浸提和液化果肉组织以及用于生产混浊果汁及带果肉的果汁等[44],具有重要的食品加工价值。因而对桃PME基因的研究能够为培育符合市场需求的桃优良品种、应用基因工程改良果实成熟特性以及桃加工方式的创新等提供重要的理论依据,具有现实的指导意义。
参考文献:
[1]Varner J E,Lin L S. Plant cell wall architecture[J]. Cell,1989,56(2):231-239.
[2]Louvet R,Cavel E,Gutierrez L,et al. Comprehensive expression profiling of the pectin methylesterase gene family during silique development in Arabidopsis thaliana[J]. Planta,2006,224(4):782-791.
[3]Castillejo C,de la Fuente J I,Iannetta P,et al. Pectin esterase gene family in strawberry fruit:study of FaPE1,a ripening-specific isoform[J]. Journal of Experimental Botany,2004,55(398):909-918.
[4]Zhu S H,Zhou J. Effects of nitric oxide on fatty acid composition in peach fruits during storage[J]. Journal of Agricultural and Food Chemistry,2006,54(25):9447-9452.
[5]Bosch M,Cheung A Y,Hepler P K. Pectin methylesterase,a regulator of pollen tube growth[J]. Plant Physiology,2005,138(3):1334-1346.
[6]Tian G W,Chen M H,Zaltsman A,et al. Pollen-specific pectin methylesterase involved in pollen tube growth[J]. Developmental Biology,2006,294(1):83-91.
[7]Francis K E,Lam S Y,Copenhaver G P. Separation of arabidopsis pollen tetrads is regulated by QUARTET1,a pectin methylesterase gene[J]. Plant Physiology,2006,142(3):1004-1013.
[8]Schmohl N J,Fisahn J,Horst W J. Pectin methylesterase modulates aluminium sensitivity in Zea mays and Solanum tuberosum[J]. Physiologia Plantarum,2000,109(4):419-427.
[9]Collmer A,Keen N T. The role of pectic enzymes in plant pathogenesis[J]. Annual Review of Phytopathology,2003,24(1):383-409.
[10]Markovic^ O,Janec^ek S. Pectin methylesterases:sequence-structural features and phylogenetic relationships[J]. Carbohydrate Research,2004,339(13):2281-2295.
[11]Gaffe J,Tieman D M,Handa A K. Pectin methylesterase isoforms in tomato (Lycopersicon esculentum) tissues (effects of expression of a pectin methylesterase antisense gene)[J]. Plant Physiology,1994,105(1):199-203.
[12]Micheli F. Pectin methylesterases:cell wall enzymes with important roles in plant physiology[J]. Trends in Plant Science,2001,6(9):414-419.
[13]Micheli F,Sundberg B,Goldberg R,et al. Radial distribution pattern of pectin methylesterases across the cambial region of hybrid aspen at activity and dormancy[J]. Plant Physiology,2000,124(1):191-199.
[14]Al-Qsous S,Carpentier E,Klein-Eude D,et al. Identification and isolation of a pectin methylesterase isoform that could be involved in flax cell wall stiffening[J]. Planta,2004,219(2):369-378.
[15]Wakeley P R,Rogers H J,Rozycka M,et al. A maize pectin methylesterase-like gene,ZmC5,specifically expressed in pollen[J]. Plant Molecular Biology,1998,37(1):187-192.
[16]Eriksson E M,Bovy A,Manning K,et al. Effect of the colorless non-ripening mutation on cell wall biochemistry and gene expression during tomato fruit development and ripening[J]. Plant Physiology,2004,136(4):4184-4197.
[17]查笑君,马伯军,潘建伟,等. 果胶酯酶的研究进展[J]. 安徽农业科学,2010,38(16):8293-8295.
[18]Yokoyama R,Nishitani K. Genomic basis for cell-wall diversity in plants. A comparative approach to gene families in rice and Arabidopsis[J]. Plant & Cell Physiology,2004,45(9):1111-1121.
[19]Jane G L,Matt G,Coutinho P M,et al. Poplar carbohydrate-active enzymes. Gene identification and expression analyses[J]. Plant Physiology,2006,140(3):946-962.
[20]Lang D,Eisinger J,Reski R,et al. Representation and high-quality annotation of the Physcomitrella patens transcriptome demonstrates a high proportion of proteins involved in metabolism in mosses[J]. Plant Biology (Stuttgart,Germany),2005,7(3):238-250.
[21]胡留申,董晓颖,李培环,等. 桃果实成熟前后细胞壁成分和降解酶活性的变化及其与果实硬度的关系[J]. 植物生理学通讯,2007,43(5):837-841.
[22]田建文,许明宪,贺普超. 柿果实采收后软化生理分析(简报)[J]. 植物生理学通讯,1991,27(2):109-111.
[23]苏素香,赵彩平,曹丽军,等. 两种不同耐贮性桃果实采后乙烯合成和果实软化相关基因表达的差异[J]. 农业生物技术学报,2015,23(4):450-458.
[24]Shulaev V,Korban S S,Sosinski B,et al. Multiple models for Rosaceae genomics[J]. Plant Physiology,2008,147(3):985-1003.
[25]Arús P,Verde I,Sosinski B,et al. The peach genome[J]. Tree Genetics & Genomes,2012,8(3):531-547.
[26]International Peach Genome Initiative,Verde I,Abbott A G,et al. The high-quality draft genome of peach (Prunus persica) identifies unique patterns of genetic diversity,domestication and genome evolution[J]. Nature Genetics,2013,45(5):487-494.
[27]Punta M,Coggill P C,Eberhardt R Y,et al. The Pfam protein families database[J]. Nucleic Acids Research,2012,40(D1):D290-D301.
[28]Ivica L. Tobias D,Peer B. SMART 7:recent updates to the protein domain annotation resource[J]. Nucleic Acids Research,2012,40(D1):302-305.
[29]Marchler-Bauer A,Lu S,Anderson J B,et al. CDD:a conserved domain database for the functional annotation of proteins[J]. Nucleic Acids Research,2011,39:D225-D229.
[30]Gasteiger E,Gattiker A,Hoogland C,et al. ExPASy:the proteomics server for in-depth protein knowledge and analysis[J]. Nucleic Acids Research,2003,31(13):3784-3788.
[31]Petersen T N,Brunak S,von Heijne G,et al. SignalP 4.0:discriminating signal peptides from transmembrane regions[J]. Nature Methods,2011,8(10):785-786.
[32]Gupta R,Brunak S. Prediction of glycosylation across the human proteome and the correlation to protein function[C]. Kauai,Hawaii,USA:Pac Symp Biocomput,2002:310-322.
[33]Hu B,Jin J P,Guo A Y,et al. GSDS 2.0:an upgraded gene feature visualization server[J]. Bioinformatics,2015,31(8):1296-1297.
[34]Bailey T L,Williams N,Misleh C,et al. MEME:discovering and analyzing DNA and protein sequence motifs[J]. Nucleic Acids Research,2006,34:W369-W373.
[35]Thompson J D,Gibson T J,Plewniak F,et al. The CLUSTAL_X Windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research,1997,25(24):4876-4882.
[36]Tamura K,Peterson D,Peterson N,et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J]. Molecular Biology and Evolution,2011,28(10):2731-2739.
[37]Saitou N,Nei M. The neighbor-joining method:a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution,1987,4(4):406-425.
[38]Giovane A,Quagliuolo L,Castaldo D,et al. Pectin methyl esterase from Actinidia chinensis fruits[J]. Phytochemistry,1990,29(9):2821-2823.
[39]And R G,C Grohmann K. Purification and characterization of a thermally tolerant pectin methylesterase from a commercial Valencia fresh frozen orange juice[J]. Journal of Agricultural & Food Chemistry,1996,44(2):458-462.
[40]尤超,刘涛,汪亚,等. 油桃嫁接繁殖育苗对比试验[J]. 江苏农业科学,2015,43(4):185-186.
[41]钱巍,严娟,马瑞娟,等. 不同成熟期黄肉桃糖酸组分的测定[J]. 江苏农业科学,2015,43(2):287-290.
[42]Huber D J. The role of cell wall hydrolases in fruit softening[J]. Horticultural Reviews,1983,5:169-219.
[43]Tieman D M,Harriman R W,Ramamohan G,et al. An antisense pectin methylesterase gene alters pectin chemistry and soluble solids in tomato fruit[J]. The Plant Cell,1992,4(6):667-679.
[44]秦星,张华方,张伟,等. 酶制剂在果汁生产中的应用研究进展[J]. 中国农业科技导报,2013,15(5):39-45.赵志常,张波,高爱平,等. 芒果UFGT基因的克隆及表达分析[J]. 江苏农业科学,2016,44(5):31-34.
