不同pH值对变异链球菌ffh基因表达的影响
2016-07-22陈泽文黎静李恺得邱传彬谯跃银薛晶李雨庆
陈泽文黎静李恺得邱传彬谯跃银薛晶李雨庆
1.口腔疾病研究国家重点实验室 华西口腔医院(四川大学);2.牙体牙髓病科,成都 610041
不同pH值对变异链球菌ffh基因表达的影响
陈泽文1黎静1李恺得1邱传彬1谯跃银1薛晶2李雨庆1
1.口腔疾病研究国家重点实验室 华西口腔医院(四川大学);2.牙体牙髓病科,成都 610041
[摘要]目的 检测变异链球菌(S. mutans)ffh基因在不同pH值条件下的表达水平,分析pH值对S. mutans ffh基因表达的影响,分析调控ffh表达的因素。方法 在不同培养时段(4 h和18 h)和不同pH值(pH值4.0~7.0)条件下培养S. mutans标准菌株UA159,提取样本,用荧光定量聚合酶链反应(qRT-PCR)法检测样本中目的基因ffh的mRNA转录水平的变化情况,分析S. mutans ffh基因在不同培养时段和pH值条件下的表达变化趋势。结果 培养4 h时,ffh基因相对表达量随着pH值的降低而减少,培养18 h时,ffh基因相对表达量随着pH值的降低而增加;相同pH值条件下,ffh基因相对表达量在培养4 h与18 h时的差异有统计学意义(P<0.05)。结论 S. mutans ffh基因的表达受细菌培养时段和pH值的影响。
[关键词]变异链球菌; ffh基因; 荧光定量聚合酶链反应
变异链球菌(Streptococcus mutans,S. mutans)和龋病及继发的牙髓炎密切相关[1]。研究[2]表明,S. mutans的致龋特性来自于较强的黏附能力,合成细胞外多糖的能力,产酸能力和耐酸能力。致龋菌均能产酸,但并非所有能产酸的细菌均能致龋,因为随着菌斑pH值的降低,一些细菌失去了产酸能力,在临界pH值时,只有少数耐酸的细菌如S. mutans和乳杆菌能够生长[3]。S. mutans的产酸能力和耐酸能力是其主要的毒力因素,该菌的产酸能力很强,在菌斑pH值下降时能自适应发生耐酸反应(acid-tolerance response,ATR),能在低至pH值为3.0的酸性条件下生存[4-5]。目前,致龋菌的耐酸性是研究抗龋病机制的重要方向。有研究[4,6]发现,在S. mutans分泌和耐酸性相关区域sat中,有5个基因操纵子,分别为ffh、ylxM、satC、satD和satE,其中ffh基因可以明显影响S. mutans的耐酸性,但目前为止对调控ffh基因表达的因素尚不清楚。
为进一步了解ffh基因的调控因素,本实验运用荧光定量聚合酶链反应(fluorescence quantitative realtime polymerase chain reaction,qRT-PCR)的方法定量检测S. mutans的ffh基因在不同培养时段以及pH值环境的表达差异,分析调控S. mutans ffh基因表达的因素。
1 材料和方法
1.1 实验菌株和培养基
实验菌株采用S. mutans标准株UA 159,培养基为7组不同pH值(分别为4.0、4.5、5.0、5.5、6.0、6.5、7.0)的TPY培养基。
1.2 细菌的培养取样
将冻存的S. mutans标准株UA 159于室温下解冻,复苏,接种于TSB培养液中,37 ℃微需氧(95%N2,5%CO2)条件下活化培养24 h,显微镜下检查排除杂菌污染。在BHI固体培养基上划线接种,37 ℃微需氧条件下培养24 h,挑取单个典型菌落,于TPY培养液中37 ℃微需氧条件下培养24 h。
取7组不同pH值的TPY培养基,每组设3只离心管,分别加入10 mL已培养好的菌液,4 000 r·min-1离心10 min。倒掉上清液,留下细菌沉淀物,每组分别加入不同pH值的TPY培养基,均匀混合后取适量不同组菌液加入96孔细胞培养板,在自动酶标仪下检测S. mutans 24 h生长曲线。剩余样本置入培养箱中37 ℃兼性厌氧培养。分别于培养4 h和18 h时取样,7组菌液样本分别标号,加入RNA保存液后放入-20 ℃冰箱冻存备用。
1.3 qRT-PCR检测
1.3.1 引物设计 结合GenBank的基因序列和引物设计原则,采用primer premier 5.0软件设计ffh基因引物,而16S rRNA引物序列参照参考文献[7]设计,委托北京赛尔星生物科技有限公司合成,引物序列如下。S. mutans ffh:上游引物5’-CGACAGGTAAGGCGACATC-3’,下游引物5’-GCGGCAAACGCAAACT-3’;S. mutans 16S rRNA:上游引物5’-AGCGTTGTCCGGATTTATTG-3’,下游引物5’-CTACGCATTTCACCGCTACA-3’。
1.3.2 qRT-PCR检测 使用超纯RNA提取试剂盒TRI-zol®Reagent提取组织样本中的总RNA,实验操作按产品说明书进行。取5 μL RNA用1%琼脂糖凝胶进行电泳。用M-MLV Reverse Transcriptase cDNA第一链合成试剂进行反转录,实验操作按产品说明书进行;用UltraSYBR Mixture(with Rox)(北京康为世纪生物科技有限公司)进行扩增,实验操作按产品说明书进行。制成qRT-PCR反应体系(10 μL UltraSYBR Mixture,0.4 μL上游引物,0.4 μL下游引物,2 μL模板,加灭菌蒸馏水至20 μL);扩增程序为变性95 ℃10 min,95 ℃10 s、60 ℃ 30 s、72 ℃ 30 s,45个循环。样品qRT-PCR分析:将各样品cDNA 10倍稀释后取2 μL作模板,分别用目的基因引物和内参基因引物进行扩增,同时在60~95 ℃进行溶解曲线分析。
1.3.3 数据处理 用Bio-Rad CFX96 qRT-PCR仪,采用2-ΔΔCT法对ffh基因mRNA的相对表达量进行数据分析。在培养4 h和18 h不同pH值条件下的ΔCt计算公式如下:ΔCt=ffh基因Ct值-16S rRNA Ct值。以4 h且pH=4.0实验样本为对照组,ΔΔCt=ΔCt处理组-ΔCt对照组,计算得出ffh基因mRNA的相对表达量。运用SPSS 17.0统计软件进行数据分析,相同pH值下不同培养时段比较采用配对t检验,相同培养时段不同pH值组间比较采用方差分析,检验水准为双侧α=0.05。
2 结果
2.1 S. mutans生长曲线
测定7组不同pH值下细菌在设定温度为37 ℃、OD600的生长曲线,结果如图1所示。4 h位于平台期开始,18 h位于平台期末。在pH=4.0条件下生长曲线变化最小,指数期略有下降,平台期细菌量最少;pH值为5.5、6.0、6.5、7.0条件下生长曲线变化大致相等,指数期增长最快,平台期细菌量最多;pH值为4.5和5.0条件下细菌生长曲线变化及平台期细胞量居两者之间。由此可知,pH<5.5时S. mutans的生长受到抑制。
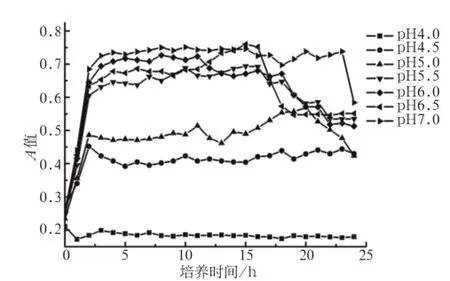
图 1 TPY培养基中S. mutans的生长曲线Fig 1 The growth curves of S. mutans in TPY culture medium
2.2 RNA电泳和溶解曲线
琼脂糖凝胶电泳结果见图2,各组细菌提取的总RNA完整无降解,总RNA纯度达到提纯要求,可将样本RNA进行后续实验。引物筛选结果得到ffh与16S rRNA基因溶解曲线都为单峰,均一性好(图3、4),实验结果有效。
图 2 RNA电泳结果Fig 2 The results of RNA electrophoresis
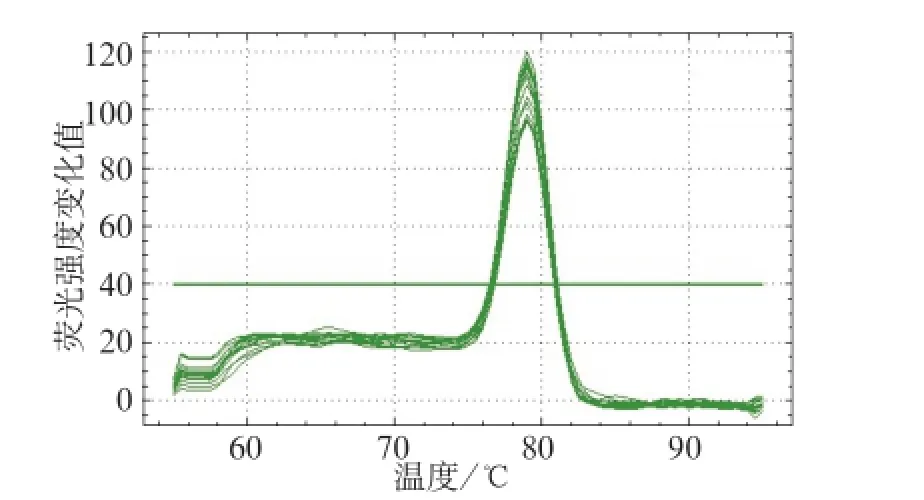
图 3 ffh基因溶解曲线Fig 3 The curves of ffh gene dissolution
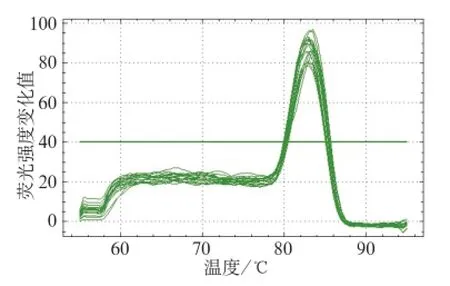
图 4 16S rRNA基因溶解曲线Fig 4 The curves of 16S rRNA gene dissolution
2.3 ffh基因mRNA相对表达量
不同pH值条件下ffh基因在培养4 h和18 h时的相对表达量见图5。

图 5 ffh基因在不同pH值培养基中的相对表达量Fig 5 The relative expression levels of ffh gene under different pH conditions
培养4 h时,ffh基因的相对表达量在pH<5.5低于pH>5.5(P<0.05),培养18 h时ffh基因的相对表达量在pH<6.0高于pH>6.0(P<0.05)。pH=4.0、pH= 4.5培养条件下,ffh基因的相对表达量在18 h时较4 h时更高(P<0.05),pH=6.0、pH=6.5、pH=7.0培养条件下ffh基因的相对表达量在18 h时较4 h时更低(P<0.05),pH=5.0、pH=5.5时ffh基因的相对表达量无明显差异(P>0.05)。
3 讨论
S. mutans是龋病的主要致龋菌[8],耐酸性在其致龋性中具有重要作用。当pH值下降到临界值(5.5)以下时,可造成羟磷灰石中钙丢失,釉质脱矿。一些在中性环境中本来可以抵抗的有益菌,如格氏链球菌[9],在临界值以下会处于弱势,对S. mutans的拮抗作用降低。有效抑制S. mutans的耐酸性是龋病研究的重点,而ffh基因能够影响致龋菌的耐酸性并参与ATR[3],若ffh基因失去活性会导致S. mutans失去耐酸性[6]。ffh基因是真核细胞信号识别颗粒(相对分子质量5.4×104)的同系物[10]。有研究[11]表明,ffh基因通过编码信号识别颗粒(signal recognition particle,SRP)的重要组成成分Ffh蛋白来参与其耐酸性。Williams等[12]发现,Ffh蛋白具有鸟苷三磷酸酶(guanosine triphosphatase,GTPase)活性且与7S或4.5S小细胞质RNA(small cytosol RNA,scRNA)形式稳定络合物SRP。S. mutans在不具有SRP通路的情况下仍可存活,但对环境适应能力减弱。Ffh蛋白和scRNA是细菌进行翻译时蛋白质运输途径中SRP的核心要素[13]。SRP通路在细菌中参与膜生物合成和分泌蛋白质等活动,而且在细胞内能引导蛋白质大分子到其相应的位点[8]。这表明SRP通路主要参与S. mutans的应激反应过程。
研究[14]表明,ffh基因突变会对酸性环境更加敏感,然而并不会使S. mutans失去生存能力,但是ffh基因的异常或缺失会极大地改变包括耐酸性在内的生理功能。ffh基因表达上调时会参与蛋白质运输和细胞膜应激反应,耐酸反应和DNA修复等。Xu等[7]通过实验也证实,ffh基因有影响膜质子泵功能的质子移位膜ATP酶(translocating ATPase,F-ATPase)活性,改建几个涉及重要代谢过程中pH稳态、丙酮酸异化和糖运输的功能。
基于ffh基因在S. mutans耐酸中表现的突出作用,本文从酸对ffh基因表达的影响进行研究。本实验运用qRT-PCR方法研究培养4 h和18 h时S. mutans在不同pH值条件下ffh基因的表达差异,结果显示,S.mutans在pH值为4.0~7.0培养4 h与18 h时的ffh基因相对表达量有明显差异。S. mutans在培养4 h时,在pH值为4.0和4.5环境中,ffh基因表达水平小于在pH值为6.0、6.5、7.0中,而在18 h时则相反,提示pH≤4.5时S. mutans ffh基因表达增加而pH>6.0时S. mutans ffh基因表达受到抑制。S. mutans在pH值为5.0和5.5 时ffh基因表达水平在培养4 h和18 h时无明显变化(P>0.05),提示这种酸性条件对ffh基因的表达无明显影响。进一步分析pH值和培养时间对ffh基因的表达影响,S. mutans在刚开始接触酸性环境时,因为有害刺激使细菌处于应激状态,生长代谢受限甚至导致部分细菌死亡,同时也抑制ffh基因表达,但随着其ATR机制的产生,细菌逐渐适应酸性环境,ffh基因表达也逐渐增加,进一步促进细菌适应酸性环境。酸性环境初期会抑制S. mutans ffh基因的表达,但随着ffh基因表达的增加,可促进细菌对酸的耐受能力,使其适应酸性环境并稳定生存。
本研究结果显示,pH值和培养时间是影响S. mutans ffh基因表达的重要因素,ffh基因在不同pH值条件下和培养时段的表达不同。本实验时间点选取偏少,也只选取了pH值为4.0~7.0的范围,下一步需排除各种因素间的相互干扰,筛选调控ffh表达的主要因素并明确其作用机制。
[参考文献]
[1]Hong SW, Baik JE, Kang SS, et al. Lipoteichoic acid of Streptococcus mutans interacts with Toll-like receptor 2 through the lipid moiety for induction of inflammatory mediators in murine macrophages[J]. Mol Immunol, 2014,57(2):284-291.
[2]王成龙, 刘佼佼, 苏东华, 等. 高致龋性变异链球菌临床分离株的初步筛选[J]. 华西口腔医学杂志, 2013, 31(2): 136-140. Wang CL, Liu JJ, Su DH, et al. Preliminary screen of high cariogenicity Streptococcus mutans strains isolated from clinical specimens[J]. West China J Stomatol, 2013, 31(2): 136-140.
[3]Matsui R, Cvitkovitch D. Acid tolerance mechanisms utilized by Streptococcus mutans[J]. Future Microbiol, 2010,5(3):403-417.
[4]Palmer SR, Crowley PJ, Oli MW, et al. YidC1 and YidC2 are functionally distinct proteins involved in protein secretion, biofilm formation and cariogenicity of Streptococcus mutans[J]. Microbiology (Reading, Engl), 2012, 158(7): 1702-1712.
[5]Guo L, Hu W, He X, et al. Investigating acid production by Streptococcus mutans with a surface-displayed pH-sensitive green fluorescent protein[J]. PLoS ONE, 2013, 8(2):e57182.
[6]Crowley PJ, Svensäter G, Snoep JL, et al. An ffh mutant of Streptococcus mutans is viable and able to physiologically adapt to low pH in continuous culture[J]. FEMS Microbiol Lett, 2004, 234(2):315-324.
[7]Xu X, Zhou XD, Wu CD. The tea catechin epigallocatechin gallate suppresses cariogenic virulence factors of Streptococcus mutans[J]. Antimicrob Agents Chemother, 2011,55(3):1229-1236.
[8]Li D, Shibata Y, Takeshita T, et al. A novel gene involved in the survival of Streptococcus mutans under stress conditions[J]. Appl Environ Microbiol, 2014, 80(1):97-103.
[9]刘敏, 闫嘉惟, 刘娅玲, 等. 生长期和pH值对格氏链球菌自溶酶atlS基因表达的影响[J]. 华西口腔医学杂志, 2015,33(1):80-83. Liu M, Yan JW, Liu YL, et al. Effects of growth stages and pH value on the expression of autolytic enzyme atlS gene of Streptococcus gordonii[J]. West China J Stomatol, 2015,33(1):80-83.
[10]Borden JR, Jones SW, Indurthi D, et al. A genomic-library based discovery of a novel, possibly synthetic, acid-tolerance mechanism in Clostridium acetobutylicum involving noncoding RNAs and ribosomal RNA processing[J]. Metab Eng,2010, 12(3):268-281.
[11]Treviño J, Perez N, Sumby P. The 4.5S RNA component of the signal recognition particle is required for group A Streptococcus virulence[J]. Microbiology (Reading, Engl),2010, 156(Pt 5):1342-1350.
[12]Williams ML, Crowley PJ, Hasona A, et al. YlxM is a newly identified accessory protein that influences the function of signal recognition particle pathway components in Streptococcus mutans[J]. J Bacteriol, 2014, 196(11):2043-2052.
[13]Liao S, Klein MI, Heim KP, et al. Streptococcus mutans extracellular DNA is upregulated during growth in biofilms,actively released via membrane vesicles, and influenced by components of the protein secretion machinery[J]. J Bacteriol, 2014, 196(13):2355-2366.
[14]Jin J, Liu S, Zhao L, et al. Changes in ffh, uvrA, groES and dnaK mRNA abundance as a function of acid-adaptation and growth phase in Bifidobacterium longum BBMN68 isolated from healthy centenarians[J]. Curr Microbiol, 2011, 62(2): 612-617.
(本文编辑 吴爱华)
[中图分类号]R 780.2
[文献标志码]A [doi] 10.7518/hxkq.2016.01.005
[收稿日期]2015-06-05; [修回日期] 2015-10-02
[基金项目]四川大学大学生创新创业训练计划省级项目(20140282)
[作者简介]陈泽文,学士,E-mail:1214150709@qq.com
[通信作者]李雨庆,副教授,博士,E-mail:liyuqing@scu.edu.cn
Effects of different pH conditions on ffh gene expression in Streptococcus mutans
Chen Zewen1, Li Jing1, Li Kaide1, Qiu Chuanbin1, Qiao Yueyin1, Xue Jing2, Li Yuqing1.
(1. State Key Laboratory of Oral Diseases, West China Hospital of Stomatology, Sichuan University, Chengdu 610041, China; 2. State Key Laboratory of Oral Diseases, Dept. of Conservative Dentistry and Endodontics, West China Hospital of Stomatology, Sichuan University, Chengdu 610041, China)
Supported by: Provincial Fundation for Innovative and Entrepreneurial Training Program for College Students in Sichuan University (20140282). Correspondence: Li Yuqing, E-mail: liyuqing@scu.edu.cn.
[Abstract]Objective This research aimed to detect the expression levels of ffh gene in Streptococcus mutans (S. mutans)UA159 under different pH conditions, analyze the effect of pH on the expression of ffh gene in S. mutans, and identify the factors regulating the ffh gene expression. Methods Samples of S. mutans were collected at different growth stages (4 h,18 h) and pH values (pH 4.0-7.0). Fluorescence quantitative real-time polymerase chain reaction (qRT-PCR) was used to measure the relative mRNA expression and trend of the target gene ffh in S. mutans at different growth stages and pH values. Results qRT-PCR results showed that the ffh gene expression decreased along with pH at 4 h, but the expression increased with decreasing pH at 18 h. Under the same pH conditions, the ffh gene expression was significantly different between 4 h and 18 h (P<0.05). Conclusion Growth stage and pH value influenced the ffh gene expression in S. mutans.
[Key words]Streptococcus mutans; ffh gene; fluorescence quantitative real-time polymerase chain reaction
