玉米Glyco-hydro-16糖苷酶家族全基因组的鉴定及其遗传分化
2016-07-18林峰,葛敏,周玲,赵涵
林 峰,葛 敏,周 玲,赵 涵
(1江苏省农业科学院农业生物技术研究所/江苏省农业生物学重点实验室,南京 210014;2上海市能源作物育种及应用重点实验室,上海 200444)
玉米Glyco-hydro-16糖苷酶家族全基因组的鉴定及其遗传分化
林峰1, 2,葛敏1,周玲1,赵涵1
(1江苏省农业科学院农业生物技术研究所/江苏省农业生物学重点实验室,南京 210014;2上海市能源作物育种及应用重点实验室,上海 200444)
摘要:【目的】全基因组水平鉴定玉米Glyco-hydro-16家族,分析该家族基因在不同组织中的表达模式以及在不同玉米杂种优势群中的遗传分化。【方法】根据 Glyco-hydro-16家族相对保守的序列及结构域,构建Glyco-hydro-16家族的隐马尔科夫模型文件(Glyco-hydro-16.hmm),利用hmmersearch程序在玉米全基因组中进行比对,获得玉米中含有该家族保守结构域的所有序列。通过Blast2GO进行功能注释,利用蛋白质序列构建该家族的系统发育进化树。使用玉米自交系B73不同组织及不同发育时期的RNA-seq数据库分析该家族基因的表达模式。根据该家族基因在染色体上的位置筛选 SNP标记,计算其在不同玉米杂种优势群间的群间遗传分化系数(genetic differentiation coefficient,Fst),分析其遗传分化。【结果】根据该家族相对保守的序列及结构域,在全基因组水平共鉴定出 34个玉米 Glyco-hydro-16家族成员,注释表明所有基因都是木葡聚糖转移酶/水解酶基因,3个保守性较高的Motif区段存在于该家族所有成员中。通过系统发育关系和序列相似性将该家族分为8个亚家族,每个亚家族有2—8个基因,分布在除第3和第6染色体外的其他8条染色体上,在第2、第5及第10染色体上成簇分布。该家族在禾本科作物中同源性较高,与拟南芥分属不同的分支,但只有3个玉米成员(AC210669.3、GRMZM2G413006和GRMZM2G166944)被划分到禾本科分支中,其他玉米成员被划分到单独的分支中。通过表达谱分析表明该家族成员在玉米中均有表达,但在不同组织中的表达水平有差异。为解析该家族基因在不同玉米种质资源中等位基因的变异,根据玉米Glyco-hydro-16家族基因在染色体上的位置筛选SNP标记,计算其在玉米杂种优势群SS及NSS间的群间遗传分化系数。结果显示,共有10个该家族基因所处位点的Fst值高于阈值0.15,达到高度分化水平,分别位于第1、第2、第4、第5、第7以及第9染色体上。其中,位于第2染色体上的GRMZM2G091118相应位点的Fst值为0.52,表明该位点在SS群和NSS群间的群间遗传分化度极大。【结论】通过全基因组扫描在玉米中鉴定出34个Glyco-hydro-16家族成员,均为木葡聚糖转移酶/水解酶基因,在不同组织中,其表达模式不同,可能参与不同生理发育过程。部分该家族成员所处位点在玉米杂种优势群SS和NSS间的等位基因分化极大。
关键词:玉米;Glyco-hydro-16;基因家族;杂种优势群;RNA-seq
联系方式:林峰,E-mail:fenglin@jaas.ac.cn。通信作者赵涵,Tel:025-84390751;E-mail:zhaohan@jaas.ac.cn
0 引言
【研究意义】糖是构成生物体的基本成份之一,既是生命活动的主要能源,又是细胞结构的重要成分,是生物体存在的基础。糖苷酶又称糖苷水解酶,是水解糖苷键酶类的总称,是糖代谢过程的重要组成部分,参与多种生物学功能[1]。随着糖类物质在生物学领域内研究的开展,关于糖苷酶的研究与应用越来越重要。因此,从全基因组水平解析糖苷酶家族的结构和功能有助于糖生物学的发展并为糖类的生物学功能研究奠定基础。【前人研究进展】糖苷酶是水解酶类中的一大类,种类繁多,主要有按底物分类和按序列相似性分类等分类方式。根据序列相似性进行分类可以反映出这些酶的结构特征,有助于分析其进化关系,并且为机制研究提供便利。根据这种分类方式,目前可将糖苷酶划分为 135个不同家族[2-3](http://www.cazy.org/Glycoside-Hydrolases.html)。Glyco-hydro-16是其中一个家族,含有多个成员,包括木葡聚糖基转移酶(xyloglucan:xyloglucosyl transferase)、硫酸角质素 β-1,4-半乳糖苷内切酶(keratan-sulfate endo-1,4-β-galactosidase)、β-1,3-葡聚糖内切酶(endo-1,3-β-glucanase)、地衣淀粉酶(licheninase)、β-琼胶酶(β-agarase)、κ-卡拉胶酶(κ-carrageenase)、木葡聚糖酶(xyloglucanase)、β-1,3-半乳糖内切酶(endo-β-1,3-galactanase)、β-紫菜聚糖酶(β-porphyranase)、透明质酸酶(hyaluronidase)、β-1,4-半乳糖苷内切酶(endo-β-1,4-galactosidase)、β-1,6-几丁质糖基转移酶(chitin β-1,6-glucanosyl transferase)等。该家族成员具有多种功能,参与不同碳水化合物复合体的新陈代谢,主要是具有转糖基酶或水解酶作用,通过水解木葡聚糖影响植物细胞壁的松驰[4]。在拟南芥中该类基因通过影响雄蕊花丝形成时细胞壁的变化,调控自花授粉[5]。木葡聚糖内糖基转移/水解酶不仅作用于木葡聚糖,而且对葡聚糖和阿拉伯木聚糖也起作用[6]。在水稻中其同源基因在生长发育和抗盐胁迫中发挥功能[7]。在甲壳类生物中与内源 β-1,3-葡聚糖酶的合成有关[8]。禾本科生物中的xyloglucan transglucosylases hydrolases主要与细胞壁的降解相关[6],并与芸芥自交亲和性的调控相关[9]。另外,玉米中的Glyco-hydro-16家族成员可能与低磷胁迫响应相关[10]。【本研究切入点】虽然在玉米中已鉴定出少量Glyco-hydro-16家族成员,但全基因组系统研究以及该家族基因在不同玉米种质资源中等位基因遗传分化的研究尚未见报道。【拟解决的关键问题】本研究通过全基因组扫描鉴定玉米Glyco-hydro-16家族,从家族成员基因结构、系统进化发育关系、成员在玉米不同组织及不同发育时期的表达谱等方面分析该家族的结构与功能,利用玉米杂种优势群SS和NSS分析该家族基因在不同玉米种质资源中等位基因的遗传分化。
1 材料与方法
1.1 玉米Glyco-hydro-16家族的鉴定
Glyco-hydro-16家族属于糖苷水解酶家族,根据其相对保守的序列信息及结构域,利用 Pfam数据库构建 Glyco-hydro-16家族的隐马尔科夫模型文件(Glyco-hydro-16.hmm),然后利用HMMER3.0软件包中的hmmersearch程序在V3版玉米自交系B73的参考基因组数据库(http://www.maizegdb.org/)中进行比对,获得玉米中含有Glyco-hydro-16家族保守结构域的所有序列。所有Glyco-hydro-16家族成员通过NCBI CDD(Conserved Domain Database)数据库进一步验证,并根据序列相似性进行去冗余处理。最后获得玉米Glyco-hydro-16家族信息。
1.2 玉米Glyco-hydro-16家族基因的功能注释
利用 Blast2GO V3.1.3[11]对所获得基因进行注释,首先运行BLASTX,阈值设为1.0E-3,然后运行mapping得到GO term,最后进行注释,阈值为1.0E-6。
1.3 玉米Glyco-hydro-16家族保守基序的鉴定与分析
为解析玉米Glyco-hydro-16家族的蛋白质结构特征,利用MEME V4.11.1[12](http://meme-suite.org/tools/ meme)对候选玉米Glyco-hydro-16家族成员的序列进行分析,鉴定保守基序。
1.4 玉米Glyco-hydro-16家族的系统发育分析
利用Clustal X程序[13]对玉米Glyco-hydro-16家族的蛋白质序列进行多重比对,然后使用MEGA6.0软件中的邻近法(Neighbor-Joining algorithm)[14]构建该家族的系统发育进化树,Bootstrap值设为1 000。
1.5 玉米Glyco-hydro-16家族基因在不同组织中的表达分析
为解析玉米 Glyco-hydro-16家族基因的表达模式,利用玉米自交系B73不同组织及不同发育时期的RNA-seq数据库(http://www.ncbi.nlm.nih.gov/Traces/ sra/?study=SRP010680)分析该家族成员在不同组织中的表达情况。B73的RNA-seq数据库由NCBI网站下载后,先利用SolexaQA[15]软件包对原始测序数据进行质量(Q20,Phred-Score≥20即1%的错误率)和测序长度(L20,长度≥20 bp)过滤,然后使用 Bowtie2 (V2.1.0)[16]将过滤后的序列锚定到B73 V3版参考基因组上。通过 Cufflinks(V2.1.1)[17]计算得到Glyco-hydro-16家族基因表达量的 FPKM 值。利用MEV4.9软件绘制Glyco-hydro-16家族基因的Heatmap图,分析其表达模式。
1.6 Glyco-hydro-16家族基因在玉米SS和NSS群中的遗传分化分析
为解析该家族基因在不同玉米种质资源中的变异,利用玉米杂种优势群SS和NSS分析该家族基因的分化。玉米是典型的异交作物,20世纪50年代美国形成了称为SS、NSS以及IDT的3个杂种优势群[18-19],群间的配组能够产生较高的杂种优势。根据前人研究结果,下载分别被划入SS群和NSS群的289个玉米自交系的基因型数据[20-21],根据Glyco-hydro-16家族基因所处位置,筛选位于该家族基因内部及上下游1 kb内的SNP位点,使用vcftools计算上述位点在2个杂种优势群间的群间遗传分化系数(genetic differentiation coefficient,Fst),window-size设为5 000,window-step设为 1 000,度量该家族基因在玉米杂种优势群 SS 及NSS中的遗传分化程度。Fst值在0—0.05表示群体间不存在分化;Fst值在0.05—0.15为中度分化;Fst值在0.15—0.25为高度分化,大于0.25表明分化度极大[22]。
2 结果
2.1 玉米Glyco-hydro-16家族的鉴定与分析
根据该家族相对保守的序列及结构域,在 Pfam数据库中构建其隐马尔科夫模型文件(Glyco-hydro-16.hmm),通过HMMER软件在B73 V3版玉米参考基因组序列中进行比对,搜索到 61条含有Glyco-hydro-16家族保守结构域的序列,经过去冗余处理,共对应34个基因。通过Blast2GO注释表明所有基因都是木葡聚糖转移酶/水解酶基因。通过MEME对上述Glyco-hydro-16家族蛋白的保守Motif进行分析,结果表明,3个保守性较高的Motif区段存在该家族所有成员中(图1)。
2.2 玉米Glyco-hydro-16家族的系统发育分析
利用Glyco-hydro-16家族蛋白质序列,通过邻近法(Neighbor-Joining algorithm)构建系统发育进化树。虽然玉米Glyco-hydro-16家族各成员具有相对保守的序列及结构域,但彼此之间仍有差异,可划分成8个亚家族,各个亚家族分别含有2—8个基因(图2-a)。为解析玉米中 Glyco-hydro-16家族与其他植物同源基因在进化上的关系,对包括拟南芥、水稻、小麦、大麦以及高粱等在内的Glyco-hydro-16家族蛋白序列进行聚类分析。可以看出该家族在禾本科作物中同源性较高,与拟南芥分属不同的分支,但只有3个玉米成员(AC210669.3、GRMZM2G413006以及GRMZM2G166944)划分到禾本科分支中,其他玉米成员的变异都较大,划分为单独的分支(图2-b)。

图1 玉米Glyco-hydro-16家族保守的MotifFig. 1 The conserved motif of Glyco-hydro-16 family in maize
2.3 玉米Glyco-hydro-16家族基因的表达模式分析
根据玉米不同组织及不同发育时期的表达数据,分析玉米Glyco-hydro-16家族基因的表达(图3)。该家族成员在玉米中均有表达,但各成员的表达模式不同。大部分成员在不同组织中的表达水平有差异,如GRMZM2G063566在播种6 d幼根中表达量最高,在其他检测组织中表达量偏低;AC210669.3只在 V5时期的叶尖中表达量较低,在其他组织中表达量都比较高;另外,GRMZM2G364748在不同组织中表达量均较高,只在萌发 24 h种子和授粉16 d后的幼胚中表达量略低;GRMZM2G319798在大部分组织中的表达均较低或不表达,只在少数组织中表达且表达水平不高。GRMZM2G845080在所有组织中的表达水平比较一致,可能是组成型表达(图3)。
2.4 玉米Glyco-hydro-16家族基因的染色体定位
根据玉米Glyco-hydro-16家族成员的基因组位置信息绘制该家族的染色体分布图(图4),除第3和第6染色体外,玉米的其他8条染色体上都有该家族基因的分布。第 10染色体上分布最为密集,其中 5个基因成簇分布。另外,在第2及第5染色体上也存在成簇分布的基因(图4)。第8染色体上只存在一个该家族基因。
2.5 玉米Glyco-hydro-16家族基因在SS及NSS杂种优势群中的遗传分化分析
共获得289个玉米自交系的数据,其中109个被划分为SS群,180个被划为NSS群,包含294 378个位点。使用vcftools计算所有位点在2个杂种优势群间的群间遗传分化系数,平均值为0.13。根据玉米Glyco-hydro-16家族基因在染色体上的位置,筛选位于 Glyco-hydro-16家族基因内部及上下游1 kb以内区段的SNP位点,分析该位点在2个杂种优势群间的遗传变异。GRMZM2G070271和GRMZM2G076157的该区段内未找到合适位点。共有 10个基因所在位点的Fst值高于 0.15,且高于各染色体位点平均Fst值,达到高度分化水平,分别位于第1、第2、第4、第5、第7以及第9染色体上(图5)。其中一个位于第2染色体上位点的Fst值为0.52,对应基因为GRMZM2G091118,表明该位点在 SS群和 NSS群的群间遗传分化度极大。
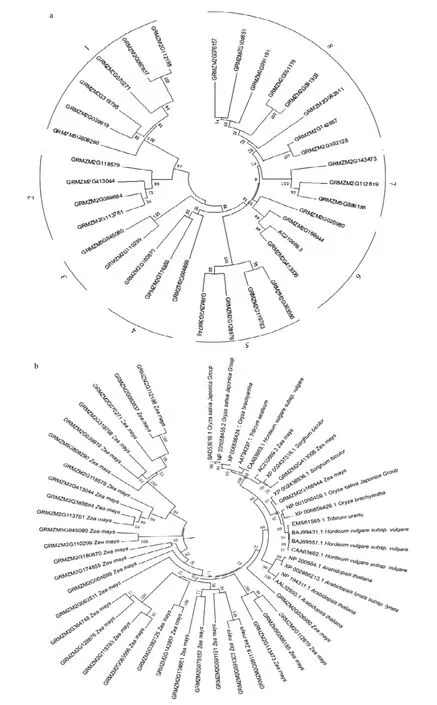
Arabidopsis thaliana 拟南芥;Arabidopsis lyrata subsp. lyrata 琴叶拟南芥;Hordeum vulgare subsp. vulgare 大麦;Oryza sativa Japonica Group 水稻粳稻;Oryza brachyantha 短花药野生稻;Sorghum bicolor 高粱;Triticum aestivum 小麦;Triticum urartu 乌拉尔图小麦;Zea mays 玉米图2 玉米Glyco-hydro-16家族蛋白系统进化关系及分类Fig.2 Phylogenetic relationships and subgroup of Glyco-hydro-16 proteins in maize
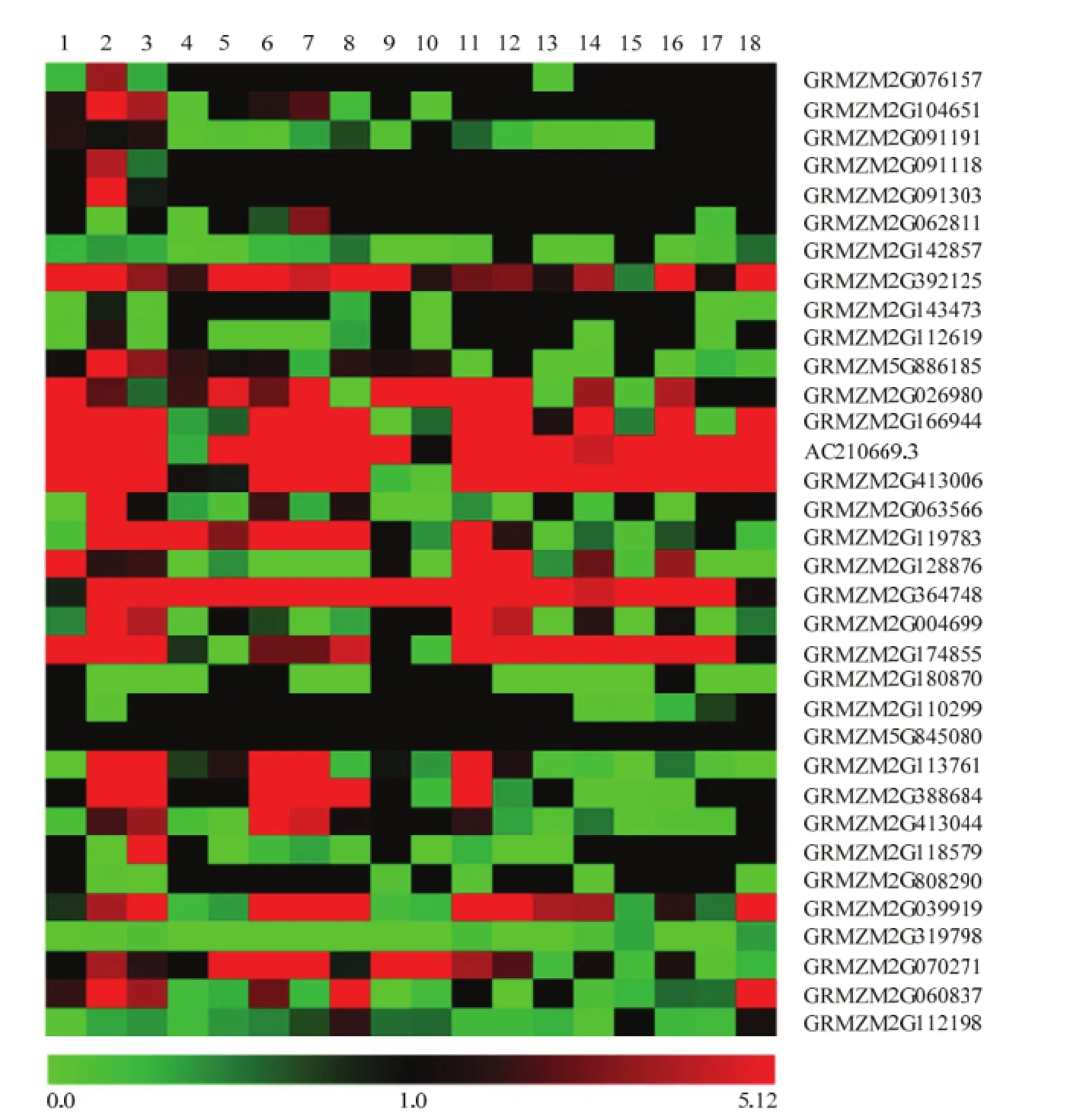
1:萌发24 h种子24 h germinating seed;2:播种6 d后幼根6 days after sowing (DAS) primary root;3:V3时期茎和顶端分生组织V3 stem and shoot apical meristem (SAM);4:V5时期叶尖V5 tip leaf;5:V9时期第8片叶V9 eighth leaf;6:V9时期第11片叶V9 eleventh leaf;7:V9时期第13片叶V9 thirteenth leaf;8:V9时期幼叶V9 immature leaves;9:VT时期第13片叶VT thirteenth leaf;10:R2时期第13片叶R2 thirteenth leaf;11:授粉10 d后种子10 days after pollination (DAP) whole seed;12:授粉12 d后种子12 DAP whole seed;13:授粉14 d后种子14 DAP whole seed;14:授粉16 d后种子16 DAP whole seed;15:授粉12 d后胚乳12 DAP endosperm;16:授粉14 d后胚乳14 DAP endosperm;17:授粉16 d后胚乳16 DAP endosperm;18:授粉16 d后幼胚16 DAP embryo图3 玉米Glyco-hydro-16家族基因的表达模式Fig. 3 Expression patterns of Glyco-hydro-16 family genes in maize
3 讨论
糖苷酶即糖苷水解酶,是使糖苷键水解的酶的总称。根据不同的分类标准可以分成不同的类型。按照序列相似性可分为135个家族,每个家族的内部成员间具有很高的结构相似度(http://www.cazy.org/ Glycoside-Hydrolases.html)。Glyco-hydro-16家族是其中一个家族,含有多个家族成员,包括半乳糖苷酶、葡聚糖酶、琼胶酶以及木葡聚糖酶等,主要涉及β-1,3 和 β-1,4糖苷键的水解。也有部分成员丧失了催化功能,而参与到免疫系统的响应过程中。

图4 玉米Glyco-hydro-16家族基因在染色体上的分布Fig. 4 Chromosomal locations of maize Glyco-hydro-16 family genes
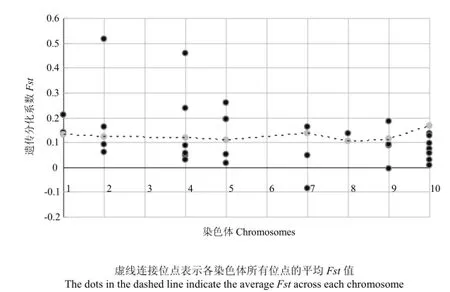
图5 玉米Glyco-hydro-16家族基因在玉米杂种优势群SS及NSS中的遗传分化系数Fig. 5 The F-statistic (Fst) of Glyco-hydro-16 family genes in heterotic group SS and NSS
本文通过该家族保守的序列信息及结构域,通过全基因组扫描,共获得34个成员。注释表明所有基因都是木葡聚糖转移酶/水解酶基因。该类基因编码蛋白具有转糖基酶或水解酶作用,通过水解木葡聚糖影响植物细胞壁的松驰[4]。在石竹开花过程中与花瓣的生长发育相关[23]。在拟南芥中该类基因通过雄蕊花丝形成时细胞壁的形成变化,调控自花授粉[5]。陈姣荣等[9]分析了芸芥木葡聚糖内糖基转移酶/水解酶基因EsXTH1,认为其与芸芥自交亲和性的调控相关。木葡聚糖内糖基转移/水解酶不仅作用于木葡聚糖,而且对葡聚糖和阿拉伯木聚糖也起作用[6]。在水稻中其同源基因在生长发育和抗盐胁迫中发挥功能[7]。糖苷酶的大部分功能与细胞壁的结构及组成相关,进而对生长发育、籽粒萌发、果实成熟脱落及细胞黏着产生调控作用[1]。根据玉米不同组织及不同发育时期的表达数据,表明该家族成员在玉米中都有表达,但表达模式各不相同,表明该类基因与多种生物学功能相关。虽然玉米中的该家族基因都是木葡聚糖相关基因,但通过构建系统发育进化树,可划分成8个亚家族(图2-a)。表明该类基因在进化过程中受到的选择压较小,分化比较大。而且玉米中该家族成员与水稻、小麦、大麦以及高粱中的同源基因分化较大(图2-b),只有亚家族6中的3个成员(AC210669.3、GRMZM2G413006以及GRMZM2G166944)划分到了禾本科分支中,三者蛋白质序列的相似度较高,分别达76%、67%和69%,并且这三个基因的表达模式也比较一致,在大部分组织中的表达量都比较高,在V5时期叶尖、V9时期第8片叶、VT时期第13片叶以及R2时期第13片叶中的表达量较低(图3),可能具有类似的功能。亚家族1包含6个基因,多数基因在不同组织中的表达量都比较低,只有GRMZM2G039919和GRMZM2G060837在某些组织中表达量较高(图3)。亚家族2中基因的表达模式比较相似,在 V3时期茎和顶端分生组织中表达量都比较高。亚家族3中2个基因在不同组织中的表达量相对比较一致,其中GRMZM2G845080在所有组织中的表达量都很接近。亚家族6和7中部分成员的表达模式也比较类似(图 3)。上述结果表明蛋白序列相似度高的玉米Glyco-hydro-16家族成员的基因表达模式也具有较高的相似性,可能参与了相同的生物学进程。
为解析该家族基因在不同玉米种质资源中的变异,利用玉米杂种优势群SS和NSS分析该家族基因的遗传分化。SS群和NSS群是20世纪50年代美国形成的2个杂种优势群[18-19],群间的配组能够产生较高的杂种优势。结果显示部分该家族成员在上述2个群体间达到了高度分化水平。其中,位于第2染色体上的一个位点的Fst值为0.52,远高于第2染色体上所有位点的平均Fst值0.12,表明该位点在SS群和NSS群间的群间遗传分化度极大。Glyco-hydro-16家族中的GRMZM2G091118位于该位点,其在大部分组织中的表达比较一致,只在播种6 d幼根中表达较高(图3)。GRMZM2G112198位于第4染色体,其所在位点的Fst为0.46,也达到了分化较大的水平,该基因在大部分组织中的表达水平都比较低,只在 V9时期幼叶、授粉12 d后胚乳以及授粉16 d后幼胚中的表达量稍高(图 3)。另外,GRMZM2G113761、GRMZM2G180870、GRMZM2G112619、GRMZM2G026980、GRMZM2G413006、GRMZM5G886185、GRMZM2G174855 和GRMZM2G039919等基因所在位点在SS及NSS群间的分化水平也较高(Fst>1.5),分别位于第1、第2、第4、第5、第7及第9染色体上。XIE等[24]鉴定了在2个水稻杂种优势群间存在差异的200个区域,发现这些差异位点的积累与产量成正相关。HUANG等[25]在水稻中鉴定出一些在杂交种中产生超显性效应的位点,认为某些位点的聚合有助于产生杂种优势。上述结果表明某些基因对产生杂交优势具有比较重要的影响,并且这些基因积累的数量与产生的优势相关。目前,已发现一些与玉米杂种优势相关的基因,如ZAR1,通过转基因试验表明,其编码的蛋白质能够提高杂交种的产量[26]。本研究鉴定出的部分Glyco-hydro-16家族基因在玉米杂种优势群SS和NSS间的分化较大,或许能为解析杂种优势的形成机制提供线索。
另外有 2个基因(GRMZM2G413044和GRMZM2G143473)所在位点的Fst值为负值,表明这两个基因在群体内部变异较大,而在群体间的分化不显著。其中,GRMZM2G413044所在位点的Fst值为-0.08,通过系统发育分析,该基因编码的蛋白被划入亚家族 2(图 2-a)。与其划入同一亚家族的GRMZM2G113761位点的Fst值为0.21,在SS和NSS群间达到较高的分化水平。虽然二者在群体间的遗传分化不同,但它们的表达模式比较相似(图 3)。可能在进化过程中虽然序列发生了变化,但功能仍然相似。
4 结论
通过对玉米全基因组扫描共鉴定出 34个Glyco-hydro-16家族成员,属于木葡聚糖转移酶/水解酶基因,可分为8个亚家族。该家族基因的表达模式不同,表明其参与不同的生物学功能。该家族成员在玉米杂种优势群SS和NSS的群间分化极大。
References
[1] MINIC Z. Physiological roles of plant glycoside hydrolases. Planta,2008, 227(4): 723-740.
[2] HENRISSAT B, CALLEBAUT I, FABREGA S, LEHN P, MORNON J P, DAVIES G. Conserved catalytic machinery and the prediction of a common fold for several families of glycosyl hydrolases. Proceedingsof the National Academy of Sciences of the USA, 1995, 92(15):7090-7094.
[3] DAVIES G, HENRISSAT B. Structures and mechanisms of glycosyl hydrolases. Structure, 1995, 3(9): 853-859.
[4] FRY S, SMITH R C, RENWICK K F, MARTIN D J, HODGE S,MATTHEWS K J. Xyloglucan endotransglycosylase, a new wallloosening enzyme activity from plants. Biochemical Journal, 1992,282(3): 821-828.
[5] KURASAWA K, MATSUI A, YOKOYAMA R, KURIYAMA T,YOSHIZUMI T, MATSUI M, SUWABE K, WATANABE M,NISHITANI K. The AtXTH28 gene, a xyloglucan endotransglucosylase/ hydrolase, is involved in automatic self-pollination in Arabidopsis thaliana. Plant and Cell Physiology, 2009, 50(2): 413-422.
[6] STROHMEIER M, HRMOVA M, FISCHER M, HARVEY A J,FINCHER G B, PLEISS J. Molecular modeling of family GH16 glycoside hydrolases: Potential roles for xyloglucan transglucosylases/ hydrolases in cell wall modification in the poaceae. Protein Science,2004, 13(12): 3200-3213.
[7] 张黎, 牛向丽, 张惠莹, 刘永胜. 水稻木葡聚糖内糖基转移酶基因OsXTH11过表达的作用分析. 中国农业科学, 2012, 45(16):3231-3239. ZHANG L, NIU X L, ZHANG H Y, LIU Y S. Functional analysis via overexpressing Xyloglucan endotransglycosylase gene OsXTH11 in rice. Scientia Agricultura Sinica, 2012, 45(16): 3231-3239. (in Chinese)
[8] LINTON S M, CAMERON M S, GRAY M, DONALD J A,SABOROWSKI R, VON BERGEN M, TOMM J M, ALLARDYCE B J. A glycosyl hydrolase family 16 gene is responsible for the endogenous production of β-1, 3-glucanases within decapod crustaceans. Gene, 2015, 569(2): 203-217.
[9] 陈姣荣, 方彦, 孙万仓, 令利军, 姜海杨. 芸芥木葡聚糖内糖基转移酶/水解酶基因 EsXTH1 的cDNA克隆和生物信息学分析. 中国油料作物学报, 2013, 35(2): 131-136. CHEN J R, FANG Y, SUN W C, LING L J, JIANG H Y. Cloning and bioinformatics of xyloglucan endotrans glycosylase and hydrolase EsXTH1 gene in Eruca sativa. Chinese Journal of Oil Crop Sciences,2013, 35(2): 131-136. (in Chinese)
[10] LIN H J, GAO J, ZHANG Z M, SHEN Y O, LAN H, LIU L, XIANG K, ZHAO M, ZHOU S, ZHANG Y Z. Transcriptional responses of maize seedling root to phosphorus starvation. Molecular Biology Reports, 2013, 40(9): 5359-5379.
[11] CONESA A, GÖTZ S, GARCÍA-GÓMEZ J M, TEROL J, TALÓN M,ROBLES M. Blast2GO: A universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics, 2005, 21(18): 3674-3676.
[12] BAILEY T L, BODEN M, BUSKE F A, FRITH M, GRANT C E,CLEMENTI L, REN J, LI W W, NOBLE W S. MEME SUITE: Tools for motif discovery and searching. Nucleic Acids Research, 2009,37(Suppl. 2): w202-w208.
[13] THOMPSON J D, GIBSON T J, PLEWNIAK F, JEANMOUGIN F,HIGGINS D G. The CLUSTAL_X windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, 1997, 25(24): 4876-4882.
[14] SAITOU N, NEI M. The neighbor-joining method: A new method for reconstructing phylogenetic trees. Molecular Biology and Evolution,1987, 4(4): 406-425.
[15] COX M P, PETERSON D A, BIGGS P J. SolexaQA: At-a-glance quality assessment of Illumina second-generation sequencing data. BMC Bioinformatics, 2010, 11(1): 485.
[16] LANGMEAD B, TRAPNELL C, POP M, SALZBERG S L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology, 2009, 10(3): R25.
[17] TRAPNELL C, WILLIAMS B A, PERTEA G, MORTAZAVI A,KWAN G, VAN BAREN M J, SALZBERG S L, WOLD B J,PACHTER L. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation. Nature Biotechnology, 2010, 28(5): 511-515.
[18] TRACY W, CHANDLER M. The historical and biological basis of the concept of heterotic patterns in corn belt dent maize//Plant Breeding:The Arnel R Hallauer International Symposium. 2006: 219-233.
[19] MIKEL M A, DUDLEY J W. Evolution of North American dent corn from public to proprietary germplasm. Crop Science, 2006, 46(3):1193-1205.
[20] ROMAY M C, MILLARD M J, GLAUBITZ J C, PEIFFER J A,SWARTS K L, CASSTEVENS T M, ELSHIRE R J, ACHARYA C B,MITCHELL S E, FLINT-GARCIA S A. Comprehensive genotyping of the USA national maize inbred seed bank. Genome Biology, 2013,14(6): R55.
[21] VAN HEERWAARDEN J, HUFFORD M B, ROSS-IBARRA J. Historical genomics of North American maize. Proceedings of the National Academy of Sciences of the USA, 2012, 109(31):12420-12425.
[22] WRIGHT S. Evolution and the Genetics of Populations: Experimental Results and Evolutionary Deductions: Vol. 3. Chicago, IL: University of Chicago Press, 1977.
[23] HARADA T, TORII Y, MORITA S, ONODERA R, HARA Y,YOKOYAMA R, NISHITANI K, SATOH S. Cloning, characterization,and expression of xyloglucan endotransglucosylase/hydrolase and expansin genes associated with petal growth and development during carnation flower opening. Journal of Experimental Botany, 2011,62(2): 815-823.
[24] XIE W, WANG G, YUAN M, YAO W, LYU K, ZHAO H, YANG M,LI P, ZHANG X, YUAN J. Breeding signatures of rice improvement revealed by a genomic variation map from a large germplasm collection. Proceedings of the National Academy of Sciences of the USA, 2015, 112(39): 5411-5419.
[25] HUANG X, YANG S, GONG J, ZHAO Y, FENG Q, GONG H, LI W, ZHAN Q, CHENG B, XIA J. Genomic analysis of hybrid rice varieties reveals numerous superior alleles that contribute to heterosis. Nature Communications, 2015, 6: 6258.
[26] GUO M, RUPE M A, WEI J, WINKLER C, GONCALVESBUTRUILLE M, WEERS B P, CERWICK S F, DIETER J A,DUNCAN K E, HOWARD R J. Maize ARGOS1 (ZAR1) transgenic alleles increase hybrid maize yield. Journal of Experimental Botany,2014, 65(1): 249-260.
(责任编辑 李莉)
Genome-Wide Identification of Glyco-hydro-16 Family in Maize and Differentiation Analysis
LIN Feng1,2, GE Min1, ZHOU Ling1, ZHAO Han1
(1Institute of Agro-Biotechnology, Jiangsu Academy of Agricultural Sciences/Provincial Key Laboratory of Agrobiology,Nanjing 210014;2Shanghai Key Laboratory of Bio-Energy Crops, Shanghai 200444)
Abstract:【Objective】Genome-wide identification was carried out for Glycoside hydrolase family 16 in maize, and theirexpression profile across tissues and differentiation between heterotic groups were analyzed. 【Method】Based on the maize V3 sequences, genome-wide survey of Glycoside hydrolase family 16 was conducted according to conserved sequences and domains through hmmersearch program. Blast2GO was used for gene annotation and phylogenetic relationships were analyzed through protein sequences. Expression profiles were examined within the whole transcriptome context at different tissues across development stages in B73. In line with the chromosome locations of the family genes, the authors screened the SNP markers and analyzed their genetic differentiation in different heterotic groups. 【Result】Totally 34 genes were identified in maize throughout genome-wide survey and annotated as xyloglucan endotransglucosylase with 3 conserved motifs discovered in all members. According to the phylogenetic relationships and sequence similarity they were divided into 8 subgroups. Glycoside hydrolase family 16 members were conserved in Gramineae, however, most maize members were not closely related with other Gramineae plants except only 3 members (AC210669.3, GRMZM2G413006, and GRMZM2G166944). The family genes in maize distributed on almost all chromosomes except on chromosomes 3 and 6 with several genes clustered on chromosomes 2, 5 and 10. Different expression profiles across tissues indicated their diversity functions. According to their chromosome location,the authors screened the SNP markers and calculated the genetic differentiation coefficient between the heterotic groups SS and NSS. Ten genes in Glycoside hydrolase family 16 differentiated significantly between the two heterotic groups, distributing on chromosomes 1, 2, 4, 5, 7 and 9. The one on chromosome 2 corresponding to GRMZM2G091118 got the highest Fst 0.52,indicating possible role contribute to heterosis. 【Conclusion】Based on the V3 sequences of maize, 34 genes were identified for Glycoside hydrolase family 16 and annotated as xyloglucan endotransglycosylase. Their expression profiles were different across tissues implying diversity functions. Some family members differentiated significantly between heterotic groups SS and NSS.
Key words:maize; Glyco-hydro-16; gene family; heterotic groups; RNA-seq
收稿日期:2016-02-22;接受日期:2016-04-20
基金项目:江苏省自然科学基金(BK20141385)、江苏省农业科技自主创新资金(CX(14)5054)、上海市能源作物育种及应用重点实验室开放课题
