植物生产层红素氧还蛋白研究进展
2016-07-16柳参奎
李 莹,柳参奎
(东北林业大学 盐碱地生物资源环境研究中心,黑龙江 哈尔滨 150040)
植物生产层红素氧还蛋白研究进展
李 莹,柳参奎
(东北林业大学 盐碱地生物资源环境研究中心,黑龙江 哈尔滨 150040)
摘要:自然界中大多数的红素氧还蛋白(rubredoxin)集中来自严格厌氧菌,包括细菌、古菌和少数微需氧细菌,而植物中该类蛋白相对较少,它的功能主要表现在作为辅因子或电子供体参与多个生化途径。也有研究显示,无论是植物中还是微生物中的红素氧还蛋白基因在协助生物抵御氧化胁迫方面均发挥着重要的作用。本文全面介绍自然界中的红素氧还蛋白基因的分布、结构以及在微生物、植物中所发挥的作用等,希望能为今后的进一步研究提供参考。
关键词:红素氧还蛋白;Fe-S蛋白;电子传递;氧化还原替代过程(SOR)
红素氧还蛋白是一类非亚铁血红素蛋白,包含一个[Fe(SCys)4]中心。它是Fe-S蛋白家族中的最简单的成员之一,它具有多变的结构。红素氧还蛋白最早于1965年由Lovenberg和Sobel[1]在研究分离梭状芽胞杆菌(Clostridiumpasteurianum)中铁氧还蛋白(ferredoxins)中分离得到了一种红色的蛋白,并命名为rubredoxins。它是一种最简单的iron-sulfur proteins(Fe-S),包含着较小的体积(由54个氨基酸残基组成)和较简单的结构。目前,关于红素氧还蛋白的报道多集中于严格厌氧细菌和少量的微需氧细菌,而关于植物红素氧还蛋白的报道更少。在美国国立生物技术信息中心(NCBI)中可以检索到的关于植物类红素氧还蛋白的文章不超过5篇。这些文章主要通过两方面来解释植物类红素氧还蛋白的功能,其中一类主要介绍植物类红素氧还蛋白在光合作用中发挥着重要的作用,另一类主要介绍红素氧还蛋白在非生物胁迫过程中发挥重要的作用。由此认为,植物类红素氧还蛋白是一类重要的胁迫相应蛋白。因此,红素氧还蛋白的发现及其结构和功能的研究对探讨植物生长发育、对环境胁迫的反应机理等方面具有重要的意义,有可能为提高植物抗逆性提供新的研究途径。本文对红素氧还蛋白基因的结构特征,及其在微生物和植物体内所发挥的功能进行详细的阐述,以期为植物抗逆性的研究提供参考。
1红素氧还蛋白在Fe-S蛋白家族中的地位
Fe-S簇在生物体内是普遍存在和必需的组成元件,包含Fe-S活性位点的铁硫蛋白首先在通过电子核磁共振指纹技术在线粒体膜中发现[2]。在根据国际生物化学联合会命名委员会(Nomenclature Committee of the International Union of Biochemistry,NC-IUB)的分类命名原则,生物体中含铁的蛋白质可以分为3类:血红素蛋白(hemeproteins),铁硫蛋白(iron-sulfur protein,Fe-S proteins)和其它包括了铁蛋白(ferritin,Fer)、转铁蛋白(transferrin)和具有电子传递功能的加氧酶(oxygenases)含铁蛋白[3]。其中的铁硫蛋白是指那些通过无机硫或半胱氨酸硫与非血红素铁交联的蛋白质,分为两类:简单铁硫蛋白和复杂铁硫蛋白。简单铁硫蛋白仅含有一个或多个Fe-S簇,而复杂铁硫蛋白还具有像核黄素和血红素一样的另外的可以激活的氧化还原中心。这些蛋白中的Fe-S簇常使用电子顺磁共振(EPR)光谱来鉴定和分类。简单铁硫蛋白又分为3类:红素氧还蛋白(rubredoxins,Rd),铁氧还蛋白(ferredoxins,Fd)和其它一些铁硫蛋白(图1)。因此,红素氧还蛋白是Fe-S蛋白家族中的最简单的成员之一,同时它具有简单多变的结构特征。
2红素氧还蛋白在自然界中的分布
研究发现,自然界中大多数的红素氧还蛋白集中来自严格厌氧菌,包括细菌和古菌(Archaea),如食油假单胞菌(Pseudomonasoleovorans)[4]、激烈火球菌(Pyrococcusfuriosus)[5]、甲烷八叠球菌(Methanosarcinamazei)[6]、闪烁古生球菌(Archaeoglobusfulgidus)[7]等均有大量的该类蛋白发现。还有一些被认为是微需氧菌(Microaerophiles),如梭状芽胞杆菌(Clostridiumpasteurianum)[1]也发现了红素氧还蛋白的存在。目前,植物类的红素氧还蛋白也陆续被发现,迄今为止已从拟南芥 (Arabidopsisthaliana)[8]、可可(Theobromacacao)[9]、碱茅(Puccinelliatenuiflora)[10]、乌拉尔图小麦(Triticumurartu)[11]、蒺藜苜蓿(Medicagotruncatula)[12]、甜瓜(Cucumismelosubsp. melo)[13]、水稻(Oryzasativa)[14]和一些藻类[15]等多个物种中分离得到该类蛋白。在Genbank中以rubredoxin为检索词可以检索到40多个植物的红素氧还蛋白类似蛋白UniGene。
3微生物体内的红素氧还蛋白的结构
红素氧还蛋白在生物界中的分布与其它蛋白比较是相当缺乏的,但红素氧还蛋白类型和结构域确实很多样化。C.pasteurianum中的红素氧还蛋白的结构早在20世纪60年代就由Lyle Jensen及其领导的研究小组利用X射线晶体学的方法得到了很好的阐释[16],并在20世纪70年代早期成为蛋白晶体学修正的测试样品,成为第1个并成功修正的蛋白质,这也引发了随后与蛋白质晶体学研究相关的计算活动的增加,并直接改善和大分子晶体学模型的精细程度。蛋白结构分析表明,C.pasteurianum的红素氧还蛋白是一类大小在45~55个氨基酸的含有氧化还原激活位点的蛋白质。具有两个保守的半胱氨酸结构域:CxxCxnCPxC(x代表任何氨基酸,n=22~29),通过4个保守的半胱氨酸来结合定位一个铁原子,铁原子通过在+2价和+3价直接的变换来作为氧化还原中心而存在。红素氧还蛋白的FeS4位点在缺乏无机硫的Fe-S激活位点中是有其独特性的,FeS4的结构单位也是所有Fe-S簇的基本构件。红素氧还蛋白[1Fe]3+2+价态变化中的氧化还原势在-100到+200 mV之间的范围。红素氧还蛋白折叠和Rieske[2Fe-2S]蛋白折叠尽管有不同的链接方式,但在活性位点区域很类似[17]。事实上一个单一连接铁的半胱氨酸的突变都可以导致红素氧还蛋中出现一个[2Fe-2S]簇,相反,Rieske位点可以通过一个三重突变使得Rieske位点转换为一个[1Fe]红素氧还蛋白类似位点[18-19]。红素氧还蛋白在其它方面也显示出来和归属于锌指蛋白家族的含锌蛋白显著的相似。它自身对铁和锌有相似的亲和性,这些金属在红素氧还蛋白中的含量已经在生物体内,至少在大肠杆菌中通过培养基中两种金属相对含量的测定得到证实[20]。重金属如镉和汞也可以和红素氧还蛋白活性位点结合[21]。包含锌和镉的红素氧还蛋白类结构域也分别在核糖核苷酸还原酶Ⅲ和蛋白激酶G中有发现[22-23]。图2为C.pasteurianum菌红素氧还蛋白的结构示意图[16]。
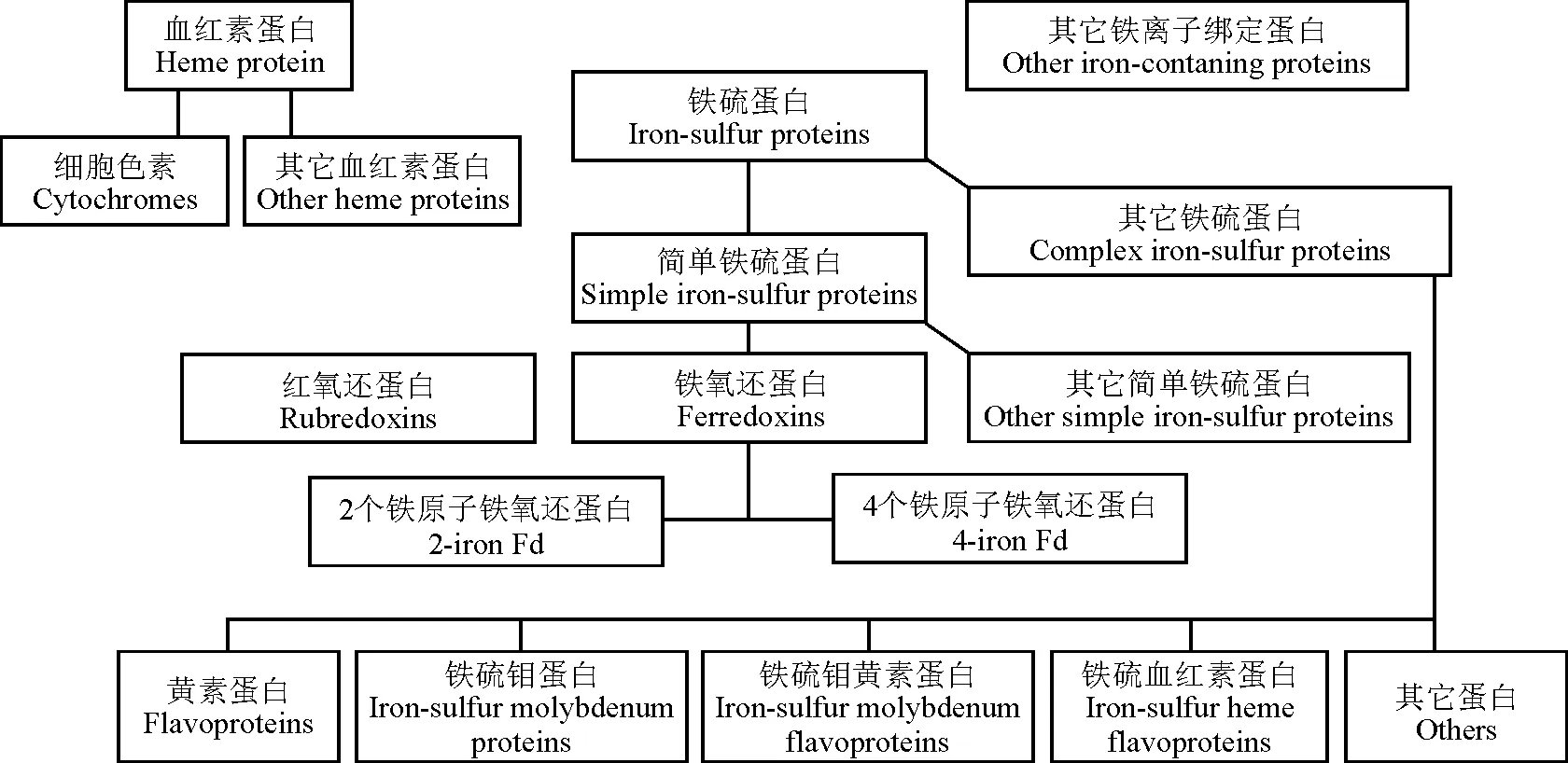
图1 含铁蛋白质的分类
注:引自Recommendations[3](1979)并加以修改。
Note: Come from Recommendations(1979)[3]and modified.
目前,在兼性厌氧菌和需氧菌中发现了一些大一点的包含红素氧还蛋白类似序列或结构域的蛋白,这些在蛋白大小、半胱氨酸结构域的位置和距离上都差别很大。这些红素氧还蛋白相关蛋白可以分为如下几类,一类是如红素氧还蛋白一样包含相同数目的半胱氨酸结构域(CxxCnCPxC,n=22~29),但由于在N和C端有10~40个额外的氨基酸分布而显得大点。如需氧菌Rhizobiumleguminosarum中的HupⅠ蛋白(GenBank accession P28151);其它几类包含相同的结构域但氨基酸数量较大,如Pseudomonasputida,该菌的红素氧还类似蛋白的氨基酸数目在105到178 (GenBank accession CAB5405l)之间;另外Pyrococcushorikoshii(GenBank accession BAA30645)的红素氧还类似蛋白氨基酸数目在279到661之间。还有一些蛋白和红素氧还蛋白的结构及其相似,只是N末端结构域的两个半胱氨酸之间的距离小于或大于两个残基。如(MethanococcusjannaschiiRd Ⅱ,GenBank accession AAB98734)、(DesulfovibriovulgarisRd-like protein,GenBank accession AAB39992)[24]。这一类大一些的蛋白中,Pyrococcusfuriosus的红素氧还蛋白是被研究最多的,其结构通过核磁共振波谱法(NMR Spectroscopy)和X 射线结晶学(X-ray Crystallography)得到了测定[25]。实际上,它是第1个得到测定的超适温性蛋白结构,这个蛋白也作为一个金属离子在4个半胱氨酸协调下在一个蛋白环境中研究的模式系统。铁、镍、锌、镉和汞等金属离子都以包含的形式通过NMR、X射线吸收和磁圆二色光谱的测定。图3为P.furiosus的红素氧还蛋白核磁共振的测定结果[25-28]。

图2 Clostridium pasteurianum的红素氧还蛋白结构示意图[25]
Note: a, Rubredoxin inClostridiumbotulinum(WP_039239571.1),Francisellanoatunensissubsp.orientalisstr. Toba 04(GenBank: AFJ44051.1);BurkholderiarhizoxinicaHKI 454(CBW75889.1),DesulfatibacillumalkenivoransAK-01(ACL06764.1),AlcanivoraxborkumensisSK2(CAL15611.1); b-d, Come from solution structure of reducedClostridiumpasteurianumrubredoxin,1998[16].

图3 Pyrococcus furiosus的红素氧还蛋白核磁共振的测定结果
注:在网站http://swissmodel.expasy.org/interactive制作。
Note: Made in the http://swissmodel.expasy.org/interactive.
红素氧还蛋白还被广泛应用在研究极端温度条件下蛋白的稳定性和蛋白结构对铁位点的还原势的影响[29]。实际上,红素氧还蛋白也是已知的热稳定性最好的蛋白之一,其融化温度估计接近200 ℃[30]。来自嗜常温菌C.pasteurianum的红素氧还蛋白在同温度下几个小时内就已经完全变性[31]。目前,导致该超适温性蛋白如此强稳定性的机理还不是很清楚。P.furiosus红素氧还蛋白的展开如其变性一样是不可逆的。其展开路径是复杂的,疏水核心松弛,但没有金属辅助因子的损失,这是决定速率关键性的一步[32]。超适温和嗜常温红素氧还蛋白在N末端和三股β折叠里有很小的结构差别,导致了其特性。这些结构差别所导致的蛋白质稳定性的差别已经通过诱变和β折叠嵌合体的研究得到了阐释。这些研究表明,尽管N末端的静电相互作用和 β折叠中的更广泛的氢键网络都对热稳定性有贡献,但这些相互作用的总体组合才使得P.furiosus红素氧还蛋白变得如此稳定[31]。
4红素氧还蛋白在微生物体内发挥的作用
研究发现,在C.pasteurianum提取物调控的几个反应中红素氧还蛋白可以作为铁氧还蛋白在电子传递过程中发挥重要作用,但在光谱和化学特性方面与其有差别[33]。 红素氧还蛋白还可以作为电子载体在多个氧化还原反应过程中发挥作用。C.pasteurianum细菌作为深入研究微生物产氢的细菌种属之一,能够在厌氧条件下,利用糖酵解(glycolysis)所产生的NADH,经由铁硫蛋白的携带,将电子传递至产氢酶(hygrogenase)中,再经由产氢酶蛋白中4个铁硫簇(Fe-S cluster)的传递,将电子送至产氢酶活性中心的H-cluster和氢气离子作用形成氢气。对绿脓杆菌(Pseudomonasaeruginosa)的研究表明,红素氧还蛋白还原酶(rubredoxin reductase,RdxRs)和Rdx组成的氧化还原反应系统是以正构烷烃(n-alkanes)为单一碳源生长所不可缺少的,这一氧化还原系统在古菌或厌氧细菌中是应对氧化应激反应中关键的一部分,这也使得绿脓杆菌在一些石油污染方面尤其潜在利用价值[34]。棕色固氮菌(Azotobactervinelandii)的红素氧还蛋白基因在H2代谢方面发挥这重要的作用[35];绿硫菌(Chlorobiumtepidum)的红素氧还蛋白基因作为丙酮酸铁氧化还原蛋白氧化还原酶(PFOR)的电子受体,在电子传递中发挥重要作用[36];食油假单胞菌(Pseudomonasoleovorans)的红素氧还蛋白作为电子传递体在NADH和氧气存在的条件下参与了烷烃、脂肪酸羟基化过程[4];巨大脱硫弧菌(Desulfovibriogigas)红素氧还蛋白基因作为电子供体将电子提供给血红素蛋白和红素氧还蛋白氧化还原酶;一些厌氧细菌的红素氧还蛋白通过快速传递代谢的还原物到超氧化还原酶(superoxide reductases)或红素氧还蛋白︰氧的氧化还原酶(rubredoxin︰oxygen oxidoreductases),来减少氧或反应性的氧化物种类的化氧化应激反应中起关键作用[37],并通过NAD(P)H和NAD(P)H︰RdxRs与主代谢系统联系起来[38]。近年来,在厌氧微生物中又提出了一个新的超氧化物还原替代途径(SOR途径),在这个过程中,红素氧还蛋白作为superoxide reductase的电子供体,在清除微生物体内有毒活性氧方面发挥了重要的作用[2,39-43]。
5植物中红素氧还蛋白的功能
目前,关于植物红素氧还蛋白的报道相对较少,在NCBI中只能检索到6种植物类的红素氧还蛋白基因的编码的完整氨基己酸序列,图4为植物类红素氧还蛋白间同源性分析结果。同时,可以检索到的报道大多数集中在对拟南芥的红素氧还蛋白的研究。2003年对拟南芥的研究发现,其叶绿体ATP合酶的γ亚基编码基因atpC1的插入突变导致叶绿体中大多数核基因下调表达,而一些光合作用基因显著上调表达,其中包含拟南芥中的ENH1 (enhancer of sos3-1,AT5G17170)基因[44]。2004年,Friso等[45]在对拟南芥叶绿体的类囊体的膜蛋白组学研究时分离得到了两个包含红素氧还蛋白结构域的红素氧还类似蛋白(At5g17170,At1g54500),其中作者推测At1g54500与Synechococcussp PCC 7002的红素氧还蛋白基因具有相似的功能。Synechococcussp PCC 7002的红素氧还蛋白与中间多肽(4Fe-4S)簇Fx的组装的关系十分密切。其中Fx为聚球藻PCC 7002光系统Ⅰ的重要组成部分,rubredoxin的缺失直接导致光系统Ⅰ功能丧失[46-47]。绿藻和高等植物叶绿体的光和类囊体膜上都包含有4个多亚基蛋白复合物(photosystem Ⅰ [PSI]、photosystem Ⅱ[PSII]、ATP synthase和cytochrome b6f complexes), 同时拟南芥红素氧还蛋白在类囊体膜上的定位表明其参与了其中的反应,但其具体功能如何,目前还需要进一步的研究。
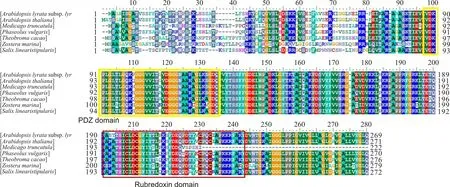
图4 7种植物红素氧还蛋白基因编码的氨基酸序列多序列比对
Note:Arabidopsislyratasubsp. lyrata(XP_002871751.1),Arabidopsisthaliana(NP_568342.1),Medicagotruncatula(XP_013453352.1),Phaseolusvulgaris(AGV54433.1),Zosteramarina(KMZ59555.1),Theobromacacao(XP_007029491.1),Salixlinearistipularis(笔者克隆获得Cloned by author)
Tran等[48]的研究表明,NAC转录因子家族中的ANAC055在转基因植物中的过量表达可以提高其耐旱性,生物芯片结果表明,逆境条件下At5g17170在转基因植物中上调表达。随后,Zhu等[8]对65 000株以sos3-1缺失突变体为遗传背景的双突变体进行筛选,试图发现可以使sos3-1缺失单突变体对NaCl的抗敏性发生改变的缺失双突变体。其中,sos3-1enh1-1双突变体加重了sos3-1突变体对NaCl的敏感性,同时也使sos3-1突变体在没有任何逆境处理的条件下生长受到抑制。研究发现,enh1-1基因为拟南芥红素氧还类似蛋白(At5g17170),它与Pyrococcusfuriosus中的红素氧还蛋白同源性最高,同时该基因还包含了一个与人类的PDZ结构域均有较高同源性PDZ结构域,该结构域的存在可能在参与蛋白质-蛋白质相互作用方面发挥重要的作用。亚细胞定位研究表明,该基因定位为拟南芥叶绿体中。在NaCl逆境存在的条件下,该基因通过调节植物体内离子平衡,来提高植物对NaCl的抗性,同时该研究提出该基因间接的与SOS途径中两个重要基因SOS2和SOS1共同作用来提高植物的抗逆性。除此之外,该基因也可通过清除植物体内活性氧来提高植物对NaCl逆境的抵御[8]。2014年Li等[10]利用酵母菌作为宿主菌株试图在碱茅(Puccinelliatenuiflora)全长cDNA文库中筛选具有抗盐性的转基因酵母时也分离得到了具有较强耐盐性的碱茅红素氧还蛋白基因。同时,该基因的过量表达显著提高了酵母菌株对NaCl、NaHCO3、Na2CO3、Sorbital和H2O2的抗性。碱茅主要分布在我国的东北、华北和西北地区,它作为一种盐碱地先锋植物,对极其恶劣的土壤也有较强的适应能力。根据以上结果推测,红素氧还蛋白基因在碱茅适应及其恶劣环境方面可能发挥着重要的作用[10,49-50]。
6小结与展望
近年来,红素氧还蛋白基因陆续被发现及研究。结果表明,微生物体内的红素氧还蛋白基因主要作为电子传递链中的重要组成部分参与各种生理生化过程。植物类红素氧还蛋白基因在光合作用和抵御非生物胁迫方面发挥着重要的作用。目前,人们初步认识了自然界中红素氧还蛋白基因在生物体生长、发育和抵御逆境胁迫过程中的作用和分子机制,但对植物类红素氧还蛋白基因的研究相对较少,除拟南芥外,其它植物的红素氧还蛋白基因是如何调控植物参与各种生理生化反应的的分子机制并不十分清楚,仍有大量问题等待着被解释。因此,可以从以下几个方面对植物类红素氧还蛋白基因进行深入的研究:1)不断从各种植物中分离挖掘具有重要功能的红素氧还蛋白基因,达到丰富该类基因资源的目的。尤其应把重点放在极端耐逆性的植物上,并深入了解这些基因的功能,对其进行详细的解析,并使这些基因的功能得到合理的应用。2)结合酵母双杂交技术,筛选与极端耐逆性植物的红素氧还蛋白的相互作用蛋白,完善植物生长发育和抵御逆境胁迫路径调控网络。通过对相互作用蛋白的研究,最终确定该类蛋白在调控通路上扮演的角色。
参考文献References:
[1]Lovenberg W,Sobel B E.Rubredoxin:A new electron transfer protein fromClostridiumpasteurianum.PNAS,1965,54(1):193-199.
[2]Beinert H,Sands R H.Studies on succinic and DPNH dehydrogenase preparations by paramagnetic resonance (epr) spectroscopy.Biochemical and Biophysical Research Communications,1960,3(1):41-46.
[3] Nomenclature of iron-sulfur proteins.Recommendations,1978.European Journal of Biochemistry,1979,93(3):427-430.
[4]Peterson J A,Coon M J.Enzymatic omega-oxidation.3.Purification and properties of rubredoxin,a component of the omega-hydroxylation system ofPseudomonasoleovorans.Journal of Biological Chemistry,1968,243(2):329-334.
[5]Bridger S L,Lancaster W A,Poole F L,Schut G J,Adams M W.Genome sequencing of a genetically tractablePyrococcusfuriosusstrain reveals a highly dynamic genome.Journal of Bacteriology,2012,194(15):4097-4106.
[6]Assis Das Gracas D,Thiago Juca Ramos R,Vieira Araujo A C,Zahlouth R,Ribeiro Carneiro A,Souza Lopes T,Azevedo Barauna R,Azevedo V,Cruz Schneider M P,Pellizari V H,Silva A.Complete genome of a methanosarcina mazei strain isolated from sediment samples from an Amazonian flooded area.Genome Announcements,2013,1(3):1140-1152.
[7]Klenk H P,Clayton R A,Tomb J F,White O,Nelson K E,Ketchum K A,Dodson R J,Gwinn M,Hickey E K,Peterson J D,Richardson D L,Kerlavage A R,Graham D E,Kyrpides N C,Fleischmann R D,Quackenbush J,Lee N H,Sutton G G,Gill S,Kirkness E F,Dougherty B A,McKenney K,Adams M D,Loftus B,Peterson S,Reich C I,McNeil L K,Badger J H,Glodek A,Zhou L,Overbeek R,Gocayne J D,Weidman J F,McDonald L,Utterback T,Cotton M D,Spriggs T,Artiach P,Kaine B P,Sykes S M,Sadow P W,D’Andrea K P,Bowman C,Fujii C,Garland S A,Mason T M,Olsen G J,Fraser C M,Smith H O,Woese C R,Venter J C.The complete genome sequence of the hyperthermophilic,sulphate-reducing archaeonArchaeoglobusfulgidus.Nature,1997,390:364-370.
[8]Zhu J,Fu X,Koo Y D,Zhu J K,Jenney F E Jr,Adams M W,Zhu Y,Shi H,Yun D J,Hasegawa P M,Bressan R A.An enhancer mutant ofArabidopsissalt overly sensitive 3 mediates both ion homeostasis and the oxidative stress response.Molecular and Cellular Biology,2007,27(14):5214-5224.
[9]Motamayor J C,Mockaitis K,Schmutz J,Haiminen N,Livingstone D,Cornejo O,Findley S D,Zheng P,Utro F,Royaert S,Saski C,Jenkins J,Podicheti R,Zhao M,Scheffler B E,Stack J C,Feltus F A,Mustiga G M,Amores F,Phillips W,Marelli J P,May G D,Shapiro H,Ma J,Bustamante C D,Schnell R J,Main D,Gilbert D,Parida L,Kuhn D N.The genome sequence of the most widely cultivated cacao type and its use to identify candidate genes regulating pod color.Genome Biology,2013,14(6):53.
[10]Li Y,Takano T,Liu S.Discovery and characterization of two novel salt-tolerance genes inPuccinelliatenuiflora.International Journal of Molecular Sciences,2014,15(9):16469-16483.
[11]Ling H Q,Zhao S,Liu D,Wang J,Sun H,Zhang C,Fan H,Li D,Dong L,Tao Y,Gao C,Wu H,Li Y,Cui Y,Guo X,Zheng S,Wang B,Yu K,Liang Q,Yang W,Lou X,Chen J,Feng M,Jian J,Zhang X,Luo G,Jiang Y,Liu J,Wang Z,Sha Y,Zhang B,Wu H,Tang D,Shen Q,Xue P,Zou S,Wang X,Liu X,Wang F,Yang Y,An X,,Dong Z,Zhang K,Zhang X,Luo M C,Dvorak J,Tong Y,Wang J,Yang H,Li Z,Wang D,Zhang A,Wang J.Draft genome of the wheat a-genome progenitorTriticumurartu.Nature,2013,496:87-90
[12]Terpolilli J,Hill Y,Tian R,Howieson J,Brau L,Goodwin L,Han J,Liolios K,Huntemann M,Pati A,Woyke T,Mavromatis K,Markowitz V,Ivanova N,Kyrpides N,Reeve W.Genome sequence ofEnsifermelilotistrain WSM1022;a highly effective microsymbiont of the model legumeMedicagotruncatulaA17.Standards in Genomic Sciences,2013,9(2):315-324.
[13]Gonzalez V M,Garcia-Mas J,Arus P,Puigdomenech P.Generation of a BAC-based physical map of the melon genome.BMC Genomics,2010,11:339.
[14]Merchant S S,Prochnik S E,Vallon O,Harris E H,Karpowicz S J,Witman G B,Terry A,Salamov A,Fritz-Laylin L K,Marechal-Drouard L,Marshall W F,Qu L H,Nelson D R,Sanderfoot A A,Spalding M H,Kapitonov V V,Ren Q,Ferris P,Lindquist E,Shapiro H,Lucas S M,Grimwood J,Schmutz J,Cardol P,Cerutti H,Chanfreau G,Chen C L,Cognat V,Croft M T,Dent R,Dutcher S,Fernandez E,Fukuzawa H,Gonzalez-Ballester D,Gonzalez-Halphen D,Hallmann A,Hanikenne M,Hippler M,Inwood W,Jabbari K,Kalanon M,Kuras R,Lefebvre P A,Lemaire S D,Lobanov A V,Lohr M,Manuell A,Meier I,Mets L,Mittag M,Mittelmeier T,Moroney J V,Moseley J,Napoli C,Nedelcu A M,Niyogi K,Novoselov S V,Paulsen I T,Pazour G,Purton S,Ral J P,Riano-Pachon D M,Riekhof W,Rymarquis L,Schroda M,Stern D,Umen J,Willows R,Wilson N,Zimmer S L,Allmer J,Balk J,Bisova K,Chen C J,Elias M,Gendler K,Hauser C,Lamb M R,Ledford H,Long J C,Minagawa J,Page M D,Pan J,Pootakham W,Roje S,Rose A,Stahlberg E,Terauchi A M,Yang P,Ball S,Bowler C,Dieckmann C L,Gladyshev V N,Green P,Jorgensen R,Mayfield S,Mueller-Roeber B,Rajamani S,Sayre R T,Brokstein P,Dubchak I,Goodstein D,Hornick L,Huang Y W,Jhaveri J,Luo Y,Martinez D,Ngau W C,Otillar B,Poliakov A,Porter A,Szajkowski L,Werner G,Zhou K,Grigoriev I V,Rokhsar D S,Grossman A R.The chlamydomonas genome reveals the evolution of key animal and plant functions.Science,2007,318:245-250.
[15]Gao C,Wang Y,Shen Y,Yan D,He X,Dai J,Wu Q.Oil accumulation mechanisms of the oleaginous microalgaChlorellaprotothecoidesrevealed through its genome,transcriptomes,and proteomes.BMC Genomics,2014,15:582.
[16]Bertini I,Kurtz Jr D M,Eidsness M K,Liu G H,Luchinat C,Rosato A,Scott R A.Solution structure of reducedClostridiumpasteurianumrubredoxin.Journal of Biological Inorganic Chemistry,1998,3(4):401-410.
[17]Iwata S,Saynovits M,Link T A,Michel H.Structure of a water soluble fragment of the ‘rieske’ iron-sulfur protein of the bovine heart mitochondrial cytochrome bc1 complex determined by mad phasing at 1.5å resolution.Structure,1996,4(5):567-579.
[18]Meyer J,Gagnon J,Gaillard J,Lutz M,Achim C,Munck E,Petillot Y,Colangelo C M,Scott R A.Assembly of a [2Fe-2S]2+cluster in a molecular variant ofClostridiumpasteurianumrubredoxin.Biochemistry,1997,36(43):13374-13380.
[19]Iwasaki T,Kounosu A,Tao Y,Li Z,Shokes J E,Cosper N J,Imai T,Urushiyama A,Scott R A.Rational design of a mononuclear metal site into the archaeal Rrieske-type protein scaffold.Journal of Biological Chemistry,2005,280(10):9129-9134.
[20]Dauter Z,Wilson K S,Sieker L C,Moulis J M,Meyer J.Zinc- and iron-rubredoxins fromClostridiumpasteurianumatatomic resolution:A high-precision model of a ZnS4 coordination unit in a protein.PNAS,1996,93(17):8836-8840.
[21]Maher M,Cross M,Wilce M C,Guss J M,Wedd A G.Metal-substituted derivatives of the rubredoxin fromClostridiumpasteurianum.Acta Crystallogr D Biol Crystallogr,2004,60(Pt 2):298-303.
[22]Logan D T,Mulliez E,Larsson K M,Bodevin S,Atta M,Garnaud P E,Sjoberg B M,Fontecave M.A metal-binding site in the catalytic subunit of anaerobic ribonucleotide reductase.PNAS,2003,100(7):3826-3831.
[23]Scherr N,Honnappa S,Kunz G,Mueller P,Jayachandran R,Winkler F,Pieters J,Steinmetz M O.Structural basis for the specific inhibition of protein kinase g,a virulence factor ofMycobacteriumtuberculosis.PNAS,2007,104(29):12151-12156.
[24]Jenney F E Jr,Adams M W.Rubredoxin fromPyrococcusfuriosus.Methods in Enzymology,2001,334:45-55.
[25]Day M W,Hsu B T,Joshua-Tor L,Park J B,Zhou Z H,Adams M W,Rees D C.X-ray crystal structures of the oxidized and reduced forms of the rubredoxin from the marine hyperthermophilic archaebacteriumPyrococcusfuriosus.Protein Science,1992,1(11):1494-1507.
[26]Blake P R,Day M W,Hsu B T,Joshua-Tor L,Park J B,Hare D R,Adams M W,Rees D C,Summers M F.Comparison of the X-ray structure of native rubredoxin fromPyrococcusfuriosuswith the NMR structure of the zinc-substituted protein.Protein Science,1992,1(11):1522-1525.
[27]Bertini I,Kurtz Jr D M,Eidsness M K,Liu G H,Luchinat C,Rosato A,Scott R.A Solution structure of reducedClostridiumpasteurianumrubredoxin.Journal of Biological Inorganic Chemistry,1998,3(4):401-410.
[28]Henehan C J,Pountney D L,Vašák M,Zerbe O.Identification of cysteine ligands in metalloproteins using optical and NMR spectroscopy:Cadmium-substituted rubredoxin as a model [cd(cyss)4]2- center.Protein Science,1993,2(10):1756-1764.
[29]Van Elp J,George S J,Chen J,Peng G,Chen C T,Tjeng L H,Meigs G,Lin H J,Zhou Z H,Adams M W.Soft X-ray magnetic circular dichroism:A probe for studying paramagnetic bioinorganic systems.PNAS,1993,90(20):9664-9667.
[30]Blake P R,Park J B,Bryant F O,Aono S,Magnuson J K,Eccleston E,Howard J B,Summers M F,Adams M W.Determinants of protein hyperthermostability:Purification and amino acid sequence of rubredoxin from the hyperthermophilic archaebacteriumPyrococcusfuriosusand secondary structure of the zinc adduct by NMR.Biochemistry,1991,30(45):10885-10895.
[31]Eidsness M K,Richie K A,Burden A E,Kurtz D M Jr,Scott R A.Dissecting contributions to the thermostability ofPyrococcusfuriosusrubredoxin:Beta-sheet chimeras.Biochemistry,1997,36(34):10406-10413.
[32] Cavagnero S,Zhou Z H,Adams M W,Chan S I.Unfolding mechanism of rubredoxin fromPyrococcusfuriosus.Biochemistry,1998,37(10):3377-3385.
[33]Park I Y,Eidsness M K,Lin I J,Gebel E B,Youn B,Harley J L,Machonkin T E,Frederick R O,Markley J L,Smith E T,Ichiye T,Kang C H.Crystallographic studies of v44 mutants ofClostridiumpasteurianumrubredoxin:Effects of side-chain size on reduction potential.Proteins:Structure,Function,and Bioinformatics,2004,57(3):618-625.
[34]Hagelueken G,Wiehlmann L,Adams T M,Kolmar H,Heinz D W,Tummler B,Schubert W D.Crystal structure of the electron transfer complex rubredoxin rubredoxin reductase ofPseudomonasaeruginosa.PNAS,2007,104(30):12276-12281.
[35]Chen J C,Mortenson L E.Two open reading frames (ORFs) identified near the hydrogenase structural genes inAzotobactervinelandii,the first ORF may encode for a polypeptide similar to rubredoxins.Biochimica et Biophysica Acta,1992,1131(1):122-124.
[36]Yoon K S,Hille R,Hemann C,Tabita F R.Rubredoxin from the green sulfur bacteriumChlorobiumtepidumfunctions as an electron acceptor for pyruvate ferredoxin oxidoreductase.Journal of Biological Chemistry,1999,274(42):29772-29778.
[37]Fareleira P,Santos B S,Antonio C,Moradas-Ferreira P,LeGall J,Xavier A V,Santos H.Response of a strict anaerobe to oxygen:Survival strategies inDesulfovibriogigas.Microbiology,2003,149(Pt 6):1513-1522.
[38]Grunden A M,Jenney F E Jr,Ma K,Ji M,Weinberg M V,Adams M W.In vitro reconstitution of an NADPH-dependent superoxide reduction pathway fromPyrococcusfuriosus.Applied and Environmental Microbiology,2005,71(3):1522-1530.
[39]Jenney F E Jr,Verhagen M F,Cui X,Adams M W.Anaerobic microbes:Oxygen detoxification without superoxide dismutase.Science,1999,286:306-309.
[40]Lombard M,Fontecave M,Touati D,Niviere V.Reaction of the desulfoferrodoxin fromDdesulfoarculusbaarsiiwith superoxide anion.Evidence for a superoxide reductase activity.Journal of Biological Chemistry,2000,275(1):115-121.
[41]Coulter E D,Kurtz D M Jr.A role for rubredoxin in oxidative stress protection inDesulfovibriovulgaris:Catalytic electron transfer to rubrerythrin and two-iron superoxide reductase.Archives of Biochemistry and Biophysics,2001,394(1):76-86.
[42]Lumppio H L,Shenvi N V,Summers A O,Voordouw G,Kurtz D M Jr.Rubrerythrin and rubredoxin oxidoreductase inDesulfovibriovulgaris:A novel oxidative stress protection system.Journal of Bacteriology,2001,183(1):101-108.
[43]Auchere F,Sikkink R,Cordas C,Raleiras P,Tavares P,Moura I,Moura J J.Overexpression and purification ofTreponemapallidumrubredoxin;kinetic evidence for a superoxide-mediated electron transfer with the superoxide reductase neelaredoxin. Journal of Biological Inorganic Chemistry,2004,9(7):839-849.
[44]Dal Bosco C,Lezhneva L,Biehl A,Leister D,Strotmann H,Wanner G,Meurer J.Inactivation of the chloroplast ATP synthase gamma subunit results in high non-photochemical fluorescence quenching and altered nuclear gene expression inArabidopsisthaliana.Journal of Biological Chemistry,2004,279(2):1060-1069.
[45]Friso G,Giacomelli L,Ytterberg A J,Peltier J B,Rudella A,Sun Q,Wijk K J.In-depth analysis of the thylakoid membrane proteome ofArabidopsisthalianachloroplasts:New proteins,new functions,and a plastid proteome database.The Plant Cell,2004,16(2):478-499.
[46]Shen G,Antonkine M L,van der Est A,Vassiliev I R,Brettel K,Bittl R,Zech S G,Zhao J,Stehlik D,Bryant D A,Golbeck J H.Assembly of photosystem Ⅰ.Ⅱ.Rubredoxin is required for the in vivo assembly of F(X) inSynechococcussp.Pcc 7002 as shown by optical and EPR spectroscopy.Journal of Biological Chemistry,2002,277(23):20355-20366.
[47]Shen G,Zhao J,Reimer S K,Antonkine M L,Cai Q,Weiland S M,Golbeck J H,Bryant D A.Assembly of photosystem Ⅰ.Ⅰ.Inactivation of therubAgene encoding a membrane-associated rubredoxin in the cyanobacteriumSynechococcussp.Pcc 7002 causes a loss of photosystem Ⅰ activity.Journal of Biological Chemistry,2002,277(23):20343-20354.
[48]Tran L S,Nakashima K,Sakuma Y,Simpson S D,Fujita Y,Maruyama K,Fujita M,Seki M,Shinozaki K,Yamaguchi-Shinozaki K.Isolation and functional analysis ofArabidopsisstress-inducible NAC transcription factors that bind to a drought-responsive cis-element in the early responsive to dehydration stress 1 promoter.The Plant Cell,2004,16(9):2481-2498.
[49]张海南,周青平,颜红波,梁国玲,刘文辉.盐胁迫对5种碱茅材料种子萌发的影响.草业科学,2013,30(11):1767-1770.
Zhang H N,Zhou Q P,Yan H B,Liang G L,Liu W H.Effects of salt stress on germination of fivePuccinelliadistansmaterials.Pratacultural Science,2013,30(11):1767-1770.
[50]金淑梅,孙丹,王吉.碱蓬金属硫蛋白基因对碳酸盐胁迫的应答.草业科学,2014,31(11):2082-2087.
Jin S M,Sun D,Wang J.Metallothionein gene(SgMT) fromSuaedaglaucaexpression under saline stress.Pratacultural Science,2014,31(11):2082-2087.
(责任编辑武艳培)
Research progress on Rubredoxin
Li Ying, Liu Shen-kui
(Alkali Soil Natural Environmental Science Center, Northeast Forestry University, Harbin 150040, China)
Abstract:Most of rubredoxin is concentrated from the strict anaerobic bacteria, including the bacteria, the ancient bacteria and a few micro aerobic bacteria in nature. In plants, these proteins are relatively few. It acts as a cofactor or electron donor to participate in multiple biochemical pathways. Researches show that these proteins both in plants, and in microorganisms play an important role in helped biological against oxidative stress. In this paper, we introduced the classification, structure and role of rubredoxinin microbes and plants, with aim to provide reference for further research in the future.
Key words:rubredoxin; Fe-S protein; electron transfer; superoxide reductase (SOR)
DOI:10.11829/j.issn.1001-0629.2015-0702
*收稿日期:2015-12-12接受日期:2016-02-24
基金项目:黑龙江省自然科学基金项目(C201406);黑龙江省博士后科研启动金(LBH-Q15010);中央高校基本科研业务费专项资金(2572014BA20);长江学者和创新团队发展计划( IRT13053);黑龙江省寒地作物种质改良与栽培重点实验开放课题资助项目(CGIC201204)
通信作者:柳参奎(1963-),男,朝鲜族,吉林省吉林市人,教授,博士,研究方向为植物抗逆育种。 E-mail:shenkuiliu@nefu.edu.cn
中图分类号:Q946.1
文献标志码:A
文章编号:1001-0629(2016)6-1118-08*
Corresponding author:Liu Shen-kuiE-mail:shenkuiliu@nefu.edu.cn
李莹,柳参奎.红素氧还蛋白研究进展.草业科学,2016,33(6):1118-1125.
Li Y,Liu S K.Research progress on Rubredoxin.Pratacultural Science,2016,33(6):1118-1125.
植物
生产层
第一作者:李莹(1979-),女,黑龙江哈尔滨人,讲师,博士,研究方向为植物抗逆育种。 E-mail:LY7966@163.com
