武陵山区四株真菌的分离与初步鉴定
2016-07-14何聪聪梅运军
何聪聪, 邓 威, 梅运军
( 武汉轻工大学 化学与环境工程学院,湖北 武汉 430023 )
武陵山区四株真菌的分离与初步鉴定
何聪聪, 邓威, 梅运军
( 武汉轻工大学 化学与环境工程学院,湖北 武汉 430023 )
摘要:从武陵山区分离到4株真菌,并对4株真菌的ITS序列进行了扩增与测序,MEGA6软件构建了真菌ITS序列系统发育树。从构建的系统发育树得知菌株HBM1、HBM2和菌株HBM3遗传关系较近,三者隶属Schizophyllumcommune,菌株HBM4隶属Irpexlacteus。
关键词:真菌;ITS;系统发育树;裂褶菌;白囊耙齿菌
1引言
真菌是自然界广泛分布的一类真核生物,是一种宝贵的自然资源,具有重要的食用及药用价值。目前已记载的食药用真菌大约有2 500种,国内报道的食药用真菌约1 000种。已探明的具有明显药效的真菌为400余种,而真正作为药物使用的仅20-30种[1-2]。因此,分离、筛选和鉴定真菌对丰富真菌种质资源具有十分重要的意义。
在真菌的分类鉴定中,传统的真菌鉴定主要依据形态学、抗原构造、生理生化等特点,由于出发点和依据不同,常出现分类难以统一;而随着真菌DNA条形码技术的出现,如将核糖体DNA内转录间隔区(ITS)引入到真菌的分类鉴定中,由于该分类鉴定不受个体形态特征、发育阶段的影响,能客观准确区分物种,且操作便捷,因此在真菌的分类鉴定中得到了广泛应用[3-6]。
本研究旨在从自然土样中分离食药用真菌,并通过真菌ITS序列对其进行分类鉴定,为丰富食药真菌的种质资源,进一步的开发利用真菌资源奠定一定的基础。
2材料与方法
2.1实验材料
2.1.1菌株来源
菌株分离自武陵山区湖南省张家界与湖北省恩施州交界处土样,分离所得4株真菌编号为HBM1、HBM2、HBM3、HBM4。
2.1.2培养基
PDA固体培养基:200 g 马铃薯、20 g 葡萄糖、12 g 琼脂、1000 mL 蒸馏水、121 ℃灭菌25 min 、pH自然。PDA液体培养基不加琼脂,其它成分与处理方式与PDA固体培养基一致。
2.1.3仪器
Eppendorf Mastercycler PCR仪(艾本德, 德国)。
2.1.4试剂
引物及PCR扩增试剂盒购于生工生物工程(上海)股份有限公司;其它试剂购自国药集团,均为分析纯。
2.2实验方法
2.2.1真菌分离与纯化
取土壤样品10 g 投入盛有90 mL无菌水的三角瓶中,并向三角瓶中投入10粒粒径大约5 mm灭菌玻璃珠,经充分震荡后梯度稀释至10-4。取上述各梯度的稀释液50 μL涂布于含PDA培养基的平板上,28 ℃倒置培养至长出单菌落。挑取单菌落再次于PDA培养基上纯化培养直至无杂菌。
2.2.2DNA提取
从平板上用接种铲接种1 cm×1 cm纯化真菌种子至盛有PDA的液体培养基三角瓶中180 r/min,28 ℃培养6 d。真菌DNA提取参照文献[7-8]。
2.2.3PCR扩增真菌ITS序列
本实验选择ITS1和ITS4为真菌ITS扩增引物[9]。以上述抽提的DNA为模板,ITS1 (5'-TCCGTAGGTGAACCTGCGG-3') 和ITS4 (5'-TCCTCCGCTTATTGATATGC-3')为引物进行PCR扩增。PCR总体系为25 μL,具体组成为10×PCR缓冲液 2.5 μL,dNTP (2.5 mmol/L) 0.5 μL,引物(5 μmol/L)各1 μL,Taq DNA聚合酶0.5 μL (5 U/μL),无菌水19 μL,模板0.5 μL。PCR反应条件依次为94 ℃预变性5 min;94 ℃变性45 s,52 ℃复性45 s,72 ℃延伸90 s,共30个循环;72 ℃终延伸10 min。PCR产物于1%琼脂糖凝胶电泳检测。
2.2.4测序与系统发育树构建
PCR扩增产物委托生工生物工程(上海)股份有限公司测序,测序结果用Blast软件比对GenBank上核苷酸序列并选择相似度高的序列。将测序得到的ITS序列与高度相似序列用CLUSTALX软件生成fasta序列格式,然后运用MEGA6软件分别构建Neighbor-Joining (NJ)系统发育树、Maximum Likelihood (ML)发育树以及Minimum-Evolution (ME)系统发育树。根据Felsenstein[10]所使用的方法将发育树拓扑结构的评估参数bootstrap值设置为1000。
3结果与分析
3.1菌株的培养特征
从武陵山区湖南省张家界与湖北省恩施州交界处土壤样品中分离到4株菌落形态各异的真菌菌株,分别编号为HBM1、HBM2、HBM3、HBM4,如图1所示。其中菌株HBM1在PDA 培养基上于28 ℃恒温条件下培养7 d 后,菌落表面为白色绒状,菌丝密集,气生菌丝明显且菌丝较长,基质白色;菌株HBM2在28 ℃下,在PDA 培养基上培养7 d之后,菌丝稀疏且在培养基中呈白色、辐射状,基质白色;HBM3菌丝生长缓慢,菌落白色绒状,基质白色;HBM4菌落白色呈绒毡状,菌丝密集较短,基质白色。
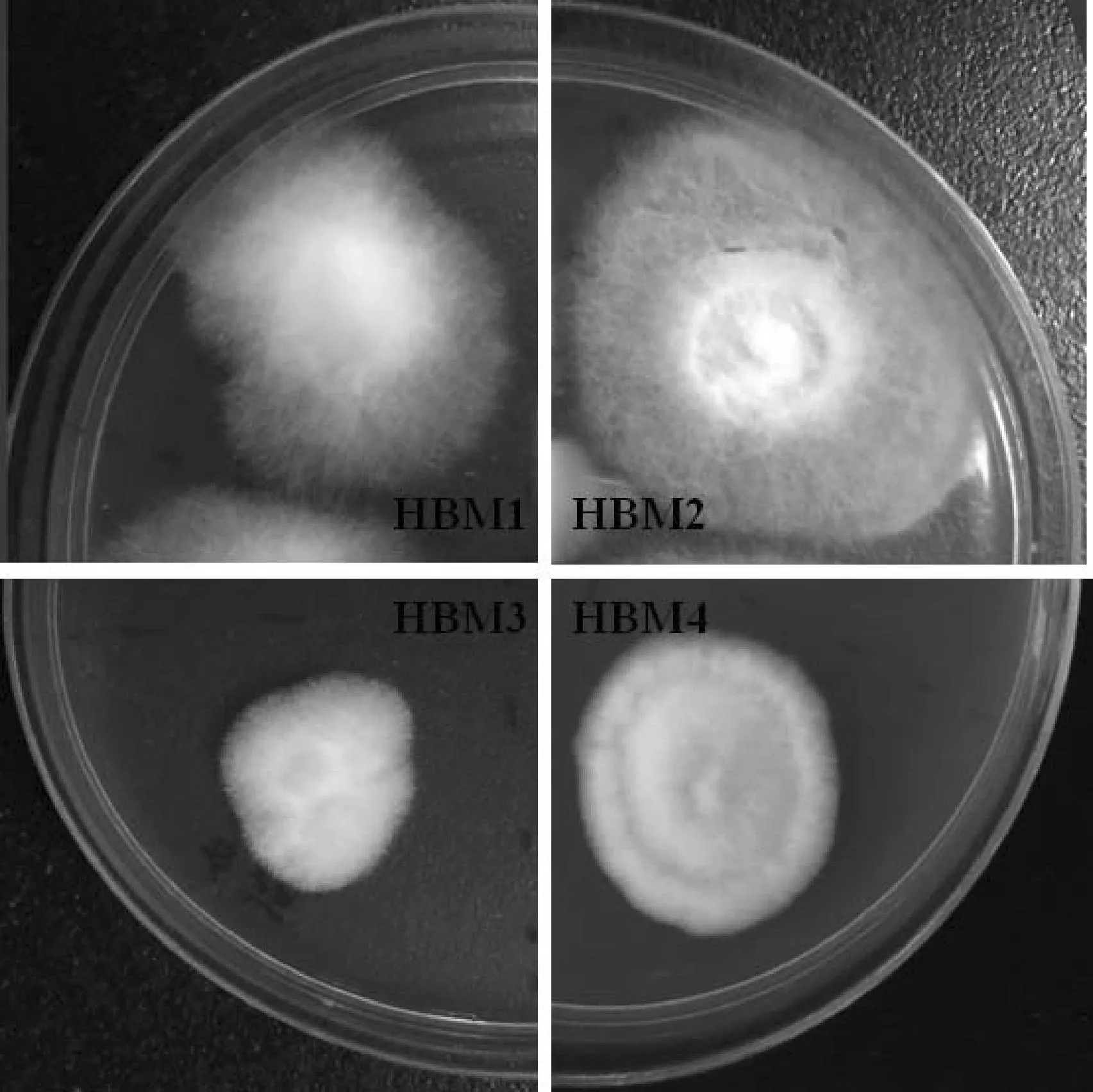
图1 4株真菌的菌落形态
3.2ITS序列系统发育树的构建
经测序后,得到菌株 HBM1、HBM2、HBM3和HBM4的ITS序列长度分别为597、597、597和635 bp,在GenBank上的登录号分别为KU726503、KU726504、KU726505、KU726506。分别将所测得的序列进行Blast比对后构建系统发育树。
3.2.1菌株HBM1、HBM2及HBM3 ITS序列系统发育树的建立及分析
菌株HBM1、HBM2及HBM3之间ITS序列经多重比对其相似性为99%,仅有1—2 个位点核苷酸不同,分别位于ITS1区和ITS2区。此外,三者ITS序列分别于NCBI上经Blast比对,结果显示HBM1、HBM2及HBM3 ITS序列与Schizophyllum属菌株的相似性最高,达到了99%。由于该属的菌种种类过少,为了加强发育树的稳定性,选取分数最高的四个ITS序列相似性为100% 的菌株Schizophyllumcommune(KJ588263.1)、Schizophyllumcommune(FJ372688.1)、Schizophyllumcommune(KP202299.1)、Schizophyllumcommune(KP202296.1),并结合该属的其他菌株构建NJ系统发育树,如图2(显示节点值≥50,图中括号内的标注为相应菌株rRNA在GenBank中的登录号)。由图2可知,菌株HBM1、HBM2及HBM3与裂褶菌Schizophyllumcommune的四个菌株形成了一个稳定的分支(节点值为85),表明这三株菌与Schizophyllumcommune亲缘关系最近;但三者在发育树中的进化地位有所差别,序列ITS序列不完全相同,这可能是存在种间变异,可能为Schizophyllumcommune的不同亚种。ML与ME系统发育树也显示HBM1、HBM2及HBM3具有类似的进化关系,如图3和图4所示。根据Renske Landeweert等对真菌的分子分类鉴定原则,即通过ITS区域比对,序列相似性≥99%,可判定为相同种;序列相似性在95%—99%之间,可判定为相同属;序列相似性≤95%,可判定为相同科[6-7]。因此根据ITS同源性及系统发育树的结果,菌株HBM1、HBM2和HBM3都隶属于S.commune。

图2 HBM1、HBM2和HBM3及其参考菌株的rDNA ITS序列的NJ系统发育树

图3 HBM1、HBM2和HBM3及其参考菌株的rDNA ITS序列的ML系统发育树
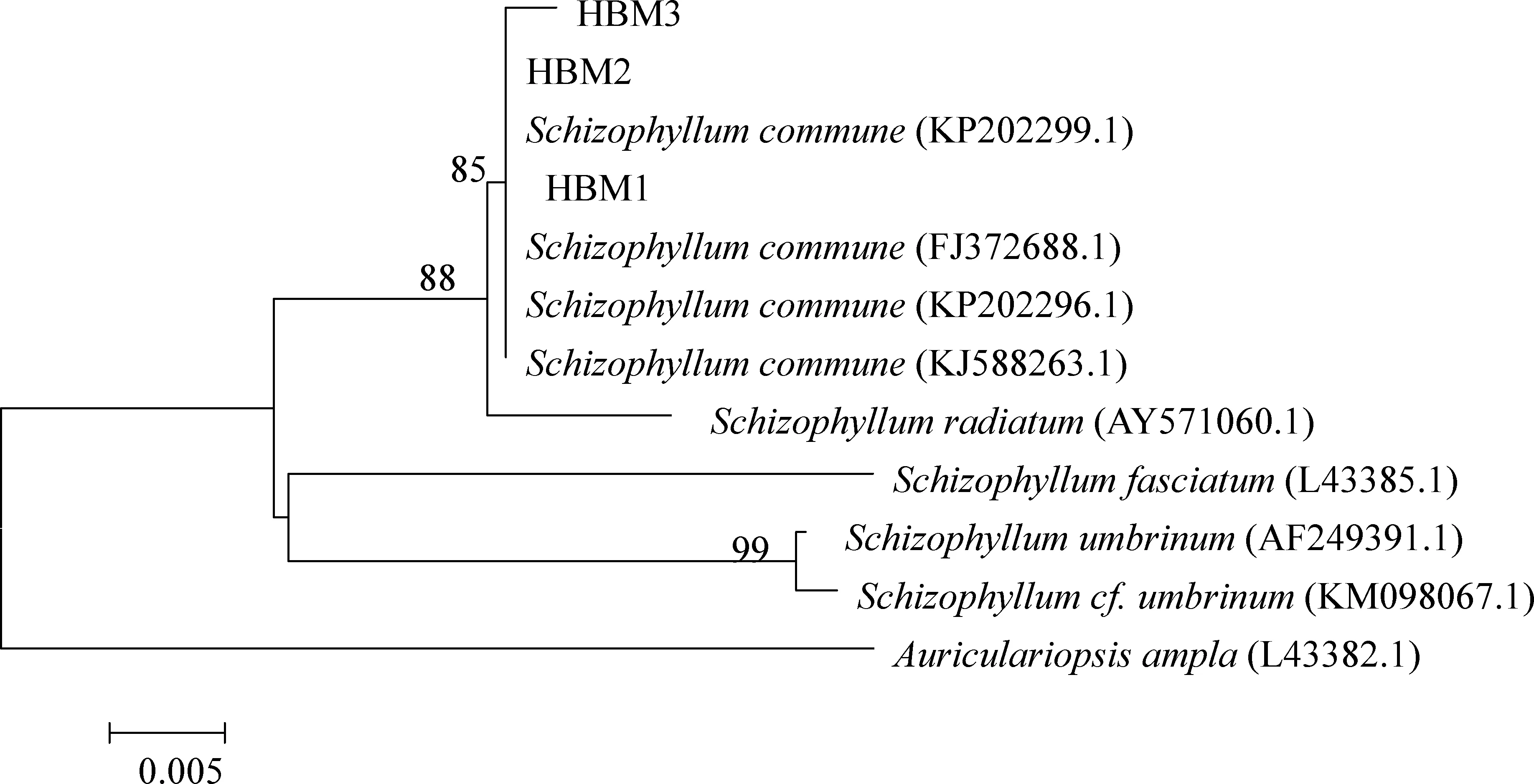
图4 HBM1、HBM2和HBM3及其参考菌株的rDNA ITS序列的ME系统发育树
3.2.2菌株HBM4 ITS序列系统发育树的建立及分析
菌株HBM4 ITS序列测序结果在NCBI Blast比对后,结果显示HBM4的ITS序列与白囊耙齿菌属Irpex的序列相似性最高(99%);在序列相似性≥99%的ITS序列中Irpex属菌株所占的比例约为99%。由于该属的菌种种类过少,为了加强发育树的稳定性,选取分数最高的四个ITS序列相似性为100%的白囊耙齿菌IrpexlacteusJX311924.1、 HM992801.1、JX290573.1及JX290578.1,并结合该属的其他菌种构建NJ系统发育树,如图5(显示节点值≥50)。由图5可知,菌株HBM4与白囊耙齿菌Irpexlacteus的四个菌株形成了一个稳定的分支(节点值为99)表明HBM4与白囊耙齿菌Irpexlacteus亲缘关系最近,在进化上应属于同一亚类群。构建的ML、ME系统发育树结果基本一致,如图6和图7。根据Renske Landeweert等对真菌的分子分类鉴定原则,结合ITS与系统发育树的结果,HBM4隶属于白囊耙齿菌Irpexlacteus。

图5 HBM4及其参考菌株的rDNA ITS序列的NJ系统发育树

图6 HBM4及其参考菌株的rDNA ITS序列的ML系统发育树

图7 HBM4及其参考菌株的rDNA ITS序列的ME系统发育树
4结束语
本研究从土壤样品中分离与纯培养到4株有益真菌,利用真菌ITS序列通过MEGA6软件分别构建了NJ、ML、ME系统发育树。初步鉴定HBM1、HBM2及HBM3三株菌隶属于裂褶菌(S.commune),而HBM4则隶属于白囊耙齿菌(I.lacteus)。该研究对丰富和发掘我国真菌资源具有一定的意义,但后续的研究和开发还需要进一步的深入。
参考文献:
[1]Brower A Z, DeSalle R, Vogler A P. Gene trees, species trees, and systematics: a cladistic perspective[J]. Annual Review of Ecology and Systematics, 1996,27(1): 423-450.
[2]Ciardo D E, Schar G, Bottger E C, et al. Internal Transcribed spacer sequencing versus biochemical profiling for indentification of medically important yeast [J]. J Clin Microbio, 2006, 44(1): 77-84.
[3]陈剑山, 郑服丛. ITS序列分析在真菌分类鉴定中的应用[J]. 安徽农业科学, 2007, 35(13): 3785-3786, 3792.
[4]Gardes M, Bruns T D. ITS primers with enhanced specificity forbasidiomycetesapplication to the identification ofmycorrhizaeadnrusts[J]. Mol Ecol, 1993, 2(2):113-118.
[5]Felsenstein J. Confidence limits on phylogenies: an approach using the bootstrap[J]. Evolution,1985,39(4):783-791.
[6]Vakalounakis D J, Fragkiadakis G A. Genetic diversity ofFusariumoxysporumisolates from cucumber: Differention by pathogenicity, vegetative compatibility, and RAPD fingerprinting [J]. Phytopa thology, 1999, 89 (2): 161-168.
[7]王琢,闫培生.真菌毒素产生菌的分子鉴定研究进展[J].中国农业科技导报,2010,12 (5):42-50.
[8]Cuongn V, Bachmann T T, Schmid R D. Development of a dipstick immunoassay for quantitative determination of 2, 4 dich- lorophenoxyacetic acid in water , fruit and urine samples[J]. Fresenius J Anal Chem, 1999, 364(6): 584-589.
[9]Campa D, Tavanti A, Gemignani F, et al. DNA microarray based on arrayed-primer extension technique for identification of pathogenic fungi responsible for invasive and superficial mycoses [J].J Clin Microbiol, 2008, 46(3): 909-915.
[10]谢丽源, 张勇, 彭金华,等. 桑黄真菌分子鉴定及遗传多样性分析[J]. 菌物学报, 2010, 29(3): 347-356.
Isolation and primary identification of four fungi from Wuling mountainous area
HECong-cong,DENGWei,MEIYun-jun
(School of Chemical and Environmental Engineering,Wuhan Polytechnic University,Wuhan 430023, China)
Abstract:Four fungi isolates were isolated from soil and their ITS sequences were amplified, sequenced and phylogenetically analyzed to identify species of the isolates. The result showed that the genetic distances of isolate HBM1, HBM2 and HBM3 were closer, all of three were belonged toSchizophyllum; the isolate HBM4 was classified asIrpexlacteus.
Key words:fungus; ITS; phylogenetic tree;Schizophyllum;Irpexlacteus
收稿日期:2016-03-01.修回日期:2016-04-20.
作者简介:何聪聪 (1990-),女,硕士研究生,E-mail: hecongcongwh@163.com. 通讯作者:梅运军 (1975-),男,博士,副教授,E-mail: meiyunjun_2000@163.com.
基金项目:湖北省教育厅科学研究计划资助项目(D20151704).
文章编号:2095-7386(2016)02-0030-06
DOI:10.3969/j.issn.2095-7386.2016.02.005
中图分类号:Q 93-331
文献标识码:A
