橡胶树羟基腈水解酶基因的克隆、表达和基因家族分析
2016-07-09丁梅吴坤鑫谭德冠张家明
丁梅 吴坤鑫 谭德冠 张家明

摘 要 植物体内羟基腈水解酶(HNL)催化羟基腈类化合物水解,释放氢氰酸,抵御害虫和病原菌的侵害。橡胶树是产氰植物,生物信息学研究表明,橡胶树有6个HNL基因,在分子进化分析中与大戟科其他植物的HNL基因聚为一类,与十字花科等植物的HNL亲缘关系较远,说明大戟科HNL基因的重复发生在与十字花科等植物分化之后,大戟科植物通过HNL基因的重复强化对病虫害的防御作用。通过RT-PCR从橡胶树热研7-33-97品系中克隆了其中最重要的1个HNL蛋白编码基因HbHNL-1。该基因cDNA序列全长1 003 bp,阅读框771 bp,编码257个氨基酸,其编码蛋白分子量为29.2 ku,理论等电点为5.7。定量PCR分析表明,HbHNL-1在初生胶乳中的表达量是次生胶乳中的100多倍,在幼嫩叶片中的表达量为成熟叶片的8倍。说明橡胶树的幼嫩组织器官是HbHNL-1的重点防御部位。
关键词 巴西橡胶树;HbHNL-1;基因克隆;基因表达分析;基因家族
中图分类号 S794.1;Q78 文献标识码 A
橡胶树(Hevea brasiliensis Müll. Arg.)属大戟科橡胶属,是重要的热带经济作物。其合成的天然橡胶占全世界天然橡胶总产量的99%以上[1-2]。我国目前植胶面积已达113.3万hm2,年产干胶85万t,位于五大产胶国之列,同时我国又是天然橡胶的最大进口国,天然橡胶对外依存度已超过80%[3-4]。因此提高天然橡胶产量成为当前我国天然橡胶研究的重要课题。病虫害是影响橡胶树产量的主要因素之一。生氰反应是橡胶树和木薯等大戟科作物防御病虫害的重要机制[5]。据不完全统计,全球有3 000多种植物利用生氰作用抵御病虫侵害[6]。生氰反应主要由两类酶完成,一种是生氰葡萄糖苷酶(Linamarase)[7],通过降解生氰葡萄苷释放氢氰酸;另一种是羟基腈水解酶[(S)-hydroxynitrile lyase,简称HNL],又名产氰醇羟基腈水解酶[(S)-cyanohydrin producing hydroxynitrile lyase]、氧腈酶[(S)-oxynitrilase]等。在植物体内,HNL所催化的产氰反应是一个可逆反应,HNL催化羟基腈类化合物水解,释放氢氰酸,以抵御害虫和病原菌的侵害[8-10]。
橡胶树的HNL能以各种脂肪链、芳香环和杂环化合物为底物合成不同的羟基腈化合物[11-12]。Hasslacher等[13]从橡胶树叶片中克隆1个HNL基因,并将其在大肠杆菌和酵母中表达,获得了有活性的重组蛋白。在原核表达的重组蛋白多积累在包含体中,而在毕氏酵母中重组蛋白表达量最高可达细胞可溶性蛋白的50%,每升培养基可产22 g重组蛋白[14]。同时通过对橡胶树HNL基因的改造,将一个酯化酶(Esterase)的活性氨基酸残基移植到HNL,可以提高HNL的反应效率[15]。综上,目前对橡胶树HNL的研究主要集中在其工业用途。但是对HNL基因在橡胶树体内的表达调控以及其在橡胶树抗病虫害中的作用机理等研究则很少报道。在本研究中,笔者通过生物信息分析,找到6个橡胶树HNL基因,分别从橡胶树叶片和胶乳中克隆了1个HNL基因的全长cDNA序列,并分析该基因的序列特征、系统进化、组织表达特点,为进一步研究HNL在橡胶树中的功能奠定基础。
1 材料与方法
1.1 材料
橡胶树品种热研7-33-97幼嫩叶片、成熟叶片,以及未开割树的初生胶乳、次生胶乳采集于中国热带农业科学院试验场三队。
1.2 方法
1.2.1 橡胶树羟基腈水解酶基因家族生物信息分析
分别利用文献报道的橡胶树羟基腈水解酶基因(U40402,命名为HbHNL-1)的DNA序列及其编码蛋白的N端120个氨基酸序列搜索GenBank数据库(http://blast.ncbi.nlm.nih.gov),选择GenBank数据库中的whole-genome shotgun contigs(wgs)项目下的橡胶树基因组数据库Hevea brasiliensis(Willd. ex A.Juss.)Müll. Arg.(taxid:3981)进行分析,获得包含该基因片段的大片段序列。用MacVector12.6(MacVector, Inc., USA)预测分析HbHNL-1基因编码蛋白质大小、等电点等信息;将HbHNL-1 5′端上游序列提交PlantCARE服务器(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)和PLACE 服务器(http://www.dna.affrc.go.jp/htdocs/PLACE/)预测启动子保守区域中潜在的顺式作用元件。同时,下载近缘物种的同源基因氨基酸序列,用ClustalX2.0进行序列比对。将比对结果输入MEGA version7.0中,用Neighbor-Joining(NJ)方法生成系统树,用Bootstrap对系统树进行检验,1 000次重复。
1.2.2 基因克隆 分别从同一棵6龄未开割树收集初生胶乳和次生胶乳,提取RNA,初生胶乳采自新抽嫩枝,次生胶乳采自离地1 m的树干。同时分别采集幼嫩叶片和成熟叶片。所有样品置于液氮中带回实验室。叶片经液氮充分研磨后,用RNA提取试剂盒(Tiangen, Beijing)提取总RNA,胶乳直接用试剂盒提取总RNA。然后用Oligotex Direct mRNA纯化试剂盒(Qiagen GmbH,Hilden,Germany)提取mRNA,再用Fermentas公司的M-MuLV反转录试劑盒把mRNA反转成cDNA。最后,用橡胶树HbHNL-1基因特异引物(F1: 5′-CATCAGAAAA
TGGCATTCGCTC-3′;R1: 5′-TACACAATGGGGGT
AGCTTTCC-3′)进行PCR扩增。25 μL扩增体系中含1 U Taq plus DNA polymerase,100 μmol/L dNTPs,0.2 μmol/L引物,0.5 μL cDNA模板。扩增程序为:95 ℃预变性3 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1.5 min,35个循环;72 ℃延伸10 min。1.0%的琼脂糖凝胶电泳分离PCR扩增产物,回收纯化目的条带,克隆到pMD19-T载体(大连宝生物)上,分别挑3个克隆测序。利用BLAST检索GenBank数据库,进行同源性分析。
1.2.3 荧光实时定量PCR分析 采用CFX96实时定量PCR仪(Bio-Rad Laboratories Inc., USA),实验操作按仪器使用说明书进行。反应试剂采用SYBR PrimeScript RT-PCR试剂盒(TaKaRa Biotechnology, Japan)。取不同实验材料RNA逆转录合成cDNA第一链, 将逆转录产物稀释5倍后做为实时定量RT-PCR分析模板。反应体系为20 μL,包括2 μL模板、10 μL 2×SYBR Premix和10 μmol/L的上、下游荧光定量特异引物各0.3 μL(终浓度为0.15 μmol/L)。PCR扩增程序如下: 94 ℃预变性30 s;94 ℃ 5 s, 58 ℃ 30 s, 72 ℃ 20 s,40个循环;72 ℃延伸7 min。每个样品重复3次。橡胶树HbHNL-1基因特异引物为F2(5′-GAGACTGTCCCA
CTTCAAATGA-3′)和R2(5′-CCAAGAGGTGGCTGAT
ACCT-3′)。内参基因采用ubiquitin-protein ligase 4(HbUBC4)基因(基因登录号:HQ323249),扩增引物为F3(5′-TTGATGGCTCACCCTGAACC-3′)和R3(5′-TGGTGACGCTGCAAGTCTAG-3′)。
2 结果与分析
2.1 橡胶树基因组中羟基腈水解酶基因家族分析
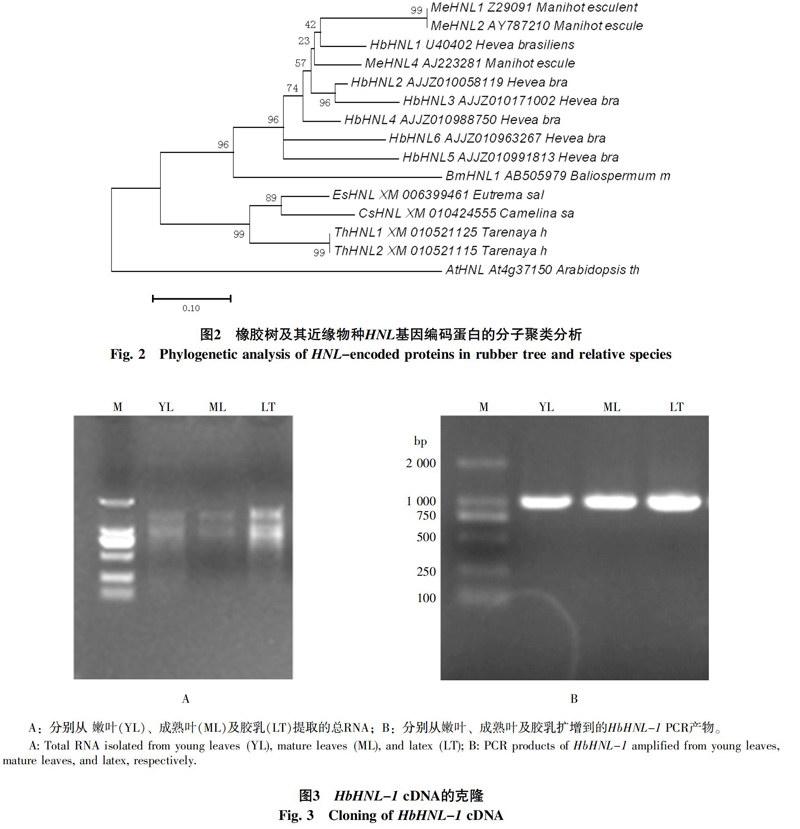
利用文献报道的橡胶树羟基腈水解酶基因(U40402,命名为HbHNL-1)搜索GenBank中橡胶树基因组数据库Hevea brasiliensis(Willd. ex A.Juss.)Müll. Arg.(taxid:3981),结果发现HbHNL-1基因位于长度为6 588 bp的基因组片段上(GenBank登录号: AJJZ010441696)。利用HbHNL-1基因编码蛋白的N端120个氨基酸序列再次搜索橡胶树基因组数据库,找到另外5个同源性较高的基因片段(基因登录号分别为:AJJZ010058119,AJJZ010171002,AJJZ010988750,AJJZ010991813,AJJZ010963267),其对应编码蛋白相似性达到80%~94%(图1)。说明HbHNL在橡胶树基因组中是一个基因家族。
2.2 橡胶树HbHNL的系统进化分析
为了明确橡胶树HbHNL基因家族的系统进化关系,从公共数据库下载典型的植物HNL蛋白及其近缘蛋白氨基酸序列,与本研究所获得的HbHNL基因的氨基酸序列进行比对,构建系统进化树。结果显示,橡胶树所有的HbHNL蛋白与大戟科植物木薯(Manihot esculenta Crantz)和斑籽(Baliospermum montanum(Willd.)Müll. Arg.)的HNL聚为一类,与拟南芥(Arabidopsis thaliana,十字花科)、醉蝶花(Tarenaya hassleriana,山柑科)、山嵛菜(Eutrema salsugineum,十字花科)、亚麻芥(Camelina sativa,十字花科)等植物的HNL类蛋白质亲缘关系较远(图2),说明大戟科植物的HNL基因家族成员的增多(基因重复)发生在与十字花科、山柑科等植物的分化之后。
2.3 橡胶树HbHNL-1的克隆及序列分析
雖然通过生物信息分析在橡胶树基因组中找到了6个HbHNL基因,但是,由于橡胶树基因组数据库还不完整,除HbHNL-1外,其他5个基因片段都不能编码完整的羟基腈水解酶;此外,根据笔者对胶乳和叶片转录组分析结果,其他5个基因的表达水平都很低,因此笔者认为HbHNL-1基因可能是橡胶树中起主要作用的羟基腈水解酶基因。于是重点研究了HbHNL-1的表达调控。
以橡胶树品种7-33-97的嫩叶、成熟叶及胶乳为材料,都提取到总RNA(图3-A)。用HbHNL-1基因特异引物进行RT-PCR扩增,在嫩叶、成熟叶及胶乳中,都扩增到1条长约1 000 bp的片段(图3-B)。测序结果表明,3种来源的羟基腈水解酶基因的cDNA序列完全相同,长度为1 003 bp,包含完整的羟基腈水解酶编码区,与已报道的橡胶树羟基腈水解酶基因(U40402)有4个碱基差异,可能是由于基因来自不同的橡胶树品种的缘故。HbHNL-1 cDNA序列的编码区长771 bp与U40402有1个碱基差异,但是两者编码的蛋白序列完全相同。用MacVector12.6预测该基因编码蛋白质含257个氨基酸残基,分子量为29.2 ku,等电点为 5.7。
2.4 HbHNL-1基因在幼嫩组织和初生胶乳中高效表达
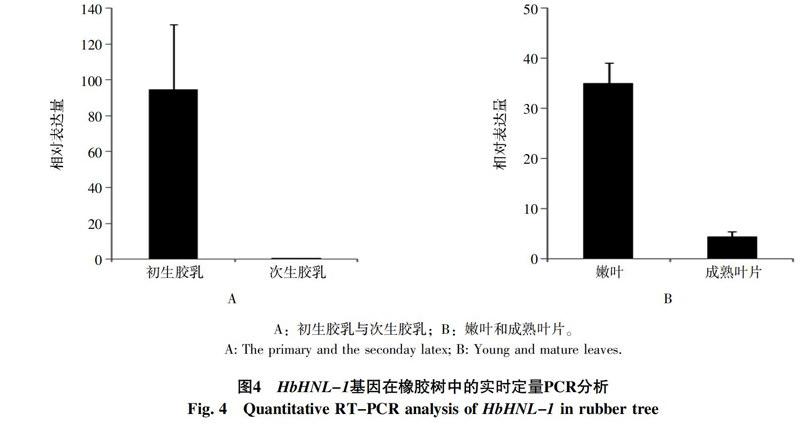
利用实时荧光定量PCR分析了HbHNL-1基因的组织表达特性。结果表明,HbHNL-1基因在初生胶乳中的表达量是次生胶乳的100多倍(图4-A)。此外,幼嫩叶片和成熟叶片实时定量PCR结果也表明,HbHNL-1基因在幼嫩叶片中的表达量也大大高于成熟叶片,约为成熟叶片的8倍(图4-B)。由于橡胶树幼嫩组织一般更容易受到病害虫的侵害,HbHNL-1基因在初生胶乳和幼嫩叶片中高效表达,可能与橡胶树对病虫害的精准防御相关。 2.5 橡胶树HbHNL-1基因启动子主要顺式作用元件预测
將HbHNL-1基因与其长度为6 588 bp的基因组片段AJJZ010441696比对,发现HbHNL-1基因编码区全长2 087 bp,含有3个外显子和2个内含子,所有内含子的剪切位点均符合真核生物“GT-AG”规则。3个外显子序列与cDNA序列一致,从而也验证了本实验扩增的HbHNL-1 cDNA序列的正确性。HbHNL-1基因翻译起始密码子ATG上游序列长度为3 607 bp。将上游长度为2 600 bp的序列提交PLACE服务器和Plant CARE服务器进行分析。由于Plant CARE只能给出1 500 bp长度序列分析结果,因此,将2 600 bp启动子序列分为两步分析,首先进行+1~1 500 bp片段序列分析,然后,对1 500~2 600 bp片段进行分析,最后,将两者结果合并统计。分析结果表明,在-80~-86位置有一个TATA Box(TATAAAT),上游-262~-265位置发现CAAT Box,推测该区域可能构成了HbHNL-1基因启动子的核心序列。除了含有高等植物启动子的保守元件TATA box和CAAT box外,HbHNL-1启动子序列还包含大量的光调控元件,如ACE、as-2-box、BoxⅠ、GATA-motif等。另外,HbHNL-1启动子序列中还有干旱诱导的MYB结合位点,以及水杨酸响应、生物节律控制、甲酯茉莉酸响应、叶形态发育等顺式调控元件(表1)。其中,除了茉莉酸和水杨酸响应与病虫抗性相关以外,其他顺式调控元件似乎与生长发育等生物过程相关。因此,除了具有防御功能外,HbHNL-1基因可能还有其他生物功能。
3 讨论
本研究通过数据库搜索在橡胶树基因组中发现了6个HbHNL家族基因。其中1个HbHNL基因(本文命名为HbHNL-1)编码完整的羟基腈水解酶,Hasslacher等[13]将该基因在酵母中表达,获得了有活性的重组蛋白。其他5个HbHNL基因在数据库中没有完整编码区。基因重复是基因家族扩增的主要来源之一,而基因重复后往往产生部分重复基因的失活或者功能异化。因此,很难断定橡胶树的其他5个HbHNL基因是否都有羟基腈水解酶基因的功能。对橡胶树叶片和胶乳转录组的分析结果表明,只有HbHNL-1具有比较高的表达量,其他5个HbHNL基因表达量都非常低,甚至不表达,因此认为HbHNL-1基因可能是橡胶树叶片和胶乳中起主要作用的羟基腈水解酶基因。当然,也不排除其他HbHNL基因在目前还没有检测的橡胶树器官,比如花、果实以及根中表达。
从橡胶树胶乳和叶片中克隆了HbHNL-1基因的全长cDNA序列。定量RT-PCR结果表明该基因在初生乳管和幼嫩叶片中上调表达,可能与幼嫩组织的防御相关。另外,HbHNL-1基因的启动子区还有大量的与光调控、干旱诱导、水杨酸和茉莉酸响应相关的调控元件(表1),说明HbHNL-1基因除具有防御功能外,可能还具有其他生物功能。
虽然同源性较高的基因可能执行相似的生物学功能,但由于HNL基因家族成员的多样性,HbHNL家族成员是否与同源性高的木薯等植物的HNL具有类似的生物功能,尚需得到遗传转化等研究的进一步分析验证。橡胶树所有的HbHNL蛋白与大戟科植物木薯和斑籽(Baliospermum montanum(Willd.)Muell. Arg.)的HNL聚为一类,与拟南芥(Arabidopsis thaliana,十字花科)、醉蝶花(Tarenaya hassleriana,山柑科)、山嵛菜(Eutrema salsugineum,十字花科)、亚麻芥(Camelina sativa,十字花科)等植物的HNL类蛋白质亲缘关系较远(图2)。说明大戟科植物的HNL基因家族成员的扩增发生在与十字花科、山柑科等植物的分化之后,推测大戟科植物可能通过增加HbHNL基因的数量来提高对病虫的抗性。 全球有3 000多种植物利用生氰作用抵御病虫侵害[6,10]。其中HNL家族蛋白在机械伤害、害虫和病原菌的侵害以及环境胁迫中发挥重要作用[8-10]。病虫害是目前影响橡胶树产量的主要因素之一。橡胶树可以通过羟基腈水解酶催化羟基腈类化合物水解,释放氢氰酸,从而抵御害虫和病原菌的侵害[6]。HbHNL-1在初生胶乳和幼嫩叶片中的表达量大大高于次生胶乳和成熟叶片中的表达量,说明橡胶树HbHNL-1主要参与幼嫩组织对病虫的防御。
参考文献
[1] Hagel J M, Yeung E C, Facchini P J. Got milk? The secret life of laticifers[J]. Trends Plant Sci, 2008, 13(12): 631-639.
[2] Verheye W. Growth and production of rubber[M]//Verheye w(ed) land use, land cover and soil sciences, (eOlSS), e.o.l.s.s., Ed. Oxford: UneScO-eOlSS Publishers, 2010.
[3] 董 昱. 2014年国内天然橡胶市场回顾与2015年展望[J]. 中国橡胶, 2015(5): 22-26.
[4] 邓海燕.全球天然橡胶产供需格局与形势分析[J].轮胎工业, 2011, 31(10): 589-596.
[5] Kadow D, Voss K, Selmar D, et al. The cyanogenic syndrome in rubber tree Hevea brasiliensis: Tissue-damage-dependent activation of linamarase and hydroxynitrile lyase accelerates hydrogen cyanide release[J]. Ann Bot, 2012, 109(7): 1 253-1 262.
[6] Wagner U G, Hasslacher M, Griengl H, et al. Mechanism of cyanogenesis: The crystal structure of hydroxynitrile lyase from Hevea brasiliensis[J]. Structure, 1996, 4: 811-822.
[7] 付莉莉, 沈培峰, 譚德冠, 等. 高氰和低氰木薯生氰葡萄糖苷酶linamarase基因的组织表达差异分析[J].中国农学通报, 2016, 32(11): 22-26.
[8] Poulton J E. Cyanogenesis in plants[J]. Plant Physiol, 1990, 94(2): 401-405.
[9] Dreveny I, Gruber K, Glieder A, et al. The hydroxynitrile lyase from almond: A lyase that looks like an oxidoreductase[J]. Structure, 2001, 9(9): 803-815.
[10] Kassim M A, Rumbold K. HCN production and hydroxynitrile lyase: A natural activity in plants and a renewed biotechnological interest[J]. Biotechnol Lett, 2014, 36(2): 223-228.
[11] Bauer M, Griengl H, Steiner W. Kinetic studies on the enzyme(S)-hydroxynitrile lyase from Hevea brasiliensis using initial rate methods and progress curve analysis[J]. Biotechnol Bioeng, 1999, 62(1): 20-29.
[12] Gartler G, Kratky C, Gruber K. Structural determinants of the enantioselectivity of the hydroxynitrile lyase from Hevea brasiliensis[J]. J Biotechnol, 2007, 129(1): 87-97.
[13] Hasslacher M, Schall M, Hayn M, et al. Molecular cloning of the full-length cDNA of (S)-hydroxynitrile lyase from Hevea brasiliensis. Functional expression in Escherichia coli and Saccharomyces cerevisiae and identification of an active site residue[J]. J Biol Chem, 1996, 271(10): 5 884-5 891.
[14] Hasslacher M, Schall M, Hayn M, et al. High-level intracellular expression of hydroxynitrile lyase from the tropical rubber tree Hevea brasiliensis in microbial hosts[J]. Protein Expr Purif, 1997, 11(1): 61-71.
[15] von Langermann J, Nedrud D M, Kazlauskas R J. Increasing the reaction rate of hydroxynitrile lyase from Hevea brasiliensis toward mandelonitrile by copying active site residues from an esterase that accepts aromatic esters[J]. ChemBioChem, 2014, 15(13): 1 931-1 938.
