人组蛋白去乙酰化酶11的克隆表达与生物信息学分析
2016-07-09
(漯河医学高等专科学校,河南 漯河 462000)
乙酰化是组蛋白共价修饰的一种方式。可逆的组蛋白乙酰化修饰发生在核心组蛋白N端的赖氨酸残基上,分别由组蛋白乙酰基转移酶(HAT)和组蛋白去乙酰化酶(HDACs)催化完成。HAT提高乙酰化水平,相反的HDACs则降低乙酰化水平[1]。HDACs是一个大家族,在哺乳细胞中已发现18个成员,根据其与酵母蛋白同源性被分为四类[2]。Ⅰ类HDACs包括HDAC1-3和HDAC8,与酵母蛋白Rpd3同源,主要存在于细胞核中,在各种组织细胞中都有表达[3]。Ⅱ类HDACs包括HDAC4-7,HDAC9和HDAC10,与酵母蛋白Hda1同源,穿梭于细胞质和细胞核之间,只在部分组织中有表达[4]。Ⅲ类HDACs与酵母蛋白Sir2同源,被称作Sirtuins。成员有7个:SIRTl-SIRT7[5],在细胞核和细胞质中都存在。HDAC11是Ⅳ类HDAC的唯一成员[6]。Ⅰ类、Ⅱ类和Ⅳ类HDAC被称作经典HDACs,经典HDACs和Sirtuins催化机制不同:经典HDACs为锌离子依赖性蛋白[7],Sirtuins为尼克酰胺腺苷酸(NAD)依赖性蛋白[8]。除了组蛋白外HDACs也能作用于非组蛋白,比如转录因子[9],从而间接影响这些转录因子所调控的基因的表达,因此HDACs在调控多种细胞进程中扮演了重要角色。相关研究表明,HDACs与癌症的发生有关[10]。目前,伏立诺他等HDAC抑制剂(HDACi)作为抗肿瘤药物进入临床并表现出了良好的治疗效果,但是现在大部分的HDACi为广谱型抑制剂,广谱HDACi可能会引起多种副作用,比如凝血障碍、心律失常等[11],这主要是因为HDACs存在多种亚型,因此针对亚型选择性的HDACi已经成为现在研究的一个热点。HDAC11是HDAC家族发现最晚的一个成员,也是研究最少的一个成员,本文通过克隆表达HDAC11并对HDAC11的序列,二级结构等进行预测分析希望对以后HDAC11的研究工作提供帮助。
一、材料与方法
1.材料
HDAC11序列(登录号:NM_024827.3)和引物交由生工生物合成,载体pGEX-6P-1为实验室保存,大肠杆菌菌株DH(5α)和BL21(DE3)感受态细胞购自北京全式金生物公司。EcoRⅠ,BamHⅠ和T4连接酶购自fermentas,DNA marker和Protein Marker购自全式金,质粒小提试剂盒和胶回收试剂盒购自莱科百奥。
2.方法
2.1 hdac11 基因的扩增
利用primer 5.0设计引物。引物为正向-CG GGATCC ATGCTACACACAACCCAGCTGT ACC, 反 向 -CG GAATTC TCAGGGCACTGCAGGGGGAA,引物交由生工生物合成。以合成的HDAC11序列为模版进行 PCR扩增,参数为:94℃预变性10min;94℃ 30s,60℃ 30s,72℃1.5min,30个循环;72℃终延伸10min。扩增产物用1%琼脂糖凝胶电泳回收。
2.2 重组质粒的构建与鉴定
将回收的目的片段与pGEX-6P-1载体分别用EcoRⅠ和BamHⅠ双酶切,并将酶切产物按一定比例16℃连接5h。将连接产物转化到大肠杆菌DH(5α)感受态细胞,氨苄青霉素筛选阳性克隆,对阳性克隆做双酶切鉴定和测序鉴定。
2.3 重组蛋白的表达
将测序正确的重组质粒转化到大肠杆菌BL21(DE3)感受态细胞中。挑取单克隆于含氨苄青霉素的LB培养基中培养,OD600=0.6时,加入IPTG使其终浓度为0.1 mM/L,16℃诱导表达12h,离心收集菌体。用PBS重悬菌体沉淀,超声破碎后高速离心,取上清和沉淀制样跑胶分析目的蛋白的表达形式。
2.4 HDAC11的生物信息学分析
利用ProtParam和Protscale对HDAC11的氨基酸序列及理化性质进行分析,blast在线分析HDAC11的同源性,SOPMA对其二级结构进行预测,NetPhos和SUMOplot对其进行翻译后修饰预测。
二、结果与分析
1.目的蛋白的克隆表达
1.1 重组质粒的构建及鉴定
构建的重组质粒用EcoRⅠ和BamHⅠ做双酶切鉴定,获得4900bp的载体片段和约1000bp的目的基因片段,结果表明成功构建重组质粒。
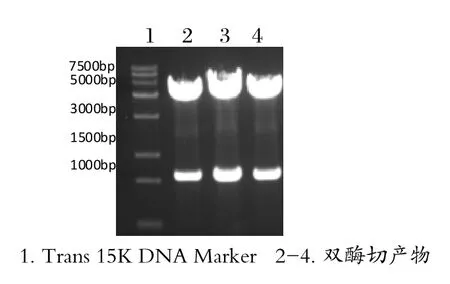
图1 重组质粒的双酶切鉴定
1.2 重组蛋白的表达
将重组质粒转入大肠杆菌BL21(DE3)中,经IPTG诱导后收集菌体,SDS-PAGE检测结果显示,加入IPTG诱导后,转入重组质粒的菌株总蛋白提取物中出现一条分子量约为65kD的蛋白条带,与重组HDAC11蛋白大小相符,表明重组表达载体经IPTG诱导后目的蛋白得到表达。但HDAC11蛋白主要以包涵体形式存在。

图2 表达蛋白的SDS-PAGE分析
2.HDAC11的生物信息学分析
2.1 HDAC11的氨基酸序列及理化性质分析
ProtParam分析表明HDAC11(UniProtKB:Q96DB2)共含有347个氨基酸残基,含量较高的是亮氨酸(10.1%)、甘氨酸(8.4%)、缬氨酸(7.8%)和精氨酸(7.8%),含量较少的氨基酸有半胱氨酸(0.6%)、色氨酸(1.4%)等。将人 HDAC11 的氨基酸序列与小鼠(UniProtKB/Swiss-Prot:Q91WA3.1),食蟹猴(UniProtKB/Swiss-Prot:Q9GKU5.2)的 HDAC11 序列进行比较发现同源性均达到90%以上。将人源HDAC11与人源HDAC1-HDAC10蛋白进行同源性比较,结果表明HDAC11与其他HDAC蛋白的同源性均不高,其与HDAC1具有31%的最高同源性,与HDAC5的同源性最低,为21%。HDAC11的分子式为C1763H2786N490O501S10,分子量为39183,酸性氨基酸有44个,碱性氨基酸有44个,理论等电点7.17为中性蛋白。在溶液中的不稳定指数为39.1,根据蛋白质的不稳定指数在40以下为稳定蛋白的标准推测该蛋白为稳定蛋白。脂肪系数为96.05。
2.2 HDAC11的二级结构预测
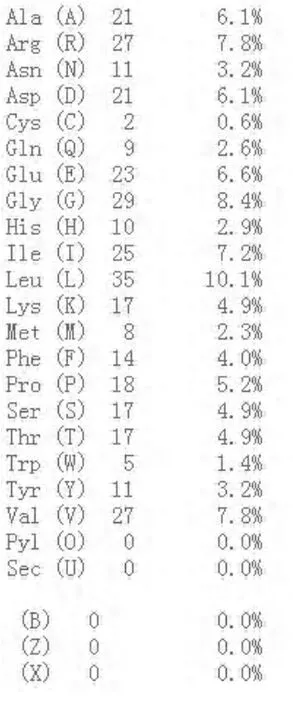
图3 HDAC11的氨基酸组成分析
利用SOPMA在线软件对HDAC11蛋白的二级结构进行预测,结果表明:该蛋白的二级结构中α-螺旋的比例最高,达到37.75%,无规卷曲次之,为30.26%,延伸链和β-转角所占比例分别为19.88%和12.10%。因此α-螺旋和无规卷曲是该蛋白的主要二级结构元件。含量最高的α-螺旋主要分布于氨基酸残基的43-66、76-92、154-170、232-247四个区域,无规卷曲、延伸链和β-转角则不均匀地分布于整个蛋白质多肽链中。

图4 HDAC11的二级结构分析
2.3 HDAC11的翻译后修饰预测
利用NetPhos对HDAC11蛋白进行磷酸化修饰预测,结果发现该蛋白拥有9个丝氨酸,2个苏氨酸和1个酪氨酸共12个潜在的磷酸化位点。利用SUMOplot对其进行SUMO化修饰预测,结果表明HDAC11有两个评分较高的修饰位点,分别为K280和K50。

图5 HDAC11的磷酸化位点分析

图6 HDAC11的SUMO化位点分析
三、讨论
本研究将hdac11基因构建到原核表达载体pGEX-6P-1中,转化入大肠杆菌BL21(DE3)中进行表达,目的蛋白有表达,但是以包涵体的形式存在。外源基因在大肠杆菌中的高表达常常导致形成无活性的包涵体,目前减少包涵体形成主要有两种策略:①降低蛋白合成速度比如降低诱导剂浓度和诱导温度[12];②加入促进可溶性表达的生长添加剂[13]。对于已经形成包涵体的蛋白可通过将包涵体蛋白体外复性得到生物活性蛋白。另外本文利用生物信息学软件如ProtParam、ProtScale、SOPMA等对HDAC11的序列,理化性质、二级结构等进行了分析预测。对HDAC11的氨基酸和理化性质分析可知,HDAC11在人、小鼠和食蟹猴中的序列非常保守,而HDAC11与其他家族成员的同源性非常低,相比较而言HDAC11更接近于Ⅰ类HDACs。对HDAC11的二级结构预测后发现无规卷曲和α-螺旋的比例较高,无规卷曲结构比较松散并随环境而改变,常构成酶活性部位和蛋白质特异的功能部位。另外HDAC11可能存在12个磷酸化位点,已有多篇文献报道HDAC1-HDAC10的10个蛋白均含有磷酸化位点,比如HDAC1 的 S421[14]、HDAC6 的 S1035[15]、HDAC7 的 T286[16]、HDAC8 的 S39[17]等,这些修饰可能会影响酶的活性或蛋白的定位。Ⅱa类HDACs在细胞内的定位主要是由核的输入输出信号和14-3-3蛋白调控,HDAC4、5、7、9含有三个保守的14-3-3结合位点,结合14-3-3后会以一种磷酸化依赖的方式将HDACs留在细胞质中[17]。比如HDAC4的S350被磷酸化后与14-3-3的结合力增强[18],HDAC5的S259和S498被磷酸化后会促进蛋白从细胞核输出到细胞质[19],因此对于Ⅱa类HDACs来说磷酸化影响了蛋白的定位。HDAC1的S421和S423被磷酸化后会促进酶活性及与NuRD及SIN3复合物的相互作用[14],而HDAC8的S39被磷酸化后会降低酶活性[17],对于HDAC11来说,磷酸化可能影响了它的酶活性。另外HDAC11还含有潜在的SUMO化修饰位点,对于HDACs家族而言,已有文献报道HDAC1、3、4、6和 9有 SUMO化修饰[20,21],HDAC1的 K444和 K476被修饰后酶的活性增强[21],SUMO化可能也影响了HDAC11的活性。
四、结论
本研究一方面将该基因构建到原核表达载体pGEX-6P-1中进行目的蛋白表达,结果以包涵体形式表达,另一方面利用生物信息学软件分析了该蛋白的氨基酸序列、理化性质、二级结构、磷酸化位点等,为进一步了解该蛋白的结构与功能以及组蛋白去乙酰化酶家族成员之间的差异打下基础。
[1]Murr R.Interplay between different epigenetic modifications and mechanisms.Adv Genet,2010,70:101-41.
[2]Xu WS,Parmigiani RB,Marks PA.Histone deacetylase inhibitors:molecular mechanisms of action.Oncogene,2007,26:5541-52.
[3]Gregoretti IV,Lee YM,Goodson HV.Molecular evolution of the histone deacetylase family:functional implications of phylogeneticanalysis.J Mol Biol,2004,338:17-31.
[4]Marks PA,Dokmanovic M.Histone deacetylase inhibitors:discovery and development as anticancer agents.Exp Opin Invest Drugs,2005,14:1497-511.
[5]Blander G,Guarente L,The Sir2 family of protein deacetylases[J].Annu Rev Biochem,2004,73:417-435.
[6]Mottet D,Castronovo V.Histone deacetylases:target enzymes for cancer therapy.Clin Exp Metastasis,2008,25:183-9.
[7]Codd R,Braich N,Liu J,et al,Pakchung AA.Zn(II)-dependent histone deacetylase inhibitors:suberoylanilide hydroxamic acid and trichostatin A.Int J Biochem Cell Biol,2009,41:736-9.
[8]Neugebauer RC,Sippl W,Jung M.Inhibitors of NAD+dependent histone deacetylases(sirtuins).Curr Pharm Des,2008,14:562-73.
[9]Yang XJ,Seto E.The Rpd3/Hda1 family of lysine deacetylases:from bacteria and yeast to mice and men.Nat Rev Mol Cell Biol,2008,9:206-18.
[10]Yang XJ,Gregoire S .Class II histone deacetylases:from sequence to function,regulation,and clinical implication.Mol Cell Biol,2005,25:2873 2884
[11]O.Bruserud,C.Stapnes,E.Ersvaer,B.et al.Ryningen,Histone deacetylase inhibitors in cancer treatment:a review of the clinical toxicity and the modulation of gene expression in cancer cell.Curr.Pharm.Biotechnol,2007,8:388-400.
[12]Xie Y,Wet laufer D B .Control of aggregation in protein refolding :the temperature leap tactic .Protein Sci,1996,5(3):517-523
[13]Georgious G,Valax P .Expression of correctly folded proteins in E .coli .Curr Opin Biot echnol,1996,7(2):190-197
[14]Pflum MK,Tong JK,Lane WS,et al.Histone deacetylase 1 phosphorylation promotes enzymatic activity and complex formation.J Biol Chem.2001,?276(50):47733-41.
[15]Bian Y,Song C,Cheng K,et al.An enzyme assisted RP-RPLC approach for in-depth analysis of human liver phosphoproteome.J Proteomics,2014?,96:253-62.
[16]Dephoure N,Zhou C,Villén J,?et al.A quantitative atlas of mitotic phosphorylation.Proc Natl Acad Sci U S A,2008,105(31):10762-7.
[17]Somoza JR,Skene RJ,Katz BA,?et al.Structural snapshots of human HDAC8 provide insights into the class I histone deacetylases.Structure,2004,12(7):1325-34
[18]Wang AH,Kruhlak MJ,Wu J,?et al.Regulation of histone deacetylase 4 by binding of 14-3-3 proteins.Mol Cell Biol,2000,20(18):6904-12
[19]McKinsey TA,Zhang CL,Olson EN.Activation of the myocyte enhancer factor-2 transcription factor by calcium/calmodulin-dependent protein kinase-stimulated binding of 14-3-3 to histone deacetylase 5.Proc Natl Acad Sci U S A,2000,97(26):14400-
[20]David G,Neptune MA,DePinho RA.SUMO-1 modification of histone deacetylase 1(HDAC1)modulates its biological activities.J Biol Chem.2002,277(26):23658-63.
[21]Petrie K,Guidez F,Howell L,et al.The histone deacetylase 9 gene encodes multiple protein isoforms.J Biol Chem,2003,278(18):16059-72.
