转基因小鼠荧光神经元结构与功能之间关联的建立
——膜片钳记录结合激光共聚焦三维重建
2016-06-16赵波何家候朱俊玲王举磊李柱一王文挺
赵波 何家候 朱俊玲 王举磊 李柱一 王文挺
710038第四军医大学唐都医院神经外科(朱俊玲、王举磊);730070兰州军区兰州总院安宁分院神经内科(赵波);710038第四军医大学唐都医院神经内科(赵波、李柱一);710032第四军医大学学员旅(何家候);710032第四军医大学基础部神经生物学教研室暨脑科学协同创新中心(王文挺)
转基因小鼠荧光神经元结构与功能之间关联的建立
——膜片钳记录结合激光共聚焦三维重建
赵波何家候朱俊玲王举磊李柱一王文挺
710038第四军医大学唐都医院神经外科(朱俊玲、王举磊);730070兰州军区兰州总院安宁分院神经内科(赵波);710038第四军医大学唐都医院神经内科(赵波、李柱一);710032第四军医大学学员旅(何家候);710032第四军医大学基础部神经生物学教研室暨脑科学协同创新中心(王文挺)
摘要:目的在细胞水平建立神经元结构和功能之间的关联。方法利用D1多巴胺受体表达红色荧光蛋白的细菌人工染色体(bacterial artifical chromosome,BAC)转基因小鼠制备脑片,对D1多巴胺受体荧光阳性纹状体中等多棘神经元(medial spiny neurons, MSNs)进行膜片钳电生理记录和细胞标记,再经激光共聚焦系统对该神经元进行结构的三维重建,观察MSN树突分支密度以及树突棘形态学特点。结果(1)电生理结果提示所标记的细胞具有偏超级化的静息膜电位(-87 mV)、较小的输入电阻(16.4 MΩ)及较长的动作电位潜伏期(172 ms)等典型的MSN电生理学特征。(2)三维重建结果可见细胞胞体略成圆锥体,树突呈放射状向四周发散。(3)进行树突棘重塑后发现MSNs平均树突棘长度(2.37±0.14)μm,平均宽度为(1.39±0.14)μm。结论描述神经元功能的膜片钳数据与三维重建的细胞形态学数据相结合的方法可建立神经元结构和功能之间的关联。
关键词:神经元;细菌人工染色体转基因技术;膜片钳;细胞标记;三维重建
神经元的结构和功能密不可分,其结构变化必然导致功能改变。因此同步记录神经元结构和功能的改变具有重要的研究意义。神经元分为多种不同类型,如纹状体的投射神经元为中等多棘神经元(medial spiny neurons, MSNs),根据表达的多巴胺受体的不同,可以将其分为表达多巴胺受体1的MSNs(D1 MSNs)和表达多巴胺受体2的MSNs(D2 MSNs)[1]。两种MSNs分别组成了基底节直接通路及间接通路,参与纹状体及整个基底神经节的功能调节。但在以往研究中,由于无法准确区分上述两类神经元,因而无法对直接及间接通路的功能进行精确研究。现有研究表明,利用细菌人工染色体(bacterial artifical chromosome,BAC)转基因技术可以将荧光蛋白标记到特定类型神经元[2],从而实现研究特定类型神经元的目的。本实验中,通过利用使D1 MSNs表达红色荧光蛋白的BAC转基因小鼠制作纹状体脑片[3],通过荧光显微镜,准确选择这些红色荧光阳性的MSNs,并利用膜片钳技术进行电生理记录及细胞标记,从而可以同时获得特定神经元的电生理学和形态特征。
1材料和方法
1.1材料
1.1.1实验动物:选取1.5~2个月龄健康D1-Td Tomato转基因小鼠,雌雄不限。该小鼠由麻省理工学院McGovern脑研究所冯国平教授赠送[3],由第四军医大学动物实验中心饲养。实验动物饲养符合国家医学实验动物管理标准,所有实验获第四军医大学医学伦理委员会批准,并遵循实验动物保护、伦理规范。
1.1.2主要试剂及仪器:切片液配方(mmol/L):氯化胆碱115,氯化钾2.5,磷酸二氢钠1.25,氯化钙0.5,氯化镁8,碳酸氢钠26,葡萄糖10,抗坏血酸0.1,丙酮酸钠0.4,渗透压 300~305 mOsm。人工脑脊液(artificial cerebral spinal fluid,ACSF)配方(mmol/L):氯化钠119,氯化钾2.3,磷酸二氢钠1.0,氯化钙2.5,氯化镁1.3,碳酸氢钠26,葡萄糖11,渗透压 300~305 mOsm。电极内液配方(mmol/L):葡萄糖酸钾128,羟乙基哌嗪乙磺酸10,磷酸肌酸钠 10,乙二醇双(2-氨基乙醚)四乙酸 1.1,三磷酸腺苷镁盐5,三磷酸鸟苷钠盐0.4,0.5%(质量分数)neurobiotin (蓝色荧光)350,用氢氧化钾调节pH至7.4,用蔗糖调节渗透压至300~305 mOsm。以上药品均购自Sigma公司。Streptavidin, Alexa Fluor®350 conjugate(S11249,Molecular probe, USA)。振动切片机(Vibratome 1000 plus)。水平程控电极拉制仪(P-97, Sutter Instruments, USA)。激光共聚焦显微镜(FV1000)及红外微分干涉相差显微镜(Olympus,Japan)。膜片钳放大器(axon 200A amplifier,Molecular Devices, USA)。电动显微三维操控器(MX7600R,SD,USA)。
1.2方法
1.2.1脑片制备:以1%(质量浓度)戊巴比妥钠按体质量(30~40)mg/kg麻醉小鼠,麻醉成功后沿两侧剪开胸腔,暴露心脏,用20~30 mL 95% O2/5% CO2(均为体积分数)混合气饱和冰浴的切片液进行心脏灌流。之后迅速断头取脑,放入冰冻切片液中1~2 min。取出脑组织,俯卧位放置在切片液湿润的滤纸上,切除大脑尾端后1/4。将脑组织以切面为底直立,用502胶粘接在切片台上,脑组织背侧对准刀片的方向。在95% O2/5% CO2(均为体积分数)混合气饱和的冰冻切片液中用振动切片机切取300~350 μm的含纹状体的冠状脑片。将脑片移至孵育槽中用32℃切片液孵育20 min后更换为室温的ACSF,继续孵育至少1 h后开始进行电生理记录,记录期间持续给予95% O2/5% CO2(均为体积分数)混合气。
1.2.2电生理记录及细胞标记:选取典型的纹状体脑片,放入记录槽中。记录槽以2~3 mL/min的流速给予ACSF灌流,利用记录槽温控装置使记录温度维持在24~28℃。红外微分干涉相差可视条件(DIC)下在40倍物镜下选择形状呈卵圆形,直径约10 μm,胞体圆润,表面清晰、柔和,未见细胞核的细胞。本实验所用转基因小鼠系将红色荧光蛋白(Td Tomato)特异性标记到D1 MSNs上,因此D1 MSNs在红色荧光通道下发出红色荧光,其余神经元无荧光。确认红色荧光阳性细胞进行全细胞膜片钳记录。记录电极电阻3~5 MΩ。电极内充灌电极内液电生理数据经膜片钳放大器放大,用Digidata 1322A数据采集系统和Clampex 9.0 软件采集、存储和分析,数据采样率为20 kHz,低频滤波为5 kHz。
在高阻封接形成后,给电极以短暂的脉冲负压吸破细胞膜后可见电容电流,代表细胞形成全细胞模式[4],即开始计时,进行电生理刺激和记录。在电流钳模式下,给予从超极化到去极化的方波电流刺激,记录其静息膜电位及动作电位以进一步明确细胞类型。随后留置电极10~15 min以使neurobiotin 350充灌到细胞远端末梢。充灌完全后在蓝色荧光通道下可见灌注到细胞内的neurobiotin 350发出蓝色荧光。完成记录后以最小档速度控制电动显微三维操控器缓慢将电极从细胞表面退出。脑片在记录槽中用10 mL/min流速冲洗30 min后放置到4%(质量浓度)多聚甲醛中固定过夜。次日将固定好的脑片用1×PBS低速振荡清洗3次,每次10 min。5%(质量浓度)BSA+0.3%(体积分数)triton室温下封闭1 h。加入streptavidin, Alexa Fluor®350 conjugate(用上述3%BSA+0.3%triton稀释至1∶600)4℃孵育过夜。第3天以1×PBS低速振荡清洗3次,每次10 min。使记录细胞层面向上放置在载玻片上,50%(体积分数)甘油封片。
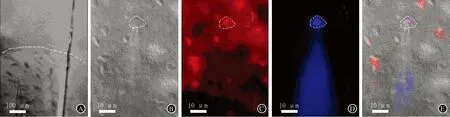
A:低倍镜下的纹状体记录区域(虚线上部分为皮层部分,之下部分为纹状体部分;物镜×4); B:IR-DIC模式下典型的MSN神经元(物镜×40);C:红色荧光通道下的图像(物镜×40);D:蓝色荧光通道下的图像(物镜×40);E:IR-DIC、红色通道和蓝色通道的融合图像
图1选择荧光标记的D1受体阳性MSNs进行电生理记录和细胞标记
用激光共聚焦显微镜观察脑片,10倍物镜下确认记录的部位,寻找蓝色荧光细胞。如蓝色荧光在位置上与红色荧光重合代表标记成功。根据蓝色细胞细胞突起的范围选择20倍或40倍物镜,确认包含所标记细胞突起的Z轴最高和最低层面,每层厚度为1~1.5 μm,选择XYZ模式对整个细胞进行逐层扫描。分析树突棘时选择树突中段(距胞体100 μm)的一段树突(长约50 μm)进行三维重建。切换到60倍油镜,确认Z轴树突最高层面和最低层面,以层厚1~1.5 μm、XYZ模式对选择的树突进行扫描。实验完毕后存储为默认格式。
1.2.3标记细胞三维重建和分析:图像文件在Imaris 7.8中打开,选择蓝色通道,将显示颜色切换到绿色。添加新的Filaments,选用自动模式对细胞整个突起进行三维重建。利用重建后的结果对整个树突的复杂度进行sholl分析,结果导入到Prism 6.0(GraphPad Software, Inc., USA)进行作图和分析。随后添加新的Filaments,选择分析树突棘,采用自动模式辨别树突棘。所得树突棘数目、长度、宽度等数据导入到Prism 6.0进行作图和分析,观察距胞体不同距离上树突分布以及树突棘数目、长度、宽度等特点。
2结果
2.1纹状体MSNs细胞标记及电生理结果 在纹状体记录区域(图1A),40倍物镜、IR-DIC模式下可见散在的MSNs(图1B,图中虚线区域所示,下同)。D1 MSNs在红色荧光通道下呈现红色荧光(图1C)。形成全细胞模式后,切换到蓝色荧光通道时可见胞体充盈蓝色荧光(图1D)。不同通道的图片经存储和后期处理可见所记录的细胞在红外微分干涉相差可视条件、红色通道和蓝色通道重合(图1E),在形态学上证明所标记的细胞即为Td Tomato所标记的D1 MSNs。在给予方波刺激后细胞静息膜电位为-87 mV,在超极化状态下输入电阻为16.4 MΩ,在去极化刺激达到阈值时动作电位潜伏期为172 ms(图2)。

图2电流钳模式下神经元动作电位模式图:在电流钳模式下,神经元静息膜电位为-87 mv(A);给予强度不断增大的方波刺激(电流强度-0.2 mA~0.38 mA)后可诱发出动作电位(B),动作电位潜伏期172 ms,输入电阻16.4 MΩ,基强度0.24 pA
2.2纹状体MSNs细胞三维重建及树突分布分析 三维重建后可见所标记细胞胞体略呈圆锥形,树突呈放射状向四周发散(图3A);对细胞进行突起追踪,平面图(图3B)可见细胞的突起向四周分散。sholl分析结果提示该细胞树突走行在距胞体100 μm的距离具有最多的分支,随着距离胞体距离增加树突分支逐渐减少,据胞体较近区域分支也较少(图4)。

A:经Imaris软件重建荧光标记神经元的3D图像;B:对图A细胞进行突起追踪所得平面图
图3膜片钳记录和标记后的整个神经元的三维重建分析图

图4经sholl分析所得荧光标记神经元的树突分支的分布曲线图
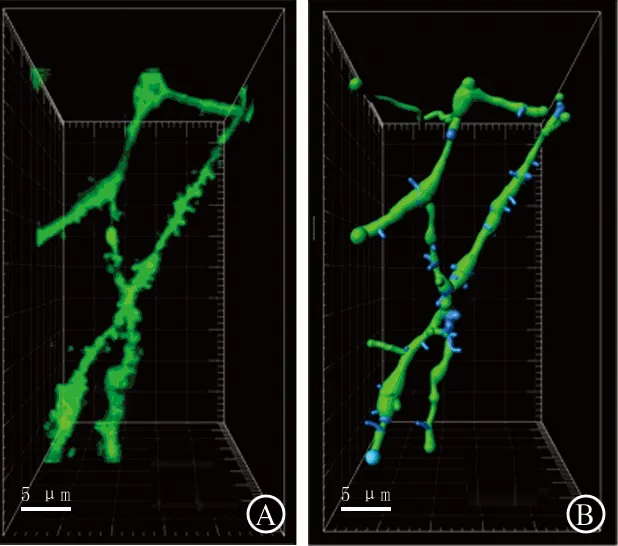
A:树突的三维重建图像;B:对A图追踪和重塑所得树突棘3D图像
图5 对距胞体100 μm的一段神经元树突经膜片钳记录和标记后的三维重建分析图
2.3标记细胞三维纹状体MSNs树突棘形态学特点 选取距胞体100 μm的一段树突进行三维重建,可见大量树突棘围绕树突段分布(图5A),在此基础上对树突棘进行标记和重塑(图5B)。所标记的树突棘计数为32个,平均突触棘长度(2.37±0.14)μm,平均宽度(1.39±0.14)μm。
3讨论
中枢神经系统包括多种类型神经元,如纹状体神经元除MSNs外还包括高频放电中间神经元、低阈值中间神经元和胆碱能中间神经元[5]。这些中间神经元和MSNs相互作用,形成复杂的神经网络,参与了纹状体及整个基底神经节的功能调节。因此研究者常常面临如何精确选择神经元的问题。本实验中,作者利用转基因技术将荧光蛋白与特定受体相结合,使表达特定受体的神经元发出特定颜色荧光,起到精确选择神经元类型的目的。
本研究中作者联合应用BAC转基因技术、全细胞膜片钳技术、免疫荧光技术以及图像三维重建和分析技术,对D1 MSNs分别进行了电生理学记录、细胞标记以及突触形态特点的研究。结果表明,实验中标记成功的细胞具有:(1)偏超级化的静息膜电位;(2)膜电位超极化时较小的输入电阻;(3)较长的动作电位潜伏期。以上特点完全符合MSN典型电生理学特点[5],表明本研究中所用方法可以在精确选择神经元类型的同时获得特定神经元电生理学指标以及形态学特性,从而有助于对神经元的结构和功能进行同步研究。
神经元的结构和功能是相互作用,相互影响的。例如树突的形态对传入的突触后神经元信息传递有很大的影响[6]。树突棘的减少可以在电生理上表现为微小突触后电流频率和幅度的降低[7],并与多种精神疾病明显相关[8]。因此,同时获得神经元的电生理学数据和形态学特征有助于了解神经元在整个神经环路中的作用,对认识疾病的病理机制具有重要的参考价值。以往研究者经常先记录电生理学特性,随后再通过免疫组化方法研究神经元形态,不仅耗费时间较长,而且无法将单个神经元的形态学数据与电生理学数据准确联系起来,因此获得的结果有较大的局限性。本实验中所用方法不仅可以同时研究确定类型的单个的神经元功能与结构,而且简便易行,节省时间,因此较以往方法有明显的进步。
此外,进一步利用图像三维重建和分析技术,可以对某类神经元在所在神经环路中的突触传递特征、树突形态对突触传递的影响,以及在疾病情况下上述指标的变化情况进行研究,使实验中获得的神经元结构信息更多、更立体[9],为探寻不同类型神经元的结构和功能相互关系提供丰富资料。
综上可见,通过基因修饰技术选择标记特定类型细胞,进而对这种类型细胞进行电生理学和形态学记录与分析,将会为特定类型神经元的生理和病理功能提供更为深入的认识。
参考文献:
[1]Gerfen CR, Engber TM, Mahan LC, et al. D1 and D2 dopamine receptor-regulated gene expression of striatonigral and striatopallidal neurons[J]. Science, 1990, 250(4986): 1429-1432.
[2]Gong S, Zheng C, Doughty ML, et al. A gene expression atlas of the central nervous system based on bacterial artificial chromosomes[J]. Nature, 2003, 425(6961): 917-925.
[3]Shuen JA, Chen M, Gloss B, et al. Drd1a-tdTomato BAC transgenic mice for simultaneous visualization of medium spiny neurons in the direct and indirect pathways of the basal ganglia[J]. J Neurosci, 2008, 28(11): 2681-2685.
[4]娄雪林, 周专,康华光. 单通道和全细胞记录技术[J]. 中国医疗器械杂志, 2000, (04): 221-226+205.
[5]Kreitzer AC. Physiology and pharmacology of striatal neurons[J]. Annu Rev Neurosci, 2009,32:127-147.
[6]Zippo AG, Biella GE. Quantifying the number of discriminable coincident dendritic input patterns through dendritic tree morphology[J]. Sci Rep, 2015, 5: 11543.
[7]Peca J, Feliciano C, Ting JT, et al. Shank3 mutant mice display autistic-like behaviours and striatal dysfunction[J]. Nature, 2011, 472(7344): 437-442.
[8]Penzes P, Cahill ME, Jones KA, et al. Dendritic spine pathology in neuropsychiatric disorders[J]. Nat Neurosci, 2011,14(3):285-293.
[9]Mancuso JJ, Cheng J, Yin Z, et al. Integration of multiscale dendritic spine structure and function data into systems biology models[J]. Front Neuroanat, 2014,8:130.
[1]Tao C, Zhang G, Xiong Y, et al. Functional dissection of synaptic circuits: in vivo patch-clamp recording in neuroscience[J]. Front Neural Circuits, 2015, 9: 23.
(本文编辑:邹晨双)
Association establishment between structure and function of fluorescent positive neurons in BAC mice——patch clamp combining confocal 3D imaging reconstruction
ZHAOBo,HEJiahou,ZHUJunling,WANGJulei,LIZhuyi,WANGWenting*.
*DepartmentofNeurobiologyandCollaborativeInnovationCenterforBrainScience,SchoolofBasicMedicine,FourthMilitaryMedicalUniversity,Xi’anShaanxi710032,China
ABSTRACT:ObjectiveTo build the relevance between structure and function of neurons at the cellular level. MethodsThe striatum slices were made with D1 Td Tomato bacterial artifical chromosome(BAC) transgenic mice.The D1 Td Tomato positive medial spiny neurons (MSN) were recorded by patch clamping and filled with neurobiotin during patch. Then the 3D image of the dendritic tree and spine were observed by confocal microscopy and Imaris software. Results(1) The electrophysiological data of the marked cell showed hyperpolarized resting membrane potential(-87 mV),lower input resistance(16.4 MΩ) and longer action potential latency(172 ms).(2) The 3Dreconstruction image also showed classical structure of MSNs. The cell body had cone shape and the dendritic spine tree radiated to spread around. (3) The further analysis also provided the average length of dendrites was(2.37±0.14)μm.The average width of dendritic spine was (1.39±0.14)μm.ConclusionsThis method can offer a way to study the function and morphology of specific neuron type and help us to understand the role of these neurons in physiological and pathophysiological conditions.
Key words:neuron; bacterial artifical chromosome transgenic technology; patch clamp; cell marker; 3D reconstruction
doi:10.3969/j.issn.1006-2963.2016.02.005
基金项目:国家自然科学基金面上项目资助(81371498, 81471342)
通讯作者:王文挺,Email,wwt0657@fmmu.edu.cn
中图分类号:Q421
文献标识码:A
文章编号:1006-2963 (2016)02-0100-05
Corresponding author:WANG Wenting,Email:wwt657@fmmu.edu.cn
(收稿日期:2015-08-31)
