篦子三尖杉牻牛儿基牻牛儿基焦磷酸合成酶基因的克隆与序列分析
2016-06-14齐小琼胡晨熙李大卫
齐小琼+胡晨熙+李大卫

摘要:三尖杉是我国特有植物,主要含有生物碱、黄酮及萜类等生物活性物质。以篦子三尖杉为试验材料,根据同源克隆的方法扩增三尖杉牻牛儿基牻牛儿基焦磷酸合酶(GGPPS)基因,并通过生物信息的方法对该基因进行鉴定和分析。结果表明,篦子三尖杉GGPPS基因全长1 217 bp,含有1个完整的1 182 bp开放阅读框(ORF),编码393个氨基酸的蛋白序列,GGPPS蛋白相对分子量为43.042 ku,理论等电点(pI)为6.57,其二级结构主要由α-螺旋和无规则卷曲组成;蛋白质同源分析表明,三尖杉GGPPS含有多聚异戊二烯基合成酶家族中保守的5个特征性结构和2个富含天冬氨酸的区域;同源模建分析显示,三尖杉GGPPS具有GGPPS蛋白典型的三维结构,特别与薄荷GGPPS蛋白的三维结构及活性位点极其相似;系统进化分析表明,三尖杉GGPPS蛋白归属植物进化支,且与加拿大红豆杉、曼地亚红豆杉、海南粗榧的GGPPS蛋白归为同一分支。
关键词:篦子三尖杉;萜类化合物;牻牛儿基牻牛儿基焦磷酸合酶(GGPPS);克隆;序列分析;同源模建;系统进化
中图分类号: S188;Q785
文献标志码: A
文章编号:1002-1302(2016)04-0079-04
萜类化合物是植物代谢中数量最多的一类代谢物,以异戊二烯为结构单元,在植物的光合作用、呼吸作用、生长、发育、繁殖、信号转导和防御中发挥重要作用。植物中存在的某些萜类具有重要的经济价值和药用价值,如红豆杉(Taxus)中的二萜紫杉醇[1]、银杏(Ginkgo biloba)中的二萜银杏内酯和倍半萜白果内酯[2-3]、青蒿(Artemisia annua)中的倍半萜青蒿素[4]等。三尖杉属(Cephalotaxus)植物主要含有生物碱、黄酮类化学成分,其中的三尖杉酯类生物碱由于具有明显的抗癌活性得到了深入研究,关于黄酮类成分的研究较少。除此之外还含有一些萜类化合物,如二萜类的海松酸、柳杉酚等,初步研究表明,该类化合物对微生物有抑制作用[5]。牻牛儿基牻牛儿基焦磷酸(geranylgeranyl diphosphate,GGPP)是包括紫杉醇、银杏内酯、海松酸、柳杉酚在内的所有二萜类化合物的共同前体,由牻牛儿基牻牛儿基焦磷酸合成酶(geranylgeranyl pyrophosphate synthase,GGPPS)催化,通过类异戊二烯合成途径生成。在该反应途径中,GGPPS催化15 C的法呢基焦磷酸(farnesylpyrophosphate,FPP)与5 C的异戊烯焦磷酸(isopentenylallyl diphosphate,IPP)缩合生成GGPP[6-7]。因此,GGPPS是红豆杉植物合成紫杉醇、银杏合成银杏内酯、三尖杉属植物合成海松酸和柳杉酚的关键酶之一。在三尖杉中克隆该基因并分析其序列的结构特征和进化模式,将为后续的功能研究和基因工程、代谢工程等技术手段大量获得有用产物奠定基础。
1 材料与方法
1.1 试验材料
1.1.1 植物材料 篦子三尖杉(Cephalotaxus oliveri)摘采于中国科学院武汉植物园,取其嫩叶立即置于液氮中保存备用。
1.1.2 主要分子生物学试剂 普通Taq DNA聚合酶、高保真 LA Taq 酶、dNTP等试剂,购自TaKaRa;DNA凝胶纯化回收试剂盒,购于Axygen生物技术有限公司。
1.2 试验方法
1.2.1 篦子三尖杉GGPPS基因的克隆
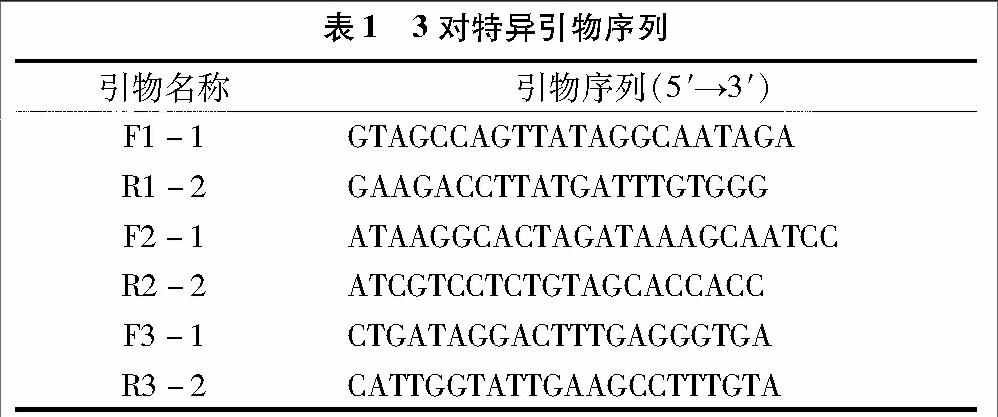
1.2.1.1 引物的设计 用加拿大红豆杉 (Taxus canadensis) GGPPS蛋白(登录号:AADl6018)作为查询序列,进行Blastp相似性搜索,在三尖杉属植物海南粗榧(C. hainanensis)中得到1条同目标序列相似性较高的序列 (登录号:GBHQ10039.1)。根据这条序列设计3对特异引物(分别位于中部和左右端部),详见表1。
1.2.1.2 篦子三尖杉GGPPS基因的分段扩增 以提取的篦子三尖杉总DNA为模板,分别以F1-1、R1-2,F2-1、R2-2,F3-1、R3-2为引物进行PCR扩增。扩增体系为25 μL,包含2.5 μL 10×PCR buffer(Mg2+ plus),2 μL dNTP混合物(每种2.5 mmol/L),各1 μL引物(10 μmol/L),0.5 μL Ex-Taq(2 U/μL),1 μL DNA模板(25 ng/μL),补加17 μL的水。反应条件为:94 ℃ 5 min;94 ℃ 1 min,53 ℃ 1 min,72 ℃ 1 min,36个循环;72 ℃延伸8 min。
PCR结束后,进行DNA琼脂糖凝胶电泳,对扩增的DNA进行检测。
1.2.2 产物回收、测序 扩增片段的凝胶回收按照试剂盒说明书进行操作,将回收的目的片段送华大基因测序。
1.3 数据分析方法
采用BioEdit(North Carolina State University) 软件对克隆测序得到的片段进行拼接。采用ORF finder进行开放阅读框确定。运用DNAstar软件推测氨基酸序列,运用分析蛋白质物理化学的Expasy Protparam软件(http://web.expasy.org/protparam)分析蛋白质的分子量、理论等电点、氨基酸组成。查询Prosite数据库查找蛋白质含有的序列模式和功能域,使用在线软件SOPMA程序(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)预测蛋白质二级结构并计算各种二级结构所占百分比。使用SWISS-MODEL服务器[8-10] (http://swissmodel.expasy.org/),基于同源建模的原理,进行篦子三尖杉GGPPS三级结构的预测,预测结果用PyMOL软件展示和编辑。
以篦子三尖杉GGPPS蛋白作为查询序列,运用Blast工具(http//blast.ncbi.nlm.nih.gov/Blast.cgi)进行序列相似性搜索,并选取相似性较高的序列用MEGA 6软件[11]中的邻接算法进行进化树的构建,其中多重序列比对使用Clustalx软件进行。
2 结果与分析
2.1 篦子三尖杉GGPPS序列的获得与分析
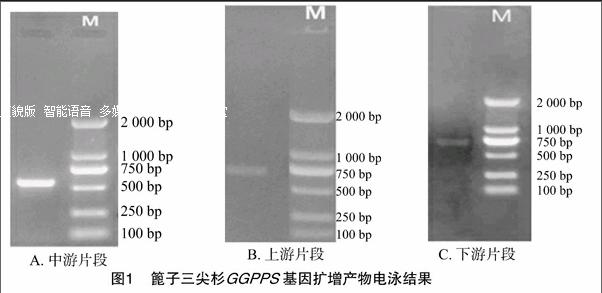
采用改良的CTAB 法获取篦子三尖杉总DNA,并通过电泳进行纯度检测,结果见图1。用引物F2-1、R2-2进行PCR扩增,最终获得560 bp左右的三尖杉GGPPS的中间片段(图1-A)。分别以引物F1-1、R1-2,F3-1、R3-2进行PCR扩增,最终分别获得 700、750 bp左右的上、下游片段(图1-B、1-C)。产物经回收纯化和测序,用BioEditor软件进行拼接,获得1条总长为1 217 bp的GGPPS DNA序列。用ORF Finder软件预测开放阅读框(ORF),结果显示:该DNA序列包含1段1 182 bp的完整编码区,编码393个氨基酸。将获得的基因组DNA序列与已报道的海南粗榧mRNA进行比对发现,篦子三尖杉GGPPS基因不含有内含子,与大多数植物一样,在外显子-内含子的进化上是保守的。
2.2 篦子三尖杉GGPPS蛋白质序列结构特征分析
篦子三尖杉GGPPS基因完整的开放阅读框(ORF)长为1 182 bp,编码393个氨基酸(图2),相对分子质量约为 43.042 ku,理论等电点为6.57。在氨基酸序列中,含量最高的为丙氨酸(Ala,39个)、亮氨酸(Leu,39个),均占9.92%;其次是赖氨酸(Lys,33个,占8.40%)、天门冬氨酸(Asp,29个,占7.28%)、苏氨酸(Thr,27个,占6.87%)、缬氨酸(Val,27个,6.87%)。酸性氨基酸[天门冬氨酸(Asp)、谷氨酸(Glu)]总数52个,碱性氨基酸[精氨酸(Arg)、赖氨酸(Lys)]50个;总亲水性平均系数为-0.147。Prosite数据库比对发现,三尖杉GGPPS蛋白序列的177~193氨基酸位(氨基酸序列:LIhDDlpcmDnddfRRG)和311~323氨基酸位(氨基酸序列:VGllFQVvDDIlD)分别是2个多聚异戊二烯基合成酶的特异序列(图3)。
SOPMA分析表明,α-螺旋(alpha helix,Hh)、无规则卷曲(random coil,Cc)是三尖杉GGPPS蛋白质二级结构的主要成分,组成α-螺旋、无规则卷曲、延伸链氨基酸比例分别为45.80%、41.48%、12.72%,不含其他结构。蛋白质三级结构是在二级结构的基础上进一步盘绕、折叠形成的,决定了蛋白质的功能。在SWISS-MODEL服务器上预测篦子三尖杉GGPPS蛋白的三维结构,结果显示,与薄荷(Menthax piperita)GGPPS蛋白同源性最高(PDB ID:3krp.2.A),为68.37%。以此结构为模型构建篦子三尖杉的三级结构,发现该蛋白具有植物GGPPS蛋白典型的三维结构,说明功能也非常类似,该蛋白在进化上很保守 (图4)。
2.3 GGPPS蛋白质同源性分析与系统进化树构建
Blast比对结果显示,三尖杉GGPPS蛋白质序列与已报道的海南粗榧(C. hainanensis)、加拿大红豆杉(T. Canadensis)、曼地亚红豆杉(Taxus xmedia)、挪威云杉(Picea abies)、银杏(Ginkgo biloba)、烟草(Nicotiana tabacum)、北美冷杉(Abies grandis)、马尾松(Pinus massoniana)、橡胶树(Hevea brasiliensis)、巴豆(Croton sublyratus)、大豆(Glycine max)、美花烟草(Nicotiana sylvestris)等12种高等植物GGPPS蛋白质的一致性分别为98.9%、89.5%、88.8%、70.4%、70.1%、54.3%、69.4%、67.6%、54.0%、54.5%、54.4%、54.5%。多序列同源比对分析结果表明,不同植物来源的GGPPS蛋白质的N末端差异很大,C末端保守性强。GGPPS蛋白在结构和功能上属于多聚异戊二烯合成酶家族,该家族具有5个保守功能域(Ⅰ~Ⅴ),5个保守区域中包含2个富含天冬氨酸区域:DDXXXXD、DDXXD。在篦子三尖杉GGPPS氨基酸序列中存在这样的5个保守结构域,结构域Ⅱ、结构域Ⅴ中也分别发现富含天冬氨酸的基序DDLPCMD、DDILD(图5)。
采用Neighbor-joining算法,在MEGA 6软件平台上构建植物GGPPS的分子系统进化树(图6)。对比植物GGPPS分子系统进化树,三尖杉GGPPS蛋白归属于裸子植物进化支,且与海南粗榧、加拿大红豆杉、挪威云杉、北美冷杉、马尾松的GGPPS蛋白归为同一小分支(图6)。被子植物、裸子植物GGPPS各形成独立的分支,两者形成姊妹关系,说明这两大分支从同一祖先平行分化而来;该基因树与物种树基本吻合,说明该基因的进化与物种的分化基本一致。
3 结论
通过PCR技术克隆获得的篦子三尖杉GGPPS DNA序列长度为1 217 bp,含有1 182 bp的完整开放阅读框。三维结构模拟表明:该蛋白具有GGPPS蛋白典型的三维结构;多重序列比对显示,三尖杉GGPPS 蛋白与其他物种来源的GGPPS蛋白相似性较高,尤其是与海南粗榧、加拿大红豆杉、曼地亚红豆杉的GGPPS相似性高达88%以上,它们都包含多聚异戊二烯合成酶家族的5个保守性结构域(图5)。在三尖杉GGPPS蛋白的氨基酸序列的177~193、311~323位分别是2个多聚异戊二烯基合成酶的特异序列LIhDDlpcmDnddfRRG、VGllFQVvDDIlD(图3),这2个保守的富含天冬氨酸的区域位于酶的催化活性位点,与IPP、烯丙基底物的结合有关。从结构到功能预测都显示,克隆的基因具有典型的GGPPS特征,因此克隆的篦子三尖杉GGPPS编码蛋白很可能参与或调节了三尖杉二萜类等物质的生物合成,但完全阐明三尖杉萜类化合物的生物合成和调控机制还需要进行进一步试验验证。
参考文献:
[1]占爱瑶,由香玲,詹亚光. 植物萜类化合物的生物合成及应用[J]. 生物技术通讯,2010,21(1):131-135.
[2]朱 俊,许 锋,廖咏玲. 银杏萜内酯调控研究进展[J]. 中国农学通报,2007,23(7):301-305.
[3]张洪娟,谭碧玥,曹福亮. 银杏GbGGPS基因的克隆及序列分析[J]. 南京林业大学学报:自然科学版,2013,37(4):8-12.
[4]陈大华,叶和春,李国凤,等. 植物类异戊二烯代谢途径的分子生物学研究进展[J]. 植物学报,2000,42(6):551-558.
[5]Politi M,Braca A,de Tommasi N,et al. Antimicrobial diterpenes from the seeds of Cephalotaxus harringtonia var. drupacea[J]. Planta Medica,2003,69(5):468-470.
[6]Lange B M,Rujan T,Martin W,et al. Isoprenoid biosynthesis:the evolution of two ancient and distinct pathways across genomes[J]. Proceedings of the National Academy of Sciences of the United States of America,2000,97(24):13172-13177.
[7]Rodríguez-Concepción M,Boronat A. Elucidation of the methylerythritol phosphate pathway for isoprenoid biosynthesis in bacteria and plastids. A metabolic milestone achieved through genomics[J]. Plant Physiology,2002,130(3):1079-1089.
[8]Biasini M,Bienert S,Waterhouse A,et al. SWISS-MODEL:modelling protein tertiary and quaternary structure using evolutionary information[J]. Nucleic Acids Research,2014,42(W1):W252-W258.
[9]Arnold K,Bordoli L,Kopp J,et al. The SWISS-MODEL workspace:a web-based environment for protein structure homology modelling[J]. Bioinformatics,2006,22(2):195-201.
[10]Benkert P,Biasini M,Schwede T. Toward the estimation of the absolute quality of individual protein structure models[J]. Bioinformatics,2011,27(3):343-350.
[11]Tamura K,Stecher G,Peterson D,et al. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution,2013,30(12):2725-2729.赵金艳,卢 宏,张振武,等. 快速PCR介导的NeuroD-3′UTR的定点突变研究[J]. 江苏农业科学,2016,44(4):83-86.
