基于psbA基因的噬藻体遗传多样性研究进展
2016-06-07刘婷婷夏雪山
李 樾,刘婷婷,刘 丽,夏雪山
(昆明理工大学生命科学与技术学院,云南 昆明 650500)
基于psbA基因的噬藻体遗传多样性研究进展
李樾,刘婷婷,刘丽①,夏雪山
(昆明理工大学生命科学与技术学院,云南 昆明650500)
摘要:噬藻体作为一类感染蓝藻的特异性病毒,广泛分布于不同水生态系统中。随着水体富营养化的加剧,蓝藻水华频繁爆发,具有潜在控藻能力的生物因子——噬藻体的研究备受关注。目前对于噬藻体的研究主要集中在其分离纯化及生物学特性等,因缺乏广泛通用的分子标记致使其遗传多样性研究受限。以编码光合作用反应中心D1蛋白的psbA基因为靶基因,综述了海洋、淡水环境中噬藻体psbA基因遗传多样性的最新研究进展,分析了噬藻体在不同自然环境中的分布及差异,同时也提出了噬藻体多样性研究中存在的一些问题。
关键词:噬藻体;psbA基因;遗传多样性
蓝藻又名蓝细菌(cyanobacteria),是一类能够进行光合作用的自养型原核生物[1]。噬藻体(cyanophage) 是一类能够专一性感染蓝藻的双链DNA病毒,是水体中最为丰富的一类浮游病毒,广泛分布于不同水生环境中。国际病毒学分类委员会(ICTV)细菌病毒分类会参照噬菌体的分类方式,根据噬藻体形态不同将其分为3个科[2]:肌病毒科(Myoviridae)有一个中央管和一条具有伸缩能力的尾巴;长尾病毒科(Styloviridae)具有一条长的、不能收缩的尾巴;短尾病毒科(Podoviridae)含有一条短尾。作为水生态系统中的重要组成因子,噬藻体与宿主蓝藻的新陈代谢和生命循环有密切的联系,噬藻体通过裂解宿主影响水中微生物及浮游植物群落结构和多样性,对调节蓝藻种群丰度、多样性及群落结构演替、介导基因水平转移等起着重要作用。此外,噬藻体是一类具有种属特异性的蓝藻致死因子,在防治蓝藻水华上潜力巨大,被认为是一种具有潜在控藻能力的生物因子而引起广泛关注。对于噬藻体的研究,目前多集中在噬藻体的分离纯化及其生理生化等生物学特性上,其遗传多样性的研究较少,主要原因在于不同形态的噬藻体之间差异性很大,目前尚未发现其基因中存在类似原核生物16S rDNA和真核生物18S rDNA的共有保守序列,因而缺乏广泛通用的遗传标记基因用于遗传多样性研究[3]。
随着分子生物学技术的发展,噬藻体的分离纯化及全基因组测序研究发现部分种的噬藻体中存在个别的共有保守基因,如G20基因、DNA聚合酶基因、G91基因、光合作用基因psbA等,针对保守基因序列设计简并性引物,采用PCR技术可直接扩增出不同噬藻体基因序列用于遗传多样性分析。笔者以能够编码光合反应中心D1蛋白的psbA基因为遗传标记基因,结合近年来有关海洋、淡水和稻田中噬藻体psbA基因遗传多样性的最新研究进展,分析噬藻体的遗传多样性,了解噬藻体及其与宿主蓝藻的关系,从而促进其对蓝藻水华的治理。
1噬藻体遗传多样性的分子标记
自然界中,某一物种的遗传多样性具有显著生态功能,遗传多样性越丰富,其对环境的适应能力和对新环境的拓展能力也越强,在自然环境中的分布范围也越广,而且某一物质遗传变异的大小与其进化速率呈正相关[4]。研究噬藻体的遗传多样性有助于分析其进化历程,完善噬藻体的分类,进一步深入探讨噬藻体间的进化关系、噬藻体与蓝藻间的相互作用关系,通过测试噬藻体基因组的大小、序列和结构,获得有关噬藻体遗传多样性的更多知识[5]。然而,噬藻体是高度差异性的病毒类群,缺乏广泛通用的分子标记。目前,噬藻体遗传多样性常用的靶基因主要有G20基因、DNA聚合酶基因、G91基因和psbA光合作用基因等。
G20基因是编码噬藻体衣壳组装蛋白的基因,与T4噬菌体编码衣壳蛋白的基因同源性很高。来源不同的噬藻体S-PM2、S-BnM1和S-WHM1中均有一段具有高度保守性的序列(G20),可用作噬藻体遗传多样性研究的分子标记物。到目前为止,依据G20基因保守序列进行PCR扩增的引物有很多,如:CPS1/CPS2[6]、CPS1/CPS8[7]、CPS1/CPS4[8]、CPS1.1/CPS8.1[9]和CPS4/G20-2[10]等。但使用G20基因作为噬藻体遗传多样性的标记基因存在一定局限性,一方面,侵染蓝藻的噬藻体广泛分布在肌尾病毒科、短尾病毒科和长尾病毒科中,而G20基因只存在于肌尾科噬藻体中;再者,G20基因与T4噬菌体编码衣壳蛋白的基因高度同源,在PCR扩增中可能出现假阳性导致最终分析结果有误。因此,G20基因主要用于肌尾科噬藻体遗传多样性研究。
G91基因是侵染铜绿微囊藻噬藻体编码衣壳尾鞘蛋白的基因,TAKASHIMA等[11]分离纯化了3株感染铜绿微囊藻的噬藻体,并与噬藻体Ma-LMM01的序列对比发现了G91基因,为噬藻体的多样性研究拓增了新的方法。随后YOSHIDA等[12]利用G91基因保守序列设计PCR引物,对日本Mikata湖进行研究,结果发现铜绿微囊藻丰度降低而噬藻体丰度增加,并且噬藻体中G91基因只能感染部分铜绿微囊藻群体。G91作为遗传分子标记物主要用于铜绿微囊藻噬藻体的研究。
DNA聚合酶(DNA polymerase)是细胞复制DNA的作用酶。DNA聚合酶的主要活性是催化DNA的合成。在噬藻体中DNA聚合酶基因主要存在于短尾病毒科中,短尾病毒科噬藻体具有严格宿主专一性和能快速裂解宿主的能力。2009年,CHEN等[13]对比已知的6种海洋短尾科噬藻体的基因序列,发现这些噬藻体中的DNA聚合酶基因具有一定的保守性,并根据该基因设计出引物,对美国切萨皮克海湾的表层、中层及底层水域中的短尾科噬藻体进行持续2 a夏、冬2季的研究。结果发现,切萨皮克海湾短尾科噬藻体具有丰富的遗传多样性,也具有明显的季节性变化,其夏季噬藻体的结构组成与冬季有所不同。由于DNA聚合酶主要存在于短尾科噬藻体中,因此将DNA聚合酶作为噬藻体多样性研究的分子标记物同样存在缺陷。
噬藻体辅助代谢基因(AMGs)是编码噬藻体的一类特殊功能基因。尽管AMGs基因普遍存在于噬藻体中,但是不同种类的噬藻体所携带的AMGs类型具有很大差异。因此将AMGs作为噬藻体分子遗传标记,对不同水域的噬藻体遗传多样性的认识还存在一些不确定性[14]。
蓝藻进行光合作用需要2个光反应系统(PSⅠ和PSⅡ),目前已发现在噬藻体基因组中存在一系列与宿主蓝藻同源的光合作用基因序列,包括psbA和psbD、petE和F以及hli等[15]。光合作用基因在噬藻体中的广泛存在,是基于噬藻体与宿主蓝藻基因片段之间、噬藻体基因片段之间的基因水平转移和交换重组[16]。psbA和psbD是编码光合系统Ⅱ反应中心蛋白主要成分D1和D2蛋白亚基的基因[17]。petE、F基因则分别编码光合电子传递的质体蓝素蛋白和铁氧化还原蛋白;hli基因编码高光诱导蛋白HLIPs。一些基因编码的功能性蛋白,对噬藻体侵染及增殖过程具有重要作用,噬藻体携带光合作用基因能够为宿主及自身提供光转化的化学能,从而增加噬藻体在蓝藻中的适应性。LINDELL等[17]研究发现,噬藻体在海洋中广泛存在,噬藻体在感染海洋蓝藻时其携带的psbA基因编码的蛋白与宿主蛋白功能相同,对光能转化为化学能有重要意义。BAILEY等[18]认为噬藻体具有保护宿主避免光抑制的作用,从而使其在侵染宿主蓝藻时确保增殖过程中能量的供应。另外,光合作用基因可使噬藻体在增殖裂解周期内基因组产量增加2.5%~4.5%,从而提高噬藻体的裂解量[19]。噬藻体由于携带有psbA、psbD光合作用基因,可以扩大其宿主范围,噬藻体基因组中是否携带psbA基因与其侵染宿主蓝藻的时间长短有关。psbA基因是光合作用系统中重要基因,广泛存在于肌尾病毒科、短尾病毒科的噬藻体中。
2噬藻体遗传多样性靶标基因psbA
psbA是编码光合系统Ⅱ反应中心(PSⅡ)D1蛋白的基因,广泛存在于藻类及绿色植物中。许多噬藻体中都包含编码光合系统Ⅱ中D1 蛋白的psbA基因,88%的感染海洋聚球藻和原绿球藻的噬藻体都含有psbA基因。全球海洋表层水中60%的psbA基因来源于病毒[20]。psbA是噬藻体中非常保守的功能基因,利用psbA基因的保守序列设计简并引物,通过PCR扩增噬藻体的基因序列并进行系统进化分析,可用于研究噬藻体的遗传多样性[21]。
根据噬藻体psbA基因扩增的引物较多。表1详细列举出历年来有关psbA的引物,不同引物扩增出的基因片段大小有一定差异。2003年,ZEIDNER等[22]首次根据噬藻体psbA基因的保守序列设计出引物58-VDIDGIREP-66/331-MHERNAHNFP-340。MILLARD等[23]根据实验需求对噬藻体S-PM2越过一个内含子并忽略一个外显子,设计引物psbA-F/psbA-R可扩增800 bp。后来,MILLARD等[24]将引物继续改进后可扩增片段长达1 080~1 291 bp。SULLIVAN等[16]根据噬藻体P-SSM2、P-SSM4、P-SSP7、S-PM2、S-WHM1、 Syn5和Syn9的psbA基因保守序列设计引物Pro-psbA-1F/Pro-psbA-1R可扩增680 bp,确定了psbA基因既存在于噬藻体中,也存在于蓝藻中,为蓝藻与噬藻体光合作用基因的转移研究奠定基础。WANG等[25]根据噬藻体psbA基因的保守序列扩增出引物psbA-93F/psbA-341R。笔者根据噬藻体psbA基因设计了巢式PCR引物psbA(1)-F/psbA(1)-R和psbA(2)-F/psbA(2)-R,对高原富营养湖泊滇池噬藻体多样性进行研究。
表1研究噬藻体分子标记psbA基因的PCR引物
Table 1PCR primers ofpsbAgene as a molecular marker for study of cyanophage diversity
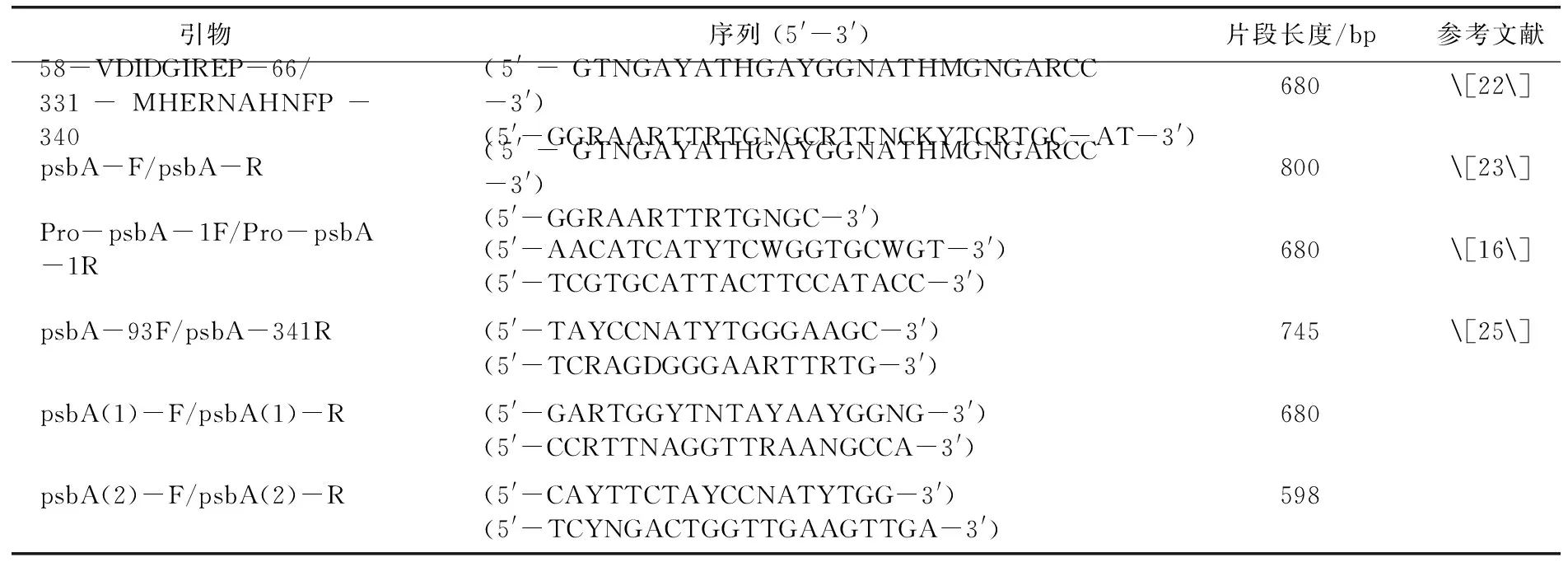
引物序列(5'-3')片段长度/bp参考文献58-VDIDGIREP-66/331-MHERNAHNFP-340(5'-GTNGAYATHGAYGGNATHMGNGARCC-3')(5'-GGRAARTTRTGNGCRTTNCKYTCRTGC-AT-3')680[22]psbA-F/psbA-R(5'-GTNGAYATHGAYGGNATHMGNGARCC-3')(5'-GGRAARTTRTGNGC-3')800[23]Pro-psbA-1F/Pro-psbA-1R(5'-AACATCATYTCWGGTGCWGT-3')(5'-TCGTGCATTACTTCCATACC-3')680[16]psbA-93F/psbA-341R(5'-TAYCCNATYTGGGAAGC-3')(5'-TCRAGDGGGAARTTRTG-3')745[25]psbA(1)-F/psbA(1)-R(5'-GARTGGYTNTAYAAYGGNG-3')(5'-CCRTTNAGGTTRAANGCCA-3')680psbA(2)-F/psbA(2)-R(5'-CAYTTCTAYCCNATYTGG-3')(5'-TCYNGACTGGTTGAAGTTGA-3')598
由于psbA基因既存在于进行光合作用的蓝藻细胞内,也存在于能够感染蓝藻的噬藻体中。从环境水样中利用PCR特异性扩增所得的psbA基因序列,其是否为噬藻体来源有必要进行探讨区分,即难点在于区分环境样品中获得的psbA基因是病毒来源还是宿主来源。CHE′NARD 等[26]提出,采集的水样先分别经 0.45和0.22 μm孔径滤膜过滤以除去藻类宿主等细胞型生物,滤液通过超滤浓缩,对浓缩液进行psbA基因PCR扩增前,先用脱氧核糖核酸酶(DNase)处理以除去浓缩水样中可能含有的游离蓝藻DNA,并用蓝藻16-23S rDNAITS基因引物进行PCR扩增结果为阴性后,再提取浓缩水样中DNA,以确保所提DNA为病毒DNA;基于噬藻体和宿主的psbA序列的鸟嘌呤和胞嘧啶所占比例(GC含量)分析(如海洋聚球藻噬藻体GC含量为46%~51%,低于其宿主的56%~62%;淡水噬藻体GC含量低于其宿主的46%);基于编码D1 蛋白的氨基酸三联体模型 R/KETTXXXSQ/H分析:SHARON等[20]通过统计并分析全球海洋样品(global ocean sampling,GOS)宏基因组数据库的psbA基因序列,发现psbA基因编码的D1蛋白中普遍存在氨基酸三联体模型R/KETTXXXSQ/H,氨基酸三联体XXX的变化可以反映出不同噬藻体所处的分类地位,其中ENE、EQE、EEE、EEV、EDE、EDV、EQV 以及EVE 都只出现在噬藻体D1蛋白中,而在宿主蓝藻中极少发现。利用以上方法能有效分析环境样品中psbA基因序列的来源。
3自然环境中噬藻体psbA基因的遗传多样性
3.1海洋水域中噬藻体psbA基因的遗传多样性
目前对于海洋中噬藻体psbA基因的研究主要集中在蒙特雷海湾、红海、地中海和热水台、夏威夷海域、墨西哥湾、切萨皮克海湾、挪威海域、北冰洋、太平洋、美国福斯特城海域及我国黄海、东海、渤海等。2003年,ZEIDNER等[22]首次根据噬藻体S-PM2的psbA基因保守序列设计引物58-VDIDGIREP-66/331-MHERNAHNFP-340,探究美国蒙特雷海湾噬藻体,利用所获序列与红海、地中海和热水台等水域的噬藻体psbA基因序列共同构建系统进化树,确定了噬藻体psbA基因的可扩增性和多样性。随后,SULLIVAN等[16]对夏威夷海域噬藻体P-SSM2、P-SSM4、P-SSP7、S-PM2、S-WHM1、Syn5和Syn9等的研究发现引物psbA-F/psbA-R扩增效果欠缺,使用改进后的引物Pro-psbA-1F/Pro-psbA-1R扩增psbA基因系列并进行系统分析,发现噬藻体具有非常丰富的遗传多样性。SANDAA等[27]采用引物psbA-F/psbA-R首次对冷水水域(挪威近岸海域)中噬藻体多样性进行研究,使用脉冲场凝胶电泳对噬藻体光合作用基因进行克隆测序,不仅可分析基因组大小,还可了解病毒的种属问题。通过与红海、地中海、美国蒙特雷湾的噬藻体和聚球藻属进行发育分析,发现psbA基因分布在不同的进化簇中,38%的psbA序列集中在同一进化簇,验证了噬藻体的丰富多样性。BENCH等[28]对切萨皮克海湾水域建立宏基因组文库,采用BLAST分类学方法对水域中psbA同源性序列进行比对,水域噬藻体中psbA基因与噬藻体序列的高比值显示psbA在噬藻体基因组中普遍存在,说明短尾噬藻体和肌尾噬藻体比较丰富;比值较低的psbD基因也表明切萨皮海湾水域中以短尾噬藻体为主。CHE′NARD 等[26]使用引物Pro-psbA-1F/Pro-psbA-1R以psbA为靶基因研究海洋及淡水生态系统中噬藻体的遗传多样性,对北冰洋、太平洋、安大略湖和德国康斯坦斯湖水域的噬藻体psbA基因进行PCR扩增,并与其他海域(夏威夷、红海、地中海、挪威海岸)噬藻体、噬藻体psbA基因、原绿球藻、聚球藻属psbA基因序列共同构建系统进化树,发现安大略湖和德国康斯坦斯湖的淡水噬藻体聚为FW1和FW2这2簇,海洋噬藻体聚为7簇(Mar1~Mar7);源自地中海的噬藻体在每一簇噬藻体中均有分布,感染聚球藻的肌尾与短尾病毒科噬藻体的psbA基因分别聚在Mar1-SM和Mar2-SP这2簇,感染原绿球藻的肌尾与短尾病毒科噬藻体的psbA基因分别聚在Mar6-PM和Mar4-PP这2簇,说明噬藻体具有丰富的遗传多样性。
光合作用基因不仅可用于噬藻体遗传多样性研究,还可用于探索分析噬藻体的进化过程及其与宿主蓝藻的关系。CHE′NARD等[26]在研究噬藻体与蓝藻的相互关系中发现,蓝藻和噬藻体的psbA基因系列属于同一进化簇的不同进化亚簇,表明噬藻体中的光合基因源自其与宿主蓝藻的基因水平交换,但噬藻体光合基因的获得不是近期发生的,其进化历史与蓝藻光合基因的进化历史不同。淡水和海洋生态系统中蓝藻、噬藻体各自聚成不同的进化簇,保持着有限的基因交流,淡水及海洋噬藻体的psbA基因系列分开聚簇,说明海洋和淡水噬藻体及宿主不同的噬藻体间光合作用基因之间的进化历史也存在差异,其进化过程相互独立。MILLARD等[23]使用引物psbA-F/psbA-R对美国福斯特城海域的噬藻体S-PM2进行PCR扩增并进行系统发育分析,发现噬藻体中psbA基因的序列与藻类等的psbA基因序列有很高的相似性。
国内对海洋噬藻体的研究起步较晚。2013年,卢龙飞[29]使用引物Pro-psbA-1F/Pro-psbA-1R对中国北黄海、渤海等海域的噬藻体多样性进行探究,通过系统发育分析发现,所获118条噬藻体psbA基因片段共聚为4簇:北黄海的psbA序列分别存在于4个簇中,而来自青岛近海海域的psbA序列只存在于2个簇中(Cluster Ⅰ和Ⅳ),来自青岛近海域和黄海水域的大部分psbA基因的片段遗传距离较近,可以共同成簇,而黄海的psbA基因遗传多样性更为丰富。2014年,ZHENG等[30]对中国东海海域的原绿球藻和侵染原绿球藻的肌尾病毒科噬藻体进行多样性分析,通过对原绿球藻和侵染原绿球藻病毒的GC含量和第3个密码子的GC含量(GC3)进行对比发现,侵染原绿球藻的噬藻体psbA基因的GC含量高于宿主原绿球藻,且原绿球藻病毒的psbA基因在GC序列重组情况下更不稳定,因此东海海域的噬藻体比原绿球藻有更高的遗传多样性。
3.2淡水水域中噬藻体psbA基因的遗传多样性
与海洋环境相比,淡水水体中噬藻体psbA基因多样性研究相对滞后,主要集中在加拿大的安吉略湖、安纳西湖、布尔热湖、日本稻田、我国的滇池和东北的稻田中等。2010年SHORT等[31]利用噬藻体psbA基因引物psbA-F/psbA-R对加拿大安吉略湖进行初步探究,通过PCR扩增并构建系统进化树分析发现冬季与初春噬藻体丰度较低,侵染藻类主要发生在春夏秋这3个季节。ZHONG等[32]对安纳西湖和布尔热湖中侵染聚球藻属的噬藻体psbA基因的结构域功能进行研究,PCR扩增采用引物Pro-psbA-1F/Pro-psbA-1R,系统发育分析表明高原湖泊明显不同于其他的淡水和海洋水体,是一个具有独特地理因素的多样化病毒群体。
2009年,WANG等[33]采用引物psbA-F/psbA-R对日本稻田水体中噬藻体psbA基因遗传多样性进行分析,使用PCR和D-DGGE技术得到37条不同的psbA基因片段,其基因序列分布在系统进化树的2个进化簇中,且噬藻体在不同环境中的进化地位不同。2014年,荆瑞勇[34]利用引物psbA-F/psbA-R 对东北(建三江、阎家岗和大安)稻田水体中噬藻体psbA基因进行PCR扩增,将得到的17条psbA基因序列与日本稻田水体psbA基因共同建树分为5个进化簇,我国扩增出的psbA基因序列主要分布在Cluster Ⅰ、Ⅲ、Ⅳ和Ⅴ中,研究表明,我国东北稻田与日本稻田水体中噬藻体的psbA基因有很大差异。笔者在对高原富营养化淡水湖泊滇池中噬藻体psbA基因多样性进行研究时发现,与海洋环境比较,滇池噬藻体群落与来自日本稻田水体和其他淡水湖泊的噬藻体psbA基因群集更为相似,淡水与海洋噬藻体psbA基因分布不同,淡水psbA序列间同源性要高于海洋系列,不同环境水域中噬藻体含有其独特的类型,其psbA基因多样性存在一定差异,显示不同自然环境中噬藻体psbA基因组成存在一定差异,psbA基因的分布与其存在的环境有一定的关系。
综合历年来对噬藻体psbA基因的研究,从Genebank下载不同自然环境中噬藻体psbA基因序列,使用MEGA 5.0软件比对确定光合作用基因psbA具有一定的特异性(图1),并构建系统进化树分析psbA基因的分布情况(图2)。噬藻体中光合作用基因psbA在海洋环境中的分布研究较多,而淡水水体中psbA基因分布有别于海洋水体。湖泊与稻田水体中噬藻体同源性很高,同时与宿主为原绿球藻、聚球藻的肌尾噬藻体或短尾噬藻体的同源性较低。海洋噬藻体具有更为丰富的遗传多样性:感染原绿球藻的短尾科噬藻体P-SSP3、P-SSP6与来自于北冰洋、墨西哥湾的海洋噬藻体聚为1簇; 北冰洋、太平洋中海洋噬藻体与宿主为聚球藻、原绿球藻的肌尾科噬藻体聚为1簇;而来自于太平洋东北部的海洋噬藻体则以宿主为聚球藻的肌尾噬藻体为主。
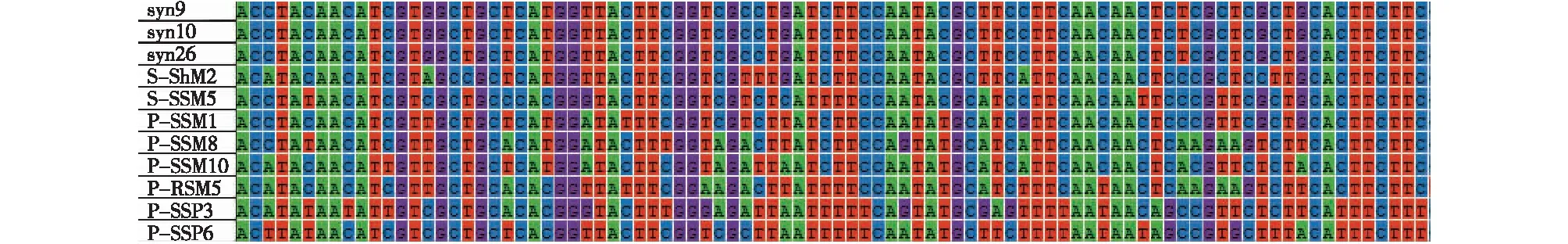
A—腺嘌呤脱氧核糖核苷酸;T—胸腺嘧啶脱氧核糖核苷酸;C—胞嘧啶脱氧核糖核苷酸;G—鸟嘌呤脱氧核糖核苷酸。
4展望
噬藻体是水体中最为丰富的一类浮游病毒,能够专一感染蓝藻,对调节蓝藻种群丰度、多样性及群落结构演替以及介导基因水平转移起着重要作用。噬藻体携带光合作用基因,介导光合作用基因在噬藻体和宿主间转移,由此影响蓝藻光合作用效率,提高蓝藻适应各种环境的能力,可能导致水域生态系统中宿主蓝藻的生态竞争优势。光合作用能力的改变也导致宿主种群生态地位的变化,从而引起整个浮游植物种群的变化。宿主蓝藻的大量繁殖同时为噬藻体提供了丰富的宿主来源以利于其自身在蓝藻细胞内增殖,实现基于病毒-宿主相互间的协同进化关系。这也预示着蓝藻水华的爆发除了受水体所含营养盐水平的影响,同时可能还受因基因转移导致的分子水平上的影响。因此,选择适合的分子标记,开展系统发育分析以及遗传多样性研究无疑具有重要意义。对于噬藻体遗传多样性研究的已有进展,主要是对G20、psbA基因的研究。在研究中发现G20基因存在于自然环境中的肌尾病毒科噬藻体中,而psbA基因也不是一个完美的分子标记物,因为在长尾病毒科噬藻体中没有发现它的存在,并且对于侵染蓝藻的噬藻体(潜伏期小于8 h)也检测不到psbA基因。但是噬藻体psbA基因是噬藻体的分子标记物中引物扩增范围最广的,具有揭示噬藻体的进化史、更好地用于研究噬藻体结构和在海洋和淡水中的遗传多样性的作用。
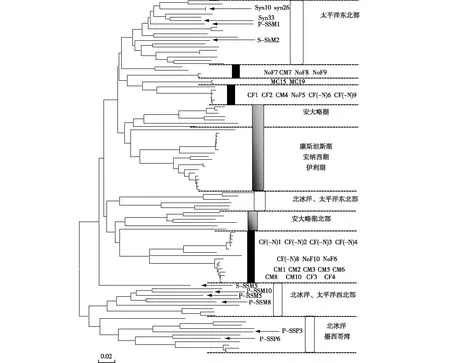
灰色代表淡水中噬藻体,白色代表海洋中噬藻体,黑色代表稻田中淡水噬藻体。
目前对于噬藻体的研究不能完全覆盖肌尾病毒科、短尾病毒科和长尾病毒科,因此迫切需要寻找覆盖面更为全面的靶标基因,或者结合噬藻体的更多分子标记基因,如psbD、petE和petF等进行综合分析。同时,相对于国外来说,我国噬藻体多样性研究起步较晚,仅限于个别海洋、湖泊和稻田水体中等,而对于其他地区噬藻体基因多样性的研究还有待深入。
参考文献:
[1]MATHIAS C B,KIRSCHNER A K T,VELIMIROV B.Seasonal Variations of Virus Abundance and Viral Control of the Bacterial Production in a Backwater System of the Danube River[J].Applied and Environmental Microbiology,1995,61(10):3734-3740.
[2]向安,刘丽,魏大巧,等.噬藻体的分子生物学研究进展[J].生物技术通报,2009(5):31-34,38.
[3]陈章然,郑伟,郑天凌.海洋藻类病毒多样性研究的现状与展望[J].地球科学前沿,2012,2:172-182.
[4]沈浩,刘登义.遗传多样性的概述[J].生物学杂志,2001,18(3):5-8.
[5]张奇亚.噬藻体生物多样性的研究动态[J].微生物学通报,2014,41(3):545-559.
[6]FULLER N G,WILSON W H,JOINT I R,etal.Occurrence of a Sequence in Marine Cyanophages Similar to That of T4G20 and Its Application to PCR-Based Detection and Quantification Techniques[J].Applied and Environmental Microbiology,1998,64(6):2051-2060.
[7]ZHONG Y,CHEN F,WILHELM S W,etal.Phylogenetic Diversity of Marine Cyanophage Isolates and Natural Virus Communities as Revealed by Sequences of Viral Capsid Assembly Protein GeneG20[J].Applied and Environmental Microbiology,2002,68(4):1576-1584.
[8]MARSTON M F,SALLEE J L.Genetic Diversity and Temporal Variation in the Cyanophage Community Infecting Marine Synechococcus Species in Rhode Island′s Coastal Waters[J].Applied and Environmental Microbiology,2003,69(8):4639-4647.
[9]JAMESON E,MANN N H,JOINT I,etal.The Diversity of Cyanomyovirus Populations along a North-South Atlantic Ocean Transect[J].The ISME Journal,2011,5(11):1713-1721.
[10]SHORT C M,SUTTLE C A.Nearly Identical Bacteriophage Structural Gene Sequences Are Widely Distributed in Both Marine and Freshwater Environments[J].Applied and Environmental Microbiology,2005,71(1):480-486.
[11]TAKASHIMA Y,YOSHIDA T,YOSHIDA M,etal.Development and Application of Quantitat Dievteection of Cyanophages Phylogenetically Related to Cyanophage Ma-LMM01 InfectingMicrocystisaeruginosain Fresh Water[J].Microbes and Environments,2007,22(3):207-213.
[12]YOSHIDA M,YOSHIDA T,KASHIMA A,etal.Ecological Dynamics of the Toxic Bloom-Forming CyanobacteriumMicrocystisaeruginosaand Its Cyanophages in Freshwater[J].Applied and Environmental Microbiology,2008,74(10):3269-3273.
[13]CHEN F,WANG K,HUANG S,etal.Diverse and Dynamic Populations of Cyanobacterial Podoviruses in the Chesapeake Bay Unveiled Through DNA Polymerase Gene Sequences[J].Environmental Microbiology,2009,11(11):2884-2892.
[14]高恶斌,宁德刚.噬藻体辅助代谢基因(AMGs)研究进展[J].微生物学通报,2014,41(8):1667-1674.
[15]SHARON I,BATTCHIKOVA N,ARO E M,etal.Comparative Metagenomics of Microbial Traits Within Oceanic Viral Communities[J].The ISME Journal,2011,5(7):1178-1190.
[16]SULLIVAN M B,LINDELL D,LEE J A,etal.Prevalence and Evolution of Core Photosystem Ⅱ Genes in Marine Cyanobacterial Viruses and Their Hosts[J].PLoS biology,2006,4(8):234.
[17]LINDELL D,JAFFE J D,JOHNSON Z I,etal.Photosynthesis Genes in Marine Viruses Yield Proteins During Host Infection[J].Nature,2005,438(7064):86-89.
[18]BAILEY S,CLOKIE M R,MILLARD A,etal.Cyanophage Infection and Photo-Inhibition in Marine Cyanobacteria[J].Research in Microbiology,2004,155(9):720-725.
[19]BRAGG J G,CHISHOLM S W.Modeling the Fitness Consequences of a Cyanophage Encoded Photosynthesis Gene[J].PLoS One,2008,3(10):3550.[20]SHARON I,TZAHOR S,WILLIAMSON S,etal.Viral Photosynthetic Reaction Center Genes and Transcripts in the Marine Environment[J].The ISME Journal,2007,1(6):492-501.
[21]王淼星,向安,魏大巧,等.噬藻体光合作用基因研究[J].生物技术通报,2010(1):14-18.
[22]ZEIDNER G,PRESTON C M,DELONG E F,etal.Molecular Diversity Among Marine Picophytoplankton as Revealed bypsbAAnalyses[J].Environmental Microbiology,2003,5(3):212-216.
[23]MILLARD A D,CLOKIE M R J,SHUB D A,etal.Genetic Organization of thepsbADRegion in Phages Infecting Marine Synechococcus Strains[J].Proceedings of the National Academy of Sciences of the United States of America,2004,101(30):11007-11012.
[24]MILLARD A D,GIERGA G,CLOKIE M R J,etal.An Antisense RNA in a Lytic Cyanophage LinkspsbAto a Gene Encoding a Homing Endonuclease[J].The ISME Journal,2010,4(9):1121-1135.[25]WANG K,CHEN F.Prevalence of Highly Host-Specific Cyanophages in the Estuarine Environment[J].Environmental Microbiology,2008,10(2):300-312.
[26]CHE′NARD C,SUTTLE C A.Phylogenetic Diversity of Sequences of Cyanophage Photosynthetic GenepsbAin Marine and Freshwaters[J].Applied and Environmental Microbiology,2008,74(17):5317-5324.
[27]SANDAA R A,CLOKIE M R J,MANN N H.Photosynthetic Genes in Viral Populations With a Large Genomic Size Range From Norwegian Coastal Waters[J].FEMS Microbiology Ecology,2008,63(1):2-11.
[28]BENCH S R,HANSON T E,WILLIANSON K E,etal.Metagenomic Characterization of Chesapeake Bay Virioplankton[J].Applied and Environmental Microbiology,2007,73(23):7629-7641.
[29]卢龙飞.中国青岛近海、北黄海及渤海噬藻体遗传多样性研究[D].青岛:中国海洋大学,2013.
[30]ZHENG Q,JIAO N,ZHANG R,etal.The Evolutionary Divergence ofpsbAGene in Synechococcus and Their Myoviruses in the East China Sea[J].PLoS ONE,2014,9(1):86644.
[31]SHORT C M,RUSANOVA O,Short S M.Quantification of Virus Genes Provides Evidence For Seed-Bank Populations of Phycodna Viruses in Lake Ontario,Canada[J].The ISME Journal,2011,5(5):810-821.
[32]ZHONG X,JACQUET S.Prevalence of Viral Photosynthetic and Capsid Protein Genes From Cyanophages in Two Large and Deep Perialpine Lakes[J].Applied and Environmental Microbiology,2013,79(23):7169-7178.
[33]WANG G,MURASE J,ASAKAWA S,etal.Novel Cyanophage Photosynthetic GenepsbAin the Floodwater of a Japanese Rice Field[J].FEMS Microbiology Ecology,2009,70(1):79-86.
[34]荆瑞勇.东北稻田水体噬藻体基因多样性与蓝藻及其伴生菌的分离鉴定[D].长春:中国科学院东北地理与农业生态研究所,2014.
(责任编辑: 陈昕)
Progress of Research on Genetic Diversity of Cyanophage Based on psbA Gene.
LI Yue, LIU Ting-ting, LIU Li, XIA Xue-shan
(Faculty of Life Science and Technology, Kunming University of Science and Technology, Kunming 650500, China)
Abstract:Cyanophages, as a kind of peculiar viruses that infect specifically cyanobacteria, exists extensively in various aquatic environments. In water bodies where water eutrophication is severe, cyanobacterial bloom outbreaks frequently. So scientists pay much attention to the study on cyanophages as a potential biological factor in controlling cyanobacteria. Currently studies on cyanophages are focused mainly on how to isolate and purify cyanophages and what its physiological and biochemical properties are. Little has been done on its genetic diversity because no universal genetic markers are available for the study. Using the photosynthesis psbA gene that encodes key photosystem II proteins (D1) of cyanophages as target, recent advances in researches on cyanophage genetic diversity in maritime and fresh water environments were reviewed and distribution and variation of cyanophages in various natural environments analyzed. Meanwhile, some tips for problems in and prospects of the researches in this field are also discussed.
Key words:cyanophage;psbA gene;genetic diversity
收稿日期:2015-08-31
基金项目:国家自然科学基金地区基金(30960005,31360065)
中图分类号:X52;Q93
文献标志码:A
文章编号:1673-4831(2016)03-0417-07
DOI:10.11934/j.issn.1673-4831.2016.03.013
作者简介:李樾(1991—),女,黑龙江铁力人,硕士生,主要从事环境微生物学方面的研究。E-mail: 184682604234@163.com
① 通信作者E-mail: Liuli2272@163.com
