基于线粒体D-loop基因的珠江翘嘴鲌遗传多样性与遗传分化研究*
2016-06-05杨子拓孙际佳李桂峰肖诗斌张鹤千
杨子拓,孙际佳,李桂峰,肖诗斌,张鹤千,
杨慧荣1,2,赵会宏1,2,刘 丽1,2
(1. 华南农业大学动物科学学院,广东 广州 510642;2. 华南农业大学动物科学学院∥广东省农业动物基因组学与分子育种重点实验室,广东 广州510642;3. 中山大学生命科学学院,广东 广州 510275)
基于线粒体D-loop基因的珠江翘嘴鲌遗传多样性与遗传分化研究*
杨子拓1,2,3,孙际佳1,2,李桂峰3,肖诗斌1,2,张鹤千3,
杨慧荣1,2,赵会宏1,2,刘 丽1,2
(1. 华南农业大学动物科学学院,广东 广州 510642;2. 华南农业大学动物科学学院∥广东省农业动物基因组学与分子育种重点实验室,广东 广州510642;3. 中山大学生命科学学院,广东 广州 510275)
为探明分布于珠江流域的野生翘嘴鲌种群遗传多样性与遗传分化状况,实验在珠江流域广东省(肇庆和韶关)、广西省(梧州、昭平、平乐、柳城、桂平、平果、田阳、龙州和扶绥)自然河流内采集野生翘嘴鲌种群。研究采用线粒体分子标记技术(D-loop基因)对珠江流域翘嘴鲌种群的遗传多样性做一个多方面的分析。利用PCR技术,得到D-loop全基因序列856 bp,通过分析发现,176个个体中合计41个单倍型;其单倍型多样性指数(Hd= 0.875 06)和核苷酸多样性指数(π= 0.007 00)显示种群具有较高的遗传多样性。11个野生地理群体翘嘴鲌遗传分化指数(FST)为0.188 28,显示珠江流域翘嘴鲌作为一个大种群已产生了中度分化,种群分化时间经计算推断为5万年前左右。其中,梧州和桂平,梧州和肇庆,昭平和平乐,田阳和平果,扶绥和大部分种群(柳城、桂平、田阳、韶关)的遗传分化程度都较低(FST<0.05),基因流Nm>5;其他种群两两之间达中度遗传分化(FST>0.05),基因流Nm<5。分子系统树和单倍型网络关系显示:单倍型Hap1和Hap2可能为单倍型先祖,其余单倍型由其进化而来。但遗传距离与地理距离并没有显著相关性,种群扩散规律需要进一步探索,说明珠江流域野生翘嘴鲌并未地理因素产生种属分化。
珠江流域;翘嘴鲌;D-loop;遗传多样性;遗传分化
翘嘴鲌Culteralburnus属鲤形目Cypriniformes鲤科Cyprinidae鲌亚科Culterinae鲌属Culter,是鲌亚科中体型较大的鱼类。翘嘴鲌的分布较广、生态作用较大、营养和经济价值较高,除在西部青藏高原少有分布外,广泛分布于我国的各大水系[1],翘嘴鲌的生态价值在于其能捕食水体中的小型鱼类,保持水体生态系统稳定[2]。但是近年来大规模捕捞导致自然种群中的翘嘴鲌资源下降,在1980年对巢湖中的翘嘴鲌种群进行资源调查发现,种群呈现严重的小型化和低龄化现象,种群结构严重不合理[3],导致现今野生存活量较少。并且在翘嘴鲌人工养殖中没有进行合理育种和繁殖,捕获野生翘嘴鲌即进行育种[4],导致种群质量衰退[5],逃逸的鱼类易破坏野生种群质量。一些研究还发现,长江中下游翘嘴鲌野生种群的的遗传多样性处于较低水平,亲缘关系较近并且分化程度较小,其原因可能是环境与人为共同作用[6];通过黑龙江、太湖、梁子湖等地采集的翘嘴鲌研究发现,翘嘴鲌种群在中国南北差异较大[7]。由于在珠江流域对翘嘴鲌的资源调查和遗传多样性分析数据空缺,对野生鱼类的遗传多样性数据并不充足[8-9]。因此急需展开此方面的调查,了解资源现状对其利用和保护有重要意义。
线粒体DNA (mitochondrial DNA,mtDNA)中的D-loop基因为非编码蛋白序列,属于线粒体基因序列的控制部分,该基因在群体遗传学中利用较多[10-12],是研究种内和种间关系的重要分子标记之一。因此,本实验应用线粒体D-loop基因对珠江流域流经的广西广东省的野生翘嘴鲌遗传多样性和种群分化动态开展深入研究。
1 材料与方法
1.1 样本采集
实验中11个野生地理种群于2014 年7月至9月分别采集于广西梧州(WZ)、昭平(ZP)、平乐(PL)、柳城(LC)、桂平(GP)、平果(PG)、田阳(TY)、龙州(LZ)和扶绥(FS),广东韶关(SG)和肇庆(ZQ)等自然珠江支流(样本数量见表1),样本分别来自珠江流域的浔江(梧州)、桂江(昭平、平乐)、黔江(柳城)、郁江(桂平)、右江(平果、田阳)、左江(龙州、扶绥)、西江(肇庆)和北江(韶关)支流。取翘嘴鲌新鲜肌肉和鳍条于野外用φ=95%酒精固定保存。运回实验室后,-20 ℃低温冰箱保存。
1.2 DNA抽提与PCR及测序
基因组DNA的提取参考《分子克隆实验指南》[13]的方法略加改进。以提取后的DNA为模板,利用线粒体DNA的D-loop区引物进行扩增,引物为DL1 (5′-ACCCCTGGCTCCCAAAGC-3′)和DH2 (5′-ATCTTAGCATCTTCAGTG-3′)[14]。PCR反应总体积为50 μL,包括5 μL 10×PCR Buffer,4 μL 25 mmol / L MgCl2,1 μL 10 mmol / L dNTP,4 μL 2.5 mmol / L 双向引物,0.4 μL Taq DNA 聚合酶(5 U/μL),3 μL DNA模板。PCR 反应条件为94 ℃预变性2 min后,再进行35 个循环,每一循环包括: 94 ℃ 45 s,58 ℃ 45 s,72 ℃ 1 min; 最后 72 ℃延伸 7 min。每次 PCR 反应均设不含模板DNA的空白对照。扩增产物经w=1.5% TAE琼脂糖凝胶电泳分离,EB 染色,紫外灯光下检测、拍照。PCR产物由中美泰和公司经切胶回收后测序。
1.3 数据处理
测得序列用SeqMan[15]软件进行比对,并辅以人工校对。使用DNAsp5.0[16]计算种群单倍型多样性Hd、核苷酸多样性π,用Arlequin 3.1.1 软件[17]计算种群遗传分化指数FST,种群间的基因流[18]由公式Nm=(1/FST-1)/4计算得出。并进行Tajima’s D中性检验(Neutrality tests)和单倍型间的分子方差分析(AMOVA)。利用该软件计算种群起始扩张时间(τ),通过公式t=τ/2uT(T为世代时间)来计算种群扩张的绝对时间。采用MEGA4.0[19]软件分析碱基组成及苷酸位点的替换数,计算种群间和种群内Kumar 2-parameter 模型的遗传距离,构建ML系统树并对拓扑图中的各个分支的支持率进行1 000 次自展法重抽样检验。使用Network程序[20]基于Median Joining方法构建单倍型之间的系统发生关系图。
2 结果与分析
2.1 翘嘴鲌线粒体D-loop序列及单倍型
本实验在11个种群176尾翘嘴鲌个体共获取长度为856bp D-loop基因序列,分析得知该段序列中含115个变异位点(占总序列长度13.43%),其中含59个简约信息位点。各碱基含量平均值分别为:A=34.0%、T=31.8%、C=20.5%和G=13.7%。
在176条序列中共得41个单倍型序列(见表1)。通过转换、颠换对遗传距离可知: 转换和颠换数没有达到饱和状态,且平均转颠换(Ts/Tv)比值为5.24,证明该序列数据可进行下一步统计分析。在11个种群间,单倍型Hap1和Hap2由10个种群共享,单倍型Hap7、Hap9和Hap11都达到5个以上种群共享,单倍型Hap5、Hap6、Hap8、Hap13、Hap19和Hap24均有2个以上种群共享。经过重复扩增和测序后,确定发现其中WZ种群独有Hap3和Hap4,PL种群独有Hap10,LC种群独有Hap12、Hap14-18、Hap20-22,GP种群独有Hap25-27,PG种群独有Hap28-31,TY种群独有Hap32,FS种群独有Hap33-36,SG种群独有Hap37-39,ZQ种群独有40-41,HY种群独有Hap42。

表1 翘嘴鲌遗传变异信息
2.2 种群遗传多样性及遗传分化
2.2.1 种群遗传多样性 在表1中,11个地理种群的单倍型多样性指数最小值0.634 0,最大值1.000 0;核苷酸多样性指数π最小值0.000 4,最大值0.020 6,两种多样性指标都显示出种群较大的变化幅度。TY种群π值为最小值0.000 4,ZQ种群为最大值0.020 6。由于部分样本数量较少导致单倍型指数不稳定原因,核苷酸多样性指数表明了珠江流域翘嘴鲌的遗传多样性在不同地理种群的差距。
2.2.2 种群结构与遗传分化 分子变异分析结果AMOVA见表2,可以看出,遗传变异主要发生在种群内(方差组分81.17%)。把所有种群作为一个整体时,总遗传分化指数(FST=0.188 3,P<0.01 ),表明珠江流域翘嘴鲌已产生中度的遗传分化。各种群翘嘴鲌的遗传距离、遗传分化指数和基因流见表3和表4。由表3可以得出各个种群翘嘴鲌之间遗传距离差异较大,其中肇庆ZQ种群与昭平ZP种群的遗传距离最大(0.020 1),肇庆ZQ和平乐PL种群的遗传距离(0.018 4)仅次于最大值,平果PG种群和田阳TY种群的遗传距离最小(0.000 3)。遗传距离表明种群间亲缘关系,说明肇庆ZQ种群与昭平ZP、平乐PL种群之间亲缘关系较远。对于整个种群而言,最大值仅为0.020 1说明珠江流域的翘嘴鲌亲缘关系并未发生大改变。

表2 珠江水系11个翘嘴鲌种群的分子方差分析结果

表3 珠江水系11个翘嘴鲌种群间遗传距离

表4 珠江水系11个翘嘴鲌种群间的FST值(对角线下方)和Nm值(对角线上方)1)
1)**P<0.01, 差异极显著;*P<0.05, 差异显著
表4中可以看出,WZ与GP和ZQ分化不显著,距离相近的TY和PG分化不显著,FS与LC、GP、TY和SG分化不显著,LZ与LC的分化不显著(遗传分化指数FST<0.25, 基因流Nm>5)。其余种群的两两组合均出现了显著遗传分化,特别是LZ种群与9个种群(除了LC)产生了极显著的遗传分化(FST大于0.05,基因流相对较低Nm<3.39)。
2.3 系统发育关系分析
用MEGA 5.0 软件的Maximum likelihood 法对11个种群翘嘴鲌mtDNA D-loop基因的41种单倍型构建系统发育树并使用草鱼CyprinusCarpioD-loop序列作为树根得到图1。由图1得出,所有种群的单倍型呈镶嵌式排列,没有形成种群特异性进化枝。使用Network对翘嘴鲌单倍型进行网络分析,经过人工简化处理后如图2,从整体上看,所有单倍型分为A、B、C三个小分支,A、B两支单倍型关系较为接近,而C支大多通过假想单倍型相连。在A支以10个种群共享的单倍型Hap1和Hap2为中央单倍型,其他单倍型环绕在四周,形成星样系统发生关系,说明种群由几个单倍型进化而来。该图像与蔡静[21]网络分析结果相似。
3 讨 论
3.1 翘嘴鲌 D-loop 基因结构与遗传多样性分析
本研究沿珠江流域采集样本,范围遍布3个省份(广东、广西和贵州)11个城市,获取共176个野生翘嘴鲌样本,相对于漫长的分子进化速率和种群迁移时间,能反映当前部分野生资源的遗传多样性状况。每个种群的样本均在5尾及以上,采集样本总数符合分析要求,采样范围广泛,得到的结果有一定的实际指导意义。D-loop基因在线粒体DNA中的变异率最高,是线粒体DNA完整分子或是其他区域的5~10倍[22],由于不编码蛋白质,所以大部分基因变异不会对机体产生重大影响,因此可以在进化过程中通过母系遗传保留下来,充分反映不同种群的进化方法。本研究中的翘嘴鲌样本D-loop基因结果与哺乳动物[23]和爬行类[24]接近,符合脊椎动物线粒体DNA序列的组合特点[25]。

图1 基于Maximum likelihood法的翘嘴鲌种群单倍型的分子系统树(Hap1-42代表42个单倍型)Fig.1 The molecular phylogenetic tree of C.alburnus from different haplotypes in the Pearl River by ML method (Hap means different haplotypes)
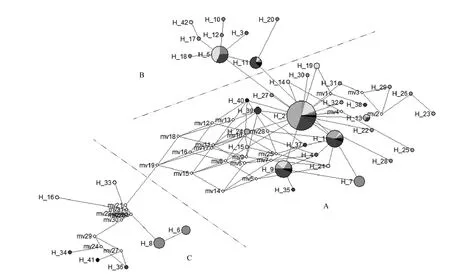
图2 41个 mtDNA D-loop 单倍型的进化网络关系Fig.2 Median joining network of 41 mtDNA D-loop haplotypes
遗传多样性程度是生物多样性的分子基础,其大小决定了该生物能否继续在生物圈繁衍和生活[26],多样性下降将会种群产生负面影响[27]。本次实验所得的珠江流域翘嘴鲌核苷酸多样性π=0.007,高于同水系中的光唇裂腹鱼[28],但低于同水系的赤眼鳟[29],对比长江水系的翘嘴鲌种群[6],本实验中珠江水系的翘嘴鲌多样性明显较高。其中的原因可能有以下两点:① 资源保护程度的差距。光唇裂腹鱼为水域中下层小鱼,在捕捞和养殖中皆不受重视,易被作为野、杂鱼而失去经济价值;赤眼鳟是全国优质经济鱼类,育苗、繁殖、生长以及野生资源的保护都有较完备的体系,不同地理种群的资源调查都在进行中,投入的保护力度较大使得多样性水平能保持较高水准。② 地理复杂性。虽然长江总面积为珠江4倍,流经的长度为珠江3倍,但是华南地区的山形地貌复杂,样本所在的珠江水系内多为山地和丘陵,一旦鱼群在某个生态系统内稳定生存,易形成孤岛与其他种群的交流降低,并根据不同的环境适应和进化,易获得相对较高的多样性。
Lan[30]的研究中表明π值低于0.005时,属于低核苷酸型。本试验中各地的核苷酸多样性差距较大,梧州、桂平、平果、田阳、龙州和韶关皆低于0.002,昭平、平乐、柳城、扶绥和肇庆的π值都大于0.005。从核苷酸多样性角度分析,尽管部分地区的种群处于低核苷酸的状态,但整体翘嘴鲌样本种群遗传多样性较高。造成该结果的原因推测是不同地理环境对翘嘴鲌种群造成的影响不同,一些π值偏低的种群,推测受到当地人为干扰如捕捞、挖沙和航运等产生变化。
本研究中11个种群翘嘴鲌的单倍型多样性指数Hd变化范围为0.634 0~1.000 0,平均值Hd=0.875 1。Nei[31]的研究认为当种群拥有较大数量,并且适应环境的种群不断增多,才可以维持较高的单倍型多样性。根据Grant分类[32],以Hd=0.5,π=0.005为界,本实验中梧州等6个种群(桂平、平果、田阳、龙州和韶关)属于高单倍型和低核苷酸多样性,推测这些种群经历瓶颈效应[33],由于受到环境的压力或者人为灾害如水体污染、水坝建设和过度捕捞而使得种群大量减少后,种群逐渐恢复但没有积累足够的核苷酸多样性而出现此结果。这部分的种群若没有受到保护很难抵御新一轮来自环境的压力,容易造成种群数量大幅减少。通过种群起始扩张时间公式得出种群整体在5 000年左右产生迁移,根据史料记载2 000多年前,两广地区开始出现经济和文化的传播,至1 000年前我国北宋时期,才将两广地区收归中央版图,根据当时的生产力状况推断,人类对该地区的野生鱼类开始初步产生影响。昭平等5个种群属于高单倍型和高核苷酸多样性,说明这些地点原本就拥有较大数量的种群,是维持高单倍型的基础,并且并没有受到环境压力影响。综合结果表明复杂的地理位置导致了样本采集地区的翘嘴鲌遗传多样性的不稳定性,需对遗传多样性较低的地区进行资源保护和管理。
3.2 翘嘴鲌种群的遗传分化
从种群遗传分化指数 (FST) 和基因流 (Nm) 两个方面对种群的遗传分化进行评估。利用软件Arlequin 得到11个种群两两之间的FST值。根据Wright研究[34]指出,当FST的值大于0.05,种群间属于无遗传分化;若FST小于0.15,则处于较小遗传分化程度;若FST小于0.25,说明种群处于中度遗传分化;若FST大于0.25,表示种群间有较大遗传分化。本研究将珠江流域2个省份11个种群翘嘴鲌作为一个整体,通过AMOVA得出其FST为0.189 72 (P<0.05),这说明将实验采集的珠江流域翘嘴鲌作为一个大种群已产生了中度遗传分化。与长江流域[6]、中国东南部翘嘴鲌[35]遗传分化结果相似,与兴凯湖、洪泽湖、洞庭湖等6野生种群翘嘴鲌线粒体ND2基因遗传分化结果[36]和黑龙江、河南水库、太湖等6个种群翘嘴鲌线粒体COII基因遗传分化结果[37]不相同(其结果均显示种群遗传分化程度较大)。根据Slatkin对基因流研究说明,当Nm值大于5时,说明种群间能保持稳定,有频繁的基因交流[38]。从表5分析,梧州和桂平、肇庆,昭平和平乐,田阳和平果,扶绥和柳城、桂平、田阳、韶关之间分化不明显。而其他种群之间产生高度的分化程度。值得注意的是,昭平与平乐种群之间的分化程度较低,但它们与其他种群却产生了高度遗传分化(FST>0.25)。其他种群之间也有中度分化现象,FST在0.1~0.3之间。从地理上分布来看,平乐与昭平在东西方向有都庞岭和大瑶山,与其他地方有明显的地理隔离。而相比分化程度较低的梧州、肇庆和桂平3个种群,它们所处的3条支流黔江、浔江和西江互相连通,因此基因交流比较丰富;同样的原因可用来解释田阳与平果种群的低遗传分化水平(均属于右江支流)。
3.3 翘嘴鲌种群遗传距离及亲缘关系
遗传距离的大小表明了种群之间亲缘关系远近,可以对种群之间的遗传关系做出预测[39]。研究中11个种群的翘嘴鲌遗传距离变异范围为0.003 0~0.020 1,遗传距离较小。根据Shaklee[40]研究发现,鱼类的遗传距离以0.9、0.3、0.05为界,区分为属、种和种群三级水平。本研究中翘嘴鲌遗传距离最高值仍低于0.05,未达亚种的分化指数。说明来自研究中来自11条不同水系的翘嘴鲌有可能为一支祖先种群繁衍而来。
由于个体数偏多,使用Maximum likelihood 法对所有个体作图后,发现不同种群间个体互相参杂。故采用对种群单倍型进行Median-joining 和 ML 作图。可看出,系统进化树与单倍型网络分析结果相似。单倍型Hap1和Hap2位于所有单倍型的中心,且这两个单倍型是除平乐外所有种群所共享,两种单倍型合计出现频率达71次,推测为所有单倍型的祖先,其余单倍型由其逐渐发展而来。且单倍型间差异并不大,A、B两支单倍型突变步骤不超过3次,说明各种群亲缘关系并不远。C支中的单倍型可能为各个种群中产生特异突变并保留下来的单倍型。
3.4 翘嘴鲌的保护管理
本研究所得的珠江流域所有翘嘴鲌种群作为一个整体来看,多样性程度并不低,但不能因此就忽视对翘嘴鲌资源的保护,仍有大量采样点的多样性处于较低水平。对比其他遗传多样性研究且本研究为第一次对珠江流域野生翘嘴鲌种群进行多样性检测,尚不能确定是否已经因受到近年来人工捕捞和放流影响。需要结合后续对珠江和长江以及其他重要经济流域的调查,开展详细的研究工作,根据遗传多样性的变化趋势采取相应的保护措施。本研究为中国翘嘴鲌资源调查补充更全面的数据,最终为翘嘴鲌野生资源提供保护和恢复,品种的选择和可持续利用提供数据和理论基础。
[1] 乐佩琦, 罗云林. 鮊亚科类系统发育初探(鲤形目:鲤科)[J]. 水生生物学报, 1996,20(2):182-185.
[2] 崔中倩. 兴凯湖翘嘴鲌消化系统及其生长的研究[D]. 哈尔滨:东北农业大学, 2013.
[3] 姚闻卿, 胡菊英. 巢湖翘嘴红鲌的生物学及其资源变动的研究[J]. 安徽大学学报(自然科学版), 1982(Z1):151-158.
[4] 杨虹. 兴凯湖翘嘴鲌生物学特性及人工繁殖研究[D]. 哈尔滨:东北农业大学, 2009.
[5] 张永正. 翘嘴红鲌人工繁殖技术研究 [C]. 杭州:浙江大学, 2005.
[6] 黄小彧. 长江水系翘嘴鲌遗传多样性研究[D]. 广州:暨南大学, 2012.
[7] 王伟. 翘嘴鲌(Culter alburnus)群体遗传多样性及鲌亚科鱼类系统发生的研究[D]. 上海:华东师范大学, 2007.
[8] 杨慧荣, 赵会宏, 蒙子宁, 等. 赤眼鳟线粒体D-loop和Cyt b基因序列的对比分析[J]. 中山大学学报(自然科学版), 2012,51(5):100-106.
[9] 杨慧荣, 欧阳徘徊, 李桂峰, 等. 用微卫星技术研究珠江流域三个野生大眼鳜群体的遗传多样性[J]. 中山大学学报(自然科学版),2015,54(1):103-109.
[10] LIU R, YANG G, LEI C. The genetic diversity of mtDNA D-loop and the origin of Chinese goats[J]. Acta Genetica Sinica, 2006,33(5):420-428.
[11] AGNèSE J, ZENTZ F, LEGROS O, et al. Phylogenetic relationships and phylogeography of the Killifish species of the subgenus Chromaphyosemion (Radda, 1971) in West Africa, inferred from mitochondrial DNA sequences[J]. Molecular Phylogenetics and Evolution, 2006,40(2):332-346.
[12] KOHLMANN K, KERSTEN P. Deeper insight into the origin and spread of European common carp (Cyprinus carpio carpio) based on mitochondrial D-loop sequence polymorphisms[J]. Aquaculture, 2013,376:97-104.
[13] SAMBROOK J, RUSSELL D W, RUSSELL D W. Molecular cloning: a laboratory manual (3-volume set)[M]. New York:Cold Spring Harbor Laboratory Press, 2001.
[14] LIU H, TZENG C, TENG H. Sequence variations in the mitochondrial DNA control region and their implications for the phylogeny of the Cypriniformes[J]. Canadian Journal of Zoology, 2002,80(3):569-581.
[15] SWINDELL S R, PLASTERER T N. SEQMAN Sequence data analysis guidebook [M]. Springer, 1997:75-89.
[16] ROZAS J, SNCHEZ-DELBARRIO J C, MESSEGUER X, et al. DnaSP, DNA polymorphism analyses by the coalescent and other methods[J]. Bioinformatics, 2003,19(18):2496-2497.
[17] EXCOFFIER L, LAVAL G, SCHNEIDER S. Arlequin (version 3.0): an integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics Online, 2005,1:47.
[18] WRIGHT S. Evolution in Mendelian populations[J]. Genetics, 1931,16(2):97.
[19] TAMURA K, DUDLEY J, NEI M, et al. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007,24(8):1596-1599.
[20] BANDELT H, FORSTER P, RÖHL A. Median-joining networks for inferring intraspecific phylogenies.[J]. Molecular Biology and Evolution, 1999,16(1):37-48.
[21] 蔡静. 基于线粒体细胞色素b基因探讨皖南地区凹耳蛙种群遗传变异及其种群历史[D]. 芜湖:安徽师范大学, 2013.
[22] 董志国, 李晓英, 王普力, 等. 基于线粒体D-loop基因的中国海三疣梭子蟹遗传多样性与遗传分化研究[J]. 水产学报, 2013,37(9):1304-1312.
[23] 张成福, 徐利娟, 姬秋梅, 等. 西藏牦牛mtDNA D-loop区的遗传多样性及其遗传分化[J]. 生态学报, 2012,32(5):1387-1395.
[24] 黄雪贞, 钱国英, 李彩燕. 中华鳖3个地理群体线粒体基因D-loop区遗传多样性分析[J]. 水产学报, 2012,36(1):17-24.
[25] BROUGHTON R E, MILAM J E, ROE B A. The complete sequence of the zebrafish (Daniorerio) mitochondrial genome and evolutionary patterns in vertebrate mitochondrial DNA[J]. Genome Research, 2001,11(11):1958-1967.
[26] 姜艳艳, 孔晓瑜, 喻子牛, 等. 黄海蓝点马鲛mtDNA D-loop序列变异分析[J]. 中国水产科学, 2003,10(3):177-183.
[27] RYMAN N, UTTER F. Population genetics and fishery management[M]. Washington:University of Washington Press, 1987.
[28] 韩虎峰, 代应贵. 珠江水系光唇裂腹鱼可渡河种群mtDNA D-loop序列多态性分析[J]. 中国水产科学, 2010,17(1):143-149.
[29] 杨慧荣, 赵会宏, 刘丽, 等. 珠江和长江水系赤眼鳟D-loop基因序列遗传变异分析[J]. 上海海洋大学学报, 2012,21(3):337-343.
[30] LAN H, SHI L. The origin and genetic differentiation of native breeds of pigs in southwest China: An approach from mitochondrial DNA polymorphism[J]. Biochemical genetics, 1993,31(1-2):51-60.
[31] NEI M. Molecular evolutionary genetics[M]. New York: Columbia University Press, 1987.
[32] GRANT W, BOWEN B W. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity, 1998,89(5):415-426.
[33] NEI M, MARUYAMA T, CHAKRABORTY R. The bottleneck effect and genetic variability in populations[J]. Evolution, 1975:1-10.
[34] WRIGHT S. The interpretation of population structure by F-statistics with special regard to systems of mating[J]. Evolution, 1965:395-420.
[35] 张宏, 赵良杰, 刘其根, 等. 翘嘴鲌线粒体控制区遗传多样性分析[C]. 2013年中国水产学会学术年会. 合肥, 2013.
[36] 伊西庆. 中国东部6个大型湖泊翘嘴鲌(Culteralburnus)遗传多样性的线粒体ND2基因序列分析[D]. 广州:暨南大学, 2009.
[37] 王伟, 陈立侨, 禹娜, 等. 应用COⅡ基因部分序列分析翘嘴鲌群体的遗传多样性[J]. 大连水产学院学报, 2008,23(5):403-408.
[38] SLATKIN M. Gene flow and the geographic structure of natural populations[J]. Science, 1987,236(4803):787-792.
[39] 陈兴汉, 郭梁, 李明明, 等. 中国南方沿海13种石斑鱼类的分子系统进化关系分析[J]. 中山大学学报(自然科学版), 2014,53(4):123-130.
[40] SHAKLEE J B, TAMARU C S, WAPLES R S. Speciation and evolution of marine fishes studied by the electrophoretic analysis of proteins[J]. Pacific Science,1982,36(2):143-157.
Genetic diversity and genetic differentiation ofCulteralburnusfrom eleven geographical populations of the Pearl River based on mitochondrial D-loop gene
YANGZituo1,2,3,SUNJijia1,2,LIGuifeng3,XIAOShibin1,2,ZHANGHeqian3,YANGHuirong1,2,ZHAOHuihong1,2,LIULi1,2
(1. College of Animal Science, South China Agricultural University, Guangzhou 510642, China;2. College of Animal Science, South China Agricultural University∥Guangdong Provincial Key Laboratory of Agro-Animal Genomics and Molecular Breeding, Guangzhou 510642, China;3. School of Life Sciences, Sun Yat-sen University, Guangzhou 510275, China )
Culteralburnusis one of the most economically important edible freshwater fish in the Pearl River and is widely distributed in the Yangtze River and the Yellow River. To assess the level of genetic diversity and genetic variation ofC.alburnuspopulations within the Pearl River,the samples were collected from the eleven geographical populations from Guangxi and Guangdong Province (Wuzhou, Zhaoping, Pingle, Liucheng, Guiping, Pingguo, Tianyang, Longzhou, Fusui). Among the 176 D-loop sequences, there were 286 polymorphic sites, including 115 variation sites and 59 parsimony informative sites. Overall, 41 haplotypes were defined among 176 Culter alburnus individuals.C.alburnuspopulations in the Pearl River were being a high haplotypic diversity index (Hd=0.875 06) and low nucleotide diversity index (π=0.007 00). The AMOVA (analysis of molecular variance) index of total populations was 0.188 28, indicating that the genetic differentiation among populations was moderate. It was predicted that differentiation time was about 50×103years.FSTindex between WZ and GP, WZ and ZQ, ZP and PL, TY and PG, especially, FS and LC, GP, TY, SG, was showed significant genetic differences (FST>0.25). The Phylogenetic tree and the Median joining drawing showed that haplotype 1 and 2 were the ancestors of other haplotypes. The relationship between genetic distance and geographical distance should be explored in the future.
The Pearl River;Culteralburnus; D-loop; genetic diversity; genetic differentiation
10.13471/j.cnki.acta.snus.2016.02.017
2015-08-31
公益性行业(农业)科研专项资助项目(201303048);国家科技基础条件平台建设项目(2008DKA30470-010)
杨子拓(1990年生),男;研究方向:水生生物学;通讯作者:刘丽;E-mail:liuli@scau.edu.cn
S932.4
A
0529-6579(2016)02-0089-08
