供体同源臂长度对ZFN介导的同源重组效率的影响*
2016-06-05乔艳乐陈瑶生何祖勇
聂 宇,乔艳乐,陈瑶生,何祖勇
(中山大学生命科学学院∥有害生物控制与资源利用国家重点实验室,广东 广州510006)
供体同源臂长度对ZFN介导的同源重组效率的影响*
聂 宇,乔艳乐,陈瑶生,何祖勇
(中山大学生命科学学院∥有害生物控制与资源利用国家重点实验室,广东 广州510006)
应用锌指核酸酶(Zinc Finger Nuclease,ZFN)和序列同源的供体(Donor)作为模板,借助DNA的同源重组修复机制能够对动物基因组实现精确的遗传修饰。目前关于供体长度与ZFN介导的同源重组修复效率相关性的报道相对较少。本研究构建了一对靶向EGFP的ZFN并鉴定了活性,同时设计了一系列不同长度的供体,应用流式细胞分析术,在稳定整合了带有移码突变的EGFP基因的CHO细胞中系统分析了供体长度对ZFN介导的同源重组修复效率的影响。结果发现当同源臂单臂仅有50 bp时,即可有效支持ZFN介导的同源同组,随着同源臂长度的延伸,同源重组的效率有所提高,但要实现高效率的同源重组(较传统方法提高104倍),同源臂单臂长度需要延长至1 000 bp以上。这为今后如何设计合适的Donor,以提高ZFN等基因组编辑工具介导的同源重组效率提供了借鉴。
锌指核酸酶; DNA双链断裂; 同源重组; 供体; 同源臂
基因组修饰在生物学、医学和农业等领域都有十分重要的应用价值,但是传统的基因打靶效率偏低使该技术的发展受到限制[1]。锌指核酸酶(Zinc Finger Nuclease,ZFN)是一种强有力的基因组编辑工具,使基因打靶的效率比传统方法提高了100~10 000倍。ZFN是由特异性识别DNA序列的Cys2-His2锌指结构域和非特异性核酸内切酶FokⅠ切割结构域组成,能够在基因组特定位点上进行切割,引起DNA双链断裂(double-strandbreak,DSB)。对ZFN引起的DSB,细胞可通过非同源末端连接(non-homologousend-joining,NHEJ)或者同源重组(homologousrecombination,HR)两种不同的途径进行修复。非同源末端连接仅促使断裂DNA片段末端进行结合,是一种易错的修复方式,通常会在ZFN切割位点处产生DNA的插入和缺失 (Indel)。同源重组可以利用序列同源的供体(Donor)作为模板修复DNA断裂,能够对基因组实现精确的遗传修饰[2-3]。在有供体存在时,特定细胞中ZFN引起的DSB最高能以50%的概率发生同源重组[4]。
目前ZFN介导的同源重组技术已应用于果蝇[5]、斑马鱼[6]、小鼠[7-8]、大鼠[9]、猪[10-11]、拟南芥[12]、人体细胞[13]等多种动植物的基因修饰。其中,介导重组所用的Donor片段长度从0.75~2.3kb不等,在不同的细胞系中获得的重组效率也不尽相同[4, 13-15]。然而关于Donor同源臂长度对ZFN介导的同源重组效率影响的研究仍较缺乏。本研究采用一系列不同长度的Donor,在稳定表达EGFP的CHO细胞中,系统比较了ZFN介导的同源重组效率,发现ZFN可以利用短至50bp的同源臂单臂来实现同源重组,但要实现较高效的同源重组,同源臂单臂长度需延长至1 000bp以上,为今后研究中如何设计Donor提供借鉴。
1 材料与方法
1.1 材料
CHO细胞购于美国ATCC生物资源中心。pEGFP-N1购于Clontech公司。
1.2 方法
1.2.1ZFN表达载体构建 本文中所采用的ZFN源自Maederetal[16]中的靶向EGFP基因的一对ZFN(EG502ApairA)。
使用VectorNTI软件根据氨基酸序列反推出ZFP的DNA序列,和FokⅠ序列拼接后,由生工生物公司进行了人源化优化后合成。用EcoRⅠ和NotⅠ对pEGFP-N1进行双酶切,将合成的DNA片段克隆进载体,获得一对ZFN的表达载体。
1.2.2EGFP移码突变载体pEGFP-N1-FS(frameshift)的构建 两对引物(表1),以pEGFP-N1为模板,扩增产生移码突变的左臂(FSLarm)和右臂(FSRarm)两个片段。其中扩增FSLarm的下游引物FSLarmR和扩增FSRarm的上游引物FSRarmF的5′端均添加了AflⅡ酶切位点。扩增得到的FSLarm和FSRarm分别克隆到T载体,用EcoR I和AflⅡ从T载体上将FSLarm片段切下,与另一个T载体上的FSRarm拼接成发生了移码的EGFP片段(EGFP-FS)。用Hind Ⅲ和XbaⅠ将EGFP-FS切下,克隆进pEGFP-N1载体,获得pEGFP-N1-FS表达载体,载体上的EGFP基因中含有8个碱基的插入(图1),使EGFP产生移码突变,无法正常表达绿光荧光蛋白。

图1 pEGFP-N1-FS表达载体图谱Fig.1 pEGFP-N1-FS vector map
1.2.3 细胞转染 CHO细胞在含有φ=10%胎牛血清和双抗(100 U/mL青霉素和100 mg/mL链霉素)的DMEM培养基中生长至80%~90%汇合度时,先用PBS洗涤两遍,然后用胰酶消化2 min,随后加入血清终止消化,将消化后的细胞收集至15 mL的离心管进行离心,弃上清后再用PBS洗涤一遍进行第二次离心。离心后弃上清,然后用适量Buffer R (Life Technology)溶液重悬细胞,使细胞密度达到每mL 1.0×107个细胞,加入2 μg质粒,使用Neon转染系统(Life Technology)对CHO细胞进行电击转染。转染条件为:电压1 650 V,脉冲10 ms,脉冲次数3次。细胞转染后接种至含血清无抗生素的DMEM培养基中培养中,24 h后更换培养基,添加双抗,在φ=5%的CO2、37 ℃条件下继续培养。2 d后进行流式分析或者收取细胞提取DNA进行后续实验。
1.2.4 稳定表达pEGFP-N1和pEGFP-N1-FS的CHO细胞的获得 CHO细胞分别电转染pEGFP-N1和pEGFP-N1-FS质粒后,在细胞培养基中添加800 ng/mL 的G418,进行为期14~20 d的筛选,分别获得稳定表达pEGFP-N1的CHOEGFP和pEGFP-N1-FS的CHOEGFP-FS细胞群。
1.2.5 T7EI酶切检测 使用组织/细胞提取试剂盒(OMEGA公司)提取细胞的基因组,使用D1024引物进行PCR扩增出包含ZFN识别位点的长度为1 024 bp的产物,采用Axgene PCR清洁试剂盒(Axgene公司)纯化PCR产物。纯化后的PCR产物先在高温下变性,然后经逐步降温退火形成异源双链DNA,使用T7EⅠ对上述产物进行酶切,如果ZFN有活性,会产生442 bp和588 bp的两个片段,可以在w=3%琼脂糖凝胶中分辨出来。最后通过Image J软件计算ZFN的切割效率,估算其活性。
1.2.6 流式分析 先用w=1%的胰蛋白酶消化贴壁的CHO细胞,用不少于300 μL PBS重悬成单个细胞,用300目滤膜将细胞过滤到流式管中,用FACSAria Ⅱ流式细胞分选仪器分选EGFP阳性细胞,接种培养。用FACScalibur流式细胞分析仪上分析细胞的荧光比例与强度。
1.2.7 不同长度Donor的获取 以pEGFP-N1为模板,使用引物对D489、D1024、D1550、D1792和D2043(数值代表扩增片段的长度)进行PCR扩增,胶回收后分别获得不同长度的Donor:Z489、Z1024、Z1550、Z1792和Z2043(图2)。
2 结果与分析
2.1 ZFN对EGFP基因的敲除效率检测
稳定转染:含在稳定表达EGFP的CHOEGFP细胞中转染ZFN表达质粒,3 d后利用荧光倒置显微镜观察荧光表达情况,与对照(图3:A)相比,转染ZFN表达质粒的CHOEGFP细胞中EGFP阴性的细胞的数量明显增多(图3:B)。流式分析表明,转染ZFN后,CHOEGFP细胞中EGFP阳性的细胞的比例降低了12.95%(图3:C)。
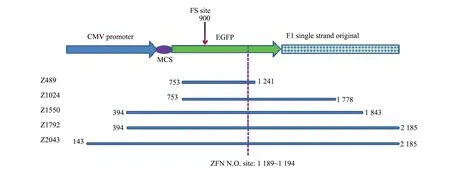
图2 Donor示意图Fig.2 Schematic diagram of disgned Donors
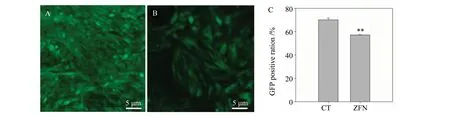
图3 ZFN对CHOEGFP细胞中EGFP基因的敲除检测Fig.3 Detection of the knock out effect of EGFP gene in CHOEGFP cells by ZFN
瞬时转染:流式上机检测,CHO细胞共转染ZFN表达质粒和pEGFP-N1质粒后,与只转染pEGFP-N1质粒的对照相比,EGFP阳性细胞的比例下降了52.3%(图4:A),细胞中EGFP的平均荧光强度也明显下降(图4:B)。表明ZFN对EGFP具有较高的敲除效率。
2.2 T7EI酶切检测ZFN的切割活性
CHO细胞瞬时转染ZFN表达质粒和pEGFP-N1质粒后48 h,以及CHOEGFP细胞转染ZFN表达质粒后48 h,分别提取基因组DNA进行T7EI酶切检测ZFN介导的EGFP突变频率。结果如图5所示, ZFN对瞬时表达EGFP和稳定表达EGFP的CHO细胞中的EGFP基因均有明显的打靶作用,其中对质粒上EGFP基因的切割效率高达67.66%,对整合在基因组中的EGFP基因的的切割效率也达11.54%,这与流式检测观察到EGFP阳性细胞比例降低的结果较为一致。

图4 CHO细胞瞬时转染GFP与ZFN后的流式检测结果Fig.4 Flow cytomety analysis of CHO cells transiently transfected with GFP and ZFN expression vectors

图5 T7EI酶切结果Fig.5 T7E1 assay reswlts
2.3 ZFN介导的同源重组
利用药物筛选的方法加入800 ng/mL G418筛选转染pEGFP-N1-FS为了检测ZFN是否可以通过同源重组修复途径将发生了移码突变的EGFP基因恢复成功能正常的EGFP基因,本研究使用长度为489 bp和1 024 bp的两个Donor Z489和Z1024,与ZFN表达质粒共转染CHOEGFP-FS细胞,7 d后通过荧光显微镜观察发现右侧同源臂长度仅为50 bp左右的Donor Z489在ZFN作用下,可以修复突变的EGFP基因,使部分CHOEGFP-FS细胞重新表达EGFP蛋白(图6:A,B),右同源臂较长的Donor Z1024在ZFN作用下也可以使部分CHOEGFP-FS细胞恢复EGFP表达(图6:C,D)。流式分析结果显示,与只转染Donor的对照相比,共转染Donor和ZFN后CHOEGFP-FS细胞群中出现EGFP阳性细胞的比例显著升高。这些结果表明,在Donor存在时,ZFN可以在CHOEGFP-FS细胞中通过同源重组修复EGFP基因。
2.4 同源臂长度对ZFN介导的同源重组效率的影响
在证明ZFN在CHOEGFP-FS细胞中具有介导同源重组的功能之后,本研究进一步通过共转染ZFN表达质粒和另外3种延长了同源臂的Donor:Z1550、Z1792和Z2043(图2),以检测同源臂长度对同源重组效率的影响。转染后7 d,通过荧光显微镜观察可以发现,与Z1024 Donor(图6:C,D)相比,Donor同源臂延长后,CHOEGFP-FS细胞群中恢复EGFP表达的细胞明显增多了(图7:A-F)。流式分析结果也证明CHOEGFP-FS细胞群中有较多的细胞恢复了EGFP表达(图7:G),且细胞中的平均荧光强度与对照相比也有显著性的提高(图7:H)。在这3种Donor中,其中Z1792 Donor相比Z1550 Donor,其右侧同源延长了242 bp,在ZFN作用下实现同源重组的效率提高了约2%(图7:G);Z2043 Donor相比Z1792 Donor,其左侧同源臂延长了251 bp,在ZFN作用下实现同源重组的效率提高了12.5%(图7:G)。Donor Z489与Z1024在ZFN介导的同源重组作用下,只能使0.8%左右的CHOEGFP-FS细胞恢复EGFP表达(图6:E),且两者之间差异不显著(图7:I)。而Donor Z1550、Z1792和Z2043在ZFN介导的同源重组作用下,分别能够8.67%,10.24%和23.15%的CHOEGFP-FS细胞恢复EGFP表达(图7:G)。综合比较5种Donor在ZFN作用下实现同源重组的效率,可以发现当同源臂单臂仅有50 bp时,即可实现有效支持ZFN介导的同源同组,随着同源臂长度的延伸,同源重组的效率有所提高,但要实现高效率的同源重组,同源臂单臂长度需要延长至1 000 bp以上(图8,表1)。
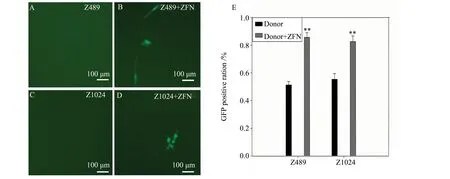
图6 CHOEGFP-FS细胞中ZFN介导的同源重组结果Fig.6 Homologows recombinaton in CHOEGFP-FS cells mediated by ZFN

图7 3种不同长度Donor在ZFN作用下实现同源重组的效率比较Fig.7 Comparison of the homologows recombinaton efficiencies of three donors with disferent length medrwted by ZFN
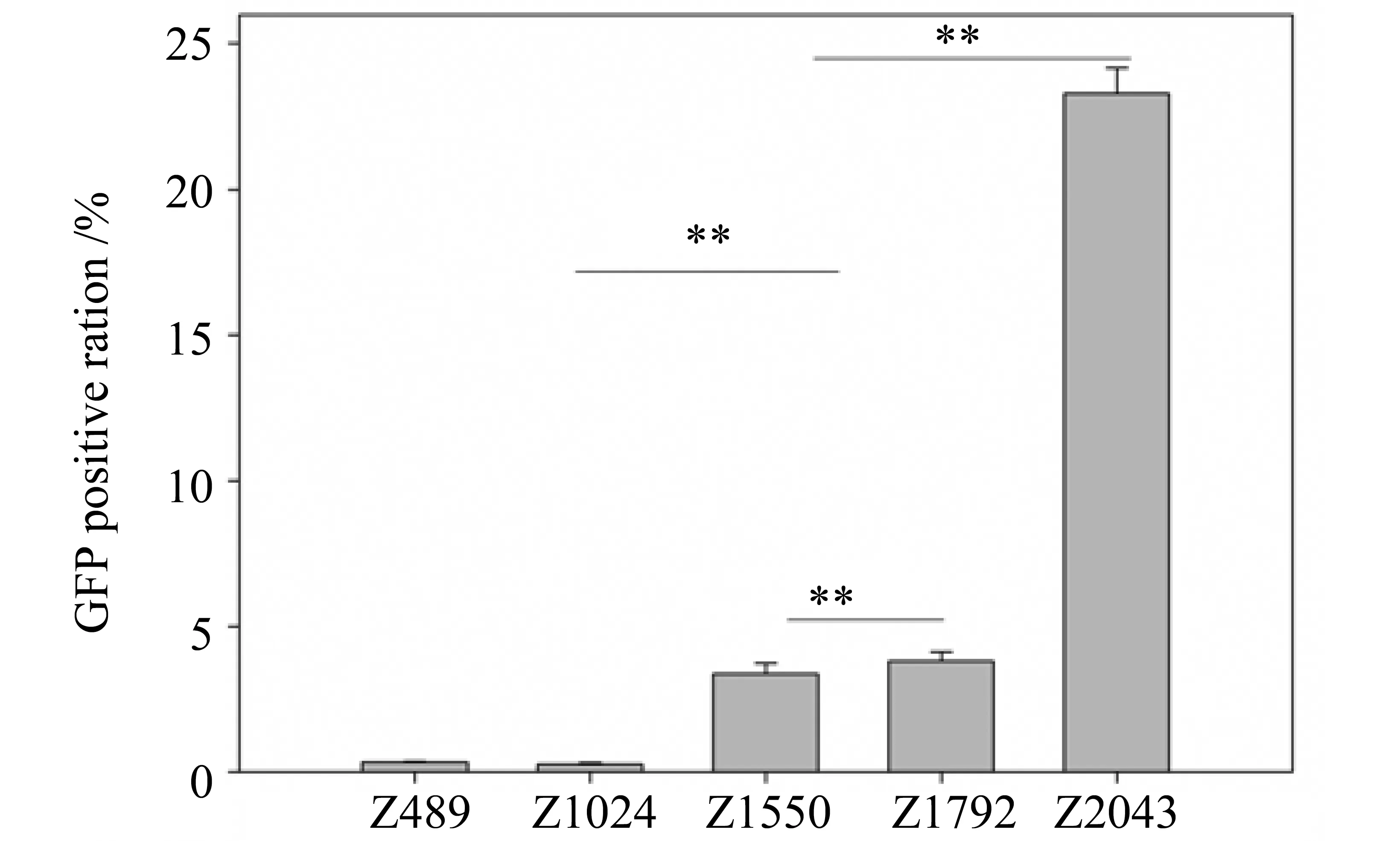
图8 不同长度的Donor在ZFN作用下实现同源重组的效率比较Fig.8 Comparison of the homologows recombination efficiencies of donors with different length mediated by ZFN**:P<0.01
表1 ZFN介导重组实验中所用的供体同源臂长度
Table 1 Length of homologous arm of donors used in homologous recombination mediated by ZFN

N供体名称L左侧同源臂长度/bpR右侧同源臂长度/bpZ48943950Z1024439585Z1550798752Z1792798994Z20431049994
3 讨 论
通过同源重组的方法在染色体DSB处实现DNA的精确修复,最关键的一个步骤是DNA异源双链结构的形成。这需要细胞募集一系列的重组蛋白来参与DNA的配对重组和修复过程。这些重组蛋白包括RecA家族DNA修复蛋白(RecA-family DNA-repairing proteins),单链DNA结合蛋白(single-stranded DNA binding proteins),重组调节蛋白(recombination mediator proteins),退火蛋白(annealing proteins)及各种核酸酶[17]。我们通常把能发生有效同源重组的最小Donor长度称为最小功能效应片段(minimum effective processing segment,MEPS)[18],它主要与稳定同源重组中产生的中间结构相关。之前的研究认为细菌中MEPS为50~100 bp,而真核生物中为200~400 bp[19]。
不同物种中同源重组修复对于Donor的长度,浓度要求不同[20]。在酵母中,可以用短至30 bp的Donor实现ZFN介导的HDR,使自发的重组处于非常低的水平[21],但针对某种NHEJ依赖性更强的酵母种属,如Yarrowia lipolytica,只有当同源臂大于1 kb时才能实现同源重组修复[22]。在多细胞真核生物中,虽然出现过利用短至25 bp同源序列实现分子间重组(intermolecular recombination)的个例[23],但是普遍认为要实现高效率的同源重组,Donor长度需大于1 kb[24],然而当同源臂达到2 kb以上时,同源重组效率不再随同源臂长度增加而提高[25-26]。与此同时,为了提高HDR效率,研究者更倾向于使用单链核苷酸作为供体[20, 25]。
本研究发现右侧同源臂仅有50 bp的Z489 Donor即可通过同源重组修复突变的EGFP基因(图6:A,B,E),表明ZFN在CHO细胞中,可以利用短至50 bp的同源臂单臂,并且在切割位点与需要修复的突变位点相距达242 bp的情况下,实现精确的同源重组修复。这可能与Rad51发挥功能需要高效搜索50 bp的同源臂相关[27-28]。
ZFN是高效的基因修饰工具。与传统的基因打靶技术相比,利用ZFN进行基因修复时,使用2 043 bp的Donor可实现23.15%的同源重组,效率提高了23 000倍(23.15% vs 10-4)证明了ZFN是一种高效的基因组编辑工具。此外,本研究发现,延长Donor的,左右同源臂长度可提高同源重组的效率,同Deng在传统基因打靶中得到的结论一致[29]。CHOEGFP-FS细胞基因组中EGFP移码突变的位点位于ZFN切割位点左侧294 bp处,我们发现Donor的左侧同源臂延长后,同源重组的效率得到大幅度的提高。这表明,在同源重组修复过程,可能需要Donor从DSB侵入一条越过突变位点的较长的单链作为模板,所以本研究中ZFN介导的同源重组对左侧同源臂的的长度具有较高的要求。当左侧同源臂向前延长时会包含CMV启动子的部分序列,尤其是Z2043 Donor的左侧同源臂包含了143~394 bp区段的CMV启动子的核心区域,导致只转染Donor的细胞,在7后的检测中,可能因为随机整合而使部分细胞表达了EGFP,然而通过扣除这些本底表达(图7:I),我们仍然可以发现左侧同源臂延长后可以提高同源重组的效率。
ZFN介导的同源重组效率随着同源臂长度的延长而增高;当同源臂单臂的长度增加至1 000 bp时,同源重组效率有较大幅度的提高。因此本研究提示在实验中需要利用ZFN等基因组编辑工具实现较高的同源重组效率,在设计Donor时,应使同源臂单臂长度应达到1 000 bp以上。
[1] BOUVIER J, CHENG J G. Recombineering-based procedure for creating Cre/loxP conditional knockouts in the mouse[J]. Curr Protoc Mol Biol, 2009, 23:13-23.
[2] WOOD A J, LO T W, ZEITLER B, et al. Targeted genome editing across species using ZFNs and TALENs[J]. Science, 2011, 6040: 307.
[3] LO T W, PICKLE C S, LIN S, et al. Precise and heritable genome editing in evolutionarily diverse nematodes using TALENs and CRISPR/Cas9 to engineer insertions and deletions[J]. Genetics, 2013, 2: 331-348.
[4] LOMBARDO A, GENOVESE P, BEAUSEJOUR C M, et al. Gene editing in human stem cells using zinc finger nucleases and integrase-defective lentiviral vector delivery[J]. Nat Biotechnol, 2007, 11: 1298-1306.
[5] BIBIKOVA M, GOLIC M, GOLIC K G, et al. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases[J]. Genetics, 2002, 3: 1169-1175.
[6] FOLEY J E, MAEDER M L, PEARLBERG J, et al. Targeted mutagenesis in zebrafish using customized zinc-finger nucleases[J]. Nature Protocols, 2009, 12: 1855-1868.
[7] MEYER M, ORTIZ O, HRABE de ANGELIS M, et al. Modeling disease mutations by gene targeting in one-cell mouse embryos[J]. Proc Natl Acad Sci U S A, 2012, 24: 9354-9359.
[8] CARBERY I D, JI D, HARRINGTON A, et al. Targeted genome modification in mice using zinc-finger nucleases[J]. Genetics, 2010, 186(2): 451-459.
[9] GEURTS A M, COST G J, FREYVERT Y, et al. Knockout rats via embryo microinjection of zinc-finger nucleases[J]. Science, 2009, 5939: 433.
[10] HAUSCHILD J, PETERSEN B, SANTIAGO Y, et al. Efficient generation of a biallelic knockout in pigs using zinc-finger nucleases[J]. Proc Natl Acad Sci U S A, 2011, 29: 12013-12017.
[11] YANG D S, YANG H Q, LI W, et al. Generation of PPAR gamma mono-allelic knockout pigs via zinc-finger nucleases and nuclear transfer cloning[J]. Cell Research, 2011, 6: 979-982.
[12] LLOYD A, PLAISIER C L, CARROLL D, et al. Targeted mutagenesis using zinc-finger nucleases in Arabidopsis[J]. Proceedings Of the National Academy Of Sciences Of the United States Of America, 2005, 6: 2232-2237.
[13] URNOV F D, MILLER J C, LEE Y L, et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases[J]. Nature, 2005, 7042: 646-651.
[14] TOWNSEND J A, WRIGHT D A, WINFREY R J, et al. High-frequency modification of plant genes using engineered zinc-finger nucleases[J]. Nature, 2009, 459(7245): 442-445.
[15] MOEHLE E A, ROCK J M, LEE Y L, et al. Targeted gene addition into a specified location in the human genome using designed zinc finger nucleases (vol 104, pg 3055, 2007)[J]. Proceedings Of the National Academy Of Sciences Of the United States Of America, 2007, 104(9): 3055-3060.
[16] MAEDER M L, THIBODEAU-BEGANNY S, OSIAK A, et al. Rapid “Open-Source” engineering of customized zinc-finger nucleases for highly efficient gene modification[J]. Molecular Cell, 2008, 2: 294-301.
[17] MORRICAL S W. DNA-pairing and annealing processes in homologous recombination and homology-directed repair[J]. Cold Spring Harb Perspect Biol, 2015, 2: a016444.
[18] SHEN P, HUANG H V. Homologous recombination in Escherichia coli: dependence on substrate length and homology[J]. Genetics, 1986, 3: 441-457.
[19] WALDMAN A S. Ensuring the fidelity of recombination in mammalian chromosomes[J]. Bioessays, 2008 (11/12): 1163-1171.
[20] SHEN B, ZHANG X, DU Y, et al. Efficient knockin mouse generation by ssDNA oligonucleotides and zinc-finger nuclease assisted homologous recombination in zygotes[J]. PLoS One, 2013, 10: e77696.
[21] REN C, YAN Q, ZHANG Z. Minimum length of direct repeat sequences required for efficient homologous recombination induced by zinc finger nuclease in yeast[J]. Mol Biol Rep, 2014, 10: 6939-6948.
[22] VERBEKE J, BEOPOULOS A, NICAUD J M. Efficient homologous recombination with short length flanking fragments in Ku70 deficient Yarrowia lipolytica strains[J]. Biotechnol Lett, 2013, 4: 571-576.
[23] AYARES D, CHEKURI L, SONG K Y, et al. Sequence homology requirements for intermolecular recombination in mammalian cells[J]. Proc Natl Acad Sci U S A, 1986, 14: 5199-5203.
[24] BASSETT A R, TIBBIT C, PONTING C P, et al. Mutagenesis and homologous recombination in Drosophila cell lines using CRISPR/Cas9[J]. Biol Open, 2014, 1: 42-49
[25] BEUMER K J, TRAUTMAN J K, MUKHERJEE K, et al. Donor DNA utilization during gene targeting with zinc-finger nucleases[J]. G3 (Bethesda), 2013.
[26] ZU Y, TONG X, WANG Z, et al. TALEN-mediated precise genome modification by homologous recombination in zebrafish[J]. Nat Methods, 2013, 4: 329-331.
[27] WALDMAN A S, LISKAY R M. Dependence of intrachromosomal recombination in mammalian cells on uninterrupted homology[J]. Mol Cell Biol, 1988, 12: 5350-5357.
[28] IRA G, HABER J E. Characterization of RAD51-independent break-induced replication that acts preferentially with short homologous sequences[J]. Molecular And Cellular Biology, 2002, 18: 6384-6392.
[29] DENG C, CAPECCHI M R. Reexamination of gene targeting frequency as a function of the extent of homology between the targeting vector and the target locus[J]. Mol Cell Biol, 1992, 8: 3365-3371.
The effect of the length of donor homologous arm on the efficiency of ZFN-induced homologous recombination
NIEYu,QIAOYanle,CHENYaosheng,HEZuyong
(State Key Laboratory of Biocontrol, School of Life Sciences, Sun Yat-sen University,Guangzhou 510006, China)
Zinc finger nuclease (ZFN) is composed of an engineered site-specific Cys2-His2zinc finger domain and the nonspecific restriction enzymeFokI cleavage domain, which is able to cut at a specified genomic locus to generate double-strand break (DSB) of DNA. The DSBs induced by ZFN are subsequently repaired through two different DNA repair mechanisms, either non-homologous end-joining (NHEJ) or homology-directed recombination (HDR). NHEJ is prone to introduce sequence insertions or deletions (indels), and can therefore produce frameshifts in open reading frames and gene loss of function. HDR requires a donor template with the sequence similar to the genome to mend a lesion. By introducing a DNA donor with desired modifications, precise genomic modifications can be achieved at a frequency improved 102-104-fold as compared to the traditional gene targeting method. Currently, most studies have focused on screening ZFN with higher activity, and improving the delivery efficiency of ZFN and donor into host cells, less studies have investigated the relationship between the homologous arm length with the efficiency of ZFN induced homologous recombination. Here, we constructed a pair of ZFN plasmids targeting to EGFP and verified its cutting activity. Then we designed a series of donors with different lengths of homologous arms. By introducing individual donor with ZFN into CHO cells harboring a frame-shift GFP gene, we measured the homologous recombination efficiencies through the flow cytometric analysis. We found that a 50 bp short homology arm was capable to support ZFN-mediated homologous recombination. Increasing the length of the homologous arms could improve the efficiency of ZFN-mediated homologous recombination. A dramatic improvement (104-fold higher than traditional method) requires a homology arm longer than 1 000 bp.
zinc finger nuclease; double-strand break; homologous recombination; donor; homologous arm
10.13471/j.cnki.acta.snus.2016.04.017
2016-01-26
国家转基因生物新品种培育重大专项资助项目(2016ZX08006003-006);NSFC-广东省联合基金资助项目(U1201213)
聂宇(1989年生),男;研究方向:动物学;通讯作者:何祖勇;E-mail:zuyonghe@foxmail.com
Q78
A
0529-6579(2016)04-0100-08
