类风湿性关节炎患者循环microRNAs生物标志物的筛选及鉴定
2016-06-03彭武建王红蕾黄建溶戴勇深圳市第三人民医院肾内科广东深圳58000南方医科大学附属第三医院肾内科广东广州50000暨南大学第二临床医学院深圳市人民医院临床医学研究中心广东深圳5800
彭武建,王红蕾,黄建溶,戴勇(.深圳市第三人民医院 肾内科,广东 深圳 58000;.南方医科大学附属第三医院 肾内科,广东广州 50000;.暨南大学第二临床医学院 深圳市人民医院 临床医学研究中心,广东 深圳 5800)
类风湿性关节炎患者循环microRNAs生物标志物的筛选及鉴定
彭武建1,王红蕾2,黄建溶1,戴勇3
(1.深圳市第三人民医院 肾内科,广东 深圳 518000;2.南方医科大学附属第三医院 肾内科,广东广州 510000;3.暨南大学第二临床医学院 深圳市人民医院 临床医学研究中心,广东 深圳 518020)
[摘 要]目的:分析类风湿性关节炎(RA)患者血浆中差异表达的microRNAs(miRNAs)。方法:采用miRNA芯片检测、筛选RA患者与正常人血浆中表达有显著变化的miRNAs,并采用RT-qPCR技术对芯片数据进行验证。数据处理采用GeneSpring GX软件,t检验和方差分析用于统计学分析。结果:RA患者与正常人间存在差异的循环miRNAs共有40个,其中18个上调,22个下调。根据循环miRNAs在芯片中表达差异的倍数和潜在的生物学价值,选取4个miRNAs进行RT-qPCR验证,候选的miRNAs与正常对照间差异有统计学意义(P<0.05),证实芯片结果可靠性。结论:RA患者体内具有差异表达的循环miRNAs,这些循环miRNAs可作为一种潜在的RA候选生物标志物。
[关键词]类风湿性关节炎;微小RNA;芯片;生物标志物
类风湿性关节炎(rheumatoid arthritis,RA)是一种以外周滑膜关节慢性炎性病变、关节软骨和骨质进行性不可逆性破坏为特征的全身性自身免疫性疾病[1]。目前RA的诊断采用1987年美国风湿病学会(ACR)修订的诊断分类标准[2],符合此标准的患者常已出现严重的关节破坏,不利于RA的早期诊断。因此,寻找RA特异性的疾病诊断生物标志物尤为重要。
微小RNA(microRNAs,miRNAs)是一类长约18~25 nt的内源性非编码RNA,在转录后水平调控基因的表达[3]。miRNAs参与免疫功能调节过程及自身免疫性疾病的发生与进展[4-6]。在循环系统中同样可以检测到miRNAs的存在,且血清/血浆中miRNAs在不同的疾病状态下存在着差异表达,可作为一类具有潜在价值的用于临床的生物标志物[7-8]。因此,本研究应用miRNA基因芯片检测RA患者与正常对照组的循环miRNAs表达谱,分析2组间差异表达miRNAs,进行生物信息学分析及实时荧光定量PCR(RT-qPCR)验证,为进一步研究RA的发病机制奠定基础。
1 对象和方法
1.1对象 收集深圳市人民医院风湿免疫科35例RA患者,其中男9例,女26例,年龄18~70岁,平均(44.9±11.5)岁。RA诊断符合1987年美国风湿协会修订的RA诊断标准[2]。RA组患者在接受大剂量糖皮质激素、细胞毒性药物及免疫抑制剂治疗前完成标本采集。正常对照组选取30例排除了自身免疫系统疾病、肝肾功能正常的健康体检者,其中男10例,女20例,年龄26~52岁,平均(42.9±8.7)岁。各组间性别、年龄差异无统计学意义,具可比性。先每组取10例,进行芯片初筛,余下病例作为RT-qPCR验证。研究计划经医院伦理委员会批准,受试对象均签署知情同意书。
1.2标本采集及总RNA的抽提和质检 EDTA抗凝管采集新鲜血液,在4 h内以2 000×g离心10 min,吸取上层血浆于冻存管中,-80 ℃冻存。按mirVana PARIS试剂盒(Ambion)说明书从600 μL的血浆中抽提总RNA,紫外分光光度计测量A260/A280值,测定浓度。
1.3混合样本池法检测循环miRNAs 按照等量混合的原则,每组10个样品各取10 ng混合到成100 ng,进行芯片检测。
1.4样品标记及杂交 本实验所用Agilent Human miRNAs microarray Rel 12.0(上海伯豪生物有限公司)涵盖886个人类相关miRNAs以及89个人类病毒相关miRNAs。100 ng总RNA经Cy3荧光基团标记及干燥处理后与芯片杂交20 h(55 ℃,20 r/min)。然后进行芯片洗涤、扫描。
1.5芯片图像采集和数据分析 Agilent Feature Extraction(FE)software version 9.5.3软件提取数据。应用GeneSpring GX软件,以Fold change ≥2,P≤0.05标准,筛选RA患者与正常人血浆中差异表达miRNAs。Fold Change指的是将2组数据均一化后,用信号值直接计算其差异的倍数。应用TargetScan、microRNA.org、PITA数据库,进行靶基因预测及GO和KEGG分析。
1.6RT-qPCR验证 应用RT-qPCR对4个显著性差异表达的miRNAs进行检测。引物序列如下:miR-16:5’-ACACTCCAGCTGGGTAGCAGCACGTAAATATT-3’;miR-155: 5’-ACACTCCAGCTGGGTTAATGCTAATCGTGATA-3’;miR-223:5’-ACACTCCAGCTGGGTGTCAGTTTGTCAAATAC-3’;miR-451:5’-ACACTCCAGCTGGGAAACCGTTACCATTACTG-3’;Cel-lin4:5’-ACACTCCAGCTGGGTCCCTGAGACCTCA AGTG-3’。反应体系:5.0 μL cDNA(1∶20),10 μL 2x SYBR Green PCR Master Mix(TOYOBO),1.0 μL的引物补充RNase-free水至20 μL,以Cel-lin4为内参,利用RT-qPCR仪(ABI PRISM 7 500 Sequence Detection System,ABI USA)进行PCR反应,反应条件:预反应95 ℃ 5 min;95 ℃ 15 s,65 ℃ 15 s,72 ℃ 32 s,共40个循环。采用2-ΔΔct法计算相对表达值[9]。
1.7统计学处理方法 采用SPSS19.0软件进行统计学处理,计量资料以±s表示,2组间比较使用t检验,以P<0.05为差异有统计学意义。
2 结果
2.1循环miRNAs在RA患者血浆中的表达 采用miRNA芯片实验,发现在RA患者和正常人血浆中存在着差异表达的miRNAs共有40个,其中18个上调,22个下调(见表1)。基于目前的研究,根据芯片结果和miRNAs潜在的价值选取4个miRNAs进行第二阶段的验证。
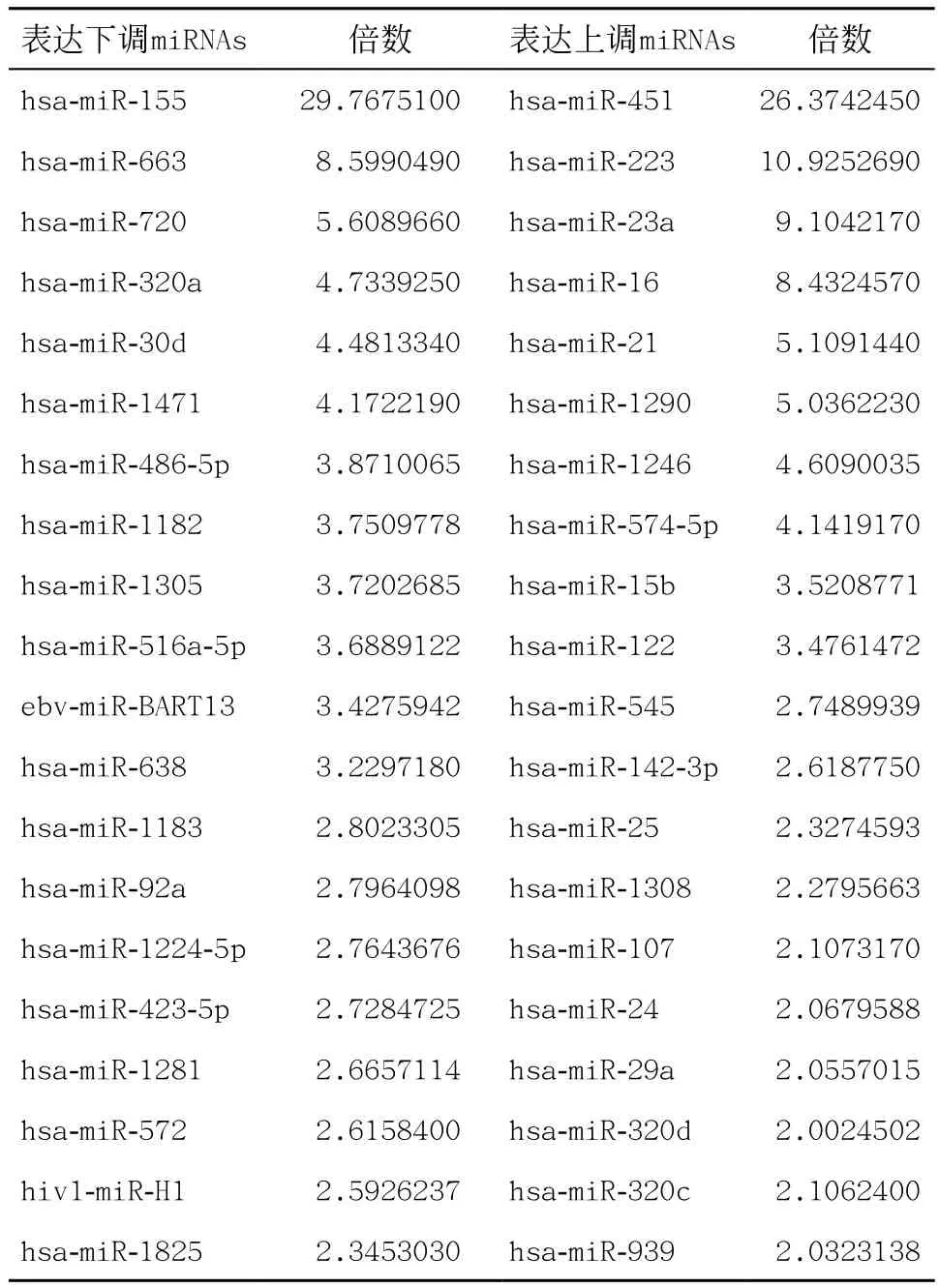
表1 RA患者与正常对照间血浆差异表达的miRNAs
2.2RT-qPCR验证差异表达的miRNAs 采用RT-qPCR检测miR-16、miR-451、miR-223、miR-155在RA患者与正常人血浆中的表达情况(见图1),其中miR-16、 miR-451和miR-223在RA患者血浆中表达量明显高于正常对照组(P<0.05),而miR-155却低于正常对照组(P<0.05)。

图1 循环miRNAs RT-qPCR验证结果
2.3生物信息学结果 为了研究miRNAs的生物学功能,用Target Scan Human Custom(http∶//www. targetsan.org)分别对差异表达的miRNAs进行靶基因预测。利用KEGG数据库对靶基因进行Pathway分析,以P<0.01,至少2个基因存在于该信号传导通路为限制条件,寻找相关信号通路,主要有:癌症相关通路、调节肌动蛋白细胞骨架通路、泛素介导的蛋白降解通路以及趋化因子信号通路等(见表2)[10]。
3 讨论
本实验利用芯片检测结合生物信息学方法,筛选了RA患者与正常人血浆中差异表达的40个miRNAs。通过对这些差异表达的循环miRNAs及可能靶基因进行功能分类,进一步推测其对RA的诊断和治疗潜在的意义。发现在RA患者和正常人血浆中存在着差异表达的miRNAs共有40个,其中18个上调,22个下调。本实验中大部分miRNAs表达与文献[11]报道相符,但有部分miRNAs,如miRNA-146a在本研究中未见表达差异。这可能与芯片敏感性可能是试验误差有关,故需要进行RT-qPCR验证芯片结果。根据芯片中miRNAs的表达倍数和miRNAs潜在表达意义,本实验选择了4个miRNAs进行验证,其中miR-16、miR-451和miR-223在RA患者血浆中的相对表达上调,而miR-155表达下调。候选循环miRNAs验证结果与芯片结果一致,证明了芯片结果的可靠性。

表2 KEGG信号通路表
目前发现miR-451在肿瘤患者中可调节细胞的增殖、浸润和凋亡。研究表明,miR-451抑制通过p38 MAPK抑制中性粒细胞的趋化性,是RA的潜在治疗靶点[12]。然而该研究中RA患者的中性粒细胞中miR-451低表达,而本研究显示循环miR-451高表达不符合,原因尚有待进一步明确。miR-16家族在特定环境中可通过调控cyclin D及CDK等细胞周期相关基因的表达诱导细胞周期G1期阻滞[13]。敲除内源性的miR-16可显著延长TNF-α mRNA的半衰期[14]。miR-16与RA成纤维细胞增殖及滑膜细胞炎症反应密切相关[11]。miR-155被证实参与炎症、免疫和肿瘤的发生、发展多种生理病理过程。miR-155抑制间质金属蛋白酶MMP-1、MMP-1的生成,缓解关节组织破坏。miR-155在RA患者滑液中表达水平升高,通过减少Toll受体配体MMP-3和细胞因子表达来达到控制慢性炎症、减少滑膜组织损害的作用[15-16]。此外,miR-155参与免疫反应,在调节造血干细胞的活化、增殖和分化,淋巴细胞发育,T、B细胞免疫应答过程中发挥作用[17]。miR-155的异常表达可导致淋巴细胞功能紊乱,发生自身免疫性疾病。RA患者滑膜组织中的miR-155高表达,它在滑膜组织中控制破骨细胞的分化,提示参与滑膜炎及骨质破坏[18]。miR-223也在造血干细胞的分化、粒细胞的分化激活中起着至关重要的作用[19]。有研究表明,miR-223参与肾移植后急性排斥反应[20]。本研究显示,RA患者中,其血浆循环miR-16、miR-45、miR-223、miR-155的表达水平显著差异,它们的表达异常与RA是密切相关的,尽管具体作用机制尚不完全明确,但有可能成为RA诊断的分子标志物及治疗靶标。
本研究同时对靶基因进行了GO分析和Pathway分析,提示RA发生发展是个复杂的过程,涉及到多种信号通路,对任一通路的调控都可能对治疗RA起到重要作用。本研究获得的差异表达miRNAs的靶基因信号通路分析显示集中在癌症相关通路、调节肌动蛋白细胞骨架通路、泛素介导的蛋白降解通路以及趋化因子信号通路等。如Focal adhesion信号通路与RA相关[21],该信号通路与黏着斑有关。黏着斑是将细胞外基质与细胞骨架联系起来的多蛋白聚集体,能进行信号传递,引发相应的生理、病理反应。黏着斑激酶与黏着斑结合,在整合素介导的信号传导中发挥作用,它与骨代谢、成纤维细胞的扩增及关节血管翳的生成有关[22]。
综上所述,本研究筛选并验证了RA患者差异表达的循环miRNAs,为进一步研究miRNAs在RA发病机制中的作用及寻找RA特异性诊断生物标志物打下了基础。但本实验采用早期芯片,许多新的miRNAs未包含在内。此外,样本数量有限,仍需要进一步扩大样本量,监测miRNAs在健康人群、RA患者血清的表达来研究验证实验结果的准确性和特异性。
参考文献:
[1]中华医学会风湿病学分会. 类风湿关节炎诊断及治疗指南[J]. 中国风湿病学杂志,2010,14(4): 265-270.
[2]ARNETT F C,EDWORTHY S M,BLOCH D A,et al. The American Rheumatism Association 1987 revised criteria for the classification of rheumatoid arthritis[J]. Arthritis Rheum,1988,31(3): 315-324.
[3]WANG Y,STRICKER H M,GOU D,et al. MicroRNA: past and present[J]. Front Biosci,2007,12:(6): 2316-2329.
[4]SIMPSON L J,ANSEL K M. MicroRNA regulation of lymphocyte tolerance and autoimmunity[J]. J Clin Invest,2015,125(6): 2242-2249.
[5]STYPINSKA B,PARADOWSKA-GORYCKA A. Cytokines and microRNAs as candidate biomarkers for systemic lupus erythematosus[J]. Int J Mol Sci,2015,16(10): 24194-24218
[6]陈芳,陈朝生,章慧娣,等. 系统性红斑狼疮患者外周血B淋巴细胞中狼疮活动相关miRNA的筛选[J]. 温州医学院学报,2013,43(2): 106-111.
[7]MACHADO M T,NAVEGA S,DIAS F,et al. MicroRNAs for peripheral blood fraction identification: Origin,pathways and forensic relevance[J]. Life Sci,2015,15(143): 98-104.
[8]DAI R,AHMED S A. MicroRNA,a new paradigm for understanding immunoregulation,inflammation,and autoimmune diseases[J]. Transl Res,2011,157(4): 163-179.
[9]SILVER N,BEST S,JIANG J,et al. Selection of housekeeping genes for gene expression studies in human reticulocytes using real-time PCR[J]. BMC Mol Biol,2006,7: 33.
[10]HUANG DA W,SHERMAN B T,LEMPICKI R A. Bioinformatics enrichment tools: paths toward the comprehensive functional analysis of large gene lists[J]. Nucleic Acids Res,2009,37(1): 1-13.
[11]冯知涛,李娟,任洁,等. 类风湿关节炎患者外周血miR-146a及miR-16的表达及与病情活动的相关性研究[J]. 南方医科大学学报,2011,31(2): 320-323.
[12]MURATA K,YOSHIYOMI H,FURU M,et al. MicroRNA-451 down-regulates neutrophil chemotaxis via p38 MAPK [J]. Arthritis Rheumatol,2014,66(3): 549-559.
[13]LIU Q,FU H,SUN F,et al. miR-16 family induces cell cycle arrest by regulating multiple cell cycle genes[J]. Nucleic Acids Res,2008,36(16): 5391-5404.
[14]JING Q,HUANG S,GUTH S,et al. Involvement of microRNA in AU-rich element-mediated miRNA instability[J]. Cell,2005,120(5): 623-634.
[15]STANCZYK J,PEDRIOLI D M,BRENTANO F,et al. Altered expression of MicroRNA in synovial fibroblasts and synovial tissue in rheumatoid arthritis[J]. Arthritis Rheum,2008,58(4): 1001-1009
[16]LIU F,FAN H,REN D,et al. TLR9-induced miR-155 and Ets-1 decrease expression of CD1d on B cells in SLE[J]. Eur J Immunol,2015,45(7): 1934-1945.
[17]XIAO B,LIU Z,LI B S,et al. Induction of microRNA-155 during Helicobacter pylori infection and its negative regulatory role in the inflammatory response[J]. J Infect Dis,2009,200(6): 916-925.
[18]SUN W,SHEN W,YANG S,et al. MiR-223 and miR-142 attenuate hematopoietic cell proliferation,and miR-223 positively regulates miR-142 through LMO2 isoforms and CEBP-β[J]. Cell Res,2010,20(10): 1158-1269.
[19]FILKOVO M,ARADI B,SENOLT L,et al. Association of circulating miR-223 and miR-16 with disease activity in patients with early rheumatoid arthritis[J]. Ann Rheum Dis,2014,73(10): 1898-1904.
[20]刘小友,徐健. Micro-RNA-223参与肾移植后急性排斥反应中的作用[J]. 细胞与分子免疫学杂志,2011,27(10):1121-1123.
[21]ATHERTON P,STUTCHBURY B,JETHWA D,et al. Mechanosensitive components of integrin adhesions: Role of vinculin[J]. Exp Cell Res,2015,S0014-4827(15): 30160-30169.
[22]ZHANG M M,JIANG Y S,LV H C,et al Pathway-based association analysis of two genome-wide screening data identifies rheumatoid arthritis-related pathways[J]. Genes Immun,2014,15(7): 487-494.
(本文编辑:吴健敏)
Screening and verification of circulating miRNA biomarkers of rheumatoid arthritis
PENG Wujian1,WANG Honglei2,HUANG Jianrong1,DAI Yong3. 1. Department of Nephrology,the Third People’s Hospital of Shenzhen,Shenzhen,518020; 2.Department of Nephrology,the Third Affiliated Hospital of Southern Medical University,Guangzhou,510000; 3.Clinical Medical Research Center of the Second Clinical Medical College ,Shenzhen People’s Hospital,Jinan University,Shenzhen,518020
Abstract:Objective: To analyze the differentl expression of circulating microRNAs (miRNAs) in patients with rheumatoid arthritis (RA). Methods: MiRNA profiles were performed to determine the differentially expressed miRNAs using RNA obtained from the plasma of patients with RA and healthy controls; to confirm array results,RT-qPCR was used for validation. GeneSpring GX software was to data processing,t-test and one way ANOVA were used for statistical analysis. Results: The array analysis identified 40 (18 upregulated miRNAs and 22 downregulated miRNAs) circulating miRNAs significantly differently expressed between RA and healthy controls,then we detected the candidate miRNAs selected based on fold-change and potential value by RT-qPCR,consistented with the data obtained from the array,the circulating level of candidate miRNAs were significantly differed in patients with RA compared with healthy controls (P<0.05). Conclusion: Differently expressed circulating miRNAs can be detected in patients with RA,this study indicates that circulating miRNAs may be used as a potential candidate biomarker of RA.
Key words:rheumatoid arthritis; microRNAs; array; biomarker
作者简介:彭武建(1976-),男,湖南永顺人,主治医师,博士。
基金项目:深圳市卫生计生系统科研基金资助项目(201402033)。
收稿日期:2016-01-22
[中图分类号]R593.22
[文献标志码]A
DOI:10.3969/j.issn.2095-9400.2016.05.005
