大豆Pre-miRNAs作为内参基因在盐碱胁迫下的表达稳定性分析
2016-06-03周永刚刘伟灿李晰亮邓文波赵利旦王兴超王法微李晓薇董园园李海燕
周永刚,王 骐,刘伟灿,邓 宇,李晰亮,靳 京,邓文波,赵利旦,王兴超,王 南,王法微,李晓薇,董园园,李海燕
(1 吉林农业大学 a生物反应器与药物开发教育部工程研究中心,b生命科学学院,吉林 长春130118;2 东北师范大学 附属中学,吉林 长春 130118)
大豆Pre-miRNAs作为内参基因在盐碱胁迫下的表达稳定性分析
周永刚1a,1b,王骐2,刘伟灿1a,1b,邓宇1a,1b,李晰亮1a,1b,靳京1a,1b,邓文波1a,1b,赵利旦1a,1b,王兴超1a,1b,王南1a,1b,王法微1a,1b,李晓薇1a,1b,董园园1a,1b,李海燕1a,1b
(1 吉林农业大学 a生物反应器与药物开发教育部工程研究中心,b生命科学学院,吉林 长春130118;2 东北师范大学 附属中学,吉林 长春 130118)
[摘要]【目的】 对比大豆Pre-miRNAs候选内参基因和传统内参基因的表达稳定性,筛选出适合盐、碱胁迫条件下RT-qPCR分析的最稳定内参基因。【方法】 选用了6个保守的Pre-miRNAs基因和4个常规的内参基因作为候选内参基因,通过RT-qPCR的方法,利用GeNorm和NormFinder软件,评价了10个候选内参基因在大豆盐、碱胁迫条件下的稳定性。【结果】 大豆盐胁迫下,叶中表达最稳定的基因组合是Pre-miR166和Pre-miR172,表达最稳定的单一基因是FboX;根中表达最稳定的基因组合是Act11和EF1A,表达最稳定的单一基因是Act11。大豆碱胁迫下,叶中表达最稳定的基因组合是Pre-miR393和Pre-miR172,表达最稳定的单一基因是Pre-miR393;根中表达最稳定的基因组合是Act11和60S,表达最稳定的单一基因是Pre-miR172。【结论】 大豆Pre-miRNAs可以与传统内参基因一样作为内参定量目的基因。
[关键词]大豆;盐胁迫;碱胁迫;荧光定量PCR;Pre-miRNAs(前体miRNAs);内参基因
荧光定量PCR是一种快捷、准确、强大的定量分析技术,但其结果的可靠性取决于内参基因是否稳定[1-2]。传统内参基因的选择通常选用构成细胞器骨架基本组分或参与生物体内基本代谢过程稳定表达的基因[3-5]。但是,近年来的研究发现,在不同类型的细胞和组织中、发育的不同阶段以及各种试验条件等情况下,这些稳定表达基因的表达水平通常变异较大。由此发现,传统的内参基因并不能完全满足定量分析对内参基因的要求[6]。因此,新种类内参基因的挖掘仍具有重要意义。
MicroRNA在生物体内存在多种形式,如Pri-miRNA、Pre-miRNA、成熟miRNA。目前已有报道表明,成熟miRNA作为内参基因比传统内参基因具有更高的稳定性,它不仅可用于定量其他的成熟miRNA,也可用于定量编码蛋白基因[6]。而目前有关成熟miRNA基因的前身Pre-miRNA相比传统内参基因稳定性的评价尚未见报道。本研究选用6个保守的大豆Pre-miRNAs基因与4个传统内参基因,利用GeNorm[7]和NormFinder[8]软件,分析了盐、碱胁迫条件下大豆根和叶2种组织中的表达稳定性,并筛选出最适合RT-qPCR的内参基因,为进一步研究大豆盐、碱2种不同胁迫条件下的基因表达差异,以及应用Pre-miRNAs作为新的内参基因资源提供参考。
1材料与方法
1.1植物材料和处理方法
本试验使用大豆(Williams 82)品种作为基础研究材料。用改良的Hoagland培养液培养大豆,待大豆第2对复叶萌发时进行如下胁迫处理:(1)盐胁迫处理:110 mmol/L NaCl;(2)碱胁迫处理:110 mmol/L NaHCO3。各处理胁迫0,3,6,9 h后,分别取根和叶片组织,在液氮中速冻后保存在-80 ℃冰箱,备用。
1.2RNA的提取
采用RNAiso plus(Takara:Code No.9108/9109)试剂对检测样本Total RNA进行提取,使用Nanodrop2000对RNA浓度和质量进行检测,利用1.0%琼脂糖凝胶电泳检测总RNA的完整性。
1.3反转录cDNA
样品反转录采用Takara公司PrimeScriptTMRT reagent Kit with gDNA Eraser-Perfect Real Time试剂盒(Takara:Code No.RR047A),具体操作依据试剂盒说明。将得到的cDNA在-20 ℃保存。
1.4候选看家基因的选择和引物设计
根据文献[9-12]中报道的与大豆相关的传统内参基因和本实验室已有的内参评价数据,选择了4个在大豆中最稳定的传统内参基因(EF1A、FboX、Act11和60S);并尝试选取了6个大豆Pre-miRNA基因(Pre-miR156、Pre-miR166、Pre-miR167、Pre-miR171、Pre-miR172和Pre-miR393)作为候选内参基因。传统内参基因的引物设计可参照常规荧光定量引物的设计原则。Pre-miRNAs基因的引物设计与常规定量引物不同的是考虑了Pre-miRNAs家族各成员的同源性,设计的引物具有家族成员通用性。所有引物均由苏州金唯智生物科技有限公司合成(表1)。
1.5荧光定量PCR
本研究采用Takara公司的SYBR Premix ExTaqTM(Tli RNaseH Plus)试剂盒,其定量部分操作均在Mx3000PTMReal time PCR仪上进行,其操作系统为Stratagene(Mx3000P)。每个样品设置2个重复和NTC对照。经优化后反应体系为:SYBR Premix ExTaqTM(2×)10 μL,PCR 正向引物(10 μmol/L)0.8 μL,PCR 反向引物(10 μmol/L)0.8 μL,Rox Reference DyeⅡ(10 μmol/L)0.4 μL,RT 反应液 2.0 μL,DEPC水 6 μL。两步法PCR扩增标准程序:预变性95 ℃ 30 s;PCR反应95 ℃ 5 s,62 ℃ 20 s,40个循环。熔解曲线95 ℃ 20 min,62 ℃ 30 s,95 ℃ 20 s。

表 1 10个候选基因引物序列及相关数据
注:由于Pre-miRNA基因家族成员间的同源性较高,设计的引物具有基因家族通用性,因此同一对引物在不同家族成员中的扩增产物长度具有差异。
Note:Since the Pre-miRNA gene homology between family members is high,universal primers were designed with gene family.Therefore there are differences in the same primers for different members of the family.
1.6数据分析
引物的扩增效率(E)和相关系数(R2)计算公式为E=10(-1/斜率)-1,可由Mx3000p系统对样本稀释曲线各稀释点的测定结果自动计算得出。候选内参基因的表达稳定性分析采用GeNorm[7]和NormFinder[8]软件。GeNorm软件可以显示逐步去除最不稳定候选内参基因后的各内参基因的平均表达稳定值M,M值越小,说明基因表达越稳定;同时,GeNorm软件可给出内参基因的配对变异数Vn/(n+1)柱形图,该图显示了准确量化时需要选择的内参基因数目。Vn/(n+1)的选择阈值为0.15,当Vn/(n+1)>0.15时,表明需选择n个以上的基因组合作为量化标准;当Vn/(n+1)≤0.15时,表明只需选择n个内参基因作为量化标准。NormFinder的运行原理与GeNorm程序类似, 也是通过计算基因表达稳定值M,然后根据稳定值M的大小排序,M值越大稳定性越差;反之,具有最小稳定值M的基因为最稳定的基因。
2结果与分析
2.1盐碱胁迫后大豆叶和根总RNA质量检测
本研究利用RNAiso plus方法对胁迫后的大豆叶和根的总RNA进行了提取,并用Nanodrop2000对其浓度和质量进行检测,保证提取的RNA OD260/OD280值在1.8~2.2,最后对提取的RNA进行1.0%琼脂糖凝胶电泳,条带清晰,质量完整,无明显降解(图1)。说明提取的RNA可以用于后续的反转录及实时荧光定量PCR,将其稀释至800 ng/μL,放入-80 ℃冰箱待用。
2.2各候选内参基因引物PCR扩增效率及特异性
为了荧光定量分析的准确性,本研究对RT-qPCR反应条件进行了优化,确保了各候选内参基因引物的扩增效率在95%~110%,稀释曲线相关系数R2>0.99(表2),符合RT-qPCR对引物扩增效率的基本要求。为了保证10个候选内参基因的引物特异性,本研究对扩增产物进行了1.0%琼脂糖凝胶电泳,结果显示扩增产物与目的片段大小一致,没有出现引物二聚体(图2);同时10个候选内参基因的熔解曲线,也都只出现单一峰(图3),且不加模板的阴性对照检测不到荧光信号,说明各候选内参基因引物的特异性好。因此,各内参基因引物的扩增特性均符合RT-qPCR的要求。

图 1 大豆总RNA样品的电泳检测结果

基因Gene扩增效率/%PCRefficiency稀释曲线相关系数R2基因Gene扩增效率/%PCRefficiency稀释曲线相关系数R2Act1198.10.997Pre-miR166108.30.996EF1A95.31.000Pre-miR16799.90.99660S100.00.998Pre-miR171106.50.987FboX100.70.999Pre-miR17296.30.999Pre-miR15697.80.990Pre-miR39399.90.995
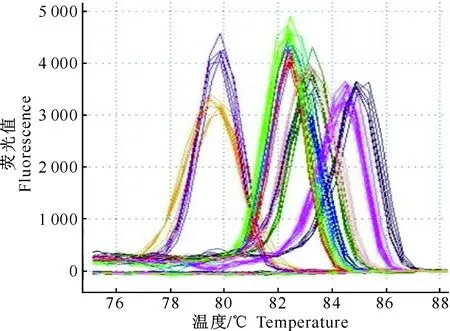
图 3 候选内参基因引物特异性扩增的熔解曲线
2.3大豆碱胁迫下候选内参基因的表达稳定性
为了发掘大豆碱胁迫下稳定表达的内参基因,本研究利用GeNorm和NormFinder软件,分别对大豆不同组织碱胁迫条件下10个候选内参基因的表达稳定性进行了评价。GeNorm分析结果(图4)显示,在碱胁迫下,各候选内参基因在大豆根、叶片中的表达稳定性有所差异。其中在叶片中候选内参基因表达稳定性(根据稳定值M大小排序,M值越小越稳定)由高到低依次为Pre-miR393/Pre-miR172(0.142)>FboX(0.154)>Act11(0.180)>Pre-miR166(0.223)>60S(0.268)>EF1A(0.301)>Pre-miR156(0.336)>Pre-miR171(0.373)>Pre-miR167(0.405);在根中候选内参基因表达稳定性由高到低依次为Act11/60S(0.125)>EF1A(0.234)>FboX(0.286)>Pre-miR167(0.346)>Pre-miR172(0.383)>Pre-miR171(0.431)>Pre-miR156(0.485)>Pre-miR166(0.528)>Pre-miR393(0.577)。
GeNorm柱形图分析结果(图5)显示,所有的Vn/(n+1)值都小于程序所推荐值0.15,说明无需加入第3个基因进行校正,最合适的内参基因数目是2个。叶中表达最稳定的基因组合是Pre-miR393和Pre-miR172,根中表达最稳定的基因组合是Act11和60S。同时,NormFinder分析结果(表3)显示,碱胁迫的叶片中Pre-miR393表达最稳定,而Pre-miR167、EF1A和Pre-miR156表达稳定性差;碱胁迫的根中Pre-miR172表达最稳定,而Pre-miR393、Pre-miR166和Pre-miR156表达稳定性差。
综上所述,通过对比GeNorm和NormFinder分析结果,发现2种软件得到的结果基本一致,不同的是GeNorm软件分析可得到内参基因的最优组合,而NormFinder软件只能选择单个最稳定的内参基因。尽管2种软件部分基因排序稍有不同,但这种差异经证实是由这2种软件计算方法不同引起的[13-18]。

图 4 GeNorm软件分析碱胁迫中内参基因的表达稳定值排序

图 5 GeNorm软件分析确定碱胁迫中用作标准化的最适内参基因数目

稳定性排序Order叶Leaves基因Gene稳定值Stability根Roots基因Gene稳定值Stability稳定性排序Order叶Leaves基因Gene稳定值Stability根Roots基因Gene稳定值Stability1Pre-miR3930.045Pre-miR1720.0956Pre-miR1660.19960S0.2772Act110.108FboX0.1667EF1A0.258Pre-miR1670.3273Pre-miR1720.109EF1A0.2138Pre-miR1560.277Pre-miR1560.3414FboX0.129Act110.2419Pre-miR1710.319Pre-miR1660.477560S0.182Pre-miR1710.26810Pre-miR1670.340Pre-miR3930.484
2.4大豆盐胁迫下候选内参基因的表达稳定性
为了发掘大豆盐胁迫下稳定表达的内参基因,本研究利用GeNorm和NormFinder软件分别对大豆不同组织盐胁迫条件下,10个候选内参基因的表达稳定性进行了评价。GeNorm分析结果(图6)显示,在盐胁迫下叶中候选内参基因表达稳定性从高到低依次是Pre-miR166/Pre-miR172(0.076)>FboX(0.097)>Pre-miR171(0.146)>Pre-miR167(0.171)>Pre-miR393(0.213)>Act11(0.248)>60S(0.275)>EF1A(0.301)>Pre-miR156(0.346);根中候选内参基因稳定性从高到低依次是Act11/EF1A(0.038)>60S(0.093)>FboX(0.122)>Pre-miR172(0.145)>Pre-miR156(0.166)>Pre-miR393(0.187)>Pre-miR166(0.240)>Pre-miR171(0.301)>Pre-miR167(0.414)。同时,GeNorm柱形图分析结果(图7)显示,所有的Vn/(n+1)值都小于程序所推荐值0.15,说明无需加入第3个基因进行校正,最合适的内参基因数目是2个。叶中表达最稳定的基因组合是Pre-miR166和Pre-miR172,根中表达最稳定的基因组合是Act11和EF1A。NormFinder分析结果(表4)显示,盐胁迫的叶片中候选内参基因FboX表达最稳定,而Pre-miR156、Pre-miR167和EF1A表达稳定性差;盐胁迫的根中Act11表达最稳定,而Pre-miR166、Pre-miR171和Pre-miR156表达稳定性差。综上所述,通过对比GeNorm和NormFinder分析结果,发现2种软件得到的结果基本一致。

图 6 GeNorm软件分析盐胁迫中内参基因的表达稳定值排序
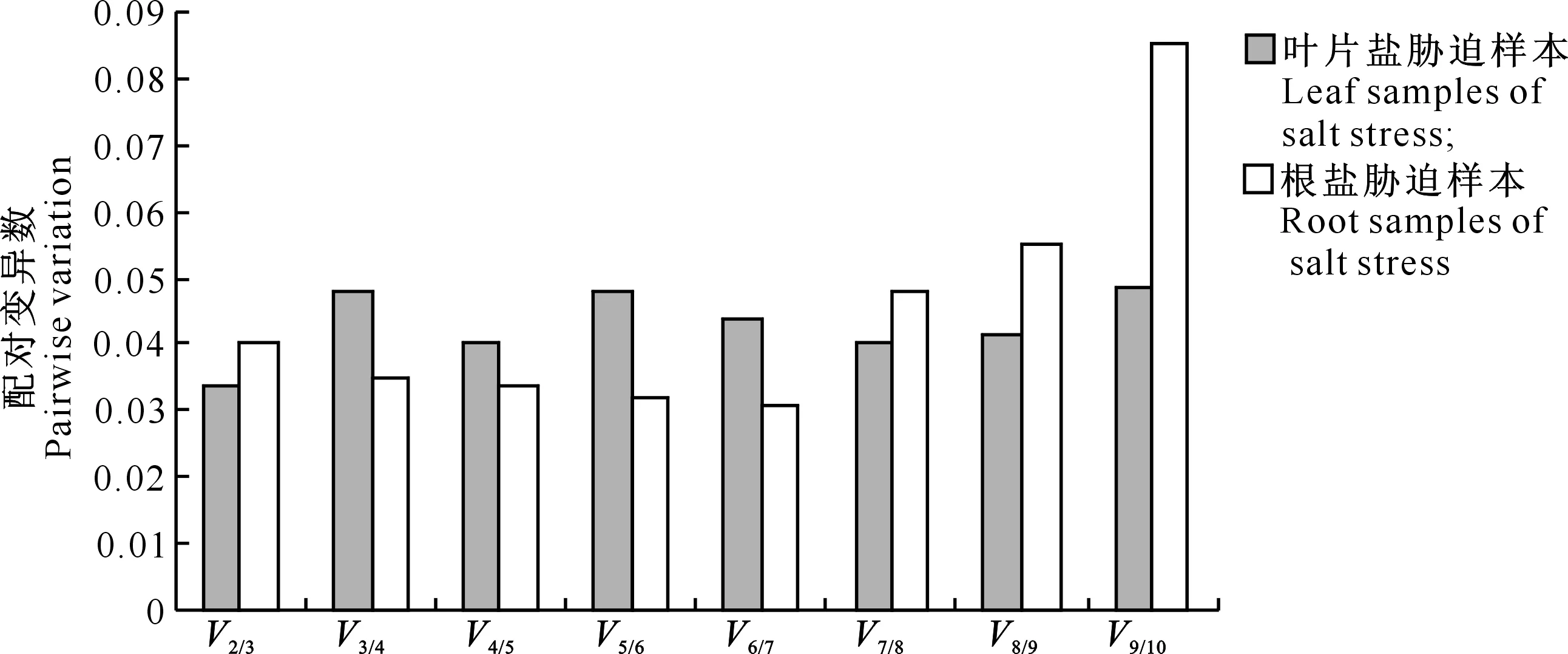

图 7 GeNorm软件分析确定盐胁迫中用作标准化的最适内参基因数目
3结论与讨论
荧光定量PCR是一个功能强大的定量分析技术,为了确保试验结果的准确性和可靠性,RT-qPCR需要选用合适的内参基因作为基因表达分析时衡量目的基因表达量的参照标准,以消除不同样品间可能存在的差异,通常选用的内参基因是表达较为稳定的看家基因[18]。但是,近年来大量的研究结果表明,并没有绝对稳定表达的基因,任何一种内参基因的稳定只是在一定试验条件下的相对稳定[6,19]。因此,如何选择一个合适的内参基因就显得尤为重要。
本研究选用了6个保守的大豆Pre-miRNAs基因和4个传统内参基因作为候选内参基因,利用GeNorm和NormFinder软件,分析了10个候选内参基因在大豆盐、碱胁迫下根和叶2种组织中的表达稳定性,结果显示,大豆盐胁迫下,叶中表达最稳定的基因组合是Pre-miR166和Pre-miR172,表达最稳定的单一基因是FboX,根中表达最稳定的基因组合是Act11和EF1A,表达最稳定的单一基因是Act11;大豆碱胁迫下,叶中表达最稳定的基因组合是Pre-miR393和Pre-miR172,表达最稳定的单一基因是Pre-miR393,根中表达最稳定的基因组合是Act11和60S,表达最稳定的单一基因是Pre-miR172。值得注意的是,Pre-miRNAs在大豆盐、碱胁迫的叶中表达稳定性优于传统内参基因,分析原因可能有以下几个方面:其一,Pre-miRNAs作为小分子RNA受RNA降解影响更小;其二,Pre-miRNAs比传统的内参基因表达丰度较低,更适用于定量分析表达丰度较低的目标基因[12,19];其三,Pre-miRNAs基因为小分子RNA,其反转录必须使用随机引物,而传统看家基因反转录常使用oligodT,这可能会使二者的反转录效率不同而影响基因表达水平的可比性。
综上所述,Pre-miRNAs基因作为新的内参基因资源为研究者提供了新的参考。另外,Pre-miRNAs作为内参基因可使用与编码蛋白基因定量分析相同的普通定量试剂体系,不增加技术困难和成本。因此,进一步开发和研究Pre-miRNAs内参基因具有重要意义。
[参考文献]
[1]Udvardi M K,Czechowski T,Scheible W R.Eleven golden rules of quantitative RT-PCR [J].Plant Cell,2008,20:1736-1737.
[2]Le D T,Aldrich D L,Valliyodan B,et al.Evaluation of candidate reference genes for normalization of quantitative RT-PCR in soybean tissues under various abiotic stress conditions [J].PLoS One,2012,7(9):e46487.
[3]Stolf-Moreira R,Lemos E G M,Carareto-Alves L,et al.Transcriptional profiles of roots of different soybean genotypes subjected to drought stress [J].Plant Molecular Biology Reporter,2011,29:19-34.
[4]Tran L S,Quach T N,Guttikonda S K,et al.Molecular characterization of stress-inducible GmNAC genes in soybean [J].Mol Genet Genomics,2009,281:647-664.
[5]Le D T,Nishiyama R,Watanabe Y,et al.Genome-wide expression profiling of soybean two-component system genes in soybean root and shoot tissues under dehydration stress [J].DNA Res,2011,18:17-29.
[6]Franceli R K,Francismar C M,Alexandre L N,et al.The use of microRNAs as reference genes for quantitative polymerase chain reaction in soybean [J].Analytical Biochemistry,2010,406(2):185-192.
[7]Jo V,Katleen D P,Filip P,et al.Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes [J].Genome Biol,2002,3(7):research0034.1-research0034.11.
[8]Andersen C L,Jensen J L.Normalization of real-time quantitative reverse transcription-PCR data:A model-based variance estimation approach to identify genes suited for normalization,applied to bladder and colon cancer data sets [J].Cancer Res,2004,64(15):5245-5250.
[9]Libault M,Thibivilliers S,Bilgin D D.Identification of four so-ybean reference genes for gene expression normalization [J].Plant Genome,2008,7:44-54.
[10]Hu R,Fan C,Li H,et al.Evaluation of putative reference genes for gene expression normalization in soybean by quantitative real-time RT-PCR [J].BMC Mol Biol,2009(10):93.
[11]Nicot N,Hausman J F,Hoffmann L,et al.Housekeeping gene selection for real-time RT-PCR normalization in potato during biotic and abiotic stress [J].J Exp Bot,2005(56):2907-2914.
[12]Dheda K,Huggett J F,Bustin S A,et al.Validation of housekeeping genes for normalizing RNA expression in real-time PCR [J].Biotechniques,2004,37:112-119.
[13]Jian B,Liu B,Yu R B,et al.Validation of internal control for gene expression study in soybean by quantitative real-time PCR [J].BMC Mol Biol,2008(9):59.
[14]Víviande J M,Roberta R C,Antnio A B V,et al.Validation of reference genes aiming accurate normalization of qPCR data in soybean upon nematode parasitism and insect attack [J].BMC Research Notes,2013,6:196.
[15]Sunkar R,Chinnusamy V,Zhu J,et al.Small RNAs as big players in plant abiotic stress responses and nutrient deprivation [J].Trends Plant Sci,2007(12):301-309.
[16]Voinnet O.Origin,biogenesis,and activity of plant microRNAs [J].Cell,2009(136):669-687.
[17]Voinnet O.Post-transcriptional RNA silencing in plant-microbe interactions:A touch of robustness and versatility [J].Curr Opin Plant Biol,2008(11):464-470.
[18]Ma S,Niu H,Liu C,et al.Expression stabilities of candidate reference genes for RT-qPCR under different stress conditions in soybean [J].PLoS One,2013,8(10):e75271.
[19]de Oliveira L A,Breton M C,Bastolla F M,et al.Reference genes for the normalization of gene expression in eucalyptus species [J].Plant Cell Physiol,2012,53(2):405-422.
Stability of Pre-miRNAs in soybean as reference genes for quantitative polymerase chain reaction under salt and alkali stresses
ZHOU Yong-gang1a,1b,WANG Qi2,LIU Wei-can1a,1b,DENG Yu1a,1b,LI Xi-liang1a,1b,JIN Jing1a,1b,DENG Wen-bo1a,1b,ZHAO Li-dan1a,1b,WANG Xing-chao1a,1b,WANG Nan1a,1b,WANG Fa-wei1a,1b,LI Xiao-wei1a,1b,DONG Yuan-yuan1a,1b,LI Hai-yan1a,1b
(1 aMinistryofEducationEngineeringResearchCenterofBioreactorandPharmaceuticalDevelopment,bCollegeofLifeSciences,JilinAgriculturalUniversity,Changchun,Jilin130118,China;2HighSchoolAttachedtoNortheastNormalUniversity,Changchun,Jilin130118,China)
Abstract:【Objective】 This study selected the appropriate reference genes for RT-qPCR by comparing the stability of candidate precursor miRNAs and traditional housekeeping genes in different tissues of soybean under different stress treatments.【Method】 Six conservative Pre-miRNAs genes and four regular housekeeping genes in soybean were selected as candidate reference genes to evaluate their expression stability under salt and alkali stresses during RT-qPCR process using GeNorm and NormFinder.【Result】 In leaves,the most stable combination of genes under salt stress was Pre-miR166 and Pre-miR172 whereas the best single gene under this stress was FboX.While,in roots,the most stable combination of genes under salt stress was Act11 and EF1A whereas the best single gene was Act11.Under alkali stress conditions,the most stable combination of genes in leaves was Pre-miR393 and Pre-miR172 whereas the best single gene was Pre-miR393.The most stable combination of genes in roots was Act11 and 60S whereas the best single gene was Pre-miR172.【Conclusion】 Precursor miRNAs reference genes can be used as reference genes together with traditional housekeeping genes.
Key words:soybean;salt stress;alkali stress;RT-qPCR;Pre-miRNAs;reference gene
[文章编号]1671-9387(2016)01-0061-07
[中图分类号]Q-03;S-3
[文献标志码]A
[通信作者]李海燕(1971-),女,吉林长春人,教授,博士,博士生导师,主要从事植物抗逆分子生物学与基因工程研究。
[作者简介]周永刚(1989-),男,吉林图们人,在读硕士,主要从事植物抗逆分子生物学与基因工程研究。
[基金项目]国家转基因生物新品种培育重大专项(2014ZX08010-002);国家自然科学基金项目(31271746,31201144);吉林省发改委项目(JF2012C002-04)
[收稿日期]2014-05-20
DOI:网络出版时间:2015-12-0214:2510.13207/j.cnki.jnwafu.2016.01.010
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20151202.1425.020.html
E-mail:31789632@qq.com
E-mail:hyli99@163.com
