水蕨血红蛋白基因的分子克隆和序列分析
2016-05-09齐小琼葛亚飞李大卫
齐小琼,葛亚飞,李大卫
(1.湖北第二师范学院化学与生命科学学院/植物抗癌活性物质提纯与应用湖北省重点实验室,武汉430205;2.中国科学院武汉植物园植物种质创新与特色农业重点实验室,武汉430074)
水蕨血红蛋白基因的分子克隆和序列分析
齐小琼1,葛亚飞1,李大卫2∗
(1.湖北第二师范学院化学与生命科学学院/植物抗癌活性物质提纯与应用湖北省重点实验室,武汉430205;2.中国科学院武汉植物园植物种质创新与特色农业重点实验室,武汉430074)
植物血红蛋白(Hemoglobin)是一类由珠蛋白(Globin)和血红素(Ferroheme)组成的结合蛋白,在植物中广泛分布,迄今已在苔藓植物、裸子植物和被子植物中克隆到血红蛋白基因序列,但在蕨类植物中相关研究还未见报道。该研究采用热不对称交错PCR(TAIL-PCR)方法克隆了水蕨血红蛋白基因的全长序列。该基因的序列总长为949 bp,包含4个外显子和3个内含子,编码189个氨基酸。预测的蛋白质(命名为CtHb)的分子量为21.14 kDa,等电点(pI)为7.81。三维结构模拟表明CtHb具有植物血红蛋白典型的三级结构:即含有A、B、C、E、F、G和H螺旋,形成了3-on-3的“三明治”结构。和水稻血红蛋白的三级结构相比,CtHb的大部分结构(包括具有远端和近端组氨酸定位的E螺旋和F螺旋的位置等)同水稻的结构极为相似。两者的不同之处主要表现在:(1)CtHb含有较长的N-端区域;(2)两者CD-loop的折叠方式不同;(3)两者螺旋B和螺旋C的连接方式不同,CtHb是通过卷曲连接的,而水稻中借助的是螺旋。结构进化分析揭示了植物血红蛋白从非共生到共生进化过程中的一些关键改变,这些改变可能有助于非共生血红蛋白向共生血红蛋白结构的转变,特别是有助于豆血红蛋白共生功能的实现。
植物血红蛋白,序列分析,三级结构模拟,结构进化,水蕨
∗通讯作者:李大卫,博士,副研究员,主要从事果树遗传育种方向,(E-mail)david.lee1983@163.com。
植物血红蛋白(Hemoglobin)是一类由珠蛋白(Globin)和血红素(Ferroheme)组成的结合蛋白,广泛存在于植物界,在植物固氮及抵御胁迫中具有重要作用。随着研究的不断深入,根据血红蛋白的序列、表达方式以及配体结合性质把植物中的血红蛋白分为三类:共生的血红蛋白(Symbiotic hemoglobin,sHb)、非共生的血红蛋白(Nonsymbiotic hemoglobin,NsHb)和截短的血红蛋白(Truncated he-moglobin,tHb)。共生的血红蛋白主要存在于豆类和一些非豆类植物(主要是放线菌结瘤植物和榆科植物某些属)固氮根瘤菌侵染的根瘤细胞中。非共生的血红蛋白比共生的血红蛋白分布更广泛,不仅存在于含有共生血红蛋白的植物中,而且也存在于其他植物中;并可在多种器官中表达,如正常生长条件和胁迫条件下植物的根、茎、叶、花和种子中(An-derson et al,1996;Lira-Ruan et al,2001;Ross et al,2004;Taylor et al,1994;Trevaskis et al,1997)。截短的血红蛋白首先在原核生物、原生动物和真核藻类中发现,后来在一些植物中也发现了该蛋白(Watts et al,2001)。其序列比其他血红蛋白短(Pesce et al,2000),三维结构是由α-螺旋通过2-on-2的方式排列而成,不同于3-on-3标准的血红蛋白折叠。通过系统发育分析表明,植物中的tHb与sHb、NsHb形成了不同的谱系(Lineages),tHb是通过基因水平转移从细菌中获得(Vinogradov et al,2006),具体的功能还不是很清楚。
血红蛋白基因在植物中普遍存在,就目前积累的数据来看,大部分序列来自于被子植物,在裸子植物和苔藓植物中有几条代表序列,但蕨类和石松类植物中尚未见有相关报道,不完全的序列数据影响了该基因家族的分子进化研究。本研究通过分子克隆手段获得蕨类植物水蕨的血红蛋白基因的完整编码区序列,并进行生物信息学分析、三级结构模拟及进化分析,试图阐明该基因在序列组成、基因结构、三级结构等方面的特征,并推断其可能的功能;通过与其他分类群中的血红蛋白基因进行比较分析,探讨其在不同分类群中较独特的进化样式。
1 材料与方法
1.1 植物材料
蕨类植物水蕨的健康幼嫩叶片采自中国科学院武汉植物园。
1.2 主要分子生物学试剂
普通Taq DNA聚合酶、高保真LA Taq酶、dNTP等试剂购自TaKaRa;DNA凝胶纯化回收试剂盒购于Axygen生物技术有限公司。
1.3 试验方法
1.3.1 水蕨血红蛋白基因部分片段的扩增 分别用拟南芥(Arabidopsis thaliana)AHB1和AHB2基因(登录号:AAD26949,AAB82770)的蛋白序列作为查询序列,通过对数据库中已报道的蕨类序列进行相似性搜索,在Ceratopteris richardii中得到一条同目标序列相似性较高的EST序列(登录号: BQ087262)。根据这条序列设计一对特异引物,进行PCR扩增。引物序列为F-cera:5′-TCACCGAGC-ATCTGCGTCAAT-3′;R-cera:5′-TCAATAGGCAGTA-CCTGACGA-3′。
PCR反应体系为20 μL,引物各40 pmol,dNTP 各1 pmol,10×PCR反应缓冲液2.0 μL,Taq酶0.2 μL(5 U/μL)。反应条件为95℃预变性5 min;接着94℃变性50 s,53℃退火50 s,72℃延伸60 s,共36个循环;最后72℃延伸8 min。
1.3.2 水蕨血红蛋白基因3′侧翼序列的扩增 以提取的总DNA为模板,用3个巢式单引物进行3轮TAIL-PCR扩增,引物序列为3′TAIL-1:5′-TCTTAAT-GTGAGTTCGCCTCTGTGA-3′;3′TAIL-2:5′-CTTTGAG-CAGCACAGGAATGGTCTC-3′;3′TAIL-3:5′-CGGTG-TAGTGGATGAGCATTATGAG-3′。
TAIL-PCR的反应体系为50 μl,简并引物AD (5′-TC(G/C)TICGNACIT(A/T)GGA-3′) (Liu and Whittier,1995)。各成分及用量如下:LA buffer (Mg2+plus)5.0 μL;dNTP Mix(2.5 mmol/L)8.0 μL;AD引物(20 μmol/L)5.0 μL;基因特异性引物(5 μmol/L)2.0 μL;LA Taq酶(5 U/μL)0.5 μL;DNA (50 ng/μL)5.0 μL;H2O 24.5 μL。
第一轮反应引物采用3′TAIL-1和AD,条件为94℃预变性1 min,98℃预变性1 min;接着94℃变性30 s,65℃退火1 min,72℃延伸2 min,共5个循环;紧接着94℃变性30 s,25℃退火3 min,在3 min内攀升到72℃,72℃延伸2 min,进行一个循环;接着94℃变性30 s,65℃退火1 min,72℃延伸2 min,94℃变性30 s,65℃退火1 min,72℃延伸2 min,94℃变性30 s,44℃退火1 min,72℃延伸2 min,共15个循环;最后72℃延伸10 min。TAIL-PCR第二轮反应引物采用3′TAIL-2和AD,将第一轮反应产物稀释50倍,取1 μL作为第二轮反应的模板,其它成分与第一轮反应相同。反应条件为94℃预变性1 min;接着94℃变性30 s,65℃退火1 min,72℃延伸2 min,94℃变性30 s,65℃退火1 min,72℃延伸2 min,94℃变性30 s,44℃退火1 min,72℃延伸2 min,共15个循环;最后72℃延伸10 min。第三轮反应引物采用3′TAIL-3和AD,将第二轮反应产物稀释50倍,取1 μL作为第三轮反应的模板,其它成分与第二轮反应相同。反应条件也同第二轮相同。
1.3.3 产物回收、连接、转化、克隆、测序 扩增片段凝胶回收采用凝胶回收试剂盒,参照说明书进行;产物连接采用TaKaRa公司pMD19-T simple Vector,参考说明书,反应体系稍作优化。大肠杆菌感受态细胞制备采用CaCl2法,将目的基因通过热激法转化到大肠杆菌感受态细胞中,阳性克隆采用蓝白斑筛选方法并进一步采用PCR方法,扩增产物长度与目的片段相当者为阳性克隆。每个目的片段,随机选取至少3个阳性克隆,送华大基因测序。序列测定采用ABI PRISM 3730 DNA测序仪用BigDye Termi-nator Kit(Applied Biosystems)进行双向克隆测序。
1.4 数据分析
采用BioEdit软件(http://www.mbio.ncsu.edu/BioEdit/bioedit.html) (North Carolina State University)对克隆测序得到的片段进行拼接。采用FGENESH和NCBI(http://linux1.softberry.com/berry.phtml)的ORF finder进行开放阅读框确定。运用DNAstar软件推测氨基酸序列,分析蛋白质的分子量、等电点和氨基酸组成。使用SignalP 3.0 Server(http://www.cbs.dtu.dk/services/SignalP/)分析蛋白N-端氨基酸序列,根据SignalP分析得到的Cmax值(the cleavage site score)、Smean(the signal peptide score)和HMM值(Hidden Markov Model)判别信号肽序列,确定是否存在信号肽。使用TargetP Server v1.01(http://www.cbs.dtu.dk/services/Tar-getP/)确定分泌蛋白的分泌途径和亚细胞定位。蛋白质保守结构功能域用PROSITE、SMART软件和PFAM数据库(http://pfam.wustl.edu/)进行预测。多条序列的一致性分析用Bioedit软件sequence i-dentity matrix进行。使用在线软件SOPMA预测蛋白二级结构并计算各种二级结构所占的百分比。将推导的氨基酸序列提交瑞士生物信息研究所的Swiss-model(http://www.isb-sib.ch/)(Arnold et al,2006;Schwede et al,2003),基于同源建模的原理,用水稻的NsHb1作为模板(PDB code:1D8UA)进行水蕨血红蛋白三级结构的预测。预测结果用Py-MOL软件展示和编辑。
运用Blast工具(http//blast.ncbi.nlm.nih.gov/Blast.cgi),以水蕨血红蛋白作为查询序列,进行序列相似性搜索,并选取相似性较高的序列用MEGA6软件(Tamura et al,2013)中的邻接算法进行进化树的构建,其中多重序列比对采取Clustalx软件进行。
2 结果与分析
2.1 水蕨血红蛋白基因的克隆
以水蕨的总DNA为模板,用一对特异引物(F-cera和R-cera)扩增得到一段约820 bp的片段(图 1:a)。经过分析,该序列的5′端是完整的,因此只需通过染色体步移技术获得3′侧翼序列。基于获得的序列片段,进一步设计了一组巢式引物通过TAIL-PCR来获得3′侧翼序列。在TAIL-PCR的第二轮和第三轮分别获得了几条清晰的条带,根据巢式引物设计的相对位置,选取这两轮反应的产物大小相差大约100 bp的条带(图1:b)作为目的条带。将第三轮的目的条带回收、克隆测序,经分析,得到的序列含有同已知序列完全一致的重叠区。将上述实验获得的序列拼接最终得到水蕨血红蛋白基因的完整编码区序列。

图1 特异PCR(a)、TAIL-PCR(b)琼脂糖电泳检测M.2000 bp分子量标记;a 1.特异引物F-cera和R-cera扩增得到的大约820 bp的产物;b II.引物3′TAIL-2通过TAIL-PCR第二轮反应得到的条带;b III.引物3′TAIL-3通过TAIL-PCR第三轮反应得到的条带。Fig.1 Agarose gel electrophoresis of the specific PCR(a),TAIL-PCR(b)products M.Molecular weight marker(DL2000);a 1.About 820 bp fragment generated by specific PCR with primer F-cera and R-cera;b II.Several bands produced by the secondary reaction of TAIL-PCR with primer 3′TAIL-2;b III.Two clear bands produced by the tertiary reaction of TAIL-PCR with primer 3′TAIL-3.
将得到的序列同C.richardii的EST(登录号: BQ087262)序列进行比对,发现本研究获得的序列比数据库中的序列多了三段区域,其它部分都完全匹配,推测这三部分是基因的内含子区域。由此获知该基因含有4个外显子,3个内含子,这与其他植物血红蛋白的基因结构相同。说明血红蛋白基因的外显子-内含子结构在植物进化中是保守的。

表1 序列一致性分析Table 1 Analysis of percent sequence identify
2.2 全长基因序列及编码蛋白的基本性质分析
对已获得的克隆基因进行分析研究后,得到水蕨血红蛋白(命名为CtHb)基因序列全长(图2)为949 bp,该基因包括4个外显子和3个内含子,编码189个氨基酸。氨基酸组成中含量最高的为丙氨酸(Ala,11.6 ),其次缬氨酸(Val,9.0 )、丝氨酸(Ser,8.5 )、亮氨酸(Leu,7.9 )和赖氨酸(Lys,7.9 )。碱性氨基酸(Arg,Lys)24个,强酸性氨基酸(Asp,Glu)23个,疏水氨基酸(Ala,Ile,Leu,Phe,Trp,Val)71个,不带电荷的极性氨基酸(Asn,Cys, Gln,Ser,Thr,Tyr)41个。预测的蛋白质分子量为21.14 kDa,理论等电点(pI)为7.81。

图2 水蕨血红蛋白基因的核酸序列和推导的氨基酸序列 推导的氨基酸序列标注在开放阅读框下面,小写字母示内含子部分。Fig.2 Nucleotide and deduced amino acid sequences of hemoglobin gene from Ceratopteris thalictroides The predicted amino acid sequence is shown below its open reading frame,and the introns are present in lowercase letters.
2.3 蛋白质的信号肽预测及亚细胞定位分析
采用SignalP对该蛋白序列及其他分类群的代表序列(包括苔藓植物2条(Physcomitrella patens和Ceratodon purpureus)、石松类1条(Selaginellae moel-lendorfii)和被子植物11条(具体的物种和序列名称见表1)进行在线搜索,发现这些蛋白存在信号肽的概率都极低,不存在信号肽酶切位点,说明这些血红蛋白很可能为非分泌蛋白。进一步分析这些蛋白的亚细胞定位,发现大部分蛋白是细胞质蛋白,只有小立碗藓和江南卷柏的血红蛋白具有较高分值的叶绿体定位信号,可能会定位于叶绿体中。
2.4 序列一致性分析
表1给出的是15条植物血红蛋白序列两两之间的一致性指数,其中包括2条苔藓植物序列(Phy- scomitrella patens和Ceratordon purpureus),1条石松类植物序列(Selaginellae moellendorfii),1条蕨类植物序列(Ceratopteris thalictroides)及11条被子植物序列。本研究发现CtHb与被子植物NsHb1的序列一致性偏高,与豆科植物sHb的序列一致性偏低,说明CtHb属于NsHb1;同时比较CtHb和其他分类群NsHb的一致性发现,与古老的苔藓、石松类植物的NsHb序列相比,CtHb在进化上更类似于被子植物NsHb。
2.5 水蕨血红蛋白高级结构预测
在二级结构预测中,CtHb主要由61.38 的α-螺旋,6.35 的延伸片层,5.82 的β-转角和26Ʊ 46 无规卷曲组成。其中α-螺旋和卷曲是CtHb二级结构的主要构成成分。
了解水蕨血红蛋白的结构性质对于阐明植物血红蛋白的进化多样性及其在植物中发挥的作用都有
着重要的意义。通过计算机模拟可以从模板预测出高质量的蛋白质三级结构,通过预测出的结构可以考察血红蛋白的进化模式,在结构和功能方面理解进化问题。由于CtHb和水稻NsHb1具有接近50的相似性,所以可以用水稻NsHb1(1D8UA)作为预测CtHb三级结构的模板。

图3 a.预测的CtHb三级结构;b.选取的CtHb血红素口袋中的氨基酸三级结构 每个螺旋分别用字母A,B,C,E,F和H表示。Fig.3 a.Predicted tertiary structure of CtHb;b.Tertiary structure of selected amino acids in heme pocket of CtHb Helices(in-cluding to prehelix A)are indicated with letters A,B,C,E,F and H.

图4 A.CtHb(灰色表示)和水稻NsHb1(黑色表示)三级结构比对图;B.选取在CtHb(灰色表示)和水稻NsHb1(黑色表示)血红素口袋中的氨基酸位点的三级结构比对图 每个螺旋分别用A,B,C,E,F和H表示,其中(a)为正面,(b)为背面。Fig.4 A.Overlay of predicted tertiary structure of CtHb(gray)and native rice NsHb1(black)tertiary structures;B.Tertiary struc-ture overlay of selected amino acids in heme pocket of CtHb(gray)and native rice NsHb1(black) Helices(including to prehelix A)are indicated with letters A,B,C,E,F and H. (a)is a front view,while(b)is a back view.
图3给出了CtHb三维结构模拟结果。图中显示,CtHb具有植物血红蛋白典型的三级结构模式,即含有A、B、C、E、F、G和H螺旋,形成了3-on-3的“三明治”结构。将CtHb和水稻NsHb1的三级结构进行overlay比较,结果如图4:A所示:预测得到的CtHb三级结构具有典型球蛋白的折叠方式,它的大部分结构(包括具有远端和近端组氨酸定位的E和F螺旋的位置等)同水稻的结构非常相似;不同之处主要表现在:(1)CtHb含有较长的N-端区域;(2)两者CD-loop的折叠方式不同;(3)两者螺旋B和螺旋C的连接方式不同,CtHb是通过卷曲连接的,而水稻NsHb1借助的是螺旋。
在比对图5中,发现苔藓植物、石松类和蕨类植物NsHb中都含有一段延长的N-端序列,Ross et al (2002)在苔藓植物NsHb中检测到了类似于前导肽的蛋白位点,推测植物NsHb的祖先可能是定位于细胞器中的,这同软件预测的小立碗藓NsHb的亚细胞定位结果一致。推测这段延长的N-端氨基酸可能有助于蛋白的转运(Garrocho-villegas&Arre-dondo-peter,2008)。在CtHb中,A螺旋之前存在较长的一段序列,可能与苔藓植物延长的N-端氨基酸有着相似的作用,即与蛋白的转运有关,但信号肽和亚细胞定位预测都没有支持这一推断。这可能与采用的生物信息学的预测方法有关,需要进一步的实验证据来证明。如果这些都被验证是正确的,那么从进化的角度讲,NsHb可能从细胞器的蛋白进化成了细胞质的蛋白(Ross et al,2002)。

图5 植物血红蛋白的多序列比对 序列的保守性通过ESPript化学等价策略进行可视化,设置相似性阈值来强调严格保守的位置;不变残基添加黑色背景,理化性质相同的残基用灰色矩形框标注;预测的CtHb二级结构元件标注在序列比对的最上面。Fig.5 Multiple alignment of plant hemoglobins Sequence conservation is visualized according to the ESPript chemical equivalence measure,with a similarity threshold set to emphasize strictly conserved positions;Invariant residues are boxed in black,physico-chemical equivalent residues are boxed in gray;The deduced secondary structure of CtHb is shown above the alignment.
本研究也检测了参与配基结合的一些重要位点的氨基酸组成和相对位置(图4:B),结果表明近端组氨酸(His),远端组氨酸,螺旋B第10位的苯丙氨酸(Phe),CD-loop第1位的苯丙氨酸及172/150的酪氨酸(Tyr)在水蕨和水稻NsHb1中都非常一致,位置完全吻合。这表明CtHb具有水稻NsHb1类似的配基结合动力学性质,即具有很低的O2解离常数,很高的O2亲和力。
4.6 血红蛋白的序列和结构进化
通过将CtHb与其他分类群的代表序列比较发现(图5):CtHb包含非常保守的近端组氨酸(H95),远端组氨酸(H130)和位于B螺旋第10位的苯丙氨酸(F62);同时也发现,在CD-loop的第1位上只有苔藓植物是酪氨酸(Tyr)而其他物种都是苯丙氨酸(Phe)。这说明CD1在非共生血红蛋白的祖先序列中是被酪氨酸占据的,而在进化过程中被苯丙氨酸所取代。
图6不同物种GGPPS蛋白序列的同源比较采用Neighbor-joining算法,在MEGA6软件平台上构建了植物血红蛋白的分子系统进化树(图6)。根据序列的差异分为两大分支—非共生血红蛋白1 (Ns1)、古老的血红蛋白(Ns)组成的分支和非共生血红蛋白2(Ns2)、共生血红蛋白(sHb)组成的分支;CtHb位于非共生血红蛋白1的基部,说明其可能是非共生血红蛋白1的共同祖先,并且其进化地位比古老的苔藓植物血红蛋白稍微高等一点,这跟物种的进化关系是吻合的。
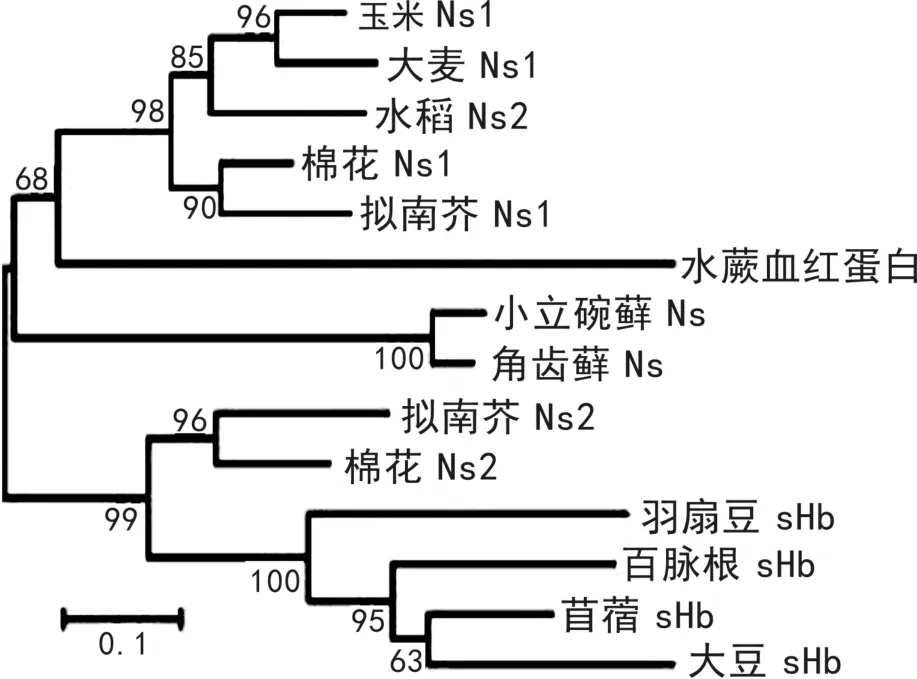
图6 不同物种的系统进化树Fig.6 Phylogenetic tree of different species

图7 角齿藓、水蕨、水稻非共生血红蛋白和大豆豆血红蛋白三级结构和表面电荷分布比较 带有正电荷和负电荷的氨基酸分别用蓝色和红色表示;小立碗藓的三级结构坐标数据(ID number:PM0075013)从蛋白质模型数据库(Protein Model Database)(http://mi. caspur.it/PMDB/)下载,水稻和大豆的三级结构坐标数据(ID numbers:1D8U和1BIN)从RCSB蛋白质数据库(RCSB Protein Data Bank)下载。Fig.7 Comparison of the predicted nonsymbiotic hemoglobin from Ceratodon purpureus,Ceratopteris thalictroides,native rice NsHb1 and soybean Lba tertiary structures and surface charge distribution Blue and red colors represent positively and negatively charged amino acid residues,respectively;Coordinates for nonsymbiotic hemoglobin from Ceratodon purpureus was obtained from Protein Model Database(http://mi.caspur.it/PMDB/)under the ID number PM0075013.Coordinates for the rice NsHb1 and soybean Lba structures were obtained from RCSB Protein Data Bank with the ID numbers 1D8U and 1BIN,respectively.
本研究也比较了角齿藓、水蕨、水稻非共生血红蛋白及大豆的豆血红蛋白三维结构的差异,揭示了植物血红蛋白从非共生到共生进化过程中的一些关键改变(图7)。这些改变包括:(1)血红素辅基从六配位体(Hexacoordinate)到五配位体(Pentacoordi-nate)的转变(Gopalasubramaniam et al,2008);(2) CD-loop,N-端和C-端区域长度的缩短;(3)蛋白结构更加紧凑,形成球状结构。这些改变可能有助于非共生血红蛋白向共生血红蛋白结构的转变,特别是有助于豆血红蛋白共生功能的实现(Gopalasubra-maniam et al,2008)。
(Continue on page 161)(Continue from page 223)
ARNOLD K,BORDOLI L,KOPP J,et al,2006.The SWISS-MODEL workspace:a web-based environment for protein structure homology modelling[J].Bioinformatics,22(2):195-201.
ANDERSON CR,JENSEN EO,LLEWELLYN DJ,et al,1996.A new hemoglobin gene from soybean:a role for hemoglobin in all plants[J].Proc Natl Acad Sci USA,93(12):5 682-5 687.
GARROCHO-VILLEGAS V,ARREDONDO-PETER R,2008.Mo-lecular cloning and characterization of a Moss(Ceratodon purpu-reus)nonsymbiotic hemoglobin provides insight into the early evo-lution of plant nonsymbiotic hemoglobins[J].Mol Biol Evol,25 (7):1 482-1 487.
GOPALASUBRAMANIAM SK,KOVACS F,VIOLANTE-MOTA F,et al,2008.Cloning and characterization of a caesalpinoid (Chamaecrista fasciculata)hemoglobin:the structural transition from a nonsymbiotic hemoglobin to a leghemoglobin[J]. Proteins,72(1):252-260.
LIRA-RUAN V,SARATH G,KLUCAS RV,et al,2001.Synthesis of hemoglobins in rice(Oryza sativa var.Jackson)plants growing in normal and stress conditions[J].Plant Sci,161(2):279-287.
LIU YG,WHITTIER RF,1995.Thermal asymmetric interlaced PCR:automatable amplification and sequencing of insert end fragments from P1 and YAC clones for chromosome walking [J].Genomics,25(3):674-681.
PESCE A,COUTURE M,DEWILDE S,et al,2000.A novel two-over-two α-helical sandwich fold is characteristic of the truncated hemoglobin family[J].EMBO J,19(11):2 424-2 434.
ROSS EJH,LIRA-RUAN V,ARREDONDO-PETER R,et al,2002.Recent insights into plant hemoglobins[J].Rev Plant Bio-chem Biotechnol,1:173-189.
ROSS EJH,STONE JM,ELOWSKY CG,et al,2004.Activation of the Oryza sativa non-symbiotic haemoglobin-2 promoter by the cytokinin-regulated transcription factor,ARR1[J].J Exp Bot,55(403):1 721-1 731.
SCHWEDE T,KOPP J,GUEX N,et al,2003.SWISS-MODEL: An automated protein homology-modeling server[J].Nucleic Acids Res,31(13):3 381-3 385.
TAMURA K,STECHER G,PETERSON D,et al,2013.MEGA6: molecular evolutionary genetics analysis version 6.0[J].Mol Biol Evol,30(12):2 725-2 729.
TAYLOR ER,NIE XZ,MACGREGOR AW,et al,1994.A cereal haemoglobin gene is expressed in seed and root tissues under anae robic conditions[J].Plant Mol Biol,24(6):853-862.
TREVASKIS B,WATTS RA,ANDERSSON CR,et al,1997.Two hemoglobin genes in Arabidopsis thaliana:the evolutionary origins of leghemoglobins[J].Proc Natl Acad Sci USA,94 (22):12 230-12 234.
VINOGRADOV S,HOOGEWIJS D,BAILLY X,et al,2006.A phylogenomic profile of globins[J].BMC Evol Biol,6(1):31.
WATTS RA,HUNT PW,HVITVED AN,et al,2001.A hemoglobin from plants homologous to truncated hemoglobins of microorganisms [J].Proc Natl Acad Sci USA,98(18):10 119-10 124.
Molecular clone and sequence analysis of hemoglobin gene in Ceratopteris thalictroides
QI Xiao-Qiong1,GE Ya-Fei1,LI Da-Wei2∗
(1.School of Chemistry and Life Sciences,Hubei University of Education/Hubei Key Laboratory of Purification and Application of
Plant Anticancer Active Ingredients,Wuhan 430205,China;2.Key Laboratory of Plant Germplasm Enhancement and
Specialty Agriculture,Wuhan Botanical Garden,Chinese Academy of Sciences,Wuhan 430074,China)
Plants,like humans,contain hemoglobins(Hbs),which consist mostly of protein subunits(the“globin”molecules)and heme groups.Phylogenomic analysis shows that Hbs are widely distributed in higher plants,and by com-paring sequences,expression patterns,and ligand-binding properties,it is evident that three distinct types of Hb exist in plants:symbiotic,non-symbiotic,and truncated Hbs.In the recent years,the genes of Hb have been cloned and identi-fied in various species of bryophytes,gymnosperms and angiosperms,but no study has been reported in the ferns until
now.In the present study,a full-length Hb gene from Ceratopteris thalictroides was cloned by Thermal Asymmetric Inter-laced(TAIL)PCR,and sequence analysis was conducted by bioinformatics.The results revealed that the gene was 949 bp long,containing 4 exons interrupted by 3 introns.The deduced protein(named CtHb)with 189 amino acid residues had a predicted isoelectric point(PI)of 7.81 and a calculated molecular mass 21.14 kDa.Modeling of the tertiary struc-ture indicated that CtHb possesses the typical tertiary structure as plant Hbs(including helices A,B,C,E,F,G and H,these together forms a“sandwich”structure of 3-on-3).Comparative structure analysis of CtHb with rice native NsHb1 revealed that most of the CtHb structure was quite similar to that of rice NsHb1,including the positions of helices E and F,where distal and proximal His were located,respectively.However,the structure differences were as follows: (1)The N-terminus region was longer in CtHb than in rice NsHb1;(2)The CD-loop folded differently;(3)Helices to coils transition at the connect regions of helices B and C were different.Structural comparison revealed major evolutionary changes during plant Hbs evolution.Some of these structural changes would be helpful for the transition from nonsymbiot-ic Hb to symbiotic Hb and for the specialization of a symbiotic function for symbiotic Hb.
plant hemoglobin,sequence analysis,tertiary structure modeling,structural evolution,Ceratopteris thalic-troides
Q943.2,Q75
A
1000-3142(2016)02-0216-08
10.11931/guihaia.gxzw201506023
齐小琼,葛亚飞,李大卫.水蕨血红蛋白基因的分子克隆和序列分析[J].广西植物,2016,36(2):216-223
QI XQ,GE YF,Li DW.Molecular clone and sequence analysis of hemoglobin gene in Ceratopteris thalictroides[J].Guihaia,2016,36(2):216-223
2015-06-24
2015-12-04
国家自然科学基金(31201242,C130405);湖北省科学研究计划项目(Q20143004);湖北省自然科学基金(2015CFB508);植物抗癌活性物质提纯与应用湖北省重点实验室开放课题(HLPAI 2014004) [Supported by the National Natural Science Foundation of China(31201242,C130405);Scientific Research Project of Educational Commission of Hubei Province(Q20143004);Natural Science Foundation of Hubei Province (2015CFB508);Open Research Fund of Hubei Key Laboratory of Purification and Application of Plant Anticancer Active Ingredients(HLPAI 2014004)]。作者简介:齐小琼(1981-),女,山东昌邑人,博士,讲师,主要从事进化遗传学研究,(E-mail)108816564@qq.com。
