全放牧牦牛与舍饲牦牛瘤胃细菌多样性比较
2016-05-03曹连宾崔占鸿孙红梅郝力壮刘书杰
曹连宾+崔占鸿+孙红梅+郝力壮+刘书杰

摘要: 以青海省黄南藏族自治州河南蒙古族自治县全放牧牦牛和青海省畜牧兽医科学院舍饲牦牛瘤胃内容物为样本,对牦牛瘤胃细菌多样性进行分析及比较。使用细菌通用引物进行瘤胃细菌16S rRNA基因序列的扩增,以16S rRNA序列分析技术分析瘤胃细菌多样性,并构建16S rRNA基因克隆文库。结果表明,放牧牦牛与舍饲牦牛瘤胃细菌都主要分为两大类:Firmicutes类和Bacteroidetes类。放牧牦牛瘤胃Firmicutes类所占比例(79.52%)比舍饲牦牛瘤胃Firmicutes类所占比例(62.79%)要高,Bacteroidetes类在瘤胃内所占比例放牧牦牛(19.27%)比舍饲牦牛(25.58%)要低。放牧牦牛与舍饲牦牛瘤胃内纤维降解菌的差别不大,都为瘤胃内优势菌群,但舍饲牦牛瘤胃内比放牧牦牛瘤胃内的蛋白降解菌和淀粉降解菌更丰富。
关键词: 牦牛;放牧;舍饲;瘤胃细菌;16S rRNA基因序列
中图分类号: S823.8+55 文献标志码: A 文章编号:1002-1302(2016)03-0242-07
青海作为中国牦牛第一大省,拥有丰富的牦牛资源,但放牧草场属于高寒草甸类型,牧草返青期时间短,枯黄期时间长,枯黄期牧草营养品质差难以消化利用,造成青海牦牛在冬季掉膘严重的现象,这些情况严重制约了青海牦牛业的发展。而牦牛舍饲情况下则能避免这一情况,但瘤胃细菌多样性受日粮结构影响显著。An等(2005年)曾报道全放牧牦牛其瘤胃细菌比之黄牛(补饲)的细菌丰富度要低[1]。但是全放牧牦牛与舍饲牦牛瘤胃细菌多样性比较的研究至今尚未见报道。
1 材料和方法
1.1 样品采集
青海省黄南藏族自治州河南蒙古族自治县牦牛屠宰厂3头4岁牦牛瘤胃内容物;舍饲牦牛瘤胃内容物取自青海省畜牧兽医科学院3头4岁舍饲牦牛瘤胃内容物。全放牧牦牛饲料为天然牧草(四川嵩草、细果苔草、高山早熟禾),舍饲牦牛饲料(精料 ∶ 粗料=3 ∶ 7,粗料为燕麦青干草,精料原料为玉米、大豆粕、菜籽粕、麸皮)。
1.2 试剂及培养基
1.2.1 试剂 引物由上海生工生物工程有限公司合成;CTAB(十六烷基三甲基溴化铵),EDTA(乙二胺四乙酸),NaCl,Tris-HCl(pH值为8.0),RNase,1 mol/L的IPTG(异丙基-β-D-硫代半乳糖苷),20 mg/μL X-gal(β-半乳糖苷酶),100 mg/mL氨苄青霉素,琼脂糖凝胶纯化回收试剂盒(QIAEX Ⅱ Gel Extraction Kit) 购于QIAGEN 公司;PMD18-T Vector购自TaKaRa公司。
1.2.2 培养基 液体LB培养基:1.0% 胰蛋白胨(tryptone),0.5% 酵母提取物(yeast extract),1.0% 氯化钠(NaCl)。
固体LB培养基:1.0% 胰蛋白胨(tryptone),0.5% 酵母提取物(yeast extract),1.0%氯化钠(NaCl),1.0% 琼脂粉。
1.3 瘤胃微生物总DNA提取方法
称取0.15 g瘤胃内容物于研钵中,加入1.5 mL 1%CTAB抽提液(100 mmol/L Tris.HCI,pH值为8.0;100 mmol/L EDTA;100 mmol/L 磷酸钠缓冲液,pH值为8.0;1.5 mol/L NaCl;1%CTAB),倒入液氮迅速研磨,始终保持研钵中存在液氮,研磨7~8次至研钵内样品成白色粉末状,等其融化后倒入2 mL离心管内,加入10 mg/mL蛋白酶K 20 μL和250 μL SDS(10%),65 ℃水浴2 h(每隔20 min颠倒混匀几次),7 000 g,离心10 min,取上清。避开上层漂浮物,取中层溶液,加入等体积酚 ∶ 氯仿 ∶ 异戊醇(25 ∶ 24 ∶ 1)抽提,12 000 g、4 ℃离心5 min,取上层溶液,重复操作1次,至上清不浑浊。然后用氯仿 ∶ 异戊醇(24 ∶ 1)抽提1次,12 000 g、4 ℃ 离心5 min,取上清。加入0.6倍体积的异丙醇,沉淀 2 h,12 000 g、4 ℃离心15 min,弃上清。用75%的乙醇洗涤沉淀,晾干,用 50 μL TE(pH值为8.0,含10 mg/μL RNase)溶解,37 ℃水浴30 min。
1.4 16S rRNA基因扩增
以瘤胃总DNA逐级稀释(稀释5、10、25、50倍)作为模板,使用Premix Taq进行PCR扩增,反应体系为50 μL,模板0.5 μL,引物(100 μmol/L)各0.1 μL,Premix Taq 25 μL,ddH2O 25 μL。引物为细菌通用引物:27 F 5′-AGAGTTTGATCMTGGCTCAG-3′和1492 R 5′-GGTTACCTTGTTACGACT T-3′。PCR反应程序:95 ℃ 5 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 90 s,30个循环;72 ℃ 10 min。产物用1%的琼脂糖凝胶电泳检测。
1.5 DNA的纯化回收、克隆以及测序
选择1.5 kb处条带最清晰的稀释倍数,使用琼脂糖凝胶纯化回收试剂盒回收、纯化其PCR产物。将纯化回收的目的DNA片段与PMD18-T Vector连接。连接体系:1 μL载体,5 μL solution 1 ,4 μL DNA样品,16 ℃条件下30 min。转化:取感受态细胞,冰上融化感受态2 min,将10 μL上述连接体系加入到感受态中,冰上放置30 min,42 ℃热激90 s,冰上放置3~5 min,加入500~700 μL液体LB培养基,37 ℃摇床培养箱培养1 h。取AMP(氨苄青霉素)抗性LB平板,提前涂布4 μL 1 mol/L的IPTG和40 μL 20 mg/μL的X-gal于平板上,无菌操作台上晾干。取转化好的感受态,4 000 g离心 3 min,弃去部分上清液,留200 μL。用移液器将感受态细胞吹打混匀,取100 μL涂布平板,37 ℃恒温培养箱内过夜培养。氨苄青霉素抗性和蓝白斑对阳性克隆进行初选,随机挑取300个白色单菌落于高频摇床上6~8 h氨苄青霉素抗性培养,使用引物M13F(-47)5′-CGCCAGGGTTTTCCCAGTCACGAC-3′和M13R(-48)5′-AGCGGATAACAATTTCACACAGGA-3′进行假阳性检验,将验证后的阳性克隆菌液送交上海生工生物工程股份有限公司进行测通,测序引物使用M13F(-47) 5′-CGCCAGGGTTTTCCCAGTCACGAC-3′和M13R(-48) 5′-AGCGGA TAACAATTTCACAC AGGA-3′。
1.6 序列分析
使用MEGA 6软件中的ClustalW程序对所有序列进行比对,去掉序列前后的载体序列。Bellerophon软件检验嵌合体序列,去除可能的嵌合体序列[2]。使用Mothur软件划分序列OTU单位[3],选取OTU代表序列,分类学上相似性大于97%的序列可看做为同一个OTU单位。使用RDP11软件[4]中的Classifer程序[5]对所有OTU代表序列进行分类。将OTU代表序列在NCBI的Blast程序中搜索相似性最高已知序列以及相似性最高的已知菌种序列[6]。将所有OTU代表序列用MEGA 6软件中的ClustalW程序重新比对,建树采用邻接法(neighbor-joining,NJ)[7]和Kimura双参数距离模型,Bootstrap设置为1 000[4],以大肠杆菌(Escherichia coli)作为外群[8]。
2 结果与分析
2.1 OTU操作单元划分
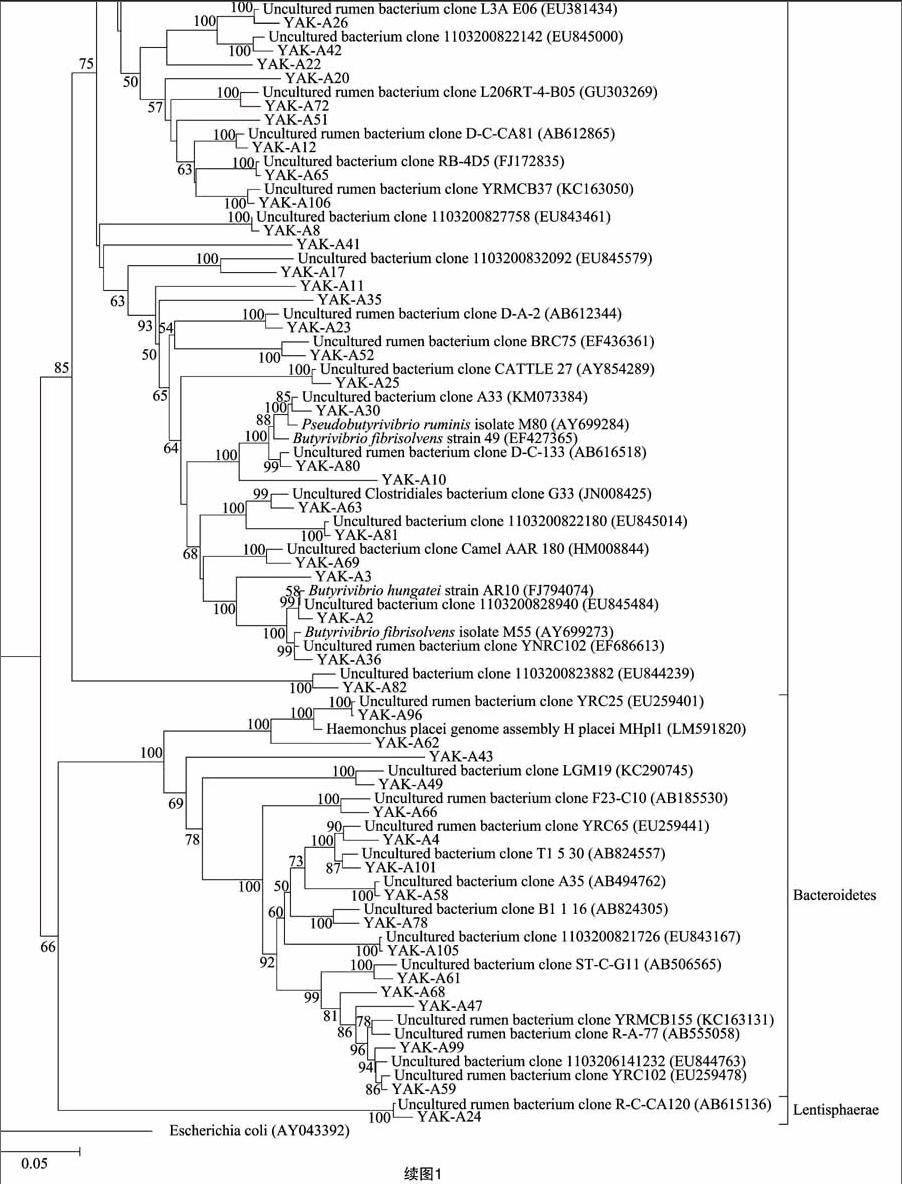
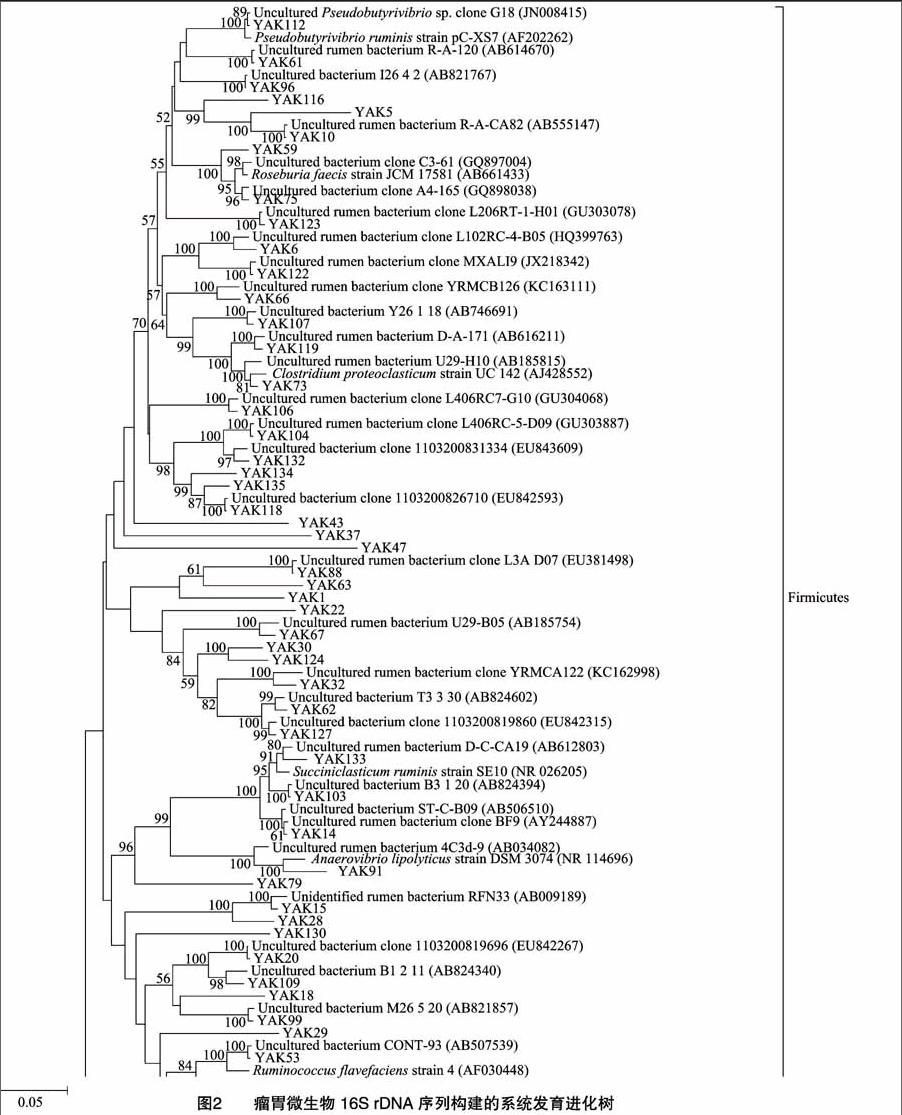
青海省河南县放牧牦牛瘤胃细菌97个序列划分为83个OTUs,青海省畜牧兽医科学院舍饲牦牛瘤胃细菌118个序列划分为86个OTUs。
2.2 RDP分类
青海省河南县牦牛瘤胃细菌有66个OTUS属于厚壁菌门(Firmicutes),占总OTUS的79.52%。其中1个OTUS属于未分类的Firmicutes门;61个OTUS属于Clostridia纲(占总OTUS的73.49%),除22个OTUS分属未分类的Clostridiales目外,剩余的39个OTUS分属于Clostridiales目的Clostridiales_Incertae Sedis ⅩⅢ科(2个OTUS)、Ruminococcaceae科(18个OTUS)、Eubacteriaceae科(1个OTU)和Lachnospiraceae科(18个OTUS);4个OTUS属于Negativicutes纲(占总OTUS的4.82%),分属于Selenomonadales目的Acidaminococcaceae科(3个OTUS)和Veillonellaceae科(1个OTU)。青海省河南县牦牛瘤胃细菌有16个OTUS属于拟杆菌门(Bacteroidetes),占总OTUS的19.27%。其中2个OTUS属于未分类的Bacteroidetes门;14个OTUS属于Bacteroidia纲(占总OTUS的16.87%),这14个OTUS中13个OTUS属于未分类的Bacteroidales目,剩余的1个OTU属于Bacteroidales目的Prevotellaceae科。青海省河南县牦牛瘤胃细菌有1个OTU属于黏胶球形菌门(Lentisphaerae),归类于Oligosphaeria纲(占总OTUS的1.20%)的Oligosphaerales目Oligosphaeraceae科。
青海省畜牧兽医科学院舍饲牦牛瘤胃细菌的86个OTUs有54个OTUs(占总OTUs的62.79%)属于Firmicutes菌门,其中48个OTUs属于Clostridia菌纲,除6个OTU属于未分类的Clostridia菌纲外,其余42个OTUs分属于Clostridiales目的Ruminococcaceae科(18个OTUs)、Lachnospiraceae科(21个OTUs)和Clostridiales_Incertae Sedis ⅩⅢ科(3个OTUs),其中5个OTUs属于Negativicutes菌纲,分属于Selenomonadales目的Veillonellaceae科(1个OTU)和Acidaminococcaceae科(4个OTUs);86个OTUs有22个OTUs(占总OTUs的25.58%)属于Bacteroidetes菌门,其中8个OTUs不能确定分类,剩余的14个OTUs分属于Bacteroidales目的Prevotellaceae科(8个OTUs)和Porphyromonadaceae科(6个OTUs);86个OTUs中剩余的10个OTUs有7个OTUs(占总OTUs的8.14%)属于Proteobacteria菌门,1个OTU属于Verrucomicrobia菌门,1个OTU属于Synergistetes菌门,还有1个OTU在细菌中不能确定其分类地位可能为尚未发现的新属。
RDP分类结果中显示,青海省河南县放牧牦牛瘤胃Firmicutes类所占比例(79.52%)比之舍饲牦牛瘤胃Firmicutes类所占比例(62.79%)要高,Bacteroidetes类在瘤胃内所占比例放牧牦牛(19.27%)比之舍饲牦牛(25.58%)要低。舍饲牦牛RDP分类结果比之放牧牦牛要丰富。
2.3 16S rRNA基因序列系统进化分析
2.3.1 青海省河南县全放牧牦牛瘤胃细菌16S rRNA基因序列系统进化树分析 83个OTUs中有6个OTUs与已培养细菌的相似性≥97%,占总OTUs的7.23%;20个OTUs与已培养细菌的相似性在90%~96%之间,占总OTUs的24.10%;57个OTUs与已培养细菌的相似性大于90%,占总OTUs的 68.67%。
将83个OTUs代表序列Blast后,取相似性≥97%的已知序列(53个序列)以及已培养瘤胃细菌序列(6个序列)与这83个OTUs代表序列以大肠杆菌(Escherichia coli)为外群构建系统发育进化树。由图1可以看出,系统发育进化树上把所有序列分为3个部分:厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、黏胶球形菌门(Lentisphaerae)。同时,83个OTUs中有66个OTUs属于Firmicutes,其中1个OTU(YAK-A91)与瘤胃解琥珀酸菌(Succiniclasticum ruminis)相似性97%,1个OTU(YAK-A30)与瘤胃假丁酸弧菌(Pseudobutyrivibrio ruminis)相似性99%,2个OTUs(YAK-A36、YAK-A80)与溶纤维丁酸弧菌(Butyrivibrio fibrisolvens)相似性 98%~99%,1个OTU(YAK-A2)与亨氏丁酸弧菌(Butyrivibrio hungatei)相似性99%;83个OTUs中有16个OTUs属于Bacteroidetes,其中1个OTU(YAK-A96)属于帕莱斯氏血矛线虫(Haemonchus placei)相似性97%;83个OTUs中有1个OTU属于Lentisphaerae。
YAK-A82序列属于Firmicutes类,但是从图1系统发育进化树上可以看出其并没有与Firmicutes类的其他序列归在一起,其进化距离较远。因此,YAK-A82可能是目前尚未发现的新的菌属。
2.3.2 青海省舍饲牦牛瘤胃细菌16S rRNA基因序列系统进化树分析 86个OTUs中有8个OTUs与已培养细菌的相似性≥97%,占总OTUs的9.30%;34个OTUs与已培养细菌的相似性在90%~96%之间,占总OTUs的39.53%;44个OTUs与已培养细菌的相似性<90%,占总OTUs的51.16%。
将86个OTUs代表序列Blast比对后,取相似性≥97%的已知序列(54个序列)以及已培养瘤胃细菌序列(9个序列)与这86个OTUs代表序列以大肠杆菌(Escherichia coli)为外群构建系统发育进化树,从系统发育关系分析上可以发现所有序列主要分为2个部分:厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)。有1个序列归类到了Verrucomicrobia菌门。YAK39、YAK58、YAK74、YAK 80、YAK 81、YAK 93、YAK 111在RDP分类中归于Proteobacteria菌门,但从进化树上可以发现,这7个序列并没有聚在一起,且进化距离较远,不能确定这7个系列的分类地位。YAK21在RDP分类中归于Firmicutes这一类,但从树图上发现,这一序列距离Firmicutes其他序列的进化距离较远,未归于Firmicutes这一类。由图2系统发育进化树可以看出86个OTUs中有54个OTUs属于Firmicutes,其中1个OTU(YAK112)与瘤胃假丁酸弧菌(Pseudobutyrivibrio ruminis)相似性99%,2个OTUs(YAK59、YAK75)与罗斯伯里氏菌(Roseburia faecis )相似性97%~98%,1个OTU(YAK73)与蛋白溶解梭菌(Clostridium proteoclasticum)相似性98%,1个OTU(YAK119)与溶纤维丁酸弧菌(Butyrivibrio fibrisolvens)相似性97%,1个OTU(YAK53)与黄色瘤胃球菌(Ruminococcus flavefaciens)相似性98%,2个OTUs(YAK103、YAK133)与瘤胃解琥珀酸菌(Succiniclasticum ruminis)相似性97%~98%,1个OTU(YAK91)与Anaerovibrio lipolyticus相似性97%;86个OTUs中有22个OTUs属于Bacteroidetes。
3 讨论
本研究发现全放牧牦牛(97个序列共属于83个OTUs),舍饲牦牛(118个序列共属于86个OYUs)说明不管放牧牦牛还是舍饲牦牛瘤胃微生物都非常丰富,比刘利等报道的广西水牛瘤胃中的细菌多样性(74个16S rRNA基因序列分属于51个OTUs分类单位)[9]和王远亮等报道的荷斯坦奶牛瘤胃细菌多样性(45个16S rRNA序列分属于28个OTUs分类单位)[10]更丰富多样。
由RDP分类结果可以看出,放牧牦牛与舍饲牦牛瘤胃细菌都主要分为2大类:Firmicutes类和Bacteroidetes类。这与Edwards 等阐述的牛瘤胃细菌[11]和Koike等阐述的羊瘤胃细菌[12]主要分为2大类LGCGPB类和CFB类结果相同。Yang等曾报道,荷斯坦奶牛、大额牛、水牛瘤胃细菌主要分为LGCGPB和CFB2大类[13]。Monica等也曾报道驯鹿瘤胃细菌主要分为LGCGPB和CFB2大类[14]。但青海省河南县放牧牦牛瘤胃Firmicutes类所占比例(79.52%)比舍饲牦牛瘤胃Firmicutes类所占比例(62.79%)要高,Bacteroidetes类在瘤胃内所占比例放牧牦牛(19.27%)比舍饲牦牛(25.58%)要低。An等曾报道的甘肃放牧牦牛(54.89%)比晋南舍饲黄牛(16.97%)瘤胃内Firmicutes类所占比例要高,而Bacteroidetes类所占比例晋南舍饲黄牛(38.99%)比甘肃放牧牦牛(34.78%)要高[1]。本试验结果与这一观点相符。Fernando 等也曾报道反刍家畜在饲喂精料时,瘤胃内Bacteroidetes类占优势[15]。同时,在RDP分类上,舍饲牦牛(归类于4个菌门)的分类结果比放牧牦牛(归类于3个菌门)更加丰富,An等曾报道由于舍饲黄牛的日粮成分比放牧牦牛日粮成分更为复杂,使其瘤胃微生物更加复杂多样[1]。
溶纤维丁酸弧菌、白色瘤胃球菌、黄色瘤胃球菌、产琥珀酸丝状杆菌是瘤胃内主要的纤维降解菌,而后三者又是公认的瘤胃内纤维降解的优势菌群;同时溶纤维丁酸弧菌还与瘤胃内蛋白质的降解有关系;瘤胃解琥珀酸菌可以将瘤胃内的琥珀酸转化为丙酸,是瘤胃内的优势菌种[15];瘤胃假丁酸弧菌、罗斯伯里氏菌的主要作用是发酵瘤胃各种碳水化合物,主产物为丁酸[16];Anaerovibrio lipolyticus菌是瘤胃内主要淀粉降解菌之一[17];亨氏丁酸弧菌能进行少量的乳酸发酵,且是瘤胃内生成乙酸的重要微生物之一[18];蛋白溶解梭菌有降解瘤胃蛋白质的作用。由图1系统进化树上可以看出,放牧牦牛瘤胃内共检测到6株已培养细菌分别为1株瘤胃解琥珀酸菌、1株瘤胃假丁酸弧菌、2株溶纤维丁酸弧菌、1株亨氏丁酸弧菌、1株帕莱斯氏血矛线虫;由图2可以看出,舍饲牦牛瘤胃内9株已培养细菌分别为1株瘤胃假丁酸弧菌、2株罗斯伯里氏菌、1株蛋白溶解梭菌、1株溶纤维丁酸弧菌、1株黄色瘤胃球菌、2株瘤胃解琥珀酸菌、1株Anaerovibrio lipolyticus。由此可以发现放牧牦牛与舍饲牦牛瘤胃内都检测到了纤维降解菌的存在,其差别不明显。但是舍饲牦牛瘤胃内检测到了1株主要蛋白降解菌(蛋白溶解梭菌)和1株降解淀粉的细菌(Anaerovibrio lipolyticus),这2种细菌在放牧牦牛瘤胃内未检测到。这是因为舍饲牦牛的日粮中添加有精料,而精料中蛋白质和淀粉含量比之天然牧草要高,致使瘤胃内蛋白质降解菌和淀粉降解菌的菌群数量增加。Coe等曾报道当给反刍家畜饲喂精料时,其瘤胃内淀粉降解菌的数量和种类会增加[19-21]。同时,舍饲牦牛瘤胃内检测到的产丁酸菌的种类(1株瘤胃假丁酸弧菌、2株罗斯伯里氏菌)比放牧牦牛瘤胃内检测到的产丁酸菌的种类(1株瘤胃假丁酸弧菌)要多。溶纤维丁酸弧菌在舍饲牦牛瘤胃内(1株溶纤维丁酸弧菌)和放牧牦牛瘤胃(2株溶纤维丁酸弧菌)内都有检测到且差别不明显,这可能是因为溶纤维丁酸弧菌除了降解纤维素外,对于瘤胃蛋白的降解也有作用有关,致使这2种条件下日粮的不同对溶纤维丁酸弧菌的影响不大。
4 结论
放牧牦牛与舍饲牦牛瘤胃细菌都主要分为2大类:Firmicutes类和Bacteroidetes类。放牧牦牛瘤胃Firmicutes类所占比例(79.52%)比舍饲牦牛瘤胃Firmicutes类所占比例(6279%)要高,Bacteroidetes类在瘤胃内所占比例放牧牦牛(19.27%)比舍饲牦牛(25.58%)要低。放牧牦牛与舍饲牦牛瘤胃内纤维降解菌的差别不大,都为瘤胃内优势菌群,但舍饲牦牛瘤胃内比放牧牦牛瘤胃内的蛋白质降解菌和淀粉降解菌更丰富。
参考文献:
[1]An D,Dong X,Dong Z. Prokaryote diversity in the rumen of yak (Bos grunniens) and Jinnan cattle (Bos taurus) estimated by 16S rDNA homology analyses[J]. Anaerobe,2005,11(4):207-215.
[2]Huber T,Faulkner G,Hugenholtz P. Bellerophon:a program to detect chimeric sequences in multiple sequence alignments[J]. Bioinformatics,2004,20(14):2317-2319.
[3]Schloss P D,Westcott S L,Ryabin T,et al. Introducing mothur:open-source,platform-independent,community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology,2009,75(23):7537-7541.
[4]Cole J R,Wang Q,Cardenas E,et al. The ribosomal database project:improved alignments and new tools for rRNA analysis[J]. Nucleic Acids Research,2009,37(Database Issue):D141-D145.
[5]Wang Q,Garrity G M,Tiedje J M,et al. Naive bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology,2007,73(16):5261-5267.
[6]Altschul S F,Madden T L,Schaffer A A,et al. Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J]. Nucleic Acids Research,1997,25(17):3389-3402.
[7]Saitou N,Nei M. The neighbor-joining method:a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution,1987,4(4):406-425.
[8]Matsui H,Kato Y,Chikaraishi T,et al. Microbial diversity in ostrich ceca as revealed by 16S ribosomal RNA gene clone library and detection of novel Fibrobacter species[J]. Anaerobe,2010,16(2):83-93.
[9]刘 利,唐纪良,冯家勋,等. 广西水牛瘤胃中的细菌多样性[J]. 微生物学报,2009,49(2):251-256.
[10]王远亮,杨瑞红,毛爱军,等. 采用未培养技术对荷斯坦奶牛瘤胃细菌多样性进行初步分析[J]. 微生物学报,2005,45(6):915-919.
[11]E Edwards J,R Mcewan N,J Travis A,et al. 16S rDNA library-based analysis of ruminal bacterial diversity[J]. Antonie Van Leeuwenhoek,2004,86(3):263-281.
[12]Koike S,Yoshitani S,Kobayashi Y,et al. Phylogenetic analysis of fiber-associated rumen bacterial community and PCR detection of uncultured bacteria[J]. FEMS Microbiology Letters,2003,229(1):23-30.
[13]Yang S,Ma S,Chen J,et al. Bacterial diversity in the rumen of Gayals (Bos frontalis),Swamp buffaloes (Bubalus bubalis) and Holstein cow as revealed by cloned 16S rRNA gene sequences[J]. Molecular Biology Reports,2010,37(4):2063-2073.
[14]Sundset M A,Praesteng K E,Cann I K,et al. Novel rumen bacterial diversity in two geographically separated sub-species of reindeer[J]. Microbial Ecology,2007,54(3):424-438.
[15]Fernando S C,Purvis H T,Najar F Z,et al. Rumen microbial population dynamics during adaptation to a high-grain diet[J]. Applied and Environmental Microbiology,2010,76(22):7482-7490.
[16]van Gylswyk N O,Hippe H,rainey F A. Pseudobutyrivibrio ruminis gen.nov.,sp.nov.,a butyrate-producing bacterium from the rumen that closely resembles butyrivibrio fibrisolvens in phenotype[J]. International Journal of Systematic Bacteriology,1996,46(2):559-563.
[17]Prins R A,Lankhorst A,van der Meer P,et al. Some characteristics of Anaerovibrio lipolytica a rumen lipolytic organism[J]. Antonie Van Leeuwenhoek,1975,41(1):1-11.
[18]Kope cˇn J,Zorec M,Mrázek J,et al. Butyrivibrio hungatei sp. nov. and Pseudobutyrivibrio xylanivorans sp. nov.,butyrate-producing bacteria from the rumen[J]. International Journal of Systematic and Evolutionary Microbiology,2003,53(Pt 1):201-209.
[19]Coe M L,Nagaraja T G,Sun Y D,et al. Effect of virginiamycin on ruminal fermentation in cattle during adaptation to a high concentrate diet and during an induced acidosis[J]. Journal of Animal Science,1999,77(8):2259-2268.
[20]Goad D W,Goad C L,Nagaraja T G. Ruminal microbial and fermentative changes associated with experimentally induced subacute acidosis in steers[J]. Journal of Animal Science,1998,76(1):234-241.
[21]Tajima K,Arai S,Ogata K,et al. Rumen bacterial community transition during adaptation to high-grain diet[J]. Anaerobe,2000,6(5):273-284.
