海巴戟ISSR遗传多样性分析
2016-04-17程江波聂风琴林道哲符文英
廖 丹 程江波 聂风琴 李 明 林道哲 符文英
(海南大学农学院,海南海口570228)
海巴戟ISSR遗传多样性分析
廖 丹 程江波 聂风琴 李 明 林道哲 符文英
(海南大学农学院,海南海口570228)
利用ISSR分子标记对202份海巴戟种质材料进行遗传多样性和聚类分析。结果表明:13条ISSR引物共扩增出65个位点,每个引物平均扩增出5个位点,其中多态性位点63个,占总数的96.92%。在相似系数为0.63时,202份海巴戟种质分为5个类,表现出一定的地域分布规律。检测到有效等位基因数Ne=1.604 2,基因多样性H=0.3 488,Shannon′s信息指数I=0.520 6,说明202份海巴戟种质间存在着较高的遗传多样性。
海巴戟;ISSR;遗传多样性;聚类分析
海巴戟(Morinda citrifolia)又名海巴戟天、海滨木巴戟、橘叶巴戟、檄树和印桑椹,俗称诺丽、萝梨等,属茜草科巴戟天属植物。国外主要分布在南太平洋诸岛屿、菲律宾、澳大利亚、柬埔寨[1]。在我国海南的万宁、文昌、儋州、陵水、三亚、西沙、南沙群岛及台湾岛有自然分布[2]。多年来,研究者对海巴戟的营养成分[3]和药理作用[4]等作了大量研究。药理实验结果表明海巴戟具有抗细菌、抗病毒、抗真菌、抗肿瘤、抗寄生虫、镇痛、降血压、消炎和提高免疫力等功效[5-6]。自美国生化学者罗夫·海尼克博士[5]发现诺丽果汁含有丰富的赛洛宁原及赛洛宁转化酶之后掀起了诺丽的栽培和研究热潮。目前,海巴戟的研究主要集中在功能性成分的提取和分析上[7-8]。
我国的海巴戟研究起步较晚,近2年海巴戟的研究倾向于海巴戟种质资源的保护和利用。2014年赖茂良等[9]开展海巴戟种质资源的遗传多样性研究,获得了最新的关于海巴戟的根、茎、叶、花、果实和种子的形态学特征特性的植物学描述结果,首次提出了海巴戟的果为聚花果而非聚合果,确定了海巴戟种质的染色体数目及组型。同年程江波等[10]研究了海巴戟核心种质的构建方法,指出海巴戟核心种质的最佳构建方法为AVERAGE系统聚类,G策略取样,15%的组内取样比例,并初步构建了由21个不同种质株系构成的海巴戟核心种质。
简单重复序列(inter-simple sequence repeats ISSR),是由Ziekiewicz等创建的一种简单重复区间扩增多态性的分子标记技术[11],该技术因样品用量少、操作简单、试验重复性好、信息量大、多态性高的特点,被广泛应用于植物遗传多样性的研究[12-13]。据检索,ISSR分子标记技术在海巴戟中的应用,除了吴田等[14]在方法上建立了海巴戟叶片基因组DNA快速提取方法和优化了ISSR-PCR体系外,其他未见报道。本研究应用ISSR分子标记对202份海巴戟种质材料进行遗传多样性研究,从分子水平揭示海巴戟种质间的遗传多样性和亲缘关系,研究将为海巴戟种质资源的保护和创新利用提供理论依据。
1 材料与方法
1.1 材料
供试材料共计202份海巴戟种质,由海南万维生物制药技术股份有限公司海巴戟种质资源圃提供。供试材料种质来源地见表1。选取新生健康嫩叶用于提取DNA,保存于-20℃备用。

表1 供试材料Tab.1 Experimentalmaterials
1.2 方法
1.2.1 引物 选用加拿大British Columbia大学设计的ISSR引物序列26条,由北京鼎国昌盛生物技术有限公司合成,再从中筛选出13条引物详细序列(表2)用于ISSR分子标记。

表2 ISSR引物序列Tab.2 Primer sequences of ISSR
1.2.2 基因组DNA的提取 参考吴田等[11]的改进方法,利用K 5500型超微量分光光度计分析仪进行DNA样品质量和浓度的检测,并稀释至50 mg/L用于ISSR-PCR扩增。
1.2.3 ISSR引物筛选及PCR扩增 PCR反应体系为20μL,其中含模板DNA 50 ng,引物浓度0.6 μmol/L,Master Mix10μL(含0.1U/μL Taq polymerase,500μmol/L dNTP each,20 mmol/L Tris-HCl(pH 8.3),100mmol/L KCl,3mmol/LMgCl2,以及其他稳定剂和增强剂),反应液用适量矿物油覆盖(表3)。PCR反应在德国产Eppendorf PCR仪上进行。PCR扩增程序:94℃预变性3 min;94℃变性45 s,56℃退火(因不同引物而有所不同)45 s,72℃延伸90 s,40个循环;72℃延伸10 min。扩增产物用2.0%琼脂糖凝胶在1×TBE缓冲液中电泳分离,以MarkerDL 2 000为分子质量标准,Bio-Rad Chemi-Doc MP全能型凝胶成像分析系统扫描分析。

表3 ISSR的PCR反应体系Tab.3 The PCR reaction system of ISSR
选用表型差异较大的17、94、193作为筛选引物的材料,按照已优化的体系,根据不同的引物设计不同的退火温度,从26条引物中筛选出带型好、多态性高、稳定性和重复性好的13条引物作为本试验的ISSR扩增引物。
1.2.4 数据处理与分析 在ISSR-PCR扩增结果电泳图谱中,根据电泳图谱中DNA条带的有无,按照Clark[15]的方法,有带记为1,无带记为0,并构建二元数据矩阵。应用POPGENE 1.31[16]软件进行遗传多样性参数分析,计算多态性引物的总等位基因数(Na)、有效等位基因数(Ne)、Nei′s基因多样性指数(H)、Shannon′s信息指数(I)、多态位点百分率(PPB)。用NTSYSpc2.1[17]进行UPGMA聚类分析。
2 结果与分析
2.1 引物筛选及多态性分析
从26条引物中筛选出13条重复性好、多态性丰富的ISSR引物对202份种质资源基因组DNA进行扩增。13条引物共扩增出65条DNA条带,其中多态性条带63条,多态性百分率为96.92%,说明海巴戟种质资源遗传多样性丰富,个体间遗传差异较大。引物UBC808的扩增结果如图1所示,扩增产物条带为100~2 000 bp,以500~1 000 bp的扩增片段较多。

2.2 聚类分析
采用UPGMA方法进行聚类,得到202份材料的聚类图,结果见图2。
从图2可看出:在遗传相似系数为0.63处,可将供试材料分为5个组,其聚类结果表现出一定的地理区域特性。第I组包含了140份种质,基本包括了来自除海南博鳌外的大部分种质。第II组包含泰国(175)、印度尼西亚(96)、大溪地(177)、海南长丰(194、195)以及海南海口(196、198~200)等共9份种质;第III组包含夏威夷(14、180)2份,1份大溪地(178)共3份种质。第Ⅳ组共48份种质,以夏威夷、印度尼西亚,新加坡海巴戟种质为主,海南海口(197)种质也聚在此类。各种质间遗传相似系数变幅较大,材料间的遗传变异相对较大,表现出极为显著的遗传多样性。第Ⅴ组群只包含了海南博鳌(201、202)2份海巴戟种质。
综上所述,本试验筛选的13条ISSR标记能很好反映出海巴戟种质间的遗传差异,并表明海巴戟种质资源间存在着丰富的遗传多样性。
2.3 遗传多样性分析
利用POPGENE对海巴戟种质材料进行各项参数统计分析,结果见表4。在物种水平上,遗传多样性各指标相对较低,多态位点百分率(PPB)达到100.00%,有效等位基因数(Ne)为1.604 2,Nei′s遗传多样性指数(H)为0.348 8,Shannon′s信息指数(I)为0.520 6。在区域水平上,Shannon′s信息指数(I)、多态位点百分率(PPB)、有效等位基因数(Ne)、Nei′s遗传多样性指数(H)最高是印度尼西亚种质,分别为0.507 9、100%、1.584 1、0.339 3;其次是新加坡种质。海南长丰、海口、博鳌等地的多态位点百分率PPB、有效等位基因数Ne、Nei′s遗传多样性指数H和Shannon′s信息指数I均低于其他区域。以Ne、H、I为标准,并综合多态位点百分率(PPB)分析结果,表明海巴戟种质资源的遗传多样性以印度尼西亚和新加坡两种质较高,海南的种质均较低。
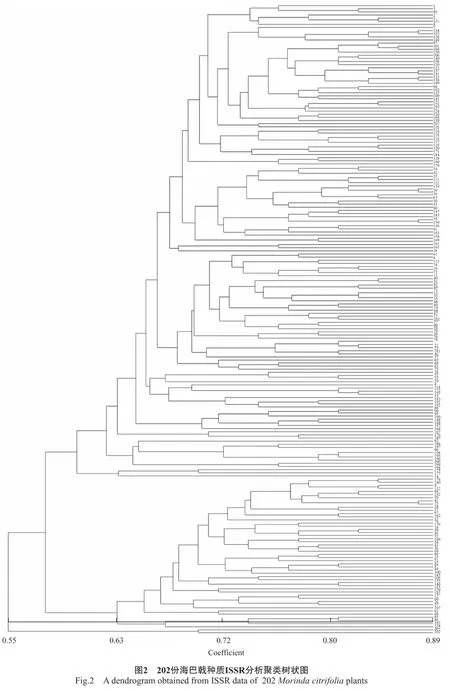

表4 ISSR分析的海巴戟种质资源的遗传多样性Tab.4 Genetic diversity of Morinda citrifolia resources detected by ISSR makers
3 结论与讨论
利用ISSR分子标记对202份海巴戟种质材料进行分析,13条ISSR引物共扩增出65个位点,其中多态性位点63个,占总数的96.92%。聚类结果表明,遗传相似系数为0.55~0.89,在相似系数为0.63时,202份海巴戟种质分聚为5类,聚类结果表现出一定的地域分布规律。检测到有效等位基因数Ne=1.604 2,基因多样性H=0.3488,Shannon′s信息指数I=0.520 6,说明202份海巴戟种质间存在着较高的遗传多样性。
分子标记技术[18-20]已广泛地应用于植物的遗传多样性分析。在海巴戟种质的遗传多样性研究中,赖茂良等[9]利用SRAP技术研究78份海巴戟种质材料获得的多态位点百分率是33.39%。巴戟天和海巴戟同属茜草科巴戟天属植物,刘颖嘉等[19]利用ISSR技术研究福建和广东7个种源的巴戟天种质遗传多样性,其多态性是78.81%。本研究13条ISSR引物共扩增出65个位点,其中多态性位点63个,占总数的96.92%,其多态位点百分率均高于前人的研究,表明海巴戟种质的遗传多样性更高。究其原因是202种质来源地不同,遗传多样性以印度尼西亚和新加坡两种质最高,且亲缘关系最近;海南的种质遗传多样性均较低。
本研究的ISSR聚类分析结果表明,海巴戟种质在亲缘关系上存在一定的地域分布规律,其聚类结果跟赖茂良等[9]的SRAP聚类结果一致,即大溪地种与夏威夷种,新加坡种与印度尼西亚种其亲缘关系最近,说明两种标记均可用于海巴戟种质的聚类分析。
[1] 彭勇,肖伟,刘勇,等.世界药用植物新宠—海巴戟果[J].国外医药(植物学分册),2007,22(3):93-96.
[2] 邢福武,周劲松,王发国,等.海南植物物种多样性编目[M].武汉:华中科技大学出版社,2012:340-341.
[3] Wangmy SU C.Cancer preventiveeffectof Morinda citrifolia(Noni)[J].Annals of the New York Academy of Sciences,2001,952:161-168.
[4] Hirazumi A,Furusawa E.An immune modulatory polysaccharide-rich substance from the fruit juice of Morinda citrifolia(Noni)with antitumour activity[J].Physiotherapy Research,1999,13:380-387.
[5] 苏文潘,吕平,韦丽君,等.海巴戟研究进展[J].广西热带农业,2006(2):37-39.
[6] 黄骐,何文锦,叶冰莹,等.海巴戟(Morinda citrifolia L.)组织培养研究[J].天然产物研究与开发,2006(18):910-913.
[7] 张洪财,王文姌,刘树民.诺丽果化学成分的研究进展[J].哈尔滨医药,2011,31(3):213-216.
[8] 龚敏,符文英,周静,等.诺丽鲜果与诺丽发酵汁的挥发性成分对比研究[J].食品科技,2009,34(9):33-35.
[9] 赖茂良.海巴戟(NONI)种质资源的遗传多样性研究[D].海口:海南大学,2014.
[10] 程江波,邢诒旺,赖茂良,等.海巴戟(NONI)核心种质构建方法[J].热带生物学报,2014,5(3):280-285.
[11] Ziekiewicz E,Rafalski A,Labuda D.Genome fingerprinting by simple sequence repeats(SSR)-anchored polymerase chain reaction amplification[J].Genomics,1994,20(2):176-183.
[12] 邹喻萍,葛颂,王晓东.系统与进化植物学中的分子标记[M].北京:科学出版社,2001.
[13] 赵谦,杜虹,庄东红.ISSR分子标记及其在植物研究中的应用[J].分子植物育种,2007,5(Z1):123-129.
[14] 吴田,蓝增全,李青红.诺丽叶片DNA的提取及ISSRPCR反应体系的建立[J].安徽农业科学,2010,38(17):8859-8860.
[15] Clark M S.Plant Molecular Biology:a Laboratory Manual[M].Beijing:China Higher Education Press,1998:3-11.
[16] Yeh FC.Population genetic analysis of codominant and dominantmarkers and quantitative traits[J].Belgian Journal of Botany,1997,129:157.
[17] Rohlf.NTSYS-pc Version 2.1.Numerical Taxonomy System and Multivariate Analysis System[M].New York:Setauket,Exeter Software,2002.
[18] 刘颖嘉,黄宇,荣俊冬,等.巴戟天遗传多样性的ISSR分析[J].福建林学院学报,2011,31(3):203-206.
[19] 于小玉,喻方圆,刘建兵,等.ISSR在油茶品种鉴别和遗传多样性分析中的应用[J].南京林业大学学报(自然科学版),2013,37(1):61-66.
[20] 李晖,张瑞,彭方仁,等.美国山核桃种质资源遗传多样性ISSR分析[J].南京林业大学学报(自然科学版),2015,39(4):7-12.
(责任编辑 张 坤)
Genetic Diversity Analysis of Morinda citrifolia by ISSR Markers
Liao Dan,Cheng Jiangbo,Nie Fengqin,Li Ming,Lin Daozhe,Fu Wenying
(College of Agricultural,University of Hainan,Haikou Hainan 570228,China)
The genetic diversity and cluster analysis of the202Morinda citrifolia germplasm resourceswere analyzed by using ISSRmolecularmarkers.The results showed that13 ISSR primerswhich amplified 65 loci,meaned the average of each primer could amplified five loci.Among them,63 bands(96.92%)were polymorphic.Cluster analysis result indicated that202 germplasm resources could be clustered into five groupswhen the genetic similarity coefficientwas0.63,revealedthe characteristicsofgeographical distribution.The average of Shannon information index(I),Nei's genetic diversity(H)and effective number of alleles(Ne)was 0.5206,0.3488 and 1.6042,respectively,which indicated there was highly genetic diversity in these 202 Morinda citrifolia resource.
Morinda citrifolia;ISSR;genetic diversity;cluster analysis

S718.46
A
2095-1914(2016)02-0072-06
10.11929/j.issn.2095-1914.2016.02.012
2016-12-03
国家自然资金项目(31360066)资助。
第1作者:廖丹(1989—),女,硕士生。研究方向:种质资源的保护和利用。Email:1015251405@qq.com。
符文英(1962—),女,硕士,教授。研究方向:作物遗传育种。Email:fwynoni@163.com。
