癌症基因组学相关数据管理与应用探析
2016-03-21,,,
,, ,
美国癌症基因组图谱(The Cancer Genome Atlas, TCGA)计划历经10年完成了阶段性任务[1],推动了癌症基因组学研究的发展,为大规模癌症基因组学研究计划的实施提供了参考。2006年,在美国国立卫生研究院(National Institutes of Health,NIH)的组织领导下,美国国立癌症研究所(National Cancer Institute,NCI)和国立人类基因组研究所(National Human Genome Research Institute,NHGRI)联合启动了癌症基因组图谱计划[2]。该计划旨在通过大规模收集特定癌症患者的临床信息、影像信息、肿瘤组织及部分对应的正常组织样本,对其进行全面的基因组数据分析,从而获得一个全面的癌症基因组“图谱”,找到癌症相关的基因组变异并为其编制目录,实现数据共享,促进癌症的早期诊断和精准治疗,并预防癌症的发生。
本文将从TCGA计划的数据管理相关机构、工作流程、数据分类及开放共享、数据应用等方面对TCGA进行调研,为建立和完善大型的开放癌症基因组学数据库及其数据开放和利用提供参考借鉴。
1 癌症基因组数据管理
1.1 数据管理相关机构
TCGA计划涉及多个负责数据收集和处理分析的相关机构(图1),具体包括组织样本采集站(Tissue Source Sites,TSSs)、样本处理中心(Biospecimen Core Resource,BCR)、基因组测序中心(Genome Sequencing Centers,GSCs)、基因组特征研究中心(Genome Characterization Centers,GCCs)、数据调度中心(Data Coordinating Center,DCC)、癌症基因组中心(Cancer Genomics Hub,CGHub)和基因组数据分析中心(Genome Data Analysis Centers,GDACs)等[2],其工作流程基本如下。
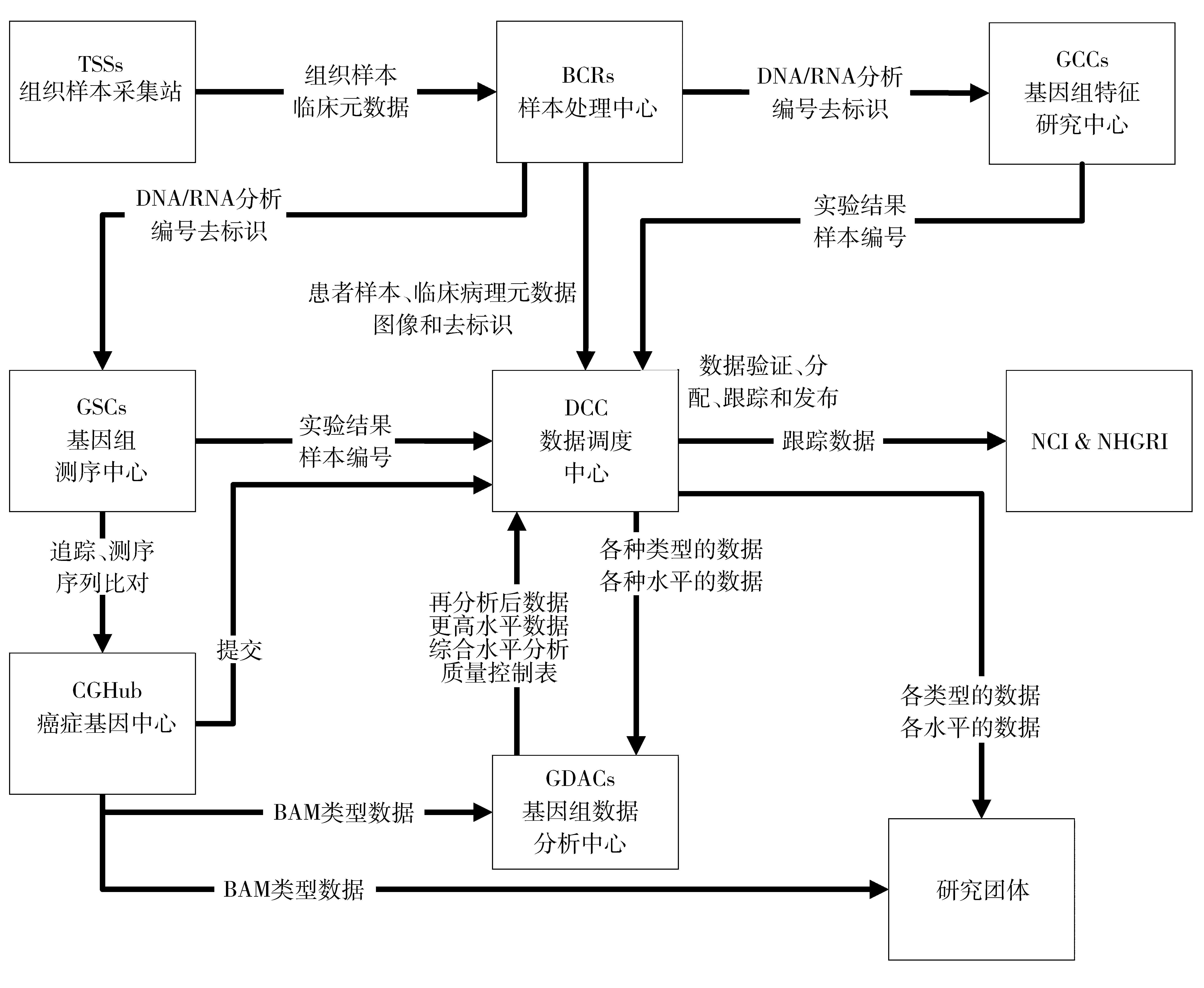
图1 美国癌症基因组图谱数据管理数据流[2]
组织样本处理:组织样本采集站(TSSs)收集志愿者的生物样本(肿瘤组织和正常组织)及临床元数据并提交给样本处理中心(BCRs),样本处理中心(BCRs)从样本中提取待分析的物质(DNA、RNA、蛋白质等),并检测以达到数量和质量的要求,同时为样本编码并去除患者隐私信息。
科学研究发现:待分析的物质由样本处理中心(BCRs)分别提交给基因组特征研究中心(GCCs)和基因组测序中心(GSCs),并分别进行基因组变异特征分析和识别特定癌症的DNA、RNA序列变化,基因组数据分析中心(GDAC)对来源于各个序列描述平台的数据进行整合,研发并提供新的信息处理、分析和可视化工具,以使癌症基因组图谱的数据得到充分利用。
科学数据共享:TCGA计划所收集和产生的各类数据由数据调度中心(DCC)集中管理,并通过数据门户(The TCGA Data Portal)等平台促进数据开放共享,使所有研究者根据其研究目的获取和利用所需数据。
数据驱动的科研协作:TCGA计划促进交叉学科研究团队从不同分子层面整合不同癌症表型信息协同开展科学研究,共同研究癌症发病机理,发现致病因素,提供精准的治疗方案,进而有效降低癌症疾病负担[3]。
1.2 数据分类
TCGA计划收集了11 000名患者、33种癌症的样本数据[4](表1)。2015年,TCGA计划所收集和产生的数据量已达20PB,其中包括1 000万个突变信息[1]。研究者可自行选择和下载所需的癌症数据并进行分析。据TCGA计划管理办公室的不完全统计,截至2014年底,已有2 700多篇已发表的研究文章使用了TCGA计划所收集和产生的数据[4]。

表1 美国癌症基因组图谱(TCGA)计划癌症样本数量分布
注:该表为截至日期为2016年1月14日
TCGA研究团队针对上述各种癌症,收集和产生了多种类型的组学和临床相关数据,主要包括基因表达,外显子表达、小RNA表达、拷贝数改变(CNV)、单核苷酸多态性(SNP)、杂合性缺失(LOH)、基因突变、DNA甲基化和蛋白质表达等组学数据,以及患者的基本资料、治疗进程、临床分期和生存状况等临床相关数据。
对于每种类型的数据,TCGA研究团队根据其加工处理程度划分为4个水平,使研究者可根据其研究需要选择不同处理水平的数据。TCGA计划的数据处理程度总体界定如表2所示。由于每一个中心和平台都会产生多种类型的数据,而各中心和平台分别根据其数据类型和所采用的分析算法对数据水平分类进行界定,因此各中心和平台之间的界定标准可能会略有不同[5]。
1.3 数据共享机制
TCGA计划根据数据粒度,将所收集和产生的数据分为汇总数据和个体数据,并分别采取不同的数据共享机制,即汇总数据可开放存取,用户使用时不需要进行认证。而个体数据须受控访问,用户须填写数据访问申请,经审核同意后方可下载使用数据。这两种数据共享机制的不同之处详见表3。
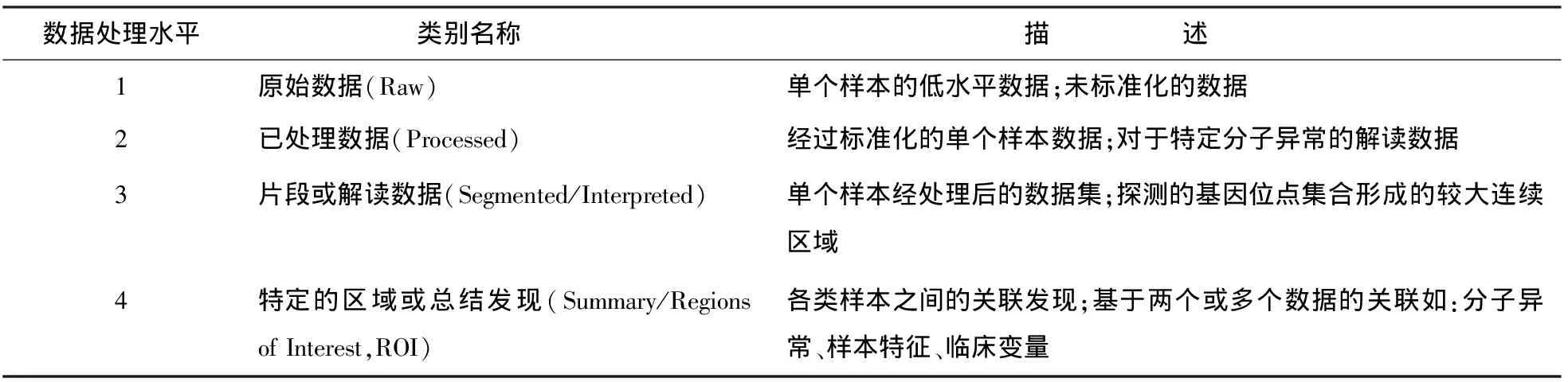
表2 美国癌症基因组图谱(TCGA)计划数据处理程度划分
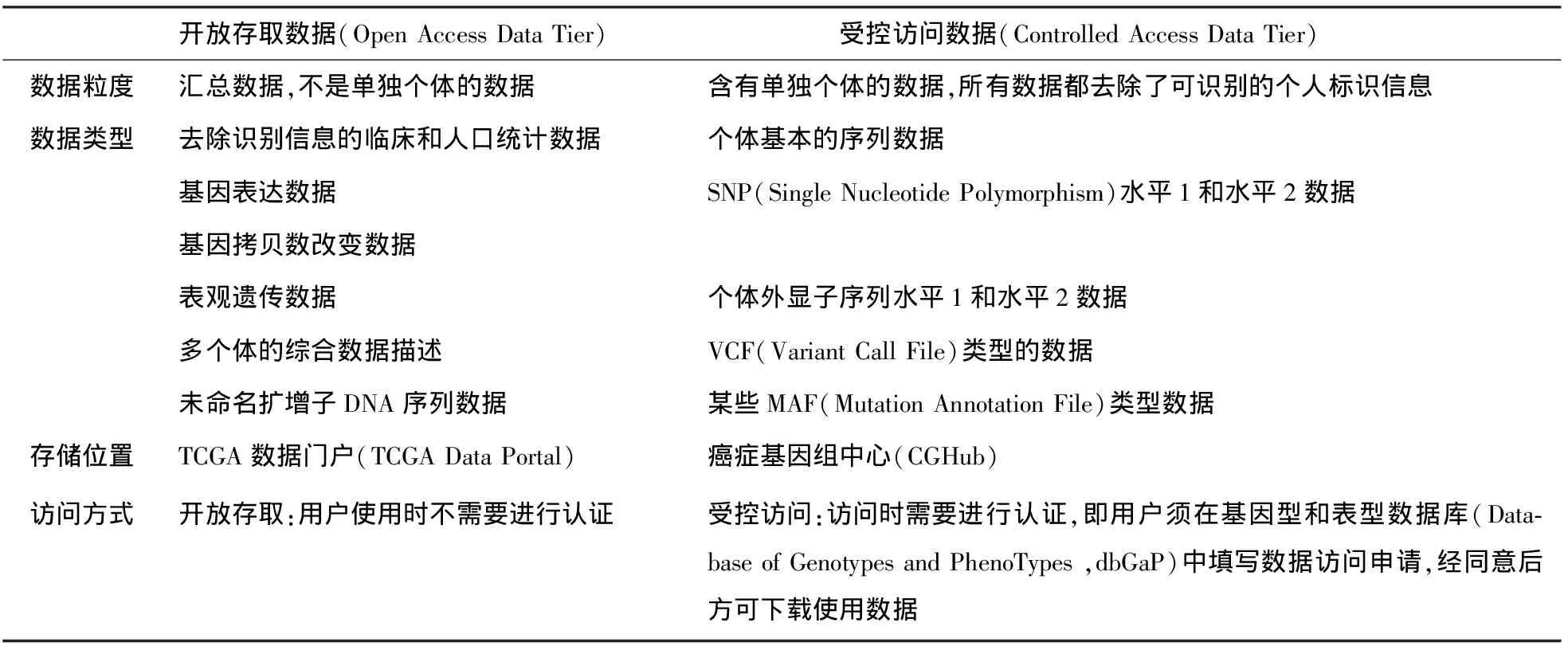
表3 美国癌症基因组图谱(TCGA)计划的数据共享机制比较
2 应用领域
TCGA计划已覆盖恶性胶质瘤、乳腺癌、卵巢癌、肺癌、结直肠癌、肾透明细胞癌、白血病、子宫内膜癌、膀胱移行细胞癌、胃腺癌等30多种癌症及其亚型(表1)。TCGA研究团队及其他相关研究者利用其共享数据开展了大量研究,包括癌症特征基因的突变、染色体扩增和缺失以及受影响的信号通路等。基于多个高通量实验平台产生的数据,开展癌症基因组学研究,为分子水平癌症分类研究开辟了新视角。下面以研究成果中的乳腺癌、前列腺癌相关发现及泛癌计划为例,对TCGA计划的数据应用情况进行介绍。
2.1 乳腺癌
2012年,TCGA研究团队通过对乳腺癌相关的基因组DNA拷贝数阵列、DNA甲基化、外显子测序、mRNA阵列、小RNA序列阵列和反相蛋白阵列等数据的整合分析,发现了4个主要的分类亚型,且每种亚型都有显著的分子异质性[6]。2015年TCGA研究团队与瑞士洛桑大学遗传学系、美国斯隆凯特林癌症中心等20多个机构的研究者合作,利用TCGA计划的多个平台的分析数据,包括817个乳腺癌样本,分析小叶样乳腺癌和导管样乳腺癌的分子差异,找到了其发病机制中的不同通路;同时根据细胞增殖及免疫相关基因的表达差异,定义了新的小叶样乳腺癌亚型(reactive-like, immune-related, proliferative),发现潜在的治疗靶点[7]。此外,研究者利用TCGA计划的数据验证其研究结果,通过整合一个大型的小叶样乳腺癌患者队列中的基因组、转录组及蛋白质组数据,找到两个生物学方面有显著差异的亚型,并利用TCGA计划乳腺癌的基因表达数据,用相同的聚类方法,显示出类似的生物学差异[8]。这些差异可通过相应靶向的化疗或免疫疗法改善治疗效果,为精准治疗方案的制定提供依据。
2.2 前列腺癌
有研究通过筛查TCGA计划所收集和产生的前列腺癌的差异表达的小RNA数据,分析靶基因的功能和信号通路,发现了6种差异表达的小RNA及它们的靶基因,可以作为前列腺癌治疗过程中的预后生物标记[9]。TCGA研究团队对原发性前列腺癌的333个样本的多个平台(包括外显子组、全基因组测序、RNA测序、小RNA测序、SNP微阵列、DNA甲基化微阵列、反相蛋白微阵列)的分析数据进行了全面的分子学分析,发现74%的原发性前列腺癌可根据基因融合和突变分为7个亚型,不同亚型之间存在表观遗传学和激素受体活性差异。该发现对前列腺癌的分子诊断与靶向治疗具有重要意义[10]。
2.3 泛癌计划
随着研究的深入和相互关联,研究者发现在不同的癌症中会存在相似的分子模式。为此,TCGA计划的研究者于2012年启动了泛癌计划(Pan-Cancer Project)。根据当时的数据可及性和完整性,计划选取多形性成胶质细胞瘤、急性骨髓性白血病、头颈部鳞状细胞癌、肺腺癌、肺鳞状细胞癌、乳腺癌、肾透明细胞癌、卵巢癌、膀胱癌、结肠腺癌、子宫颈与子宫内膜癌、直肠腺癌等12种癌症,共计3 000多个样本的基因突变(包括单核苷酸变异和结构变异)、DNA拷贝数改变、基因表达、DNA甲基化、小RNA测序、反向蛋白阵列等组学数据和临床相关数据进行整合分析,探寻不同癌症的相似生物通路[11]。
在该计划中,来自30多个机构的250位研究者开展协作研究,进行数据的处理、分析和知识发现[12]。该计划的开展,为大型协作研究提供了一个可行的模式。此外,泛癌计划的研究结果为不同病发部位肿瘤的系统生物学研究提供了可行性。
美国斯隆凯特林癌症中心的Giovanni Ciriello等人利用生物信息学算法对12种癌症的3 299个癌症样本进行了层次分类,将这些癌症分为原发性体细胞变异类型(M类)和原发性拷贝数改变类型(C类),揭示了癌症形成中不同的致癌过程。研究发现的层次分类结果表明肿瘤形成过程中存在不同的致癌标记,为不同阶段的癌症治疗提供了新思路[13]。另外有研究分析了泛癌数据集中11种癌症的4 934个原位癌样本的体细胞拷贝数改变(somatic copy number alteration,SCNA)情况,发现了不同癌症有着相同的SCNA模式,约37%的癌症有全基因增倍(whole-genome doubling)同时伴有相当高的体细胞拷贝数改变[14]。
Nature杂志于2013年创建了 TCGA泛癌分析(TCGA Pan-Cancer Analysis)专栏,总结了TCGA泛癌计划包括突变驱动、网络模型、暴露与致病因素、数据发现、未来方向等方面的研究成果[15]。
3 对我国的启示
目前,我国的基因组学等生物科研数据共享与数据汇交工作已启动[16],但仍缺少与之匹配数据管理制度和技术支撑[17]。TCGA计划的数据管理经验可为国家级大型的癌症基因组学相关数据资源管理提供参考。
3.1 加强多中心合作,落实项目管理制度
人类基因组计划开启了多中心、多机构合作模式,之后团体合作的基因组工程随之而来,包括千人基因组计划、TCGA计划和人类微生物组计划等。合作模式下的科学研究将会使更多人获益,各个参与其中的中心或机构,利用相同的基础设施、分析工具,遵循统一制定的政策及数据标准,用一致的共享技术开放数据,保证研究后期能够在最大程度上实现数据的统一管理[18]。
3.2 做好数据分析计划,建立全链条的数据管理流程
TCGA计划建立了组织样本采集、处理、质量控制、序列测定、变异特征分析、数据共享与研究应用等全链条的癌症基因组图谱数据管理流程。在建立大型相关数据资源时,需要对数据分析早期进行统筹规划,确保不同科研中心的数据产生、传递、存储、共享及利用等操作的相互衔接与规范化,保证数据的完整性和准确性。在大规模癌症基因组学研究计划的实施过程中,可参考其数据管理相关机构的合作方式,各个科研中心负责链条中的某项特定工作,最终数据汇总呈现于数据调度中心实现数据共享。
3.3 加强分级分类管理,促进数据开放共享
实现生物科研数据共享是一个系统工程,需进行需求分析、资源调查和分级分类等研究。TCGA计划从所属癌症、数据类型、处理水平、数据粒度等角度对数据进行精细分类,根据数据类型定义不同用户的数据访问权限以及开放共享数据的内容。
TCGA计划采用了两级数据发布系统,一部分数据全面开放,另一部分仅可用于研究性目的,研究人员和机构得到授权后才可使用相应数据。在充分保护患者隐私的情况下实现癌症基因组数据的优化与共享。在此方面,我国需要加大科学数据精细标识与分级分类管理,在保障个人隐私和信息安全的前提下,实现数据的开放共享。
4 结语
通过分析癌症基因组信息了解癌症发生发展机理,发现癌症标志物和药物作用基因靶点,可为癌症的精准诊断和治疗提供支撑。TCGA计划收集了大量癌症基因组与临床表型的数据,其中蕴藏着潜在的癌症的分子标记物和药物靶点有待挖掘,科学的数据管理方案为癌症基因组研究提供了保障。癌症基因组图谱计划在数据管理方面的实践探索可为精准医学等大科学计划的开展和实施、为数据驱动的协作研究模式提供参考[19]。
