利用靶拟态合成和Gateway技术构建水稻miR164c沉默突变体
2016-03-09吴多周艳宋傲群刘浏史筱靓
吴多++周艳++宋傲群++刘浏++史筱靓++姜孝成

摘 要 以水稻基因组DNA为模板, PCR扩增得到与miR399互补的OsMIM399后,改造成与miR164c互补的靶拟态序列OsMIM164c,经测序分析后连入pGWC载体,获得入门载体pGWCOsMIM164c;利用Gateway技术,将入门载体pGWCOsMIM164c经LR反应连接到植物表达载体GCS中,获得GCSOsMIM164c表达载体,经农杆菌介导将该表达载体导入水稻品种Kasalath的愈伤组织中,组织培养后获得再生植株并进行目的基因的PCR鉴定, Real Time PCR方法检测阳性突变体植株中miR164c的表达量. 研究结果表明,miR164c在突变体植株中的表达量显著低于野生型,说明miR164c沉默突变体构建成功.
关键词 靶拟态; Gateway技术; miR164c; 沉默突变体; 水稻
中图分类号 Q785; Q943.2 文献标识码 A 文章编号 10002537(2016)01002406
Construction of Silence Mutant of miR164c in Oryza Sativa L.
by Target Mimicry and Gateway Technology
WU Duo**, ZHOU Yan**, SONG Aoqun, LIU Liu, SHI Xiaoliang, JIANG Xiaocheng*
(College of Life Sciences, Hunan Normal University, Changsha 410081, China)
Abstract Genomic DNA of rice (Variety ZR02) was used as template to amplify OsMIM399 by PCR which has a region complementary to miR399, and OsMIM399 was reconstructed into the target mimicry OsMIM164c with a region complementary to miR164c. After sequencing, the right fragment was ligated into pGWC vector to construct pGWCOsMIM164c, and OsMIM164c was then transferred to the plant expression vector GCS to prepare GCSOsMIM164c through LR reaction. GCSOsMIM164c was finally transformed into rice Kasalath calli by the improved Agrobacteriummediated method and regenerated plantlets were obtained by tissue culture technique. After target gene OsMIM164c was identified by PCR, the expression of miR164c in positive mutants was analyzed by RT qPCR. The results showed that the expression of miR164c exhibited a distinct reduction in mutant plants compared to the wild type, indicating that the miR164c silence mutant has successfully been prepared.
Key words target mimicry; gateway technology; miR164c; silence mutant; rice
microRNA(miRNA)是一类长度为20~24 nt的非编码小分子RNA,在转录后水平对靶mRNA进行剪切或抑制其翻译,继而调控靶基因的表达[1]. 每个miRNA家族中有多个成员,各成员核苷酸序列极其相似,对基因的调控作用表现出冗余效应. 因此,沉默其中一个miRNA很难出现预期的表型;通过常规的构建基因沉默载体方法来研究miRNA的功能丧失(loseoffunction)存在很大困难[2]. 2007年,Jose等在研究低磷诱导下miR399的功能时,发现一个非编码的基因IPS1,它是TPS1大家族中的一员. TPS1家族成员都含有一段非保守的短开放阅读框(ORF),具编码四肽(MetAlaIlePro)的功能[36];另一个共同特征是含有一个保守的23 bp的基序,该基序与miR399可形成不完美配对,即配对时在第10~11座位错配而出现一个突起环,致使IPS1可以与miR399特异结合而不被切割,因此,过表达IPS1可导致miR399的功能沉默,从而导致miR399的靶基因PHO2的表达上调. Jose把这个抑制miRNA活性的机制称为“靶拟态(target mimicry)”,并通过制造人工靶拟态抑制了相关miRNA的活性[7]. “靶拟态”机制使得专一性抑制目的miRNA的活性成为可能,为研究miRNA的功能提供了新的策略和方法.
目前普遍采用高通量基因克隆技术(Gateway Cloning Technology,简称Gateway技术)以获得目的基因的沉默突变体[8]. 该技术在被称为“入门克隆(Gateway Entry Clone)”中目的DNA片段的插入位置两端分别整合att L1和att L2侧翼重组序列(Flanking Recombination Sequence),形成一个类似通道的结构. 入门克隆与表达载体可以通过LR反应(即在LR反应酶作用下入门克隆的目的基因两端的attL1和attL2位点与表达载体的attR1和attR2位点发生定向重组),从而将目的基因转移到表达载体. 该方法无需进行限制性内切酶酶切等操作,简易高效.
在前期研究中,作者发现人工老化处理的水稻种子随着活力下降,其胚中miR164c的表达量上调,因此推测miR164c可能参与水稻种子活力的调控[9]. 作者试图整合靶拟态合成技术和Gateway技术,制备水稻miR164c沉默突变体,为进一步研究miR164c调控种子活力的分子机制奠定基础.
1 材料与方法
1.1 材料
水稻Kasalath、水稻ZR02、大肠杆菌(Escherichia coli)TOP10、农杆菌(Agrobacterium tumefaciens)Ag10和EHA105(湖南师范大学植物发育与分子生物学实验室提供);pGWC Vector,GCSAsRedpCambia1301UbiN Vector(北京大学生命科学学院邓兴旺实验室惠赠). 限制性内切酶AhdⅠ(Fermentas公司),Gateway LR Clonase Enzyme Mix(Invitrogen),反转录系统及AllinOne miRNA Detection Kit(GeneCopiea公司),PrimeSTAR HS DNA聚合酶、质粒提取及胶回收试剂盒(上海生工生物公司),TRIzol及引物的合成(Invitrogen),其他为国产或进口分析纯试剂.
1.2 方法
1.2.1 OsMIM164c的制备
1)引物设计 以水稻基因组数据库中序列IPS1为模板,运用primer 5.0软件设计引物OsIPS1F(Primer A)和OsIPS1R(Primer B),见表1;将IPS1中本来与miR399互补的序列(图1)替换为与miR164c互补的序列(图2),在IPS1的上、下游分别设计引物OsIPS1miR164cF(Primer C)和OsIPS1miR164cR(Primer D),然后在引物 C的5′端加上需要改造的碱基TCTA,引物 D的5′端加上需要改造的碱基TAGA(TAGA与TCTA彼此反向互补配对),见表1.
注:带下划线部分为与miR164c互补的序列;引物设计(背景颜色对应于上述OsIPS1的相应部位):
OsIPS1F(Primer A) 5′GCAGAAAGCCCCTCAAATCTCAAAGAGGCACCAATAC3′
OsIPS1R(Primer B) 5′GGCACACACAATCTTAATTACTAATATCAAATTCATC3′
OsIPS1miR164cF(Primer C) 5′TGCACGTACC TCTA TGCACGTACCATTATTCGGTGGATG3′
OsIPS1miR164cR(Primer D) 5′TGGAGAAGCA TAGA GG TAC GTG CA CCTTAGTAGAGGTAA3′
其中,带“ ”部分为改造的与目的miRNA互补配对的片段; 带“ ”部分为不与miRNA互补配对而设计的碱基
图2 改造的与miR164c互补的 IPS1序列
Fig.2 The remoulded sequence of IPS1 complementary to miR164c
注:带“ ”部分为与miR164c互补配对的序列, 为不与miR164c匹配的部位,带波浪线部分为BglⅡ的切割识别部位
图3 OsMIM164c序列
Fig.3 The sequence of OsMIM164c
2)重叠PCR制备OsMIM164c
按陈锐等的方法[10]进行.以水稻ZR02叶片为材料,CTAB法[11]提取基因组DNA,以引物(OsIPS1F与OsIPS1R )PCR获得OsIPS1片段;以OsIPS1为模板,引物(OsIPS1miR164cF与OsIPS1R)PCR获得OsIPS13′端片段;用引物(OsIPS1miR164cR与OsIPS1F)PCR扩增获得OsIPS15′端片段;(扩增用pfu酶);以OsIPS13′端片段和OsIPS15′端片段为模板,加入引物(OsIPS1F与OsIPS1R)和dNTP及pfu酶,进行30循环PCR,胶回收获得OsMIM164c片段.
1.2.2 pGCWOsMIM164c入门载体构建 构建带OsMIM164c的pGCW入门载体步骤如下: (1)酶切反应:pGCW空载体 7 μL,Buffer 0.2 μL,ddH2O 10 μL,AhdⅠ1 μL,混匀后37 ℃水浴 5 h. (2)电泳:1%琼脂糖凝胶电泳,回收大片段. (3)连接:用回收纯化后的带有AhdⅠ酶切位点的OsMIM164c PCR产物与pGWC酶切片段通过快速连接酶在16 ℃水浴锅中连接1 h.(4)转化:连接后转导入感受态细胞TOP10中,混匀,冰浴 2~3 min;42 ℃热激 90 s,再冰浴 2~3 min;加入2 mL LB 培养基,于 37 ℃振荡培养 45 min.(5)培养:取适量菌液均匀涂布于 LB 平板(含氯霉素12.5 mg/L),培养皿倒置于培养箱中,37 ℃培养过夜,再挑取菌落划线培养.(6)PCR鉴定:挑选阳性克隆进行PCR鉴定后,铂尚生物技术(上海)有限公司测序确认.
选取测序正确的克隆制备质粒. 挑取相应的阳性菌落,分别接种于5 mL LB液体培养液中 (含氯霉素125 mg/L),37 ℃振荡培养过夜后,参照SanPrep柱式质粒DNA小量抽提试剂盒说明提取质粒. 1%琼脂糖凝胶电泳检测,-20 ℃保存.
1.2.3 构建GCSOsMIM164c表达载体 LR反应创建表达载体GCSOsMIM164c. 在0.2 mL的离心管中加入以下反应体系(10 μL):3.5 μL入门载体pGWCOsMIM164c,1.5 μL植物空表达载体GCS,1 μL Buffer,15 μL LR酶,2.5 μL ddH2O. 混匀,瞬时离心后,25 ℃水浴1 h;取3 μL 反应液转导入感受态细胞TOP10中(方法同1.2.2),取适量菌液涂布的LB平板(含潮霉素30 mg/L)上,涂布均匀后,培养皿倒置于培养箱中,37 ℃ 恒温箱中过夜培养. 转化后挑取菌落划线培养,筛选阳性克隆,提取质粒(方法同1.2.2).
植物表达载体GCSOsMIM164c酶切鉴定: 反应体系20 μL:GCSOsMIM164c 7 μL,BglⅡ1 μL, Buffer 02 μL, ddH2O 10 μL. 37 ℃ 水浴锅,酶切6~8 h,1%琼脂糖电泳(5 V/cm)检测.
1.2.4 农杆菌介导GCSOsMIM164c遗传转化水稻Kasalath 参照周艳[12]的方法.
1.2.5 突变体植株的分子鉴定 选取T0代抗性植株,采用CTAB[11]法提取水稻基因组DNA,根据GCSIPS1的嵌合片段设计上游引物PGCSF,根据目的基因设计下游引物PIPS1R,引物序列见表1,进行PCR扩增.
1.2.6 RTqPCR分析 取水稻组织(叶、茎或种子等)样品100 mg,液氮研磨至粉末状,迅速转移至预冷的1.5 mL EP管中,采用TRIzol法提取总RNA. 非变性琼脂糖凝胶电泳及紫外分光光度计检测后,按AllinOne miRNA Detection Kit(GeneCopoeia)说明操作,合成cDNA第一链. 根据PremiR164c序列设计RTqPCR正向引物OsmiR164cF,并以Os5.8s为内参基因(正向引物为Os5.8sF),引物序列见表1,反向引物采用试剂盒中的通用引物. 以反转录得到的cDNA为模板,按Thermo Scientific PCR0096 Realtime PCR系统的说明操作,PCR反应重复3次. RTqPCR检测的反应体系10 μL:2×AllinOne qPCR Mix 5 μL,正、反向引物(02 μmol/L)各1 μL,diluted cDNA(1∶5)1 μL,ddH2O 2 μL. 荧光定量PCR扩增程序:95 ℃预变性10 min;95 ℃变性10 s,60 ℃退火20 s,72 ℃延伸15 s,40个循环,最后以每6 s上升0.5 ℃的速度从65 ℃到95 ℃记录熔解曲线,实验数据用Piko Real Software2.0分析.
2 结果和分析
2.1 OsMIM164c目的片段和GCSOsMIM164c载体的鉴定
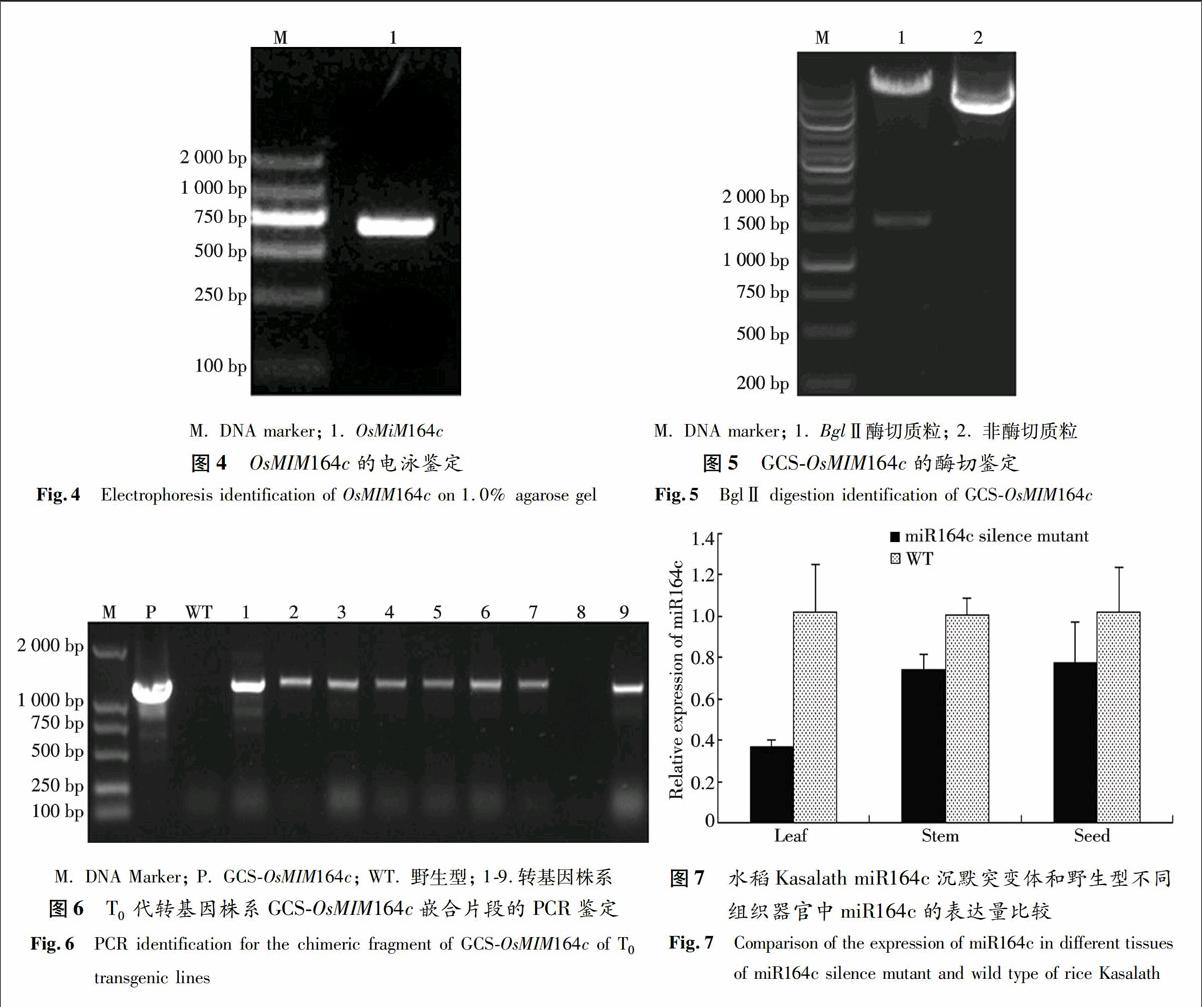
以水稻ZR02的基因组DNA为模板扩增得到的OsIPS1片段预期大小656 bp. 运用重叠PCR技术将OsIPS1改造为与miR164c不完全互补的片段OsMIM164c,改造后片段与IPS1大小一致(图4), 测序结果符合预期. 将GCSOsMIM164c用BglⅡ进行酶切后电泳检测,结果如图5所示,图中1 580 bp左右的条带与预期一致,表明目的片段已经成功连入载体,表达载体构建成功.
2.2 miR164c沉默突变体的分子鉴定
挑取9个转基因株系,分别提取其叶组织的基因组DNA,以GCSOsMIM164c做阳性对照,野生型做阴性对照. 结果表明,其中8个株系扩增出特异性目的条带(图6),说明已成功获得了miR164c沉默突变体.
2.3 沉默突变体的miR164c表达量分析
运用SYBR Green I染料法对miR164c沉默突变体的叶、茎和种子中的miR164c表达量进行RTqPCR分析(分别以野生型的叶、茎和种子作对照). 结果表明,miR164c在沉默突变体的三类组织器官中的表达量均比野生型低(图7).
3 讨论
miRNA的作用方式主要是转录后调控靶基因,广泛参与植物生长发育、抗逆性等方面. miRNA靶拟态(target mimicry)能够有效地抑制相关miRNA的活性. 通过定向改造IPS1基因构建人工miRNA靶拟态(Target mimicry)来下调miRNA表达,已成为研究miRNA功能的一种重要方法. Todesco等[13]利用靶拟态技术已经成功构建了在拟南芥中表达的MIM156,MIM157,MIM159,MIM160,MIM164,MIM167,MIM170,MIM172和MIM319等靶拟态突变体株系,使相应的靶miRNA表达量显著下调并获得明显表型. 水稻是单子叶模式植物,吕明芳[14]用靶拟态技术获得了沉默miR156功能的植物转化载体. 本研究改造水稻的IPS1序列,使其与miR164c配对时形成一个错配环,获得了OsMIM164c. 荧光定量PCR结果表明,突变体植株中miR164c的表达量在叶、茎和种子中均明显下调.
Gateway技术是invirtogen公司开发的一种高通量克隆技术,已被广泛运用. 比如,徐化学等构建了水稻基因OsDAD1基因的RNA干扰载体[15];耿卫东等构建了棉花GhSAMDC基因RNA干扰载体和过表达载体[16];郭姗姗等构建了植物表达载体p1104D[17];高东尧等构建了含有双35S启动子的高效表达载体以验证小麦穗发芽抗性相关Vp1基因的功能[18];何敏等构建了靶向人Akt基因的shRNA表达载体[19]等等. 该技术可以将入门克隆的重组片段(目的基因)转移到各种不同的表达载体中,由于在重组时DNA片段的阅读框和方向保持不变,因而不影响同步表达克隆的测序结果;在使用每一种新的表达系统时,能节省更多的时间. 此外,Gateway构建过程没有限制性内切酶和连接酶的参与,比普通酶切法的转化效率高. 因此,该方法具有操作简便、反应条件容易控制、反应后阳性克隆易于检测的特点,适合大规模的载体构建,为高通量地研究植物基因功能提供了有力支持. 另外,利用该技术构建的是一种二元载体,可以直接用于农杆菌介导的植物转化.
本文通过整合靶拟态技术和Gateway技术,快速高效获得了miR164c的靶拟态沉默突变体,通过PCR检测,阳性率高达89%, 为进一步探讨miR164c对水稻种子活力调控的分子机制及其他生物学功能奠定了基础.
参考文献:
[1] LEWIS B P, SHIH I H, JONESRHOADES M W, et al. Prediction of mammalian microRNA targets [J]. Cell, 2003,115(7):787798.
[2] 陈龙俊, 侯 蕾, 纪鸿飞. 盐芥miRNA393沉默载体的构建[J]. 现代农业科技, 2010(23):1114.
[3] BURLEIGH S H, HARRISON M J. The downregulation of Mt4like genes by phosphate fertilization occurs systemically and involves phosphate translocation to the shoots[J]. Plant Physiolo, 1999,119(1):241248.
[4] LIU C, MUCHHAL U S, RAGHOTHAMA K G, et al. Differential expression of TPS11, a phosphate starvationinduced gene in tomato[J]. Plant Mol Biol, 1997,33(5):867874.
[5] MARTIN A C, POZO J C, IGLESIAS J, et al. Influence of cytokinins on the expression of phosphate starvation responsive genes in Arabidopsis[J]. Plant J, 2000,24(5):559567.
[6] SHIN H, SHIN H S, CHEN R, et al. Loss of At4 function impacts phosphate distribution between the roots and the shoots during phosphate starvation[J]. Plant J, 2006,45(5):712726.
[7] FRANCOZORRILLA J M, VALLI A, TODESCO M, et al. Target mimicry provides a new mechanism for regulation of microRNA activity [J]. Nat Genet, 2007,39(8):10331038.
[8] 王聪颖, 陈书霞, 陈 巧, 等. 利用Gateway技术构建黄瓜HPL基因的RNA干扰载体[J]. 西北农业学报, 2013,22(2):152158.
[9] 成海兰. miRNA与水稻种子活力的相关性研究[D]. 长沙: 湖南师范大学, 2011.
[10] 陈 锐, 胡 正, 张 辉. 小分子RNA表达载体构建的新方法—MicroRNA前体PCR置换法[J]. 生物技术通报, 2010(7):101105.
[11] 敬国兴, 赖小玲, 李伏钦, 等. 植物基因组DNA提取—本科分子生物学实验方法改进[J].中国教育技术装备, 2008(20):127128.
[12] 周 艳, 吴亚先, 姜孝成, 等. 农杆菌介导的遗传转化籼稻Kasalath的条件优化[J]. 湖南农业大学学报, 2013,39(5):471477.
[13] TODESCO M, RUBIOSOMOZA I, PAZARES J, et al. A collection of target mimics for comprehensive analysis of microRNA function in Arabidopsis thaliana[J]. PLoS Genet, 2010,6(7):e1001031.
[14] 吕明芳. 水稻miR156基因克隆及其功能初步分析[D]. 杭州: 浙江师范大学, 2011.
[15] 徐化学, 熊建华, 傅彬英. 应用Gateway技术构建水稻OsDAD1基因的RNA干涉载体[J]. 分子植物育种, 2007,5(1):133136.
[16] 耿卫东, 李艳军, 张新宇, 等. Gateway技术构建棉花GhSAMDC基因植物表达载体[J]. 石河子大学学报:自然科学版, 2012,30(2):138141.
[17] 郭姗姗, 张 蒙, 单卫星. 基于Gateway技术的植物表达载体的构建[J].西北农林科技大学学报:自然科学版, 2010,38(11):161166.
[18] 高东尧, 夏兰琴, 徐兆师, 等. 小麦WBp1基因表达载体的Gateway技术构建及其遗传转化[J]. 麦类作物报, 2009,29(2):189194.
[19] 何 敏, 赵学芹, 李 曼, 等. 应用Gteway技术构建pLenti6Akt shRNA真核表达载体[J]. 国际检验医学杂志, 2012,33(15):17981720.
(编辑 WJ)
