茶树中性/碱性转化酶基因CsINV10的克隆与表达分析
2016-03-03钱文俊曹红利郝心愿王玉春黄玉婷王新超杨亚军西北农林科技大学园艺学院陕西杨凌700中国农业科学院茶叶研究所国家茶树改良中心农业部茶树生物学与资源利用重点实验室浙江杭州30008
钱文俊 岳 川 曹红利 郝心愿 王 璐 王玉春黄玉婷 王 博 王新超 肖 斌 杨亚军,*西北农林科技大学园艺学院, 陕西杨凌 700;中国农业科学院茶叶研究所 / 国家茶树改良中心 / 农业部茶树生物学与资源利用重点实验室, 浙江杭州 30008
茶树中性/碱性转化酶基因CsINV10的克隆与表达分析
钱文俊1,2岳川2曹红利2郝心愿2王璐2王玉春1,2黄玉婷2王博2王新超2肖斌1,*杨亚军1,2,*
1西北农林科技大学园艺学院, 陕西杨凌 712100;2中国农业科学院茶叶研究所 / 国家茶树改良中心 / 农业部茶树生物学与资源利用重点实验室, 浙江杭州 310008
摘要:基于课题组前期对茶树冷驯化系统的转录组测序分析结果, 从中挑选出6条与中性/碱性转化酶基因高度相似的EST序列, 电子拼接和RT-PCR验证后获得一条全长为2101 bp的核酸序列。该基因包含1923 bp的ORF, 编码640个氨基酸, 蛋白分子量为71. 8 kD, 理论等电点为5.69。根据BlastX同源性比对显示, 该基因与荔枝LcNI相似性最高(80%), 为G100家族成员, 属于中性/碱性转化酶基因, 将其命名为CsINV10 (GenBank登录号为KT359348)。对CsINV10氨基酸序列的系统进化树分析显示, 其与木薯MeNINV8亲缘关系最近。进一步分析显示, CsINV10的氨基酸序列无N端信号肽, 无跨膜结构域, 属于亲水性蛋白, 并定位在叶绿体上。荧光定量PCR分析表明, CsINV10具有组织表达特异性, 在茶树叶和花中的表达量最高, 根系中最低。分析发现, 低温(4℃)、干旱和盐胁迫分别处理茶树1 d后, 成熟叶片中CsINV10的表达呈逐渐上升趋势; 而在ABA条件处理下, 该基因呈先升高后降低趋势, 在处理5 d后基本不表达, 表明该基因可能参与茶树对多种逆境胁迫的响应, 这为后续进一步研究转化酶基因在茶树抗寒等逆境胁迫中的作用奠定基础。
关键词:茶树; 中性/碱性转化酶基因; 定量分析
本研究由国家自然科学基金项目(31170650), 国家现代农业产业技术体系建设专项(CARS-23), 浙江省自然科学基金项目(LY14C160001),浙江省农业新品种选育重大专项(2012C2905-3)和中国农业科学院科技创新工程项目(CAAS-ASTIP-2014-TRICAAS)资助。
This study was supported by the National Natural Science Foundation of China (31170650), the Earmarked Fund for China Agriculture Research System (CARS-23), the Natural Science Foundation of Zhejiang Province (LY14C160001), the Major Project for New Agricultural Varieties Breeding of Zhejiang Province (2012C2905-3) and the Chinese Academy of Agricultural Sciences through an Innovation Project for Agricultural Sciences and Technology (CAAS-ASTIP-2014-TRICAAS).
第一作者联系方式: E-mail: jajyqian9066@mail.tricaas.com
蔗糖作为非还原型双糖, 在大多数植物中基于异养型组织的生理和生化需要, 被运送到不同的亚细胞区域, 水解为葡萄糖和果糖, 参与到三羧酸循环和糖酵解途径中, 以维持植物正常生长发育和对逆境胁迫的抗性。双糖中的碳可用于生物合成原初代谢, 对植物生长发育非常重要。同样, 蔗糖可能转化为聚合物, 如淀粉、三酸甘油酯或多肽用于长期储存; 或者形成次级化合物使植物能够适应各种生物和非生物逆境胁迫[1]。蔗糖作为碳水化合物和能量的来源, 关键在于其能够水解为己糖, 在植物中主要靠两类蔗糖水解酶来催化这一反应: (i)蔗糖合成酶(Suc, EC2.4.1.13), 是一类糖基转移酶, 能够可逆水解蔗糖形成尿苷二磷酸葡萄糖和果糖, 主要在器官成熟过程中参与维持库强的稳固和合成库产物方面起作用; (ii)转化酶(INV, EC3.2.1.26), 能够不可逆地将蔗糖水解为葡萄糖和果糖, 在蔗糖分布和调控“库—源”关系方面具有十分重要的作用[2-4], 同时,该类酶能广泛参与调控植物生长、信号响应、原初碳代谢和逆境胁迫响应等方面[5-8]。
根据不同的细胞定位, 转化酶可以分为胞质转化酶(cytoplasmic invertase, CIN)、液泡转化酶(vacuolar invertase, VIN)和细胞壁结合转化酶(cell wall invertase, CWIN)三类, 其中CIN又可分为α和β亚族, α亚族主要定位于细胞器, 如线粒体和叶绿体; β亚族则主要定位于核内。而根据其生化活性和pH的不同又可分为酸性转化酶(acid invertase, AI)和中性/碱性转化酶(alkaline/neutral invertase, A/N-Inv)[9],前者pH在5.0左右时有较高的酶活性, 而后者pH 6.5~8.0左右时具有较高的转化酶活性。A/N-Inv还存在2种不同的异构体, 最佳酶活pH为6.5时为中性转化酶, pH为8.0时为碱性转化酶[10]。
目前, 关于转化酶在植物中作用的研究已经非常广泛, 特别是对酸性转化酶的研究, 涉及到酸性转化酶对植物生长发育、开花结果、生物和非生物逆境胁迫响应等各方面[11-18]。然而, 由于A/N-Inv并未被糖基化, 蛋白很难纯化, 具有不稳定性和较低的酶活性, 使得目前关于这类酶的研究较之酸性转化酶少。近年来, 陆续在胡萝卜[4]、小麦[10]、拟南芥[19]、水稻[20]、葡萄[21]、杨树[22]、百脉根[23]、甘蔗[24]、番茄[25]、木薯[26]等中克隆出A/N-Inv基因。最近的研究表明, A/N-Inv在植物生长和发育、能量代谢、碳水化合物平衡和环境胁迫响应方面也发挥着重要作用[10, 27-30]。借助于植物中的A/N-Inv基因突变, 证明A/N-Inv基因在植物生长发育中发挥重要作用。例如, 在拟南芥中有研究表明A/N-Inv功能的丧失能够降低根系原初生长[28,31], 另外, 敲除拟南芥叶绿体中一个At-A/N-InvE能够降低拟南芥中A/N-Inv活性, 并且在突变体植株中既检测不到A/N-Inv活性也检测不到含有A/N-Inv蛋白, 同时还伴随着淀粉含量的减少[27]。在水稻中突变一个与拟南芥同源的A/N-Inv基因OsCyt-inv1会导致水稻突变体根系变短, 推迟开花和部分不育, 然而通过外源施用葡萄糖又能修复根系的生长[32]。相似地, 在百脉根中将一个A/N-Inv基因LjINV2突变后发现其A/N-Inv活性和表型都不受影响, 然而, 对另一个A/N-Inv基因LjINV1突变处理发现, 其能够降低A/N-Inv活性, 且极大降低根系和茎干生长, 同时阻遏花粉形成和损害开花。有趣的是, 这种突变体能够形成功能性根瘤, LjINV1对植物整体生长具有重要作用, 但对茎节的形成和功能却并不重要[23]。然而, 目前依然未知A/N-Inv是如何调控细胞的生长, 同时, 有关A/N-Inv基因在响应植物逆境方面的作用研究也报道较少。
茶树作为多年生常绿叶用植物, 其生长和分布受到温度、光照、土壤、水分等多种环境因子的影响, 其中温度是限制茶树自然分布和产量的主要因素。近年来, 茶树低温危害成为茶叶生产最常见的环境逆境, 严重影响了茶叶的产量与经济效益。为此, 如何提高茶树抗寒性以抵御低温伤害已是近几年众多研究者们所关注的热点问题。众多研究表明,植物抗寒性的提高需要经过一定时间和程度的低温冷驯化才能表现出来[33]。Wang等[34]对冷驯化条件下的茶树进行转录组全局分析发现, 茶树在冷驯化条件下总共有1770个差异表达基因, 这些差异基因主要包括低温感应或信号转导基因、低温响应转录因
子基因、渗透响应相关基因等, 此外, 通过对这些基因的KEGG途径搜索发现, 碳水化合物代谢途径和钙离子信号途径在茶树响应低温胁迫方面起主要作用。Yue等[35]进一步研究发现, 随着茶树的冷驯化,茶树成熟叶中的可溶性糖含量逐渐增加, 其中蔗糖(Suc)、葡萄糖(Glc)、果糖(Fru)和半乳糖(Gal)的含量在抗寒性阶段达到最大, 随着脱驯化又逐渐降低;且蔗糖等的含量变化与茶树抗寒能力密切相关。A/N-Inv作为蔗糖水解的关键酶类, 其在茶树冷驯化诱导抗寒性方面也可能起重要作用, 但关于该类基因在茶树中的研究报道较少。本研究在课题组前期转录组测序的基础上, 拼接克隆一个CsINV10,通过对该基因生物信息学分析和表达模式的研究,旨在为深入研究A/N-Inv在响应茶树逆境胁迫方面的作用和探索冷驯化诱导茶树抗寒的分子机制奠定理论基础。
1 材料与方法
1.1材料与试剂
试验材料为2年生国家级茶树良种龙井43扦插苗, 种植于中国农业科学院茶叶研究所内实验基地。试验试剂主要有SYBR Premix Ex Taq (Perfect Real-time)试剂盒、Prime Script RT Reagent Kit、克隆载体pMD18-T、大肠杆菌DH5α感受态细胞和rTaq酶均购自TaKaRa公司(大连); PCR SuperMix、逆转录酶SuperScript III和PCR Cleanup Kit购自Invitrogen公司(Carlsbad, USA); DNA Marker购自Axygen公司(California, USA); RNA提取试剂CTAB、EDTA、LiCl、β-巯基乙醇、无水乙醇等均为国产分析纯。引物合成和基因测序由上海华津生物有限公司完成。
1.2总RNA提取及cDNA的合成
参照岳川等[36]所述方法提取不同处理样品的总RNA, 然后分别参照SuperScript III试剂盒和Prime Script RT Reagent试剂盒说明书反转录合成基因扩增用cDNA和表达分析用cDNA。
1.3CsINV10基因克隆
基于课题组前期对冷驯化茶树的转录组测序结果[34], 从中筛选出6条与其他物种中A/N-Inv基因同源的EST序列, 在Seqman软件中对这些序列进行拼接, 将拼接好的序列在NCBI中进行BlastX同源比对分析, 最后根据拼接好的序列设计相应引物进行RT-PCR验证(引物如表1所示)。PCR反应含PCR SuperMix 45 μL、cDNA 1.0 μL、上下游引物各1.0 μL, 加ddH2O至终体积50 μL; 反应程序为94℃ 5 min; 94℃ 30 s, 55℃ 30 s, 72℃ 2.5 min, 35个循环; 72℃10 min; 4℃保存。PCR反应后, 利用1%琼脂糖凝胶电泳, 将获得的条带利用PCR Cleanup Kit回收纯化, 取2 μL回收产物与pMD18-T载体后转化到大肠杆菌DH5α中进行蓝白斑筛选, 菌液PCR后挑选阳性单克隆测序。
1.4生物信息学分析
利用DNASTAR软件包中的Seqman软件对CsINV10全长序列进行拼接, Editseq进行ORF查询; 在NCBI数据库中分别用BlastN和BlastX对核酸序列和氨基酸序列进行同源性分析; 在ClustalX 2.0软件中对多序列比对后, 利用DNAMAN和MEGA 5.0输出同源比对和进化树构建结果[37]。用ProtParam预测蛋白分子量、等电点、氨基酸组分等[38]; SignalP 4.1 Server进行信号肽预测[39], TMHMM Server v. 2.0预测蛋白的跨膜结构, ProtScale Sever在线分析分析蛋白疏水性, GOR4在线分析CsINV10蛋白的卷曲螺旋结构, TargetP1.1预测该蛋白的亚细胞定位[40], SMART服务器分析CsINV10蛋白的结构功能, NetPhos 2.0 Server[41]和YinOYang 1.2 Server[42]预测蛋白磷酸化和糖基化位点, SWISS-MODEL预测该蛋白的三级结构, 通过Phymol软件编辑得到蛋白三维结构模型[43]。
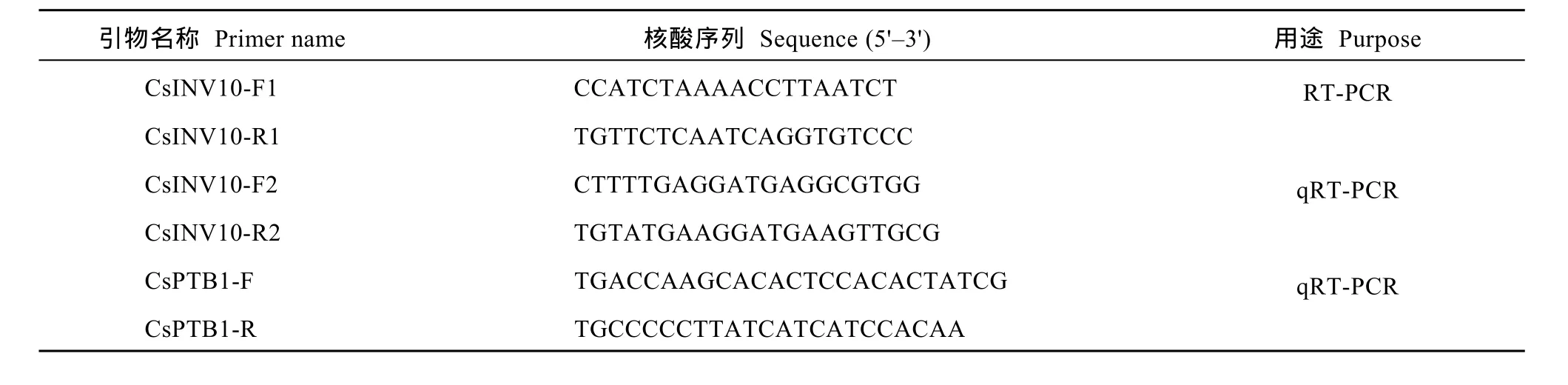
表1 RT-PCR和qRT-PCR引物对Table 1 RT-PCR and qRT-PCR primers
1.5CsINV10不同组织中的表达分析
以实验室保存的龙井43茶树不同组织(根、茎、叶、花)为材料进行qRT-PCR分析, 参照岳川等[36]所述方法取样, 以生长于露天茶园的大小和长势基本一致的2年生盆栽龙井43品种茶树为材料, 在开花季(10月)分别取茶树顶端成熟叶、茎干、根系和花在液氮中速冻后–80℃保存备用。以茶树PTB为内参基因[44], 荧光定量反应体系为SYBR Premix Ex Taq 10 μL、上下游引物(10 μmol L–1)(表1)各1 μL、ROX Dye II 0.6 μL、cDNA 2 μL, 加水至终体积20 μL。充分混匀, 短暂离心后, 于ABI PRISM 7500实时定量PCR 仪上进行PCR扩增, 反应程序为95℃15 s; 94℃ 5 s, 60℃ 34 s进行40个循环后增加熔解曲线。每个处理进行3次生物学重复和3次技术学重复, 最后采用2–ΔΔCT法[45]分析结果, 并用SPSS18.0软件分析差异显著性。
1.6CsINV10不同非生物胁迫下的表达分析
对不同胁迫处理(低温、盐分、干旱、ABA)茶树成熟叶片进行CsINV10的qRT-PCR分析。参照曹红利等[46]所述, 选取6盆大小和长势基本一致的盆栽茶树于LED冷光源植物气候箱中(24℃, 16 h光照/8 h黑暗, 湿度65%)培养两周后进行4℃低温处理,分别在处理后0 h、1 h、3 h、9 h、1 d、2 d、3 d和4 d取茶树顶端第3片成熟叶进行基因表达分析。在温室中分别随机选取3盆大小和长势基本一致的茶树, 用250 mmol L–1NaCl溶液以浇灌的方式进行盐分处理; 同样, 另取3盆茶树用100 mmol L–1的ABA溶液均匀喷洒在茶树新稍上, 在处理后0 h、1 h、3 h、9 h、1 d、3 d和5 d分别取茶树顶端第3片成熟叶进行基因表达分析。参照岳川等[36]所述方法, 将3盆大小和长势基本一致的茶树从盆中取出, 用自来水清洗根系并在纯净水中平衡15 min后, 将茶树放入10% (w/v) PEG-6000溶液中进行干旱胁迫处理, 处理时间和取样方法同ABA处理。以茶树PTB为内参基因, qRT-PCR程序及结果分析参照1.5方法。
2 结果与分析
2.1CsINV10的克隆
基于课题组前期对茶树冷驯化进行的转录组测序结果, 从中挑选出与A/N-Inv基因高度同源的6条EST序列, 利用Seqman对这些序列进行拼接获得一条全长为2101 bp的核酸序列, 利用NCBI数据库中的ORF Find查找该序列的ORF, 然后进行BlastX比对发现该序列包含完整的开放阅读框, 全长为1923 bp, 编码640个氨基酸。根据序列拼接和比对结果设计1对RT-PCR引物进行全长验证, 通过1%琼脂糖凝胶电泳获得2.5 kb左右的单一条带(图1),将其回收后和pMD18-T连接转化到大肠杆菌DH5α进行蓝白斑筛选, 挑阳性单克隆摇菌送测序, 测序结果和之前的拼接结果基本一致(图2), 提交GenBank, 登录号为KT359348。

图1 茶树中性/碱性转化酶基因CsINV10 RT-PCR电泳结果Fig. 1 Electrophoresis result of neutral/alkaline invertase gene CsINV10 in tea plant
2.2序列结构和同源性分析
在NCBI数据库中对CsINV10的氨基酸序列进行Blastx比对, 显示其与荔枝中已报道的中性/碱性转化酶LcNI (JQ773414)氨基酸序列相似性最高, 达80%, 与橡胶树HbNIN3 (AGU19630)、木薯MeNINV8 (AFH77954)相似性分别为78%和76%。用相邻连接法构建进化树显示CsINV10与木薯中性/碱性转化酶家族中的MeNINV8 (AFH77954)亲缘关系最近, 与定位于叶绿体上的拟南芥At-A/N-InvE (At5g22510)、水稻OsNIN3 (AK121301)聚为一类(图3)。因而, 推测CsINV10蛋白可能也定位于叶绿体, 属于A/N-Inv α亚家族成员。
结合聚合进化树分析结果, 从中挑选部分At-A/ N-Inv蛋白氨基酸序列进行同源性比对(图4)。所有比对序列中间大约450 bp左右的氨基酸序列具有较高的保守性。另外, 在这些保守氨基酸序列中发现有38个位点的氨基酸残基能用于将At-A/N-Inv分成不
同的亚家族, 其中30个氨基酸位点能将At-A/N-Inv分成α和β亚家族(红色框内所示), 4个位点能将定位于线粒体的At-A/N-Inv与叶绿体、细胞质中的At-A/N-Inv区分开(橙黄色框内所示), 3个位点能将定位于叶绿体上的At-A/N-Inv与定位于线粒体、细胞质中的At-A/N-Inv分开(黄色框内所示), 还有1个氨基酸位点能分别将定位于叶绿体、线粒体和细胞质的At-A/N-Inv区分开(绿色框内所示)。利用这些特异的氨基酸位点可将不同的At-A/N-Inv定位于不同的亚细胞中, 这种分类结果和利用系统进化树的分类结果完全一致。

图2 CsIINV10的编码序列Fig. 2 Encoding sequence of CsIINV10下画线部分代表CsINV10的开放阅读框, 粗体字分别代表起始子和终止子, 阴影部分代表保守结构域。The underlined part represents open reading frame of CsINV10, the shadow represents conserved domains and the bolds represent start codon and terminator respectively.
2.3蛋白序列基本性质分析
2.3.1理化性质分析利用ProtParam预测CsINV10蛋白分子量为71.8 kD, 理论等电点pI为5.69, 总共包括10034个原子, 分子式为C3223H4981N857O944S29。在组成CsINV10蛋白的20种氨基酸中,亮氨酸(Leu)所占比例最高, 达到9.7%, 而组氨酸(His)所占的比例最低, 为1.7%; CsINV10蛋白的不稳定指数为43.81, 脂肪指数为87.61, 总平均亲水性(GRAVY)为–0.166, 根据Guruprasad法判断该蛋白不稳定。
2.3.2信号肽分析SignalP 4.1 Server预测该蛋白信号肽显示S平均值(mean S-score)为0.237<0.5,因而预测为不包含信号肽, 属于非分泌蛋白和非N端糖基化蛋白。
2.3.3疏水性分析利用ProtScale Sever 在线分析软件中的Hyhob/Kyte & Doolittle 法对该基因的亲水性和疏水性预测显示(图5), 最大正值为2.467,最小负值为–3.267, 表明该蛋白属于亲水性蛋白。2.3.4跨膜结构预测利用在线软件TMHMM Server v. 2.0对CsINV10进行跨膜区分析显示, 该蛋白不存在跨膜螺旋区, 不属于跨膜蛋白(图6), 这和该蛋白的疏水性区域分析结果(图5)基本一致。
2.3.5亚细胞定位TargetP1.1在线软件预测该蛋白的亚细胞定位显示目的蛋白最可能定位于叶绿体上, 这与系统进化树预测结果(图3)一致。
2.3.6卷曲螺旋(Coil区)分析GOR4在线软件分析CsINV10蛋白的卷曲螺旋结构显示, CsINV10
蛋白由26.88% α-螺旋、21.56% β-折叠和51.56%无规则卷曲组成。
2.3.7蛋白磷酸化和糖基化位点预测利用NetPhos 2.0 Server对CsINV10蛋白的磷酸化位点预测显示, 该蛋白在翻译后可能存在28个磷酸位点,其中包含14个丝氨酸(Ser)位点、4个苏氨酸(Thr)位点和9个络氨酸(Tyr)位点(图7)。利用YinOYang 1.2 Server进行蛋白糖基化位点预测, 结果显示该蛋白氨基酸序列中包含11个潜在的O-GlcNAc位点, 包括Ser (2、3、4、24、83、183、295、466、519、634)残基和一个Thr (182)残基, 其中丝氨酸(519)可能是YinYang位点(图8)。
2.4结构域及motif搜索
用SMART对CsINV10蛋白的结构功能预测显示, CsINV10蛋白氨基酸序列156~599位之间高度保守(图2), 属于糖苷水解酶家族100 (G100)成员共有的典型结构域, 可能具有糖肽α-N-乙酰半乳糖胺酶活性。
2.5CsINV10三级结构预测
通过SWISS-MODEL在线软件对CsINV10三级结构预测后, 利用PhyMol软件编辑获得图9所示结果, CsINV10 (194-552)三级结构主要由14个α螺旋、17个无规则卷曲和第2和第3个α螺旋之间的2个小的β折叠构成。

图3 CsINV10与其他物种中的A/N-Inv氨基酸序列聚合进化树分析结果Fig. 3 Phylogenetic tree of amino acid of CsINV10 and A/N-Inv in other plants参与构建系统进化树的A/N-Inv分别为: 拟南芥A/N-InvA~I (At1g56560, At4g34860, At3g06500, At1g22650, At5g22510, At1g72000, At1g35580, At3g05820, At4g09510)、水稻OsNIN1~3, 7~8 (AK103334, AK120720, AK121301, AK065562, AK102741)、木薯MeNINV1~10 (JN616390, JQ339931, JQ339932, JQ782220, KF533729, KF533730, JN801148, JQ339933, KF533731, KF533732)、小麦Ta-A/N-INV AM295169)、甜菜BvINV (AJ422050)、胡萝卜DcINV (Y16262)、百脉根LjInv1 (AJ717412)、毒麦LtINV (AJ003114)、甘蔗SoNIN1 (JX109944)、橡胶树HbNIN1 (GU573728)和HbNIN2 (GU573727)、茶树CsINV10 (KT359348)。The Phylogenetic tree grouped by A/N-InvA–I (At1g56560, At4g34860, At3g06500, At1g22650, At5g22510, At1g72000, At1g35580, At3g05820, At4g09510), Arabidopsis thaliana; OsNIN1–3, 7–8 (AK103334, AK120720, AK121301, AK065562, AK102741), Oryza sativa; MeNINV1–10 (JN616390, JQ339931, JQ339932, JQ782220, KF533729, KF533730, JN801148, JQ339933, KF533731, KF533732), Manihotesculentacrantz; Ta-A/N-INV (AM295169), Triticum aestivum; BvINV (AJ422050), Beta vulgaris; DcINV (Y16262), Daucuscarota; LjInv1 (AJ717412), Lotus japonicus; LtINV (AJ003114), Lolium temulentum; SoNIN1 (JX109944), Saccharum officinarum; HbNIN1 and HbNIN2 (GU573728, GU573727), Hevea brasiliensis; CsINV10 (KT359348), Camellia sinensis.

图4 CsINV10与其他物种中的A/N-Inv氨基酸序列同源比对结果Fig. 4 Homologous analysis of amino acid sequences among different plants参与同源比对的A/N-Inv分别为茶树CsINV10 (KT359348)、拟南芥A/N-InvA~I (At1g56560, At4g34860, At3g06500, At1g22650, At5g22510, At1g72000, At1g35580, At3g05820, At4g09510)、水稻OsNIN1~3, 7~8 (AK103334, AK120720, AK121301, AK065562, AK102741)、木薯MeNINV1~4, 6~8, 10 (JN616390, JQ339931, JQ339932, JQ782220, KF533730, JN801148, JQ339933, KF533732)。红色框用于区分α和β亚族, 黄色框用于区分叶绿体定位的A/N-Inv, 橙色框用于区分线粒体定位的A/N-Inv, 绿色框用于区分细胞质、叶绿体、线粒体定位的A/N-Inv; 红色箭头代表可能的催化位点。The homologous sequences include A/N-InvA–I (At1g56560, At4g34860, At3g06500, At1g22650, At5g22510, At1g72000, At1g35580, At3g05820, At4g09510), Arabidopsis thaliana; OsNIN1–3, 7–8 (AK103334, AK120720, AK121301, AK065562, AK102741), Oryza sativa; MeNINV1–4, 6–8, 10 (JN616390, JQ339931, JQ339932, JQ782220, KF533730, JN801148, JQ339933, KF533732), Manihot esculenta Crantz; CsINV10 (KT359348), Camellia sinensis. The red boxes are used to separate α and β subfamily, orange boxes to separate A/N-Invs located in mitochondria, yellow boxes to separate A/N-Invs located in chloroplast, and green box to identify different A/N-Invs located in cytoplasm, chloroplast and mitochondria. The red arrow represents candidate catalytic residues.
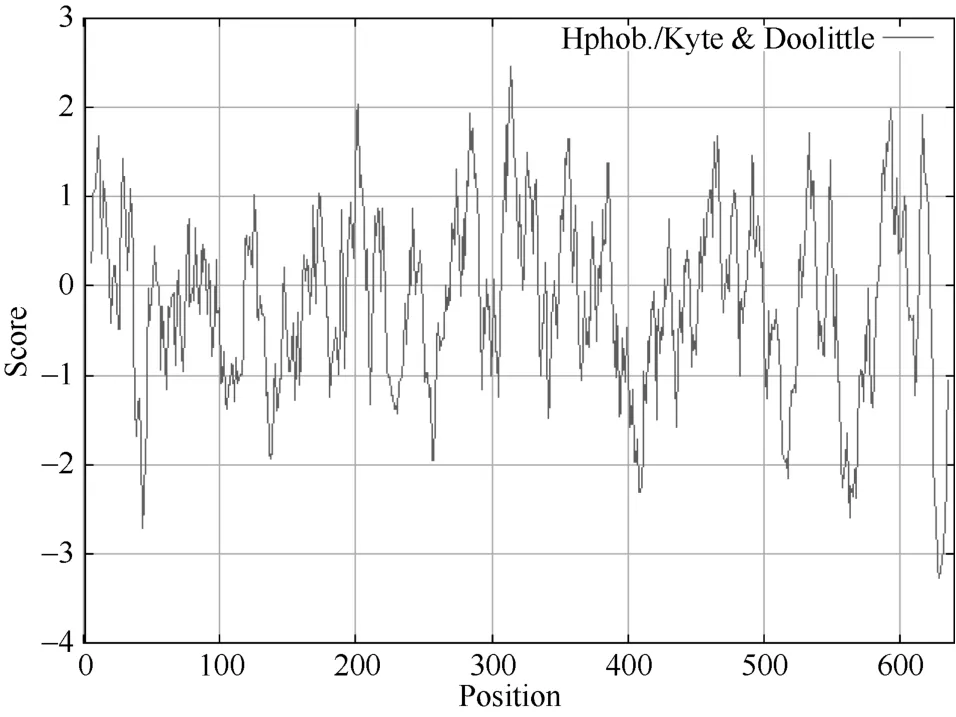
图5 CsINV10亲水性/疏水性分析Fig. 5 Hydrophobicity/hydrophilicity analysis of CsINV10

图6 CsINV10蛋白跨膜结构域预测Fig. 6 Prediction model for transmenbrane domain of CsARF1 protein
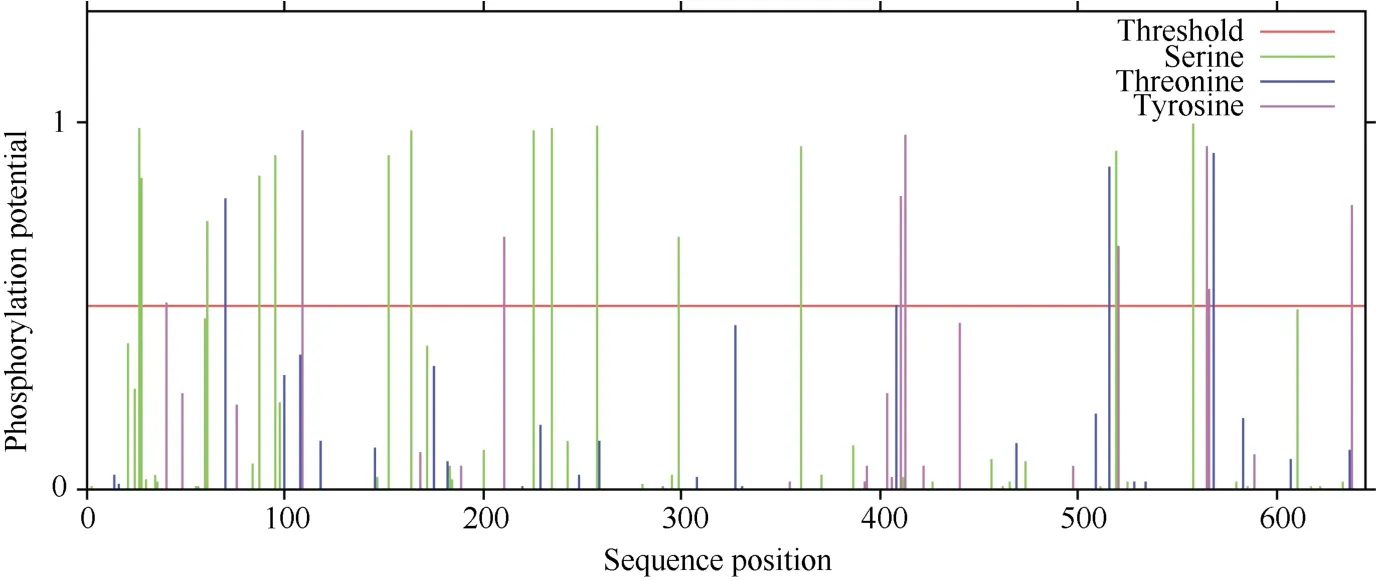
图7 CsINV10磷酸化位点预测Fig. 7 Phosphorylation sites prediction of CsINV10 protein

图8 CsINV10糖基化位点预测Fig. 8 Glycosylation sites prediction of CsINV10 protein
2.6CsINV10基因的表达模式分析
2.6.1组织表达差异性图10表明, CsINV10基因在成熟叶片和花中的表达要明显高于根系中的表达, 茎干中的表达量较成熟叶片和花都要低, 但也比根系中高, 说明该基因在茶树中具有组织表达特异性。
2.6.2不同非生物胁迫下的表达模式NaCl处理下, 茶树成熟叶片中CsINV10基因表达量随着处理时间的延长呈逐渐升高趋势(图11), 在处理5 d后,其表达量为未处理前的13倍, 差异达到显著水平。PEG条件下, 叶片中该基因的表达量在处理后3 h达到最大值, 是处理前的5.3倍, 随后又逐渐降低并在处理后1 d达到最小值, 而后随着处理时间的延长, 表达又逐渐升高, 在处理5 d后其表达量又基本恢复到处理3 h的水平。ABA处理下, 该基因表达量在处理后3 h达到最大值, 随后随着处理时间的延长呈现逐步降低的趋势。低温条件下, CsINV10基因在茶树成熟叶中的表达随着处理时间的延长呈现
先下调后上调的趋势, 在处理3 h后表达量达最小值, 随后逐渐升高, 在处理4 d后为处理前的5.7倍。表明CsINV10基因的表达受多种逆境胁迫诱导, 同时ABA能够瞬时诱导该基因的表达, 而低温、PEG和盐分处理下该基因表达量在处理1 d后较对照都要高, 说明该基因可能作为一个长效表达基因参与响应茶树的逆境胁迫。

图9 CsINV10蛋白三维结构预测Fig. 9 Three-dimension structure of CsINV10 protein

图10 同一品种不同组织器官中CsINV10的表达分析Fig. 10 Expression analysis of CsINV10 in different organs of a tea plant柱上小写字母不同表示差异达到显著水平(P < 0.05)。The columns with different small letters are significantly different (P < 0.05).
3 讨论

图11 CsINV10在不同非生物胁迫下的表达分析Fig. 11 Expression analysis of CsINV10 in various abiotic stresses柱上小写字母不同表示差异达到显著水平(P < 0.05)。The columns with different small letters are significantly different (P < 0.05).
在氨基酸序列中, CWIN和VIN在接近成熟蛋白N端处都有一个β-呋喃果聚糖苷酶基序(NDPD/ NG)[1], 而在接近C端则有一段保守的(WECV/PD),该序列被认为是酸性转化酶蛋白家族中保守结构域和催化活性所必须的序列[47], 而A/N-Inv缺乏N端信号肽, 既不是糖基化蛋白, 也不是β-呋喃果糖苷酶, 具体催化位点依然未知。酸性转化酶能够水解包含有β-呋喃果糖苷基的低聚糖糖如蔗糖、棉子糖和水苏糖等, 而A/N-Inv只能特异水解蔗糖。本研究从茶树成熟叶片中克隆的转化酶基因, 通过与其他物种中已报道的A/N-Inv进行氨基酸比对显示, 这类转化酶氨基酸序列中间的450多个氨基酸具有较高的保守性, 而N端和C端的同源性非常低, 在这段保守结构域中存在多个能将不同亚细胞定位的A/N-Inv区分开的氨基酸残基位点, 同时还包含Ji 等[19]报道的12个保守结构域和两个潜在的催化残基Asp88和Asp149 (图4红色箭头所示)。此外, CsINV10的氨基酸序列与茶树中已报道的酸性转化酶氨基酸序列基本没有同源性, 而与另外几个预测为A/N-Inv基因的序列具有较高同源性, CsINV10氨基酸序列不包含酸性转化酶的特征五肽(NDPNG)和(WECV/PD), N端不含跨膜结构域和信号肽, 且具有较高的亲水性。CsINV10蛋白氨基酸序列性质和胡萝卜[4]、小麦[10]、甘蔗[24]等的A/N-Inv蛋白性质
相似, 说明我们所克隆获得的CsINV10也属于A/N-Inv基因。根据进化树分析显示, CsINV10与水稻OsNIN3和拟南芥A/N-InvE聚为一大类, 而OsNIN3和A/N-InvE均已报道定位于叶绿体, 因而推测CsINV10也可能定位于叶绿体, 这与TargetP1.1预测结果一致。然而, 本研究所报道的CsINV10在酶学水平上, 具体属于中性还是碱性转化酶, 最佳pH值为多少; 它的底物特异性如何, 米氏常数(Km)值为多少, 催化位点在哪等众多问题还需要我们后期更深入的研究后才能确定。
目前, 很多研究认为A/N-Inv在调控植物根系生长和生殖发育方面具有重要作用。Qi等[28]对拟南芥Atcyt-inv1突变体研究发现, Atcyt-inv1能够通过控制细胞内己糖浓度来抑制侧根发育, 拟南芥中另外2个A/N-Inv基因AtCIN7和AtCIN9 (cINV1/cINV2)双突变的突变体中A/N-Inv活性较对照降低了40%,抑制了根系生长, 同时还伴随着细胞的畸形扩增[48]。相似地, 百脉根中的LjCIN1 (LjINV1)突变后降低了根系和茎干的生长、阻遏花粉形成和损害开花[23],水稻中OsCIN8 (OsCyt-INV1)的突变造成突变体植株根系变短, 推迟开花和部分不育[32]。植物中组织器官的生长不仅需要己糖作为能量和碳的来源, 还需要一个细胞伸长的驱动力, 即相对稳定的细胞渗透压和一个细胞壁伸展性的增加[1]。在上述突变体中根系缩短可能是由于转化酶活性的降低导致己糖含量匮乏使得细胞壁元件合成减少和细胞延长所需的驱动力降低, 最终降低细胞长度。碳水化合物在花药和花粉发育中具有重要作用, 对马铃薯[49]和烟草[50]花药组织中的胞外转化酶研究发现蔗糖的胞外水解和花药、花粉之间存在紧密的联系, A/N-Inv活性的变化会影响花粉的育性。本研究通过对茶树不同组织中CsINV10的表达进行qRT-PCR分析发现在茶树成熟叶和花中其表达量最高, 茎干中的表达量则低于二者, 这可能与CsINV10蛋白亚细胞定位于叶绿体有关。茶树作为常绿植物, 叶片中较高的A/N-Inv活性能够充分水解光合产物——蔗糖产生大量葡萄糖和果糖参与到茶树生长发育和对逆境胁迫的响应过程, 而叶片中的叶绿体作为植物参与光合作用的主要细胞器, 其内部可能存在较高的A/N-Inv活性以参与蔗糖水解过程。茎干作为蔗糖运输通道, 较低的A/N-Inv活性可能更利于蔗糖通过产生浓度梯度从源器官向库器官中运输。而花作为快速生长分化组织, 较高的A/N-Inv活性能够促进蔗糖的水解合成己糖, 为开花过程提供必要的碳水化合物来源。基于CsINV10基因在茶树成熟叶和花中较高的表达量, 推测该基因可能主要在参与响应茶树体内蔗糖从“源器官”到“库器官”的分配、生长发育、开花结果等方面具有重要作用, 但关于A/N-Inv基因是如何介导响应植物生长发育过程, 比如是通过糖信号途径还是通过不同信号途径(如赤霉素、细胞分裂素和磷脂酰肌醇等)之间互作等众多问题都还有待更深入的研究。另外, 本研究发现CsINV10基因在茶树根系中的表达量较其他组织都低, 推测该基因在响应茶树根系生长方面的作用可能并不明显, 原因可能也和CsINV10的亚细胞定位有关, 该蛋白的亚细胞定位与上述报道的AtCIN7、AtCIN9、LjCIN1、OsCIN8不同, 后四者都被报道定位于细胞质中, 属于β亚家族, 同时四者相互之间具有较高的同源性。因此, 我们推测在茶树中可能还存在能特异调控茶树根系生长发育的转化酶基因, 但这还需我们后续深入研究。
最近有研究表明植物体内的A/N-Inv基因被敲除后, 突变体植株中由于A/N-Inv活性的降低或缺失对植株造成了叶片、茎干、根系生长不良和花期推迟等不良影响, 说明A/N-Inv是植物生长和发育不可或缺的部分[29]。此外, 植物A/N-Inv在胁迫条件下还伴随着酶活性的升高[32,48], 其可能通过调控细胞质或细胞器中蔗糖浓度, 或是通过调控能被线粒体中相关己糖激酶响应或作为己糖激酶底物的葡萄糖浓度来调节线粒体活性氧的平衡。Xiang等[29]分别敲除拟南芥中定位于细胞质的At-A/N-InvG和线粒体中的At-A/N-InvA基因后发现, 缺失突变体的生长明显受到抑制, 两种突变体中的A/N-Inv活性都降低了30%以上; 另外, 他们对野生型拟南芥进行过氧化氢处理后发现, At-A/N-Inv基因和HXK1在野生型中的表达量都明显增加, 因而推测At-A/N-Inv可能作为抗氧化系统的成员参与调控胞内活性氧的平衡。Vargas 等[10]对小麦一个碱性转化酶在不同环境胁迫中活性变化情况的研究表明, 在低温和甘露醇处理小麦72 h中, 小麦体内A/N-Inv活性随着处理时间的延长呈逐渐升高的趋势, 在处理24 h后达到最高, 随后缓慢降到一定程度后便维持在一个相对稳定水平, 同时, 一个碱性转化酶基因Ta-A-Inv在低温和甘露醇处理72 h后表达量较对照要高, 推测小麦幼苗叶片中A/N-Inv水解蔗糖可能作为植物常规代谢机制的开端, 通过提供较高的能量和生物合成化合物来抵
御逆境胁迫。本研究发现, 在4℃低温条件下, 茶树成熟叶片中CsINV10基因的表达呈先降后升趋势, ABA和PEG处理下则呈现瞬时增加后急剧降低趋势,但与ABA不同的是, PEG处理下该基因的表达量在降到一定程度后又随着处理时间的延长逐渐升高。而在NaCl处理条件下, 该基因随着处理时间的延长逐渐上调表达, 在处理5 d后, 表达量较处理前提高了13倍, 呈极显著水平。总体而言, 无论是低温、盐分还是PEG胁迫处理茶树1 d后, CsINV10的表达量都较对照要高, 并随处理时间延长逐渐升高, 说明该基因能够响应茶树非生物逆境胁迫, 其在参与茶树抵御环境胁迫方面可能具有重要作用。然而, 王菲[51]报道的一个枳叶片中的中性转化酶基因PtrNINV在低温、干旱、盐胁迫下的表达模式却与CsINV10基因在相同逆境胁迫下的表达相反, 这些A/N-Inv基因在不同物种中表达模式的差异可能受物种间差异限制, 同时也可能与它们的亚细胞定位或最佳酶活pH不同有关。PtrNINV被报道定位于细胞质中的线粒体上, 属于中性转化酶基因[51], 而我们所克隆的CsINV10根据亚细胞定位预测显示定位于叶绿体上。由于时间和空间的差异使得不同亚细胞定位的A/N-Inv基因对逆境胁迫的响应可能有不同的时空表达模式。
本研究明确了该类酶编码基因在茶树响应逆境胁迫中的表达模式, 但关于它们在茶树抗逆中的作用机制仍有待进一步深入研究。例如: (i)茶树逆境胁迫下A/N-Inv活性变化与A/N-Inv基因表达之间的对应关系如何; (ii)胁迫条件下, A/N-Inv与激素之间是否具有对应关系, 又是通过什么信号途径参与调控茶树体内活性氧的平衡; (iii)不同细胞器中A/N-Inv和AI基因之间在茶树逆境下是如何协同表达; (iv)逆境下, A/N-Inv基因表达的高低是否由相关转录因子来调控。同时, A/N-Inv活性变化除被相应A/N-Inv基因调控外, 其他影响因素又是什么等问题,有待在以后的研究中逐一揭开。
4 结论
克隆获得一个全长ORF为1923 bp, 编码640个氨基酸的A/N-Inv基因CsINV10, 该基因编码的蛋白氨基酸序列无N-端信号肽, 无跨膜结构域, 属于亲水性蛋白并定位于叶绿体。茶树中CsINV10的表达具有组织表达差异性, 该基因的表达受多种逆境胁迫诱导, 表明它与茶树抗逆响应密切相关。
References
[1] Sturm A. Invertases. Primary structures, functions, and roles in plant development and sucrose partitioning. Plant Physiol, 1999, 121: 1–8
[2] Tang G Q, Luscher M, Sturm A. Antisense repression of vacuolar and cell wall invertase in transgenic carrot alters early plant development and sucrose partitioning. Plant Cell, 1999, 11: 177–189
[3] Roitsch T. Source-sink regulation by sugar and stress. Curr Opin Plant Biol, 1999, 2: 198–206
[4] Sturm A, Hess D, Lee H S, Lienhard S. Neutral invertase is a novel type of sucrose-cleaving enzyme. Physiol Plant, 1999, 107: 159–165
[5] Koch K. Sucrose metabolism: regulatory mechanisms and pivotal roles in sugar sensing and plant development. Curr Opin Plant Biol, 2004, 7: 235–246
[6] Ruan Y L. Goldacre Paper: Rapid cell expansion and cellulose synthesis regulated by plasmodesmata and sugar: insights from the single-celled cotton fibre. Funct Plant Biol, 2007, 34: 1
[7] Pugh D A, Offler C E, Talbot M J, Ruan Y L. Evidence for the role of transfer cells in the evolutionary increase in seed and fiber biomass yield in cotton. Mol Plant, 2010, 3: 1075–1086
[8] Ruan Y L, Jin Y, Yang Y J, Li G J, Boyer J S. Sugar input, metabolism, and signaling mediated by invertase: roles in development, yield potential, and response to drought and heat. Mol Plant, 2010, 3: 942–955
[9] Tymowska-Lalanne Z, Kreis M. The plant invertases: physiology, biochemistry and molecular biology. Adv Bot Res, 1998, 28: 71–117
[10] Vargas W, Pontis H, Salerno G. Differential expression of alkaline and neutral invertases in response to environmental stresses: characterization of an alkaline isoform as a stress-response enzyme in wheat leaves. Planta, 2007, 226: 1535–1545
[11] Weber H, Borisjuk L, Wobus U. Molecular physiology of legume seed development. Annu Rev Plant Biol, 2005, 56: 253–279
[12] Hayes M A, Davies C, Dry I B. Isolation, functional characterization, and expression analysis of grapevine (Vitis vinifera L.) hexose transporters: differential roles in sink and source tissues. J Exp Bot, 2007, 58: 1985–1997
[13] Kim J Y, Mahe A, Guy S, Brangeon J, Roche O, Chourey P S, Prioul J L. Characterization of two members of the maize gene family, Incw3 and Incw4, encoding cell-wall invertases. Gene, 2000, 245: 89–102
[14] Kang B H, Xiong Y, Williams D S, Pozueta-Romero D, Chourey P S. Miniature1-encoded cell wall invertase is essential for assembly and function of wall-in-growth in the maize endosperm transfer cell. Plant Physiol, 2009, 151: 1366–1376
[15] Jin Y, Ni D A, Ruan Y L. Posttranslational elevation of cell wall invertase activity by silencing its inhibitor in tomato delays leaf senescence and increases seed weight and fruit hexose level. Plant Cell, 2009, 21: 2072–2089
[16] Chourey P S, Li Q B, Cevallos-Cevallos J. Pleiotropy and its dissection through a metabolic gene Miniature1 (Mn1) that encodes a cell wall invertase in developing seeds of maize. Plant Sci, 2012, 184: 45–53
[17] Schaarschmidt S, Kopka J, Ludwig-Muller J, Hause B. Regulation of arbuscular mycorrhization by apoplastic invertases: enhanced invertase activity in the leaf apoplast affects the symbiotic interaction. Plant J, 2007, 51: 390–405
[18] Sun L, Yang D L, Kong Y, Chen Y, Li X Z, Zeng L J, Li Q, Wang E T, He Z H. Sugar homeostasis mediated by cell wall invertase GRAIN INCOMPLETE FILLING 1 (GIF1) plays a role in pre-existing and induced defence in rice. Mol Plant Pathol, 2014, 15: 161–173
[19] Ji X, Van den Ende W, Van Laere A, Cheng S, Bennett J. Structure, evolution, and expression of the two invertase gene families of rice. J Mol Evol, 2005, 60: 615–634
[20] Murayama S, Handa H. Genes for alkaline/neutral invertase in rice: alkaline/neutral invertases are located in plant mitochondria and also in plastids. Planta, 2007, 225: 1193–1203
[21] Nonis A, Ruperti B, Pierasco A, Canaguier A, Adam-Blondon A F, Di Gaspero G, Vizzotto G. Neutral invertases in grapevine and comparative analysis with Arabidopsis, poplar and rice. Planta, 2008, 229: 129–142
[22] Bocock P N, Morse A M, Dervinis C, Davis J M. Evolution and diversity of invertase genes in Populus trichocarpa. Planta, 2008, 227: 565–576
[23] Welham T, Pike J, Horst I, Flemetakis E, Katinakis P, Kaneko T, Sato S, Tabata S, Perry J, Parniske M, Wang T L. A cytosolic invertase is required for normal growth and cell development in the model legume, Lotus japonicus. J Exp Bot, 2009, 60: 3353–3365
[24] 牛俊奇, 王爱勤, 黄静丽, 杨丽涛, 李杨瑞. 甘蔗中性/碱性转化酶基因SoNIN1的克隆和表达分析. 作物学报, 2014, 40: 253–263
Niu J Q, Wang A Q, Huang J L, Yang L T, Li Y R. Cloning and expression analysis of sugarcane alkaline/neutral invertase gene SoNIN1. Acta Agron Sin, 2014, 40: 253–263 (in Chinese with English abstract)
[25] Ruan Y L. Sucrose metabolism: gateway to diverse carbon use and sugar signaling. Annu Rev Plant Biol, 2014, 65: 33–67
[26] Yao Y, Geng M T, Wu X H, Liu J, Li R M, Hu X W, Guo J C. Genome-wide identification, expression, and activity analysis of alkaline/neutral invertase gene family from cassava (Manihot esculenta Crantz). Plant Mol Biol Rep, 2014, 33: 304–315
[27] Vargas W A, Pontis H G, Salerno G L. New insights on sucrose metabolism: evidence for an active A/N-Inv in chloroplasts uncovers a novel component of the intracellular carbon trafficking. Planta, 2008, 227: 795–807
[28] Qi X, Wu Z, Li J, Mo X, Wu S, Chu J, Wu P. AtCYT-INV1, a neutral invertase, is involved in osmotic stress-induced inhibition on lateral root growth in Arabidopsis. Plant Mol Biol, 2007, 64: 575–587
[29] Xiang L, Le Roy K, Bolouri-Moghaddam M R, Vanhaecke M, Lammens W, Rolland F, Van den Ende W. Exploring the neutral invertase-oxidative stress defence connection in Arabidopsis thaliana. J Exp Bot, 2011, 62: 3849–3862
[30] Martin M L, Lechner L, Zabaleta E J, Salerno G L. A mitochondrial alkaline/neutral invertase isoform (A/N-InvC) functions in developmental energy-demanding processes in Arabidopsis. Planta, 2013, 237: 813–822
[31] Lou Y, Gou J Y, Xue H W. PIP5K9, an Arabidopsis phosphatidylinositol monophosphate kinase, interacts with a cytosolic invertase to negatively regulate sugar-mediated root growth. Plant Cell, 2007, 19: 163–181
[32] Jia L, Zhang B, Mao C, Li J, Wu Y, Wu P, Wu Z. OsCYT-INV1 for alkaline/neutral invertase is involved in root cell development and reproductivity in rice (Oryza sativa L.). Planta, 2008, 228: 51–59
[33] Thomashow M F. PLANT COLD ACCLIMATION: Freezing tolerance genes and regulatory mechanisms. Annu Rev Plant Physiol Plant Mol Biol, 1999, 50: 571–599
[34] Wang X C, Zhao Q Y, Ma C L, Zhang Z H, Cao H L, Kong Y M, Yue C, Hao X Y, Chen L, Ma J Q, Jin J Q, Li X, Yang Y J. Global transcriptome profiles of Camellia sinensis during cold acclimation. BMC Genom, 2013, 14: 415
[35] Yue C, Cao H L, Wang L, Zhou Y H, Huang Y T, Hao X Y, Wang Y C, Wang B, Yang Y J, Wang X C. Effects of cold acclimation on sugar metabolism and sugar-related gene expression in tea plant during the winter season. Plant Mol Biol, 2015, 88: 591–608
[36] 岳川, 曹红利, 周艳华, 王璐, 郝心愿, 王新超, 杨亚军. 茶树谷胱甘肽还原酶基因CsGRs的克隆与表达分析. 中国农业科学, 2014, 47: 3277–3289
Yue C, Cao H L, Zhou Y H, Wang L, Hao X Y, Wang X C, Yang Y J. Cloning and expression analysis of glutathione reductase genes (CsGRs) in tea plant (Camellia sinensis). Sci Agric Sin, 2014, 47: 3277–3289 (in Chinese with English abstract)
[37] Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol, 2011, 28: 2731–2739
[38] Gasteiger E, Hoogland C, Gattiker A, Duvaud S, Wilkins M R, Appel R D, Bairoch A. The proteomics protocols handbook. Biochemistry, 2005, 71: 696
[39] Petersen T N, Brunak S, von Heijne G, Nielsen H. SignalP 4.0: discriminating signal peptides from transmembrane regions. Nat Meth, 2011, 8: 785–786
[40] Emanuelsson O, Nielsen H, Brunak S, von Heijne G. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence. J Mol Biol, 2000, 300: 1005–1016
[41] Zhou X Z, Lu P J, Wulf G, Lu K P. Phosphorylation-dependent prolyl isomerization: a novel signaling regulatory mechanism. Cell Mol Life Sci, 1999, 56: 788–806
[42] Jochmann R, Holz P, Sticht H, Sturzl M. Validation of the reliability of computational O-GlcNAc prediction. Biochim Biophys Acta, 2014, 1844: 416–421
[43] Arnold K, Bordoli L, Kopp J, Schwede T. The SWISS-MODEL workspace: a web-based environment for protein structure homology modelling. Bioinformatics, 2006, 22: 195–201
[44] Hao X Y, Horvath D P, Chao W S, Yang Y J, Wang X C, Xiao B. Identification and evaluation of reliable reference genes for quantitative real-time PCR analysis in tea plant (Camellia sinensis (L.) O. Kuntze). Int J Mol Sci, 15: 22155–22172
[45] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-delta delta C(T)) method. Methods, 2001, 25: 402–408
[46] 曹红利, 岳川, 郝心愿, 王新超, 杨亚军. 茶树胆碱单加氧酶
CsCMO的克隆及甜菜碱合成关键基因的表达分析. 中国农业科学, 2013, 46: 3087–3096
Cao H L,Yue C, Hao X Y, Wang X C, Yang Y J. Cloning of choline monooxygenase (CMO) gene and expression analysis of the key glycine betaine biosynthesis-related genesin tea plant (Camellia sinensis). Sci Agric Sin, 2013, 46: 3087–3096 (in Chinese with English abstract)
[47] Sturm A, Chrispeels M J. cDNA cloning of carrot extracellular beta-fructosidase and its expression in response to wounding and bacterial infection. Plant Cell, 1990, 2: 1107–1119
[48] Barratt D H, Derbyshire P, Findlay K, Pike M, Wellner N, Lunn J, Feil R, Simpson C, Maule A J, Smith A M. Normal growth of Arabidopsis requires cytosolic invertase but not sucrose synthase. Proc Natl Acad Sci USA, 2009, 106: 13124–13129
[49] Maddison A L, Hedley P E, Meyer R C, Aziz N, Davidson D, Machray G C. Expression of tandem invertase genes associated with sexual and vegetative growth cycles in potato. Plant Mol Biol, 1999, 41: 741–751
[50] Goetz M, Godt D E, Guivarc’h A, Kahmann U, Chriqui D, Roitsch T. Induction of male sterility in plants by metabolic engineering of the carbohydrate supply. Proc Natl Acad Sci USA, 2001, 98: 6522–6527
[51] 王菲. 枳中性转化酶基因PtrNINV克隆及其功能分析. 华中农业大学硕士学位论文, 湖北武汉, 2013. pp 35–37
Wang F. Cloning and Functional Analysis of PtrNINV, a NeutralInvertase, in Poncirus trifoliata. MS Thesis of Huazhong Agricultural University. Wuhan, China, 2013. pp 35–37 (in Chinese with English abstract)
URL: http://www.cnki.net/kcms/detail/11.1809.S.20151218.0915.008.html
Cloning and Expression Analysis of a Neutral/alkaline Invertase Gene (CsINV10) in Tea Plant (Camellia sinensis L. O. Kuntze)
QIAN Wen-Jun1,2, YUE Chuan2, CAO Hong-Li2, HAO Xin-Yuan2, WANG Lu2, WANG Yu-Chun1,2, HUANG Yu-Ting2, WANG Bo2, WANG Xin-Chao2, XIAO Bin1,*, and YANG Ya-Jun1,2,*1College of Horticulture, Northwest A&F University, Yangling 712100, China;2Tea Research Institute of Chinese Academy, Agricultural Sciences / National Center for Tea Improvement / Key Laboratory of Tea Biology and Resources Utilization, Ministry of Agriculture, Hangzhou 310008, China
Abstract:Based on the comprehensive RNA-Seq analysis of tea plant during cold acclimation stage, we picked out six ESTs sequences with a high similarity to neutral/alkaline invertase gene to get a splicing. As a result, a full-length of 2101 bp nucleotide sequence was obtained from tea plant after validated by using RT-PCR technique. Bioinformatics analysis showed that the sequence containing 1923 bp ORF (Open Reading Fram) and encoding 640 amino acid residues with a putative molecular mass of 71.8 kD and theoretical isoelectric point of 5.69, was named as CsINV10 (GenBank accession number: KT359348). BlastX and phylogenetic analysis indicated that the protein encoded by CsINV10 shared the highest identity (80%) with LcNI in Litchi, and had a closest genetic relationship with MeNINV8 in Manihot esculenta Crantz. Also, CsINV10 as a neutral/alkaline invertase gene could be classified into G100 family. The protein had no N-terminal signal peptide and transmembrane domain, and was predi-
cated to be a hydrophilic protein localized in chloroplast. The expression analysis of CsINV10 under different abiotic stress conditions showed that cold, PEG and salt stresses could gradually promote the expression of CsINV10 when treated for one day, however, it had a transient increase when treated by ABA for three hours. Consequently, we speculated that CsINV10 might be involved in the tea plant response to abiotic stresses. Moreover, we found that CsINV10 had a tissue-specific expression pattern in leaf and flower. These results provide a theoretical basis for the functional analysis of invertase genes of tea plant in response to various stresses.
Keywords:Tea plant (Camellia sinensis); Neutral/alkaline invertase gene; Expression analysis
收稿日期Received(): 2015-08-12; Accepted(接受日期): 2015-11-20; Published online(网络出版日期): 2015-12-18.
通讯作者*(Corresponding authors): 杨亚军, E-mail: yjyang@mail.tricaas.com, Tel: 0571-86653162; 肖斌, E-mail: xiaobin2093@sohu.com
DOI:10.3724/SP.J.1006.2016.00376
