花生长链酰基辅酶A合成酶6基因(LACS6)的克隆、鉴定与组织表达
2016-02-13徐日荣陈湘瑜唐兆秀
徐日荣,陈湘瑜,唐兆秀*
(1.福建省农业科学院作物研究所,福建 福州 350013; 2.国家农业部闽台农作物种质资源利用重点开放实验室,福建 福州 350013)
花生长链酰基辅酶A合成酶6基因(LACS6)的克隆、鉴定与组织表达
徐日荣1,2,陈湘瑜1,2,唐兆秀1,2*
(1.福建省农业科学院作物研究所,福建 福州 350013; 2.国家农业部闽台农作物种质资源利用重点开放实验室,福建 福州 350013)
长链酰基辅酶A合成酶(long chain acyl-CoA synthetase:LACS)是油脂代谢的重要催化酶。为揭示花生脂肪酸代谢机理,采用RT-PCR技术,首次从花生ArachishypogaeaL.克隆到LACS6(GenBank登录号:KU301860),分析该基因的结构组成,预测编码氨基酸与其他植物的同源性,采用Real-Time PCR技术对LACS6的组织表达进行研究。结果显示,花生LACS6基因全长2 116 bp,包含2 088 bp的ORF,编码695个氨基酸,有23个外显子和22个内含子。氨基酸序列比对显示花生LACS6有真核生物酰基辅酶A合成酶保守结构域,并含有保守的激活位点和绑定位点。同源性分析发现花生LACS6与鹰嘴豆、绿豆、大豆、梅等13种物种的氨基酸一致性在79%~87%,进化树分析显示,花生LACS6与鹰嘴豆等豆科植物亲缘较近。实时荧光PCR分析表明,花生LACS6在花生根、茎、叶、子房柄、仁和花等组织均有表达,且差异明显。子房柄和花的表达量极高,与根、茎、叶和仁等组织有极显著差异,花生LACS6组织的表达量大小排序为花>子房柄>叶>仁>茎>根。本研究结果为揭示花生脂肪酸代谢和品质改良提供理论依据。
花生;长链酰基辅酶A合成酶;组织表达;油脂
花生是世界范围内广泛栽培的油料与经济作物,是重要的油脂和蛋白质来源[1]。中国是世界花生的主产国之一,根据中国统计局数据显示,2014年中国花生种植面积为460.39万hm2,总产1 648.17万t。花生脂肪酸是食用植物油的重要来源,有学者研究认为,花生原料含油量每提高1个百分点相当于产量提高2个百分点,加工企业的效益可提高7个百分点以上[2]。因此,揭示花生脂肪酸代谢机理,提高花生品种含油量,对增加单位面积油产量、提高企业效益具有重要意义。
油料作物种子中的油脂即三酰基甘油(triacylglycerol: TAG)[3],在TAG的合成和降解代谢中,长链酰基辅酶 A合成酶(long chain acyl-CoA synthetase: LACS)都起着重要的作用。TAG的合成底物脂肪酰辅酶A是由LACS催化脂肪酸和辅酶A而成的,TAG分解成的脂肪酸必须由LACS催化后形成脂肪酰基辅酶A后才能进入β-氧化途径,脂肪酸最终降解成乙酰辅酶 A的形式供给幼苗的生长[4]。
在植物中,LACS基因的研究率先在模式植物拟南芥中开展,2002年已经证实有9个LACS基因存在,大多数LACS酶与脂肪酸组成的结构和存储脂肪具有高的活性[5]。AtLACS1和AtLACS2可能参与角质和蜡的合成[6-7],AtLACS1、AtLACS2和AtLACS3在酵母中可以促进脂肪酸的摄取[8],AtLACS1和AtLACS4共同参与花粉外壁的形成[9],AtLACS6和AtLACS7编码氧化物酶体LACS蛋白并与脂肪酸β-oxidation[10-11],AtLACS9是一个叶绿体蛋白,和AtLACS1参与种子油的生物合成[12-13]。从油菜克隆了BnACS6基因,在脂肪合成的组织中高度表达,可能参与了脂质合成[14]。从大豆的过氧酶体中分离到GmACSL2基因,显示参与脂肪酸和脂质的降解[15]。从棉花分离到GhACS1,是早期花粉发育的重要基因[16]。此外, LACS酶存在于蓖麻[17]、向日葵[18]、山羊草[19]和小麦[20]。
本研究对花生LACS6基因进行cDNA克隆及序列分析,并利用实时荧光定量PCR技术检测花生根、茎、叶、子房柄、仁和花不同组织的表达情况,为深入研究LACS6基因的生物学功能提供理论依据。
1 材料与方法
1.1 材料
1.1.1 植物材料与样品采集 供试材料为福花8号,以幼苗期健康的叶片作为LACS6基因克隆的材料,在花生下针40 d,采取植株根、茎、叶、子房柄、仁和花等组织,作为LACS6基因组织表达的研究材料,材料采集后放入液氮罐转入-80℃冰箱保存。
1.1.2 主要试剂与仪器TaqDNA聚合酶、pMD18-T vector、RNA反转录试剂盒(Prime ScriptII 1st Strand cDNA Synthesis Kit)均购自TaKaRa公司(大连),荧光定量PCR试剂TransStart Top Green qPCR SuperMix购于全式金公司,其他生化试剂购于鼎国生物公司。ABI7500实时定量PCR仪(ABI公司,德国)。
1.2 方法
1.2.1 总RNA提取及cDNA合成 按熊发前等[21]的CTAB方法提取花生各个组织的总RNA。由Prime Script II 1st Strand cDNA Synthesis Kit反转录试剂盒合成cDNA第一链。
1.2.2 花生LACS6基因的克隆 以GenBank大豆LACS6 mRNA (登录号: XM_004496693)为探针,搜索并拼接数据库中花生的EST序列,用primer软件设计全长引物AhLACS6-F(AGC TTT CGC GTT AAT GGA TT)和AhLACS6-R(ATA GGG CCT TCA GCA TCA CA)。采用CTAB法提取花生幼苗叶片的总RNA并反转录得到cDNA,以此为模板进行PCR扩增。PCR反应体系25 μL,其中cDNA模板1 μL、10×TaqBuffer 2.5 μL、dNTPs 2 μL、10 mmol·L-1正反引物各0.5 μL、TaqDNA聚合酶0.5 μL、ddH2O 18 μL。PCR反应条件:94℃ 5 min;94℃ 30 s,60℃ 30 s,72℃ 1 min,共35个循环;最后72℃延伸10 min。PCR产物凝胶电泳检测后,选择与预测大小一致的目的条带切胶回收并连接载体pMD19-T,经热击转化、扩大培养、质粒提取、质粒PCR鉴定,选择阳性质粒,送铂尚生物技术(上海)有限公司进行测序。
1.2.3 花生LACS6的鉴定 根据测序获得的核苷酸序列预测编码氨基酸,利用NCBI blastp和CCD在线程序分析该氨基酸序列与其他物种LACS6的相似性及功能结构域,下载大豆、绿豆和鹰嘴豆等13个物种的氨基酸序列,用Clustalx2、Genedoc和Mega6.0软件对AhLACS6与其他物种进行蛋白同源性和进化树分析。
1.2.4 花生LACS6的实时荧光定量PCR分析 根据获得花生LACS6基因序列重新设计表达引物RT- AhLACS6-F(TTT GGT GGG CTA AAG GGC TGA AGA TTA CTG) 和RT- AhLACS6-R(ACG ACT AAT TGT GGT AGT TGG AGG CAT TGA),以‘福花8号’下针40 d的叶片、茎、花、子房柄、仁和根cDNA为模板,进行实时荧光定量PCR扩增,以内参基因ACTIN(RT-ACTIN-F: GTC ATC GTC ATC CTC TTC TC, RT- ACTIN-R: CAT TCC TGT TCC ATT GTC AC)为对照,分析花生LACS6基因在植株各部位的表达情况。反应体系总体积为25 L,包括12.5 μL TransStart Top Green qPCR SuperMix,1 μL cDNA,正反向引物各0.5 μL(浓度为10 μmol·L-1),10.5 μL蒸馏水。反应程序为:95℃预变性10 min;95℃变性15 s,60℃退火1 min,共40个循环。每个反应3次重复。
2 结果与分析
2.1 花生LACS6基因的克隆
2.1.1 序列拼接 以大豆登录号: XM_004496693的LACS6 mRNA为电子探针,利用NCBI blastn在线工具,搜索出2个花生的EST序列,登录号分别为:JK157315.1和ES759871.1,他们与大豆的LACS6一致性在83%和90%。用ES759871.1继续查询花生EST文库,获得100%一致性的ES723932序列,2条EST拼接成1 159 bp的3端,长度为520 bp的JK157315.1序列查询没有获得其他花生EST序列,利用两端获得的并拼接的片段设计引物,克隆花生LACS6基因。
2.1.2 基因的克隆 利用CTAB法提取健康的花生叶片总RNA,经反转录获得cDNA,以此为基因克隆模板,用设计的引物AhLACS6-F和AhLACS6-R PCR扩增,1%琼脂糖凝胶电泳结果显示,在DNA Marker 2 000 bp上面有1条明亮的条带(图1),与预测的大小一致。经切胶回收后,连接T载体,热击转化大肠杆菌DH5α感受态细胞,挑取单菌落扩大培养,提取质粒,质粒电泳后,选取阳性克隆测序。铂尚生物技术(上海)有限公司的测序结果表明,目的片段的长度为2 116 bp,经过NCBI网址BLAST比对,该序列与大豆Glycinemax、鹰嘴豆Cicerarietinum和绿豆Vignaradiatavar. radiata等植物的LACS6 基因有很高的同源性,确认为花生的LACS6基因,命名为AhLACS6(Arachis hypogaea long chain acyl-CoA synthetase)。
2.1.3 序列分析 根据DNASTAR软件分析,AhLACS6序列包括13 bp的5′UTR区、15 bp的3′UTR区以及2 088 bp的完整CDS区,预测编码695个氨基酸(图2),该氨基酸分子量为76.5 kD,理论等电点为7.13。花生基因组比对发现,该基因与Arachisipaensis第9和第5染色体分别有68.2%和66.24%的相似性,与Arachisduranensis第5和第9染色体分别有96.36%和62.50%的相似性,该结果显示AhLACS6是单拷贝基因,可能存在同源基因。AhLACS6具体分布在Arachisduranensis第5染色体19030831~19036754区域,总共有5 923 bp,含有23个外显子和22个内含子(图3)。基因该序列已提交到GenBank,登录号为:KU301860。
2.2 花生AhLACS6基因的鉴定
2.2.1 花生AhLACS6的功能结构域分析 花生AhLACS6基因编码695个氨基酸,利用NCBI保守结构域数据库(Conserved Domain Database,CDD)分析预测,花生LACS6的含有1个真核生物酰基辅酶A合成酶保守结构域(Eukaryotic long-chain fatty acid CoA synthetase :LC-FACS),序列上包含保守的激活位点和绑定位点,该结构域属于AFD_class_I超级家族。在花生AhLACS6 261~272的氨基酸序列具有 [LIVMFY]-X-X-[STG]-[STAG]-G-[ST]-[STEI]-[SG]-X-[PASLIVM]-[KR]12个酰基激活酶(acyl-activating enzyme:AAE)高度保守序列,具体为ICYTSGTTGTPK氨基酸序列(图4下划线部分)。
2.2.2AhLACS6基因同源性分析 通过NCBI的blastp在线搜索,利用ClustalX2软件将花生与其他物种的LACS6氨基酸序列进行同源性比对,鹰嘴豆、绿豆、大豆、梅、葡萄、蓖麻、白梨苹果、枣、野草莓、杨、黄瓜和欧洲油菜等13种物种与花生AhLACS6氨基酸序列一致性在79%~87%(表1),其中与鹰嘴豆、绿豆和大豆等豆科植物一致性在86%以上。用GeneDoc软件形成多重比较,LACS6氨基酸序列整体同源性较高,在对比方阵第0~35、91~98和695~703氨基酸残基有较大差异(图4)。用Mega6.0绘制进化树显示,花生和鹰嘴豆、绿豆和大豆等豆科植物处在一个分支上,它们的遗传距离较近(图5)。
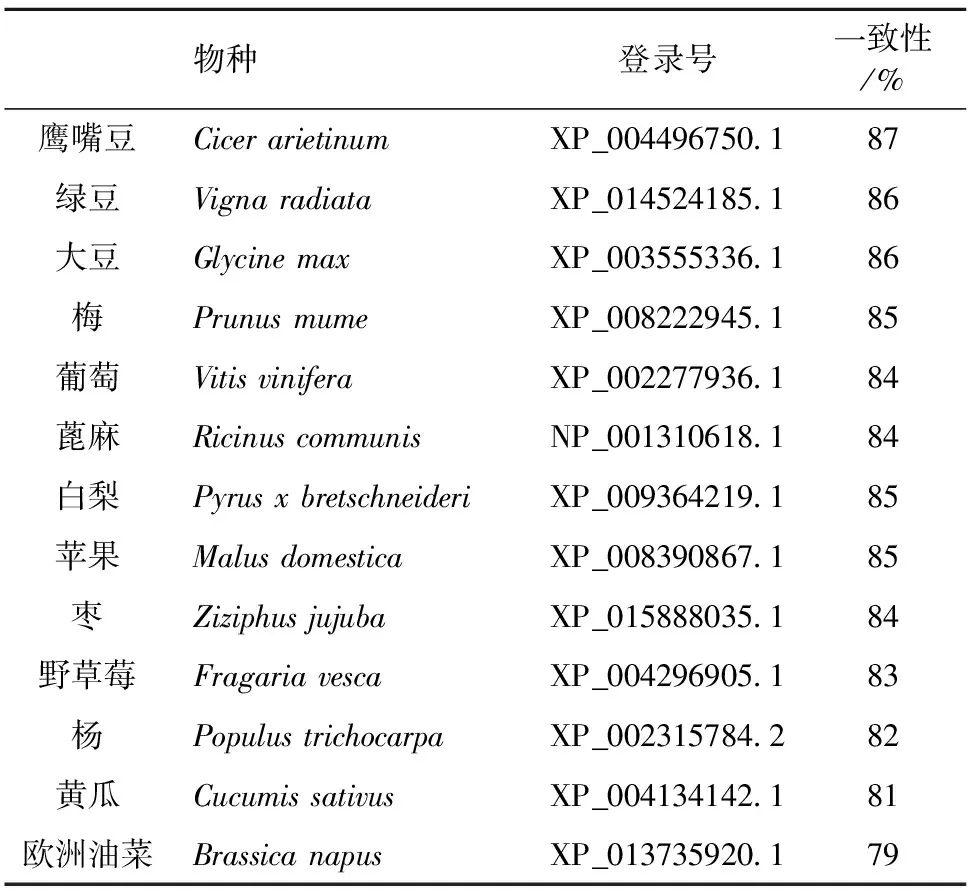
表1 AhLACS6与其他植物LACS6的一致性Table 1 Homology of AhLACS6 gene and other plant LACS6 genes
2.3 AhLACS6基因花生组织的表达
利用定量PCR技术分析AhLACS6基因花生不同组织的表达,以各部位内参基因ACTIN为对照,设定茎的相对表达为1.00,结果表明在花生根、茎、叶、子房柄、仁和花组织均有检测到AhLACS6的表达,但不同部位表达差异明显(图6)。在根有轻微表达,相对表达量分别为0.21,茎部表达有所增加,仁和叶片表达明显,相对表达量分别为2.60和4.74,前4个组织相对表达量无显著差异,子房柄和花的表达量极高,相对表达量分别为30.49和53.21,它们与根、茎、叶和仁等组织的表达量有极显著差异。AhLACS6在花生组织表达量大小排序为花>子房柄>叶>仁>茎>根。
3 讨 论
长链酰基辅酶 A合成酶(LACS)在TAG的合成和降解代谢中起着重要的作用。本研究首次从花生克隆获得AhLACS6基因,该基因cDNA全长2 116 bp,包含2 088 bp的完整开放阅读框,编码695个氨基酸,含有1个真核生物酰基辅酶A合成酶(LC-FACS)保守结构域,并有[LIVMFY]-X-X-[STG]-[STAG]-G-[ST]-[STEI]-[SG]-X-[PASLIVM]-[KR]12个酰基激活酶(acyl-activating enzyme:AAE)保守序列[22-24]。AhLACS6氨基酸与GenBank登录的鹰嘴豆(GenBank登录号:XP_004495889.1)、绿豆(GenBank登录号:XP_014514389.1)、大豆(GenBank登录号:XP_003518711.1)、梅(GenBank登录号:XP_008222945.1)、葡萄(GenBank登录号:XP_002277936.1)、蓖麻(GenBank登录号:NP_001310618.1) 、白梨(GenBank登录号:XP_009364219.1) 、苹果(GenBank登录号:XP_008390867.1) 、枣(GenBank登录号:XP_015888035.1) 、野草莓(GenBank登录号:XP_004296905.1) 、杨(GenBank登录号:XP_002315784.2) 、黄瓜(GenBank登录号:XP_004134142.1)和欧洲油菜(GenBank登录号:XP_013735920.1)等预测的693~703个氨基酸极其相近,氨基酸序列同源性的多重分析也显示与这些物种具有很高的保守性,进化树分析显示了与豆科植物LACS6有较好的亲缘关系,这些结果很好地表明本研究成功克隆到花生的AhLACS6基因。
AhLACS6的组织表达量大小排序为花>子房柄>叶>仁>茎>根,与拟南芥的组织表达类似[5],LACS6在拟南芥中表达顺序为花>叶>茎>根>发育种子,但在发育的种子中,拟南芥LACS6表达量很低,这可能与种子的含油量有关。
[1]SARVAMANGALA C, GOWDA M V C, VARSHNEY R K. Identification of quantitative trait loci for protein content, oil content and oil quality for groundnut(ArachishypogaeaL.) [J]. Field Crops Research, 2011, 122(1):49-59.
[2]王移收. 工业用油料植物脂肪酸及其改良[J].中国油料作物学报, 2006, 28(4): 498-502.
[3]SHEN B, SINKEVICIUS K W, SELINGER D A, et al. The homeobox gene GLABRA2 affects seed oil content inArabidopsis[J]. Plant Molecular Biology, 2006, 60(3):377-387.
[4]HILLS M J, MURPHY D J, BEEVERS H. Inhibition of neutral lipase from castor bean lipid bodies by coenzyme A (coA) and oleoyl-coA[J]. Plant Physiology, 1989, 89(3):1006-1010.
[5]SHOCKEY J M, FULDA M S, BROWSE J A.Arabidopsiscontains nine long-chain acyl-coenzyme a synthetase genes that participate in fatty acid and glycerolipid metabolism[J]. Plant Physiology, 2002, 129(4): 1710-1722.
[6]L S, SONG T, KOSMA DK, et al.ArabidopsisCER8 encodes long-chain acyl-coA synthetase 1(LACS1) that has overlapping functions withLACS2 in plant wax and cutin synthesis [J]. The Plant Journal, 2009, 59: 553-564.
[7]SCHNURR J A, SHOCKEY J M, BROWSE J. The acyl-coA synthetase encoded byLACS2 is essential for normal cuticle development inArabidopsis[J]. Plant Cell, 2004, 16(3): 629-642.
[8]PULSIFER I P, KLUGE S, ROWLAND O.Arabidopsislong-chain acyl-CoA synthetase 1 (LACS1),LACS2, andLACS3 facilitate fatty acid uptake in yeast[J]. Plant Physiology & Biochemistry, 2012, 51(2):31-39.
[9]JESSEN D, OLBRICH A, KNUFER J, et al. Combined activity ofLACS1 andLACS4 is required for proper pollen coat formation inArabidopsis[J]. The Plant Journal, 2011, 68: 715-726.
[10]FULDA M, SHOCKEY J, WERBER M, et al. Two long-chain acyl-CoA synthetases from Arabidopsis thaliana involved in peroxisomal fatty acid β-oxidation [J]. The Plant Journal, 2002, 32(1):93-103.
[11]MARTIN F, JUDY S, AMINE A, et al. Peroxisomal acyl-CoA synthetase activity is essential for seedling development inArabidopsisthaliana[J]. Plant Cell, 2004, 16(2):394-405.
[12]SCHNURR J A, SHOCKEY J M, DE BOER G J, et al. Fatty acid export from the chloroplast. Molecular characterization of a major plastidial acyl-coenzyme A synthetase fromArabidopsis[J]. Plant Physiology, 2002, 129(4):1700-1709.
[13]ZHAO L, KATAVIC V, LI F, HAUGHN GW, et al. Insertional mutant analysis reveals that long-chain acyl-CoA synthetase 1 (LACS1), but notLACS8, functionally overlaps withLACS9 inArabidopsisseed oil biosynthesis [J]. The Plant Journal, 2010, 64: 1048-1058.
[14]PONGDONTRI P, HILLS M. Characterization of a novel plant acyl-coA synthetase that is expressed in lipogenic tissues ofBrassicanapusL [J]. Plant molecular biology, 2001, 47(6): 717-726.
[15]LILI Y, XIAOLI T, BINGJUN J, et al. A peroxisomal long-chain acyl-CoA synthetase from Glycine max involved in lipid degradation[J]. Plos One, 2014, 9(7):e100144-e100144.
[16]WANG X L, LI X B. TheGhACS1 gene encodes an acyl-CoA synthetase which is essential for normal microsporogenesis in early anther development of cotton [J]. Plant Journal, 2009, 57(3):473-486.
[17]JIA J, ZHAO S, KONG X, et al.Aegilopstauschiidraft genome sequence reveals a gene repertoire for wheat adaptation[J]. Nature, 2013, 496(7443):91-95.
[18]AZNAR-MORENO J A, CALER N M V, MART NEZ-FORCE E, et al. Sunflower(Helianthusannuus) long-chain acyl-coenzyme a synthetases expressed at high levels in developing seeds[J]. Physiologia Plantarum, 2014, 150(3):363-373.
[19]LING H Q, ZHAO S, LIU D, et al. Draft genome of the wheat A-genome progenitorTriticumurartu[J]. Nature, 2013, 496(7443):87-90.
[20]CHAN A P, CRABTREE J, ZHAO Q, et al. Draft genome sequence of the oilseed speciesRicinuscommunis[J]. Nature biotechnology, 2010, 28(9):951-956.
[21]熊发前, 刘俊仙, 王丛丛, 等 mCTAB-dLiCl法高效提取花生各组织部位RNA及其验证[J]. 南方农业学报, 2013, 44(11):1781-1784.
[22]CONTI E, FRANKS N P, BRICK P. Crystal structure of luciferase throws light on a superfamily of adenylate-forming enzymes[J]. Structure, 1996, 4(3):287-298.
[23]CONTI E, STACHELHAUS T, MARAHIEL M A, et al. Structural basis for the activation of phenylalanine in the non-ribosomal biosynthesis of gramicidin S[J]. Embo Journal, 1997, 16(14): 4174-4183.
[24]SHOCKEY J M, FULDA M S, JOHN B.Arabidopsiscontains a large superfamily of acyl-activating enzymes. Phylogenetic and biochemical analysis reveals a new class of acyl-coenzyme A synthetases[J]. Plant Physiology, 2003, 132(2):1065-1076.
(责任编辑:柯文辉)
Cloning, Verification and Tissue Expression of Long-chain Acyl-CoA Synthetase 6 (LACS6) Gene in Peanut (ArachishypogaeaL.)
XU Ri-rong1,2, CHEN Xiang-yu1,2, TANG Zhao-xiu1,2*
(1.InstituteofCropSciences,FujianAcademyofAgriculturalSciences,Fuzhou,Fujian350013,China; 2.KeyLaboratoryofCropGermplasmUtilizationbetweenFujianandTaiwan,MinistryofAgriculture,Fuzhou,Fujian350013,China)
Long-chain acyl coenzyme A synthetase (LACS) is one of the important catalytic lipid metabolic enzymes in peanut plants. To understand the mechanism of the fatty acid metabolism in peanut (ArachishypogaeaL.), the cDNA ofLACS6 gene was cloned by RT-PCR (GenBank accession number: KU301860). Its structure was analyzed, homology with other plants compared, and mRNA expression in different tissues obtained using real-time PCR. The results showed that the cDNA was 2,116 bp including an open reading frame of 2,088 bp, encoded 695 amino acids, and had 23 exons and 22 introns. Its amino acid sequence alignment showed a conserved domain of eukaryotic long-chain fatty acid CoA synthetase, as well as conserved activation and binding sites. The homologies ofLACS6 gene with the genes fromCicerarietinum,Vignaradiate,Glycinemax,Prunusmumeand others were between 79% and 87%. The evolutionary tree indicated that the gene was close to those from leguminous plants, such as chickpeas. The real-time PCR analysis showed that it was differentially expressed in the ovary stalks, roots, stems, leaves, flowers and kernels on a peanut plant. The ovary stalks and flowers had the highest expression among all,and with significant differences,The order of expression ranked as flowers>ovary stalks>leaves>kernels>stems>roots. The information obtained would benefit further studies on the fatty acid metabolism and quality improvement for peanut.
peanut (ArachishypogaeaL.); long-chain acyl CoA synthetase; tissue expression; lipid
2016-05-12初稿;2016-09-19修改稿
徐日荣(1979-),男,硕士,副研究员,研究方向:花生遗传育种(E-mail:rirongxu@163.com) *通讯作者:唐兆秀(1958-),男,研究员,研究方向:花生遗传育种(E-mail:tzxfz@163.com)
福建省科技计划项目——省属公益类科研院所基本科研专项(2014R1026-4)
S 565.2
:A
:1008-0384(2016)11-1164-07
徐日荣,陈湘瑜,唐兆秀.花生长链酰基辅酶A合成酶6基因(LACS6)的克隆、鉴定与组织表达[J].福建农业学报,2016,31(11):1164-1170.
XU R-R,CHEN X-Y,TANG Z-X.Cloning, Verification and Tissue Expression of Long-chain Acyl-CoA Synthetase 6 (LACS6) Gene in Peanut (ArachishypogaeaL.)[J].FujianJournalofAgriculturalSciences,2016,31(11):1164-1170.
