基于系谱和SSR标记的高山杜鹃杂交种亲缘关系分析
2016-02-06王继华解玮佳蔡艳飞彭绿春李树发李世峰
张 露,王继华,解玮佳,蔡艳飞,宋 杰,彭绿春,李树发,李世峰
(云南省农业科学院花卉研究所,国家观赏园艺工程技术研究中心, 云南省花卉育种重点实验室,昆明 650205)
基于系谱和SSR标记的高山杜鹃杂交种亲缘关系分析
张 露,王继华,解玮佳,蔡艳飞,宋 杰,彭绿春,李树发,李世峰*
(云南省农业科学院花卉研究所,国家观赏园艺工程技术研究中心, 云南省花卉育种重点实验室,昆明 650205)
该研究对高山杜鹃(RhododendronL.)的总DNA提取方法进行了改良,然后综合利用高山杜鹃EST数据库和该实验室的马缨杜鹃高通量测序数据,引用已发表文献的SSR引物,从154对SSR引物中筛选出了26对多态性高、重复性好、条带清晰的SSR引物。再从中随机选择10对SSR引物进行荧光标记,对69份不同高山杜鹃的种质进行遗传多样性分析。结果表明,平均有效等位基因数6.959 2个;位点多态性信息含量(PIC)、观测杂合度(HO)、期望杂合度(HE)和Nei’s基因多样性(H)分别为0.795 2、0.543 5、0.826 5和0.820 2;杂交种间的非加权配对算术平均(UPGMA)聚类结果与其谱系分析结果基本一致,可实现对一些未知来源的育成品种资源进行祖先亲本类型的推测。
高山杜鹃;DNA提取;SSR引物筛选;系谱;亲缘关系
杜鹃花(RhododendronL.)为世界著名的园林观赏花卉,是中国传统十大名花和云南八大名花之一。高山杜鹃是一些常绿阔叶杜鹃原种及其杂交后代的总称,因其株型优美、花大色艳,四季常绿,逐渐成为中国年宵花和高档园林绿化的新秀,在西方素有“没有杜鹃不成园林”的说法[1]。中国西南部及毗邻的东喜马拉雅地区为常绿杜鹃亚属的分布中心和分化起源中心[2],有杜鹃属植物320余种,占世界杜鹃属植物的1/3[3]。欧美国家对中国杜鹃的引进及培育利用已有300多年历史[4],已培育出上万园艺品种并广泛应用到城市园林中,而作为杜鹃资源大国的中国却相形见绌,不仅选育的品种很少,城市园林建设中也鲜见杜鹃属植物的踪影[5]。由于高山杜鹃育种工作的滞后,导致中国高山杜鹃种质利用率低下,产业发展受制于人的问题日益凸显[6],挖掘和引入本土种质资源进行育种开发对于产业发展与提升具有重要意义。现如今,高山杜鹃的杂交育种研究工作将面临以下两大问题:1)高山杜鹃杂交育种历史悠长,诸多品种资源的系谱关系混乱不清,造成对其进一步的利用存在很大程度上的盲目性;2)中国高山杜鹃的野生种质资源虽丰富多样化,但极度缺乏对优异种质资源的搜集和研究关注度,导致在进一步扩宽种质遗传基础的实际工作中缺乏科学的理论指导,对优异新种质的评价及利用程度非常低。在解决此两大问题的基础上,中国有关高山杜鹃的杂交育种研究将迎来一个全新的局势。本研究针对已搜集到的国外引进高山杜鹃种质进行遗传多样性分析,对未来高山杜鹃的杂交育种研究具有重要的理论和现实指导意义。
1 材料与方法
1.1 实验材料及样品采集
本研究采用的实验材料均采自云南省农业科学院花卉研究所晋宁市大春河实验基地杜鹃花种质资源圃,62份高山杜鹃杂交种资源均为2013年先后引种于比利时和德国,基本信息详见表1;7份野生种资源分别是大白杜鹃(R.decorum)、大喇叭杜鹃(R.excellens)、马缨杜鹃(R.delavayi)、露珠杜鹃(R.irroratum)、云南杜鹃(R.yunnanense)、爆杖花杜鹃(R.spinuliferum)和富源杜鹃(R.fuyuanense),样品编号依次为A1、A2、A3、A8、A10、A14、A16。均于2014年6月采集样品新抽枝条上的幼嫩叶片,置于变色硅胶中用自封袋密闭干燥,室温保存备用。
1.2 方 法
1.2.1 高山杜鹃杂交种的谱系分析 根据62个高山杜鹃杂交种的系谱资料(主要参考网站http://hirsutum.info/,另有网站http://ip-173-201-58-163.ip.secureserver.net/RhododendronHybrids.htm作资料补充)分别列出其祖先亲本,将祖先亲本未知的单独列为“未知”类型,然后参考崔章林[19]的方法计算每个杂交种的祖先亲本遗传贡献值。凡由祖先亲本经自然变异选择法育成的品种其祖先亲本的遗传贡献值为1;凡由杂交育成的品种其双亲的遗传贡献均为0. 5。每一亲本再按均等分割方法上推其双亲,直至终极的祖先亲本,这样每一育成品种的各祖先亲本的遗传贡献值总和应等于1。同时根据《中国花卉品种分类学》[20]中对高山杜鹃杂交种的分类方法,对62份杂交种进行归类。
1.2.2 高山杜鹃总DNA提取 经过反复实验,对Zeng 等[21]及王书珍等[22]的方法做了稍许修改。同时对2×CTAB法、4×CTAB法和改良的4×CTAB法的总DNA提取效果进行比较分析。4×CTAB法主要是在2×CTAB法的基础上提高CTAB浓度;改良后的4×CTAB法则是在4×CTAB法的基础上增加了冰浴去多糖的步骤,其具体操作步骤如下:1)称取约100 mg硅胶干燥叶片于液氮中研磨成粉末,立即转移至装有1.5 mL Buffer Ⅰ (含pH 8.0的200 mmol/L Tris-HCl、pH 8.0的50 mmol/L EDTA、250 mmol/L NaCl、1%β-巯基乙醇和1%PVP) 的2.0 mL离心管中,待溶解后涡旋混匀并冰浴10 min,期间不时摇匀;2)4 ℃、7 000 r/min离心10 min,倒掉上清液(上清液如若仍粘稠,根据情况可重复加Buffer Ⅰ 进行冰浴处理),加入800 μL 经65 ℃预热的4×CTAB裂解液(含pH 8.0的100 mmol/L Tris-HCl、pH 8.0的25 mmol/L EDTA、1.5 mol/L NaCl 和 4.0 % CTAB),再置于65 ℃水浴40~60 min,期间不时混匀;3)加入等体积Tris饱和酚∶氯仿∶异戊醇(25∶24∶1),涡旋混匀,静置5 min后于12 000 r/min,离心8 min后用剪过枪尖的枪头转移上清(避免吸到下层杂质)至新离心管;4)往上清液中加入10 mg/mL RNase A 5 μL,室温静止处理20 min。然后再往里面加入等体积的氯仿∶异戊醇(24∶1),颠倒混匀,静置5 min后于12 000 r/min,离心8 min。小心转移上清至新离心管;5) 加入4 ℃预冷的异丙醇(上清液体积的2/3)上下颠倒混匀后于-20 ℃沉淀1 h以上,然后4 ℃、12 000 r/min离心10 min后弃上清;6)用75%酒精洗涤DNA沉淀2次。控干酒精后,置于通风橱内晾干,最后加ddH2O室温溶解5 min。所得DNA溶液经琼脂糖凝胶或微量核酸检测仪(NanoDrop 2000,美国Thermo Scitific corporation)检测后置于-20 ℃下长期保存备用。
通过琼脂糖凝胶电泳和核酸微量检测仪2种检测方法进行DNA质量检测。要求DNA琼脂糖凝胶电泳呈现明显主带,无弥散,且对应点样孔中无杂质滞留;同时OD260/280值在1.8~2.0之间, OD260/230值在1.8以上,且浓度大于100 ng/μL,即可直接用于下游的SSR扩增。
在2016年12月7日至8日举行的全国高校思想政治工作会议中,习近平总书记强调,“要坚持不懈地培育和弘扬社会主义核心价值观,引导广大师生做社会主义核心价值观的坚定信仰者、积极传播者、模范践行者”,“要运用新媒体新技术使工作活起来,推动思想政治工作传统优势同信息技术高度融合”[1]。 新媒体具有传播的迅速性、方式的多样性、手段的兼容性等传播特点,其方式符合大学生的生活习惯,易被大学生接受,它为社会主义核心价值观全面深入、迅速有效地传播带来了机遇。
1.2.3 引物来源及设计 引物主要来源于: 1)引用文献中已开发的杜鹃属植物SSR引物。其中来自杜鹃(R.simsii)的是14对[9-10],来自马缨杜鹃的是15对[12],共29对SSR引物;2)利用NCBI上已公布的杜鹃属植物杜鹃[23]和山石南杜鹃(R.catawbiense)[24]的EST文库。采用SSR Hunter软件[25]对文库序列进行2~6个重复碱基微卫星序列的搜索,设置单元重复次数≥6,且上下游序列长度≥150 bp作为筛选条件,得到105条微卫星EST序列并进行引物设计;3)利用本课题组已获得的马缨杜鹃基因组文库,从中随机选择20条含微卫星的DNA序列进行引物设计。因此,可用于本研究的SSR引物共有154对,利用软件Primer Premier 5.0[26]进行引物设计,引物序列交由上海捷瑞生物工程有限公司合成。
1.2.4 SSR扩增及检测 从69份材料中随机选择12份样品进行引物筛选,包含A3、A10、A16等3个野生种和D1、D6、D11、D20、W5、W8、W10、W19、R1等9个杂交种。PCR扩增试剂为2×EasyTaq PCR SuperMix for PAGE(全式金生物技术有限公司,含buffer、dNTP、Mg2+及rTaq酶),引物终浓度为0.2 μmol/L。PCR反应扩增程序为:95 ℃预变性3 min;95 ℃变性30 s,退火温度下退火30 s,72 ℃延伸30 s,32个循环;72 ℃延伸8 min。并以PAGE胶条带亮度和条带特异性作为评判依据,对每对引物的DNA模板浓度和退火温度进行分别优化。
引物多态性筛选时,SSR扩增产物在8%PAGE胶上进行电泳检测,对照Marker为20 bp DNA Ladder(上海捷瑞生物工程有限公司生产)。
随机选择的10对多态性SSR引物序列交由上海捷瑞生物工程有限公司进行荧光标记的合成,并分别在69份样品DNA中进行多态性扩增, SSR扩增产物则由硕擎生物科技有限公司进行毛细管电泳检测(机型为3730XL)分析,机器自动化判读条带。
1.2.5 数据统计与分析 1)多态性SSR引物筛选。根据扩增条带的迁移率,以20 bp DNA ladder作为Marker参照,统计每个位点在12个样品DNA中扩增片段大小。片段大小相同则认为无多态性,大小不同则视为等位基因,组成共显性标记数据矩阵。然后采用软件DataFormater 2.7[27]进行格式统一,再利用POPGENE version 1. 32软件[28]计算各个位点的等位基因数(Na)、有效等位基因数(Ne)、观察杂合度(HO)、期望杂合度(HE)、香农多样性指数(I)及Nei’s 基因多样度(H)等指标;各位点的多态性信息含量(PIC)则由软件PowerMarker version 3. 25[29]进行计算;2) 遗传多样性分析。按照软件DataFormater 2.7的格式要求,在Excel表格中统计整理10对荧光标记引物在69份样品中的扩增条带数据,然后同上分别采用POPGENE version 1. 32软件和PowerMarker version 3. 25软件计算以上各多样性信息指标,最后采用软件NTSYS 2.10e[30]进行非加权配对算术平均法(UPGMA)的聚类分析。
2 结果与分析
2.1 62份高山杜鹃杂交种的谱系分析
采用Excel表记录每个杂交种的各祖先亲本遗传贡献值(所有祖先亲本的遗传贡献值总和为1),然后根据各祖先亲本在62份杂交种中的遗传贡献值大小,取遗传贡献值排名在前十二的12个祖先亲本作为本研究的重要祖先亲本(“未知” 类型除外)。表1中依次列出了12个重要祖先亲本在62个高山杜鹃杂交种中的遗传贡献值,12个重要祖先亲本分别是:土耳其杜鹃(R.ponticum,A)、黄杯杜鹃(R.wardii,B)、高加索杜鹃(R.caucasicum,C)、山石南杜鹃(R.catawbiense,D)、两色杜鹃(R.dichroanthumspp.dichroanthum,E)、东石楠杜鹃(R.degronianumspp.yakushimanum,F)、朱红大杜鹃(R.griersonianum,G)、不丹杜鹃(R.griffithianum,H)、云锦杜鹃(R.fortuneispp.fortunei,I)、树形杜鹃(R.arboreum,J)、似血杜鹃(R.haematodesspp.haematodes,K)和绵毛房杜鹃(R.facetum,L),遗传贡献值总和为30.813 75,遗传贡献值从高到低依次为:D(7.593 75)>A(3.75)、F(3.75)>G(3.062 5)>H(2.907 5)>I (2.625)>E(2.125)>B(1.875)>C(1.375)>J(0.875)>L (0.625) >K(0.25)。
根据《中国花卉品种分类学》中对高山杜鹃杂交种的分类方法,62份高山杜鹃杂交种主要归类于美国山石南杜鹃花杂种群(Catawba Hybrids Group)、高加索杜鹃花杂种群(Caucasicum Hybrids Group)、云锦杜鹃花杂种群(Fortunei Hybrids Group)和腺柱杜鹃花杂种群(Griffithianum Hybrids Group)等4个杂种群。另外,本研究还将以R.degronianumspp.yakushimanum为亲本杂交而得的杂种群暂定名为‘东石楠杜鹃花杂种群’(Degronianum Hybrids group),其余则定为‘其它杂交种群’或‘亲本不详的杂交种群’,具体归类情况见表2。
2.2 总DNA提取
图1为3种提取方法效果对比,可以看出:由2×CTAB法提取所得DNA对应泳道点样孔中有明显杂质残留;提高CTAB浓度至4×CTAB可在一定程度上提高DNA得率,但仍有杂质残留;改良4×CTAB法所得DNA主带明亮、无弥散,点样孔干净,表明DNA完整且纯度高;OD260/280值均在1.8~2.0之间,OD260/230值均大于1.9,浓度大于300 ng/μL,浓度和纯度上均能满足下一步SSR实验要求。
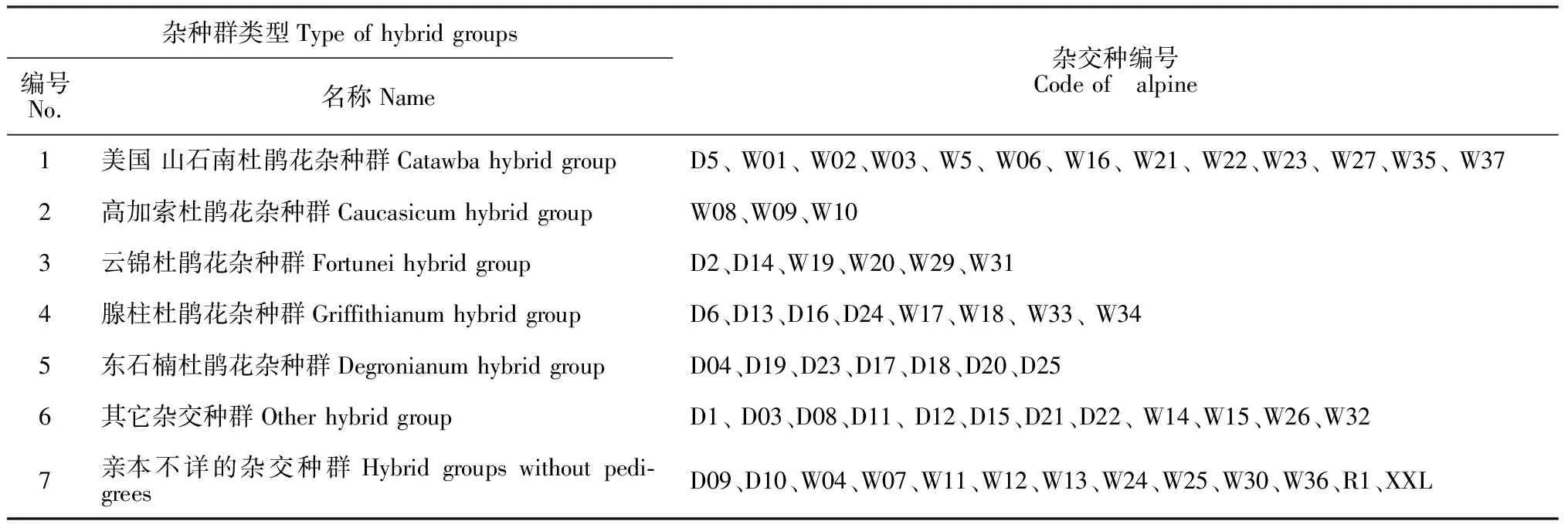
表2 62份高山杜鹃杂交种的分类情况
注:杂交种编号同表1
Note: Codes of 62 alpine same as Table 1

2.3 多态性SSR引物的筛选
采用优化的SSR体系,在12个样品中对154对SSR引物进行筛选,根据SSR扩增条带的有无、特异性、多态性和可重复性原则,最终从154对SSR引物中筛选出26对条带清晰、特异性好、重复性好、多态性较高的SSR引物。其中从山石南杜鹃(R.catawbiense)的99个EST序列中筛选得14对SSR引物;从杜鹃(R.simsii)的6个EST序列中筛选出了1对SSR引物;从马缨杜鹃的20个基因组DNA序列中筛选出了6对SSR引物;从29对文献引用的SSR引物中筛选得5对多态性SSR引物[9-10]。26对引物的基本信息详见表3;各位点的多态性分析结果详见表4。
随机选取一份DNA样品作为SSR扩增模板,对筛选的26对SSR引物进行PCR扩增,图2,A展示了26份扩增产物的电泳结果,表现为条带清晰、特异性好,基本无杂带。从26对SSR引物中随机选择2对引物(Rho-13和Rho-26)对课题组高山杜鹃资源圃内的21份高山杜鹃(含以上所用12份材料)的DNA样品进行多态性扩增,从图2,B可以看出它们在21份不同样品中均呈现出较高的多态性。

M1. 15K DNA marker;M2. 2K Plus DNA marker; Ⅰ.2×CTAB法;Ⅱ.4×CTAB法;Ⅲ.改良4×CTAB法图1 3种不同CTAB法提取高山杜鹃总DNA琼脂糖凝胶电泳M1 represents 15K DNA marker; M2 represents 2K Plus DNA marker; Ⅰ. 2×CTAB method;Ⅱ. 4×CTAB method; Ⅲ. Improved 4×CTAB method; The description texts on the top white line represent the corresponding extracting methodFig.1 Agarose gel image showing Rhododendron total DNA samples extracted by three different CTAB methods

M.20 bp DNA ladder; A.26对SSR引物扩增结果; B.1~21.引物Rho-13 扩增条带;22~42.引物Rho-26扩增条带图2 PCR扩增产物的PAGE胶图M. 20 bp DNA ladder; A. Amplified PCR bands using 26 SSR primer pairs; B. 1~21. PCR bands of primer Rho-13; 22~42. PCR bands of primer Rho-26Fig.2 PAGE image of PCR products
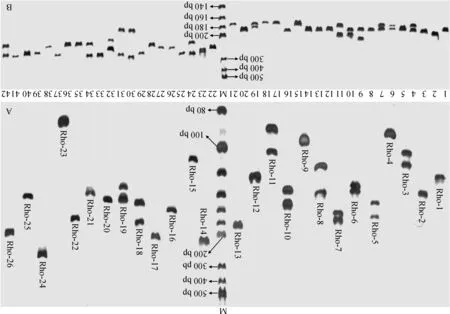
表4 各位点信息
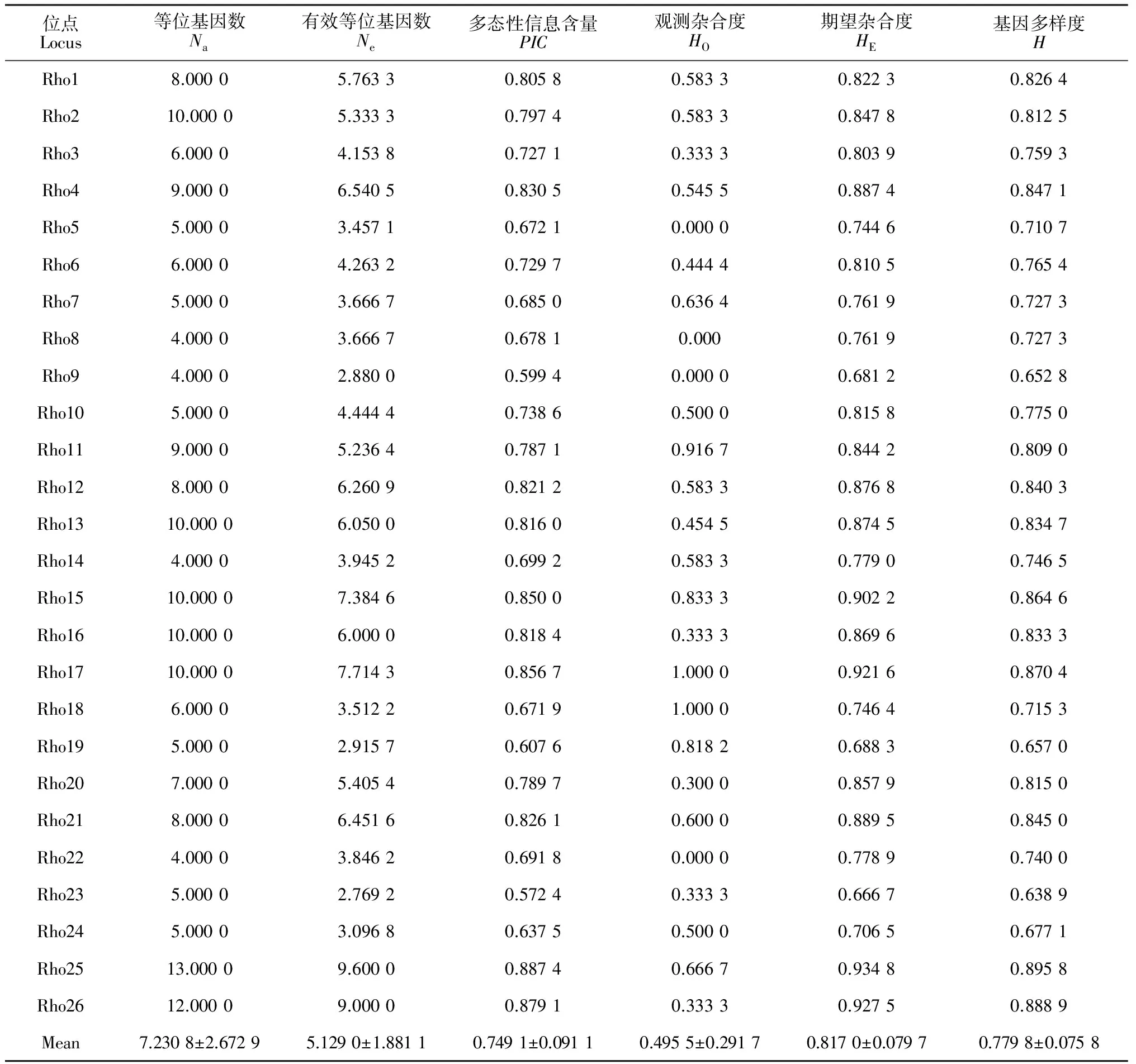
位点Locus等位基因数Na有效等位基因数Ne多态性信息含量PIC观测杂合度HO期望杂合度HE基因多样度HRho18.00005.76330.80580.58330.82230.8264Rho210.00005.33330.79740.58330.84780.8125Rho36.00004.15380.72710.33330.80390.7593Rho49.00006.54050.83050.54550.88740.8471Rho55.00003.45710.67210.00000.74460.7107Rho66.00004.26320.72970.44440.81050.7654Rho75.00003.66670.68500.63640.76190.7273Rho84.00003.66670.67810.0000.76190.7273Rho94.00002.88000.59940.00000.68120.6528Rho105.00004.44440.73860.50000.81580.7750Rho119.00005.23640.78710.91670.84420.8090Rho128.00006.26090.82120.58330.87680.8403Rho1310.00006.05000.81600.45450.87450.8347Rho144.00003.94520.69920.58330.77900.7465Rho1510.00007.38460.85000.83330.90220.8646Rho1610.00006.00000.81840.33330.86960.8333Rho1710.00007.71430.85671.00000.92160.8704Rho186.00003.51220.67191.00000.74640.7153Rho195.00002.91570.60760.81820.68830.6570Rho207.00005.40540.78970.30000.85790.8150Rho218.00006.45160.82610.60000.88950.8450Rho224.00003.84620.69180.00000.77890.7400Rho235.00002.76920.57240.33330.66670.6389Rho245.00003.09680.63750.50000.70650.6771Rho2513.00009.60000.88740.66670.93480.8958Rho2612.00009.00000.87910.33330.92750.8889Mean7.2308±2.67295.1290±1.88110.7491±0.09110.4955±0.29170.8170±0.07970.7798±0.0758
2.4 种质遗传多样性分析
2.4.1 SSR引物多态性分析结果 统计10对荧光标记SSR引物在69份高山杜鹃种质中的扩增条带数据,计算各位点的有效等位基因数、多态性信息含量、观测杂合度、期望杂合度及基因多样度等指标,其平均值分别为6.959 2、0.795 2、0.543 5、0.826 5和0.820 2(表5)。
2.4.2 SSR聚类结果 对69份高山杜鹃种质进行UPGMA聚类分析,结果分为三大支(图3):第一大支(Clade Ⅰ)主要由常绿杜鹃组中的3个野生种(A01、A03、A08)和云锦杜鹃花杂种群(D2、D14、W20、W29、W19、W31)所组成;第二大支(Clade Ⅱ)主要由同属糙叶杜鹃亚属的2个野生种(A14和A16)和同属杜鹃花组的2个野生种(A2、A10)及东石楠杜鹃花杂种群(D04、D19、 D23)组成;第三大支(Clade Ⅲ)的聚类成员最多,主要包含美国山石南杜鹃花杂种群(W35、D5、W5、W16、W22、W21、W23、W01、W06、W37、W02、W03、W27)、高加索杜鹃花杂种群(W08、W09、W10)、腺柱杜鹃花杂种群(D06、D13、D16、D24、W17、W18、W34、W33)和东石楠杜鹃花杂种群(D17、D18、D20、D25)。
从聚类结果可以看出,系谱资料与聚类结果间的一致性较好,提示聚类结果可靠。亲本未知类群在系统树上的聚类位置,可以作为我们推测其祖先亲本类型的科学依据。D12和D15作为外类群分支到三大支之外,推测它们的祖先亲本可能为常绿杜鹃亚属、杜鹃亚属及糙叶杜鹃亚属之外的杜鹃属某亚属内的杜鹃种类。

表5 10对荧光标记SSR引物的多态性分析结果

图中高山杜鹃样品编号所指同文中表1图3 基于SSR分子标记的69份高山杜鹃种质资源系统树图Sample codes in this image represent the same Rhododendron samples as Table 1Fig.3 Dendrogram of 69 alpine Rhododendron germplasms based on SSR markers
3 讨 论
3.1 DNA提取方法的优化改良
高山杜鹃植物体内的次生代谢物质丰富,为典型的多糖多酚植物类型。从多糖多酚植物类群中提取高质量的DNA是许多研究领域例如居群生物学、生物多样性、分子标记辅助育种等研究中遇到的普遍难题。本实验室的前期实验表明,高山杜鹃的DNA 质量关乎下游SSR-PCR实验的成败。有关改良的关键步骤和效果主要有:1)利用去多糖buffer I有效地洗去了植物细胞匀浆中大部分多糖和多酚等次生代谢物质;2) 提高细胞裂解液中的CTAB浓度,可以使植物细胞裂解更加充分,在一定程度上提高DNA得率;3)先将细胞裂解混合液进行高速离心后取上清,再对上清液进行抽提,可有效地提升抽提效果,提高DNA得率;4)对进行了1次抽提后的上清液中加入Rnase A处理,可基本去除RNA的污染。以上改良方法有效解决了用常规方法提取高山杜鹃DNA的杂质较多、纯度较低、DNA完整性较差等技术问题。此改进方案简单、经济、可靠、针对性强,DNA不需纯化可直接用于下游的分子实验,可能也适用于内源次生代谢物质丰富的其他植物种类。
3.2 多态性SSR引物的筛选
本研究用于引物筛选的12份材料包括3份高山杜鹃野生种和9份高山杜鹃杂交种,材料间来源差异较大,涉及不同野生种和品种。因此,筛选得的微卫星标记将具有更好的种间通用性,可应用于更大范围内的杜鹃属植物的遗传多样性等研究。总体引物筛选比率达17%(26/154),其中已发表引物的筛选比率约为17%(5/29),EST序列所设计引物筛选比率约为14%(15/105),基因组文库中的DNA序列所设计引物筛选比率最高达30%(6/20)。由此笔者认为,来源于基因组文库的DNA序列较EST序列更适合用于遗传多样性分析的SSR引物开发。
3.3 系谱关系与聚类结果间的比照分析
从69份高山杜鹃种质的UPGMA聚类结果来看,各分支中的野生种和杂交种的聚类结果与其分类地位或系谱资料间基本吻合。通过二者之间的比照分析,可以对系谱资料不完全或缺失的高山杜鹃杂交种的祖先亲本来源作出科学性解释或推测。
第一大支中(Clade Ⅰ)分属不同的常绿杜鹃组亚组的3个野生种(大白杜鹃、马缨杜鹃和露珠杜鹃)聚在了一起,此结果可以通过它们之间存在频繁自然杂交现象的分子证据来解释[31-32];其它则主要为云锦杜鹃花杂种群类型(D2、D14、W20、W29、W19、W31)。基于此,虽然杂交种D2和W31基于祖先亲本的遗传贡献值应归类到六大类之外,但是云锦杜鹃(R.fortunei)作为其祖先亲本之一,在两品种中的遗传贡献值均达到了0.25,且根据聚类结果来看,它们确与其它云锦杜鹃花杂种群聚在了一起。同理,杂交种D24和W33在系谱资料和SSR聚类间也存在此类矛盾。笔者认为遗传贡献值的计算方法在亲缘关系分析上仅具有一定的参考价值,毕竟遗传贡献值只是一个理想的假设,并未考虑到性状连锁遗传和细胞质遗传等情况。
第二大分支(Clade Ⅱ)则主要集结了常绿杜鹃亚属之外的杜鹃亚属和糙叶杜鹃亚属内的4个野生种,分别为大喇叭杜鹃(A2)、云南杜鹃(A10)、爆杖花杜鹃(A14)和富源杜鹃(A16),其它主要为东石楠杜鹃花杂种群(D04、D19、 D23)。据此,我们推测R.degronianumspp.yakushimanum也可能是D09或D11的祖先亲本之一。
第三大支(Clade Ⅲ)主要集结了美国山石南杜鹃花杂种群(W35、D5、W5、W16、W22、W21、W23、W01、W06、W37、W02、W03、W27)、高加索杜鹃花杂种群(W08、W09、W10)、腺柱杜鹃花杂种群(D06、D13、D16、D24、W17、W18、W34、W33)及东石楠杜鹃花杂种群(D17、D18、D20、D25)。基于此结果,我们推测W14的母本可能为R.catawbiense而并非R.ponticum,且R.catawbiense也可能是W11、W12和XXL的亲本之一;R.caucasicum可能是W13、W30和W36的亲本之一;R.griffithianum可能为D01、D03、D08、W07、W24的亲本之一,还可能是亲本未知类群D10、W25、W04、R1的亲本之一。
根据62个引进高山杜鹃育成品种的重要祖先亲本组成类型及其遗传贡献值,可以看出这些国外引进品种的祖先亲本主要来源于中国,且亲本来源范围相对狭窄,主要集中来源于常绿杜鹃亚属(Subgen.Hynenaanthes)常绿杜鹃组(Sect. Ponticum)。比照62份高山杜鹃种质的系谱资料和SSR聚类分析结果,二者之间基本吻合。本研究主要对系谱资料不详的编号为D1、D03、D8、D09、D10、D11、W04、W07、W11、W12、W13、W14、W24、W25、W30、W36、R1、XXL的高山杜鹃杂交种进行了亲本类型推测,研究结果可为未来高山杜鹃杂交育种的亲本选配提供科学的理论参考。
[1] 冯正波, 庄 平, 张 超, 等. 野生杜鹃花迁地保护适应性评价[J].云南植物研究, 2004, 26(5): 497-506.
FENG Z B, ZHUANG P, ZHANG C,etal. An adaptive evaluation of wildRhododendronunder ex-situ conservation [J].ActaBotanicaYunnanica, 2004, 26(5): 497-506.
[2] 闵天禄, 方瑞征. 杜鹃属(RhododendronL.)的地理分布及其起源问题的探讨[J]. 云南植物研究, 1979, 1(2): 17-28.
MIN T L, FANG R Z. On the origin and geographic distribution of genusRhododendronL. [J].ActaBotanicaYunnanica, 1979, 1(2): 17-28.
[3] 方瑞征, 闵天禄. 杜鹃属植物区系的研究[J]. 云南植物研究, 1995, 28(4): 359-379.
FANG R Z, MIN T L. The floristic study on the genusRhododendron[J].ActaBotanicaYunnanica, 1995, 28(4): 359-379.
[4] 武建勇,薛达元,周可新. 皇家爱丁堡植物园引种中国植物资源多样性及动态[J].植物遗传资源学报, 2011, 12(5):738-743.
WU J Y,XUE D Y,ZHOU K X. Diversity and dynamics of introduced plant from China in Royal Botanic Garden Edinburgh [J].JournalofPlantGeneticResources, 2011, 12(5):738-743.
[5] 李志斌, 白霄霞, 李 萍. 高山杜鹃将成为我国高档园林绿化的新秀——高山杜鹃研究的过去·现在·将来[J]. 农业科技与信息: 现代园林, 2007, 7: 82-85.
LI Z B, BAI X X, LI P. The alpine rose will become our country top grade landscaping new favorite—The past, now, future of alpine rose studies [J].ModernLandscapeArchitecture, 2007, 7: 82-85.
[6] WU H, YANG X, SHAO H,etal. Germplasm resource base for rhododendron horticulture: status, problems and countermeasures [J].BiodiversityScience, 2014,21(5): 628-634.
[7] DIKSHIT H K, HANG T J, SINGH N K,etal. Genetic differentiation of Vigna species by RAPD, URP and SSR markers [J].BiologiaPlantarum, 2007,51: 451-457.
[8] NAITO K, ISAGI Y, NAKAGOSHI N. Isolation and characterization of microsatellites ofRhododendronmetternichiisieb. et zucc. var.hondoensenakai[J].MolecularEcology, 1998,7: 927-928.
[9] DENDAUW J, DE RIEK J, ARENS P,etal. Development of sequenced tagged microsatellite site (STMS) markers in azalea [J].ActaHorticulturae, 2001,546: 193-197.
[10] TAN X X, LI Y, GE X J. Development and characterization of eight polymorphic microsatellites forRhododendronsimsiiPlanch (Ericaceae) [J].ConservationGenetics, 2009,9(1): 326-329.
[11] WANG X Q, YUAN H, LONG C L. Isolation and characterization of twenty-four microsatellite loci forRhododendrondecorumFranch. (Ericaceae) [J].Hortscience, 2009,44(7): 2 028-2 030.
[12] WANG N, QIN Z C, YANG J B,etal. Development and characterization of 15 microsatellite loci forRhododendrondelavayiFranch.(Ericaceae) [J].Hortscience, 2010,45(3): 457-459.
[13] LI L F, YIN D X, SONG N,etal. Genomic and EST microsatellites forRhododendronaureum(Ericaceae) and cross-amplification in other congeneric species [J].AmericanJournalofBotany, 2011,98(9): 250-252.
[14] CHOUDHARY S, THAKUR S, SAINI R G,etal. Development and characterization of genomic microsatellite markers inRhododendronarboretum[J].ConservationGeneticsResources, 2014,6(4): 1 029-1 035.
[15] YAN L J, ZHANG Z R, LI D Z,etal. Isolation and characterization of microsatellite markers for the endemic speciesRhododendronspinuliferum(Ericaceae) [J].PlantDiversity&Resources, 2014,36(1): 41-46.
[16] CHARRIER O, DELMAS C E, PORNON L A,etal. Development of 18 microsatellite markers inRhododendronferrugineum(Ericaceae) for investigating genetic structure at margins [J].ConservationGeneticsResources, 2013,5(2): 473-477.
[17] KWAK M, WON H, HONG J,etal. Isolation and characterization of 19 novel microsatellite loci inRhododendronaureum, andRhododendronbrachycarpum(Ericaceae) [J].BiochemicalSystematics&Ecology, 2015,61: 520-523.
[18] WANG S Z, LI Z L, JIN W B,etal. Development and characterization of polymorphic microsatellite markers inRhododendronsimsii(Ericaceae) [J].PlantSpeciesBiology, 2016, DOI: 10.1111/1442-1984.12125.
[19] 崔章林, 盖钧镒, CARTER T E, 等.中国大豆育成品种及其系谱分析[M]. 北京: 中国农业出版社, 1998: 23-311.
[20] 陈俊愉. 中国花卉品种分类学[M]. 北京: 中国林业出版社, 2000:197-198.
[21] 曾 杰, 邹喻苹, 白嘉雨, 等. 顽拗植物类群的总DNA制备[J]. 植物学报, 2002, 44(6): 694-697.
ZENG J, ZOU Y P, BAI J Y,etal. Preparation of total DNA from ‘recalcitrant plant taxa’ [J].ActaBotanicaSinica, 2002, 44(6): 694-697.
[22] 王书珍, 张传进, 查三省, 等. 杜鹃基因组 DNA 提取方法的研究及应用[J]. 林业科技通讯, 2015, 2: 5-8.
WANG S Z, ZHANG C J, ZHA S S,etal. Genomic DNA extracting and its application inRhododendron[J].ForestScienceandTechnology, 2015, 2: 5-8.
[23] DE KEYSER E, DE RIEK J,etal. Discovery of species-wide EST-derived markers inRhododendronby intron-flanking primer design [J].Molecularbreeding, 2009, 23(1): 171-178.
[24] WEI H, DHANARAJ A L, ROWLAND L J,etal. Comparative analysis of expressed sequence tags from cold-acclimated and non-acclimated leaves ofRhododendroncatawbienseMichx [J].Planta, 2005, 221(3): 406-416.
[25] 李 强, 万建民. SSRHunter, 一个本地化的SSR位点搜索软件的开发[J]. 遗传, 2005, 27(5): 808-810.
LI Q, WAN J M. SSRHunter: development of local searching software for SSR sites [J].Hereditas, 2005, 27(5): 808-810.
[26] CLARKE K R, GORLEY R N. Primer v5: user manual/tutorial. PRIMER-E Ltd. [J],Plymouth, 2001.
[27] 樊文强, 盖红梅, 孙 鑫,等. SSR数据格式转换软件DataFormater [J]. 分子植物育种, 2016, 14(1): 265-270.
FAN W Q, GE H M, SUN X,etal. DataFormater, a software for SSR data formatting to develop population genetics analysis [J].MolecularPlantBreeding, 2016, 14(1): 265-270.
[28] YEH F C, BOYLE T J B. Population genetic analysis of co-dominant and dominant markers and quantitative traits [J].BelgianJournalofBotany, 1997, 129: 157.
[29] LIU K, MUSE S V. PowerMarker: an integrated analysis environment for genetic marker analysis [J].Bioinformatics, 2005, 21(9): 2 128-2 129.
[30] ROHLF F J. NTSYS-pc: numerical taxonomy (VitisviniferaL.) varieties using morphological data and multivariate analysis system. Exeter Software, and AFLP markers [J].ElectronicJournalofBiotechnology, 2000, 6: 37-45.
[31] ZHA H G, MILNE R I, HANG S.. Morphological and molecular evidence of natural hybridization between two distantly relatedRhododendronspecies from the Sino-Himalaya [J].BotanicalJournaloftheLinneanSociety, 2008, 156(1):119-129.
[32] ZHA H G, MILNE R I, HANG S. Asymmetric hybridization inRhododendronagastum: a hybrid taxon comprising mainly F1s in Yunnan, China [J].AnnalsofBotany, 2010, 105(1): 89-100.
(编辑:宋亚珍)
Ancestor Parents Speculation for AlpineRhododendronHybrids Based on the Pedigree and SSR Markers
ZHANG Lu, WANG Jihua, XIE Weijia, CAI Yanfei,SONG Jie, PENG Lüchun, LI Shufa, LI Shifeng*
(Flower Research Institute, Yunnan Academy of Agricultural Sciences, National Engineering Research Center for Ornamental Horticulture, Key Lab of Yunnan Flower Breeding, Kunming 650205, China)
In this study, the total DNA extraction method was modified based on the conventional CTAB method forRhododendron, and then many microsatellite sequences were chosen from the publishedRhododendronEST database and unpublished genome database ofR.delavayiin our lab to design SSR primer pairs. Moreover, adding many SSR primer pairs referred to the references, 154 pairs of SSR primers were used to be screened. Finally, 26 pairs of polymorphic primers were screened out, and all of PCR amplicons were specific with clearer band and better repeatability. Furthermore, 10 pairs of polymorphic SSR primer fluorescently-labeled were used to genetic diversity analysis on 69Rhododendrongermplasms. The results as following: mean effective allele number (Ne), polymophism information content (PIC), observed heterozygosity (HO) , expective heterozygosity (HE), andNei’sgene diversity(H) for 10 SSR locus are 6.959 2, 0.795 2, 0.543 5, 0.826 5 and 0.820 2, respectively. Based on the consistency between the UPGMA clustering results and their pedigree analysis ofRhododendronhybrids, a speculation of ancestor parent types will be made forRhododendronhybrids with unknown or unclear pedigree.
alpineRhododendron; DNA extraction; SSR primer screening; pedigree; genetic relationship
1000-4025(2016)12-2421-12
10.7606/j.issn.1000-4025.2016.12.2421
2016-10-17;修改稿收到日期:2016-11-29
国家自然科学基金(31460217,31560225);云南省应用基础研究计划(2016FB058);云南省花卉育种重点实验室开放基金(FKL-201501);云南省科技计划(2015BB013)
张 露(1984-),女,博士,助理研究员,主要从事高山杜鹃植物的遗传育种研究。E-mail: changjiangyulu@163.com
*通信作者:李世峰,硕士,研究员,主要从事高山杜鹃种质创新及繁育研究。E-mail:452977351@qq.com
Q346+.5;Q789
A
