应用PCR⁃DGGE技术比较绵羊瘤胃、网胃、瓣胃和皱胃菌群的多样性
2016-01-19东倪学勤张唐雨蕊四川农业大学动物医学院动物微生态研究中心动物疫病与人类健康四川省重点实验室雅安625014
曾 燕 曾 东倪学勤张 艳 祝 辉 唐雨蕊(四川农业大学动物医学院动物微生态研究中心,动物疫病与人类健康四川省重点实验室,雅安625014)
应用PCR⁃DGGE技术比较绵羊瘤胃、网胃、瓣胃和皱胃菌群的多样性
曾 燕 曾 东∗倪学勤∗张 艳 祝 辉 唐雨蕊
(四川农业大学动物医学院动物微生态研究中心,动物疫病与人类健康四川省重点实验室,雅安625014)
摘 要:本研究采用聚合酶链式反应-变性梯度凝胶电泳(PCR⁃DGGE)技术结合DGGE图谱条带的克隆和测序比较了绵羊瘤胃、网胃、瓣胃和皱胃菌群的多样性,同时对菌群进行了聚类分析和主成分分析。结果表明:绵羊瘤胃、网胃、瓣胃和皱胃内容物DGGE图谱的平均条带数分别为18、13、16和15条;瘤胃和瓣胃内菌群的多样性指数、均匀度和丰富度均较高,分别为2.83、0.79、17.00和2.82、0.79、16.80,而网胃和皱胃内容物较低,分别为2.52、0.70、12.60和2.73、0.76、15.40;聚类分析结果显示不同胃的内容物菌群具有一定差异,但不同动物个体相同胃的内容物相似性系数均高于0.63;PCR⁃DGGE图谱中测序的条带大多数归为拟杆菌门(Bacte⁃roidetes)和厚壁菌门(Firmicutes),优势菌群为厚壁菌门(Firmicutes)细菌、拟杆菌门(Bacte⁃roidetes)细菌、未培养瘤胃细菌(uncultured rumen bacterium)、未培养细菌(uncultured bacterium)和韦荣球菌科(Veillonellaceae)细菌,特异性细菌为不动杆菌属(Acinetobacter sp.)细菌和瘤胃球菌属(Ruminococcaceae)细菌。结果提示,绵羊4个胃中均含有种类丰富和数量巨大的细菌,且随着消化道部位由前往后的顺序,菌群的多样性呈现先高后降低再升高的趋势。
关键词:绵羊;胃;细菌;PCR⁃DGGE;聚类分析;主成分分析
成年哺乳动物胃肠道栖息着种类繁多且数量巨大的微生物,其数量是动物体细胞数的10倍左右[1]。宿主可为微生物提供良好的生长环境,而微生物通过促进胃肠道对营养物质的消化吸收维持动物的健康[2]。近年来,反刍动物依赖瘤胃微生物能将纤维素等物质有效地转化为动物产品成为了研究热点[3],由于采样的难易程度不同,大多数都集中在对瘤胃和粪中微生物的研究[4-6]。然而,网胃、瓣胃和皱胃也是反刍动物的标志性消化器官,其中栖息的微生物及这些微生物对宿主的作用等信息大多数仍处于未知状态。因此,反刍动物微生物的研究,尤其是4个胃的微生物的研究,对畜牧生产均具有指导意义,具有广阔的前景。
目前,利用纯培养技术,仅有11%左右的动物胃肠道微生物被分离培养[7],仍有大量的微生物信息处于未知状态。PCR⁃DGGE技术1993年首次被Muyzer等[8]应用于研究微生物菌群结构和多样性,目前已被广泛用于胃肠道、土壤、葡萄酒和海洋微生物的研究,能全面准确地反映微生物菌群的结构和组成。因此,本研究应用PCR⁃DGGE技术结合DGGE图谱中共性条带和特异性条带的克隆和测序,全面评估绵羊4个胃细菌菌群的结构和组成,为今后反刍动物胃肠道微生物的研究提供有价值的理论指导。
1 材料与方法
1.1 试验动物
试验动物来自四川省雅安市名山县屠宰场,选择5只同批次成年雄性蒙古羊作为采样对象,体重为(48.16±1.48)kg。
1.2 试验材料
1.2.1 样品的采集
无菌操作采集试验羊瘤胃、网胃、瓣胃和皱胃新鲜内容物,各采集3份,每份采集5 g,分装至灭菌EP管中至液氮速冻,-80℃保存。
1.2.2 主要试剂和仪器
1.3 细菌总DNA的提取及16S rDNA V3区的扩增
参照Li等[9]方法提取细菌总DNA。用核酸蛋白浓度测定仪测定细菌总DNA浓度后置-20℃保存备用。大肠杆菌16S rRNA V3区通用引物[10]的上、下游序列分别为:5′-CGC CCG GGG CGC GCC CCG GGC GGG GCG GGG G CA CGG GGG GAC TCC TAC GGG AGG CAG CAG T-3′和5′-GTA TTA CCG CGG CTG CTG GCA C-3′(带下划线部分为“GC”架子序列)。PCR扩增体系(25 μL):2×Taq MasterMix 12 μL,上、下游引物(10 pmol/μL)各1.0 μL,模板总DNA 1.0 μL,ddH2O 10 μL。扩增条件为94℃4 min;94℃30 s,58℃30 s,72℃2 min,35个循环;72℃延伸10 min。PCR产物片段大小用1.0%琼脂糖凝胶电泳检测。
1.4 PCR⁃DGGE分析及条带的克隆测序
参照倪学勤等[11]方法进行样品细菌的PCR⁃DGGE。选取35%~65%的凝胶电泳梯度(100%的变性剂包括7 mol/L尿素和40%甲酰胺),变性方向与电泳方向一致。电泳缓冲液为1×TAE,电压为100 V,温度为60℃,电泳时间为15 h。电泳结果经硝酸银染色后用Bio⁃Rad GS800 Calibrated Densitometer扫描成像。同时,回收DGGE图谱上的共性条带和特异性条带,进行克隆测序。测定序列采用Chromas 2软件进行编辑,非嵌合体序列在GenBank数据库(http://www.ncbi.nlm.nih.gov/BLAST/)中进行比对分析,寻找亲缘关系最近的细菌或克隆。
1.5 计算公式

式中:H为多样性指数(Shannon diversity in⁃dex);Hmax为当i为所有样品总物种数时的多样性指数;pi为物种i的相对丰度比例;E为均匀度(evenness);R为丰富度(richness);s为样品在DGGE的条带数。
1.6 数据分析
运用SAS 9.1软件、Excel 2013和NTSYS 2.10软件对PCR⁃DGGE图谱数据进行菌群多样性分析、主成分分析(PCA)和聚类分析。
2 结果与分析
2.1 细菌16S rDNA V3区PCR⁃DGGE图谱分析
PCR⁃DGGE图谱中,强的电泳条带代表了优势菌群,条带位置和数量分别代表细菌种类和丰富度。由图1可见,绵羊4个胃内容物样品均产生了丰富的电泳条带,且相邻胃的菌群结构和组成相似,不相邻胃的菌群结构和组成存在一定差异。绵羊瘤胃、网胃、瓣胃和皱胃内容物的平均条带数分别为18、13、16和15条。与瘤胃相比,瓣胃内容物菌群的丰富度更高;与瓣胃相比,皱胃内容物菌群丰富度更低。此结果可能是由于不同胃对营养物质的吸收不同,使其定植的细菌菌群结构和组成存在差异。
2.2 PCR⁃DGGE图谱多样性分析
由表1可知,绵羊不同胃内容物菌群的多样性指数存在差异。瘤胃、瓣胃和皱胃内容物菌群的多样性指数、均匀度和丰富度均较高,分别为2.83、0.79、17.00和2.82、0.79、16.80及2.73、0.76、15.40,网胃内容物菌群的多样性指数、均匀度和丰富度较低,为2.52、0.70、12.60。
2.3 PCR⁃DGGE图谱的聚类分析
PCR⁃DGGE图谱结果(图1)表明绵羊4个胃内容物中均含有物种丰富且数量巨大的细菌,但聚类分析结果(图2)显示,绵羊瘤胃和网胃内容物聚为一簇,相似性系数为0.62;瓣胃和皱胃内容物聚为一簇,相似性系数为0.56。绵羊不同胃内容物菌群具有一定差异,但不同动物个体、相同胃内容物的相似性系数均高于0.63,表明动物个体差异对来自相同消化道部位样品细菌的影响较小。
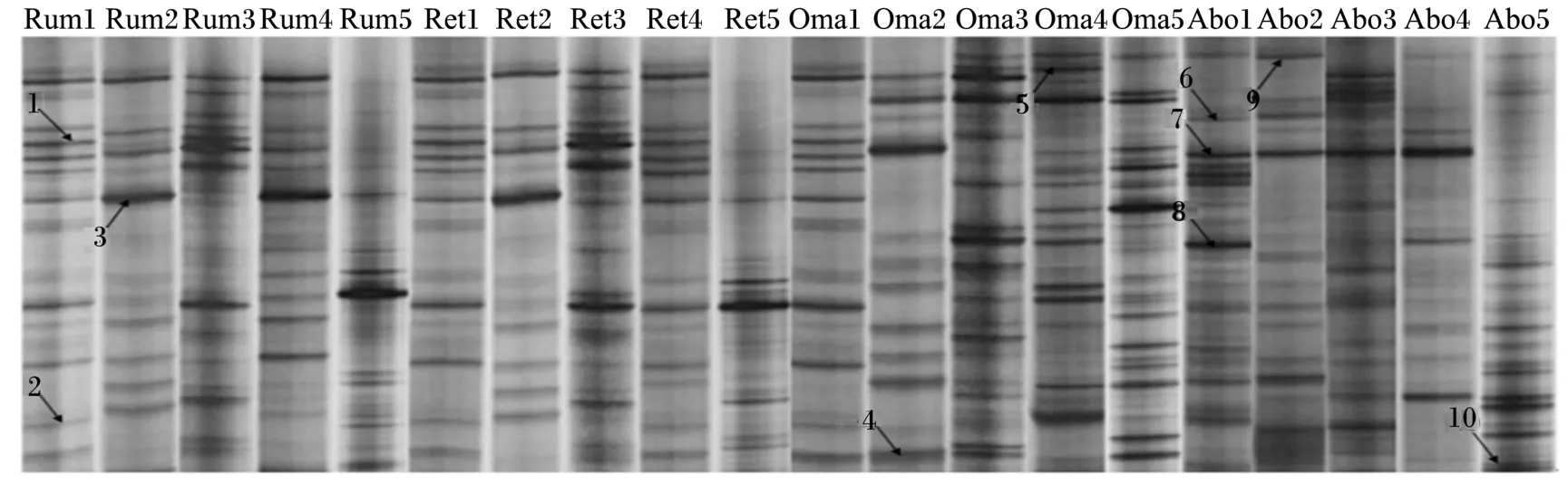
图1 绵羊胃细菌16S rDNA V3区PCR⁃DGGE图谱Fig.1 PCR⁃DGGE profiles of 16S rDNA V3 region of stomach bacteria from sheep

图2 PCR⁃DGGE图谱的聚类分析Fig.2 Cluster analysis of PCR⁃DGGE profiles
2.4 PCR⁃DGGE图谱的PCA
PCR⁃DGGE图谱的PCA(图4)同聚类分析的结果一致。主成分因子1(PC1)的贡献率为21.37%,主成分因子2(PC2)的贡献率为15.11%;PC1明显地将样品分成2个部分,来自绵羊瘤胃和网胃内容物主要分布在图的左边,瓣胃和网胃内容物主要分布在图的右边;同样,PC2明显地将瘤胃、瓣胃内容物同网胃、皱胃内容物区别开来。由此可看出,绵羊4个胃内容物菌群结构和组成既有相似之处,但又相互区别。

图4 PCR⁃DGGE图谱的PCAFig.4 PCA of PCR⁃DGGE profiles
2.5 绵羊胃的共性细菌和特异性细菌
从PCR⁃DGGE图谱上共回收10个条带(图1箭头所指),测序结果在GenBank数据库中比对,结果见表2。绵羊的4个胃中均栖息着数量庞大且菌群结构丰富的细菌,含有大量的厚壁菌门(Firmicutes)细菌、拟杆菌门(Bacteroidetes)细菌、未培养瘤胃细菌(uncultured rumen bacterium)、未培养细菌(uncultured bacterium)和韦荣球菌科(Veillonellaceae)细菌。而特异性条带较少,如不动杆菌属(Acinetobacter sp.)细菌和瘤胃球菌属(Ruminococcaceae)细菌。
由表2可看出,有70%的测序结果与Gen⁃Bank数据库中微生物的同源性高达100%,而条带1、8和9在数据库中比对分析后的相似性均达到了97%以上,说明试验中所测的DGGE条带与已鉴定的微生物亲缘性非常高。
3 讨 论
本研究应用PCR⁃DGGE技术分析绵羊胃的菌群。一般而言,动物在出生时其胃肠道几乎未发育完全,但随着消化器官的发育,其中的微生物种类和数量逐渐增多[12],而瘤胃因其庞大的体积和严格的厌氧环境含有丰富的微生物。PCR⁃DGGE图谱(图1)显示,绵羊的4个胃均具有种类复杂且数量巨大的细菌。菌群的多样性指数结果(表1和图2)表明,瘤胃、瓣胃和皱胃内容物菌群的多样性指数、均匀度和丰富度均较高,且随着消化道部位由前往后的顺序,呈现出先高后降低,再升高的趋势,这与胃的功能和环境密切相关。
PCR⁃DGGE图谱中条带的测序结果(表2)显示,瘤胃、网胃、瓣胃和皱胃中均存在大量的厚壁菌门细菌、未培养拟杆菌门细菌、未培养瘤胃细菌、韦荣球菌科细菌、不动杆菌属菌和未培养瘤胃球菌属细菌,说明绵羊胃中的细菌大多数来自厚壁菌门和拟杆菌门。同样,大量研究表明,反刍动物胃肠道内存在种类繁多且数量巨大的微生物,主要是厚壁菌门菌和拟杆菌门菌[13-15]。拟杆菌门菌是动物胃肠道的一类革兰氏阴性菌,该类群在降解和发酵植物细胞壁多糖等有机物方面具有重要的作用[16]。本研究中,PCR⁃DGGE图谱条带的测序结果大多数归为拟杆菌门菌,而厚壁菌门细菌相对较少,说明绵羊的4个胃的细菌在分解和消化吸收植物细胞壁多糖等方面发挥了重要的作用。Cunha等[17]研究巴西山羊的瘤胃液相和固相微生物,结果表明类杆菌门和厚壁菌门为优势菌群。而Singh等[18]采用454 GS FLX高通量测序技术研究了饲喂4种不同精粗比饲粮的水牛瘤胃微生物,发现厚壁菌门菌和变形菌均为优势菌。这均与本研究中的测序结果存在一定的差异,可能是由于动物品种及饲粮不同等因素引起的。结合PCR⁃DGGE图谱和条带的测序结果可看出,仅在瘤胃和网胃中存在大量的未培养瘤胃细菌;而瓣胃和皱胃中存在大量的韦荣球菌科细菌,可能是由于各个胃在消化道的位置不同而引起的。有研究表明,瘤胃中存在大量球菌,尤其是白色瘤胃球菌(Ruminococcus albus)和黄色瘤胃球菌(Rumi⁃nococcus flavefaciens),在降解植物纤维素方面具有不可替代的作用[19-20]。而在本研究结果中,仅在皱胃检测出瘤胃球菌,进一步说明反刍动物消化道的其他部位也存在大量的未培养瘤胃球菌。
聚类分析(图3)及PCA(图4)表明,不同动物个体间相同消化道部位菌群的结构和组成具有较高的相似性;同一动物个体不同消化道部位细菌菌群的结构和组成存在差异。PC1将瘤胃和网胃内容物同瓣胃和皱胃内容物分开,PC2将瘤胃和瓣胃内容物同网胃和皱胃内容物分开,可能是由相邻消化道部位含有相似的营养物质和营养物质在胃中的停留时间不同导致的。本研究的样品虽然来自同一批次的绵羊,但由于动物个体的差异和饲粮组成的差异,所以来自相同消化道部位的不同动物间的菌群存在差异。Jami等[21]对健康荷斯坦奶牛个体间瘤胃微生物的研究发现所有动物个体均有共同的细菌,但个体间仍存在一定差异,这与本研究结果一致。本研究仅对绵羊的4个胃的细菌进行了初步的研究,并不能代表全部的细菌信息,因此,每种细菌的数量差异还有待进一步研究。

表2 PCR⁃DGGE图谱共性条带和特异性条带Table 2 Common bands and special bands in PCR⁃DGGE profiles
4 结 论
①绵羊瘤胃、网胃、瓣胃和皱胃优势菌群为厚壁菌门细菌、拟杆菌门细菌、未培养瘤胃细菌、未培养细菌和韦荣球菌科细菌,特异性细菌为不动杆菌属(Acinetobacter sp.)细菌和瘤胃球菌属(Ru⁃minococcaceae)细菌。
②绵羊4个胃中均含有种类丰富和数量巨大的细菌,且随着消化道部位由前往后的顺序,菌群的多样性呈现先高后降低再升高的趋势。
参考文献:
[1] BACKHED F,LEY R E,SONNENBURG J L,et al. Host-bacterial mutualism in the human intestine[J].Science,2005,307(5717):1915-1920.
[2] DETHLEFSEN L,MCFALL⁃NGAI M,RELMAN D A.An ecological and evolutionary perspective on hu⁃man⁃microbe mutualism and disease[J].Nature,2007,449(7164):811-818.
[3] 刘开朗,王加启,卜登攀.2008—2009年反刍动物营养研究进展Ⅰ.瘤胃微生物多样性与功能[J].中国畜牧兽医,2010,37(2):5-14.
[4] PEI C X,LIU Q,DONG C S,et al.Diversity and a⁃bundance of the bacterial 16S rRNA gene sequences in forestomach of alpacas(Lama pacos)and sheep (Ovis aries)[J].Anaerobe,2010,16(4):426-432.
[5] FOUTS D E,SZPAKOWSKI S,PURUSHE J,et al.Next generation sequencing to define prokaryotic and fungal diversity in the bovine rumen[J].PLoS One,2012,7(11):e48289.
[6] LIN B,LU Y,SALEM A Z M,et al.Effects of essen⁃tial oil combinations on sheep ruminal fermentation and digestibility of a diet with fumarate included[J].Animal Feed Science and Technology,2013,184(1/2/3/4):24-32.
[7] EDWARDS J E,MCEWAN N R,TRAVIS A J,et al.16S rDNA library⁃based analysis of ruminal bacterial diversity[J].Antonie van Leeuwenhoek,2004,86 (3):263-281.
[8] MUYZER G,DE WAAL E C,UIRRERLINDEN A G.Profiling of complex microbial population by dena⁃turing gradient gel electrophoresis analysis of polymer⁃ase chain reaction-amplified genes encoding for 16S rRNA[J].Applied and Environmental Microbiology,1993,59(3):695-700.
[9] LI M,GONG J H,COTRILL M,et al.Evaluation of
DNA Stool Mini Kit for ecological studies of gut microbiota[J].Journal of Microbiological Methods,2003,54(1):13-20
[10] WALTER J,HERTEL C,TANNOCK G W,et al.De⁃tection of Lactobacillus,Pediococcus,Leuconostoc and Weissella species in human feces by using group⁃spe⁃cific PCR primers and denaturing gradient gel electro⁃phoresis[J].Applied and Environmental Microbiolo⁃gy,2001,67(6):2578-2585.
[11] 倪学勤,GONG J,YU H,等.采用PCR⁃DGGE技术分析蛋鸡肠道细菌种群结构及多样性[J].畜牧兽医学报,2008,39(7):955-961.
[12] 张柏林,秦贵信,孙泽威,等.仔猪胃肠道微生物菌群定植规律及其功能的研究进展[J].中国畜牧杂志,2009,45(19):66-69.
[13] CALLAWAY T R,DOWD S E,EDRINGTON T S,et al.Evaluation of bacterial diversity in the rumen and feces of cattle fed different levels of dried distillers grains plus solubles using bacterial tag⁃encoded FLX amplicon pyrosequencing[J].Journal of Animal Sci⁃ ence,2010,88(12):3977-3983.
[14] ROSS E M,MOATE P J,BATH C R,et al.High throughput whole rumen metagenome profiling using untargeted massively parallel sequencing[J].BMC Genetics,2012,13(1):53.
[15] THOETKIATTIKUL H,MHUANTONG W,LAOTHANACHAREON T,et al.Comparative analy⁃sis of microbial profiles in cow rumen fed with differ⁃ent dietary fiber by tagged 16S rRNA gene pyrose⁃quencing[J].Current Microbiology,2013,67(2):130-137.
[16] KURITZA A P,SHAUGHNESSY P,SALYERS A A.Enumeration of polysaccharide⁃degrading Bacteroides species in human feces by using species⁃specific DNA probes[J].Applied and Environmental Microbiology,1986,51(2):385-390.
[17] CUNHA I S,BARRETO C C,COSTA O Y A,et al.Bacteria and Archaea community structure in the ru⁃men microbiome of goats(Capra hircus)from the semiarid region of Brazil[J].Anaerobe,2011,17(3):118-124.
[18] SINGH K M,AHIR V B,TRIPATHI A K,et al.Met⁃agenomic analysis of Surti buffalo(Bubalus bubalis)rumen:a preliminary study[J].Molecular Biology Re⁃ports,2012,39(4):4841-4848.
[19] MOSONI P,MARTIN C,FORANO E,et al.Long⁃term defaunation increases the abundance of cellulolyt⁃ic ruminococci and methanogens but does not affect the bacterial and methanogen diversity in the rumen of sheep[J].Journal of Animal Science,2011,89(3):783-791.
[20] CARBERRY C A,KENNY D A,HAN S,et al.Effect of phenotypic residual feed intake and dietary forage content on the rumen microbial community of beef cattle[J].Applied and Environmental Microbiology,2012,78(14):4949-4958.
(编辑 王智航)
[21] JAMI E,MIZRAHI I.Composition and similarity of bovine rumen microbiota across individual animals [J].PLoS One,2012,7(3):e33306.
A Comparison Study on Bacteria Diversity of Rumen,Reticulum,Omasum and Abomasum from Sheep Using PCR⁃DGGE Analysis
ZENG Yan ZENG Dong∗NI Xueqin∗ZHANG Yan ZHU Hui TANG Yurui
(Institute of Animal Microecology,College of Veterinary Medicine,Sichuan Agricultural University,Key Laboratory of Animal Disease and Human Health of Sichuan Province,Ya’an 625014,China)
∗Corresponding author,ZENG Dong,professor,E⁃mail:zend@sicau.edu.cn;NI Xueqin,professor,E⁃mail:xueqinni@foxmail.com
Abstract:In order to compare the bacterial flora diversity of rumen,reticulum,omasum and abomasum from sheep,polymerase chain reaction⁃denaturing gradient gel electrophoresis(PCR⁃DGGE)analysis compounded with cloning and sequencing of bands in DGGE profiles were used,and cluster analysis and principal constitu⁃ent analysis(PCA)of bacterial flora were carried out.The results showed as follows:the average number of DGGE bands of contents in rumen,reticulum,omasum and abomasum was 18,13,16 and 15,respectively;Shannon diversity index,evenness and richness of bacteria in rumen and omasum were higher,which were 2.83,0.79 and 17.00,and 2.82,0.79 and 16.80,respectively,however,those of reticulum and abomasum were lower,which were 2.52,0.70 and 12.60,and 2.73,0.76 and 15.40,respectively;clustering analysis results suggested that there were some differences in bacterial flora among the contents in different stomachs,but the similarity coefficients from the same segment of stomach of different animals were all above 0.63;most of the sequenced bands belonged to Bacteroidetes and Firmicutes,and Bacteroidetes,Firmicutes,uncultured rumen bacterium,uncultured bacterium and Veillonellaceae were predominant in stomach,while the special bacteria were Acinetobacter sp.and Ruminococcaceae bacteria.In conclusion,there are rich kinds and large a⁃mount of bacteria in the four stomachs of sheep.With rearward moving of position of intestinal tract,the diver⁃sity increased firstly and then decreased and finally increased.[Chinese Journal of Animal Nutrition,2015,27 (1):298⁃304]
Key words:sheep;stomach;bacteria;PCR⁃DGGE;cluster analysis;PCA
通信作者:∗曾 东,教授,博士生导师,E⁃mail:zend@sicau.edu.cn;倪学勤,教授,博士生导师,E⁃mail:xueqinni@foxmail.com
作者简介:曾 燕(1987—),女,四川内江人,硕士研究生,从事动物微生态研究。E⁃mail:zengyanA@foxmail.com
基金项目:教育部留学回国人员科研启动基金资助项目(200835⁃1);四川省学术带头人培养基金资助
收稿日期:2014-07-08
doi:10.3969/j.issn.1006⁃267x.2015.01.036
文章编号:1006⁃267X(2015)01⁃0298⁃07
文献标识码:A
中图分类号:S826
