香樟延伸因子EF1a基因片段的克隆及表达分析
2015-12-20张力维李勇鹏
张力维,李勇鹏,姚 瑶,梁 优,杜 丽
(南阳师范学院 生命科学与技术学院,河南 南阳 473000)
香樟延伸因子EF1a基因片段的克隆及表达分析
张力维,李勇鹏,姚 瑶,梁 优,杜 丽
(南阳师范学院 生命科学与技术学院,河南 南阳 473000)
通过同源克隆的方法从香樟Cinnamomum camphora中分离到一个编码延伸因子EF1a的cDNA序列片段。根据已经在GenBank中登录的其它物种的EF1a基因的保守序列设计一对兼并引物,通过反转录PCR技术获得一个1 297 bp的基因片段。同源比对表明:CcEF1a与GenBank中注册的其它植物EF1a基因核苷酸序列的相似性均在80%以上,氨基酸序列的相似性高达96%以上。将所获得基因片段命名为CcEF1a并提交GenBank,登录号为KM086740。实时定量PCR结果显示,CcEF1a在低温胁迫下的叶片中表达量稳定,可以在实时定量PCR分析中作为内参基因。
香樟;延伸因子;EF1a基因片段; 克隆; 表达分析
香 樟Cinnamomum camphora, 樟 科Lauraceae,常绿乔木,是贵重家具、高级建筑、造船和雕刻等的理想用材,近年来,香樟在园林绿化中的应用越来越多,成为城市重要的园林绿化树种[1]。香樟在抗有害气体中也发挥重要作用,是城区抗有害气体的骨干树种[2]。但是耐寒性差这一不良性状严重限制了香樟的地理分布及其在园林绿化中的应用,香樟抗寒新品种的培育有利于香樟的推广,丰富北方冬天的园林植物组合。研究香樟抗寒相关基因从而通过基因工程技术获得抗寒性增强的香樟新品种是一个有效的途径,基因工程育种与传统育种相比具有较强的目的性、育种周期短和克服远缘杂交不亲和的优点。
近 年 来,CBF/DREB1(CRT/DRE binding factor 1)转录因子被认为是对植物冷驯化机制具有重要调节作用的一类基因,该基因能感受上游传递的低温信号,在低温诱导下迅速合成,并在转录水平上调控多个抗逆基因,从而提高植物的耐寒性。周静等[3]获得了蓝花楹JaCBF1基因的拟南芥转基因植株,经耐寒性分析,结果显示转基因拟南芥抗寒性较对照组增强,因而对该家族基因的克隆和分子重组为实现育种目标提供了新思路。CBF/DREB1途径广泛存在于各个物种中,近年来科学家们相继从平榛、巨桉、梅花等木本植物中分离到CBF/DREB1家族基因[4-6],本实验室也已经从香樟中克隆到2个CBF/DREB1家族基因(数据未公布),要深入研究这些基因的抗寒相关性及表达模式,一般通过实时定量PCR手段对基因的表达量进行定量分析。
实时定量PCR(realtime quantitative reverse transcription polymerase chain reaction )即RT-qPCR由于其再现性和灵敏度(reproducibility and sensitivity)高而成为对基因的表达量进行定量的最佳选择,并广泛应用于植物的表达分析中[7-8]。RT-qPCR技术需要合适的内参基因才能使对基因的定量分析有意义[9]。研究抗寒相关基因的表达模式需要一个在低温条件下表达量相对恒定的持家基因作为内参,从而实现该基因的定量和标准化。近年来关于各个物种实时定量PCR分析中内参基因筛选的报道很多,如牡丹、马铃薯、柳枝稷等[10-12],传统持家基因像甘油醛-3-磷酸脱氢酶(GAPDH)、肌动蛋白(ACT)和延伸因子(EF1a)因为其组成型表达而被广泛认为是比较可靠的内 参 基 因[13]。EF1a基 因(elongation factor 1 alpha)编码真核生物翻译延伸因子(eEF1)的一个亚基,在细胞中表达丰都较高,真核生物的延伸因子是一类在蛋白质合成中发挥作用的组成型蛋白,帮助肽链的延伸,其中eEF1由4个亚基构成[14]。EF1a被认为在黄瓜、矮牵牛和大白菜中最为稳定的内参基因[15-17]。很多研究者在研究抗寒相关基因DREB1家族基因的表达模式中应用了EF1a基因作为内参基因[6,18],因而,本研究也首选EF1a基因作为后续实时定量PCR工作的内参基因。然而,目前为止,国内外还没有见到香樟延伸因子EF1a相关文献的报道,GenBank中也没有与香樟延伸因子EF1a有关的基因序列登录。本研究从香樟中克隆了EF1a基因片段,并设计了RT-qPCR引物,利用实时定量PCR技术,研究了其在低温胁迫0~72 h下叶片中的表达量变化,并利用软件对该基因的稳定性做了评估。研究结果可以为开展香樟抗寒或其它相关基因的表达分析和调控等研究工作提供内参基因,也为其他科研工作者们研究香樟提供了优良的候选内参基因,同时为其他物种EF1a基因的克隆提供参考。
1 材料和方法
1.1 材 料
1.1.1 实验材料
供试材料为实验室保存的香樟无性系组培苗,继代培养基为 MS+2.0 g·L-1BA+0.5 g·L-12,4-D,生根培养基为MS基本培养基,其中蔗糖30 g·L-1,琼脂8.0 g·L-1,培养温度 (25±1) ℃,光照强度 2 500 lx,光照/黑暗时间16/8 h。待材料在生根培养基中生长至有5~7片叶片时,对材料进行低温处理,处理温度为4 ℃,分别于0、0.5、1、2、4、6、12、24、48和72 h 随机取叶片,所有的材料平行取4个重复,收获后立即用液氮速冻,-80℃保存直至RNA提取;大肠杆菌TOP10原菌株为实验室保存。
1.1.2 主要试剂
EasyTaq DNA Polymerase购自北京全式金(Transgen)生物技术有限公司;dNTP Mixture、DL2000 DNA Maker、PMD19-T Vector、PrimeScriptII 1st strand cDNA Synthesis Kit购自大连宝生物工程(TaKaRa)有限公司;CTAB植物基因组DNA快速提取试剂盒、EASYspin Plus植物RNA快速提取试剂盒购自北京艾德莱(Aidlab)生物科技有限公司;琼脂糖凝胶纯化回收试剂盒购自苏州康宁(Axygen)生命科学有限公司;0.2 mL EU材质薄壁8连管购自荷兰Bioplastics公司;RT-qPCR反应试剂FastStart Universal SYBR Green Master(ROX)购自德国罗氏(Roche)公司;PCR引物合成及测序工作由上海生工生物工程(Sangon Biotech)股份有限公司完成;其他生化试剂采用国产或者进口分析纯。
1.2 方 法
1.2.1 总RNA的提取
根据北京艾德莱EASYspin Plus植物RNA快速提取试剂盒的操作说明提取香樟叶片总RNA。
1.2.2 香樟延伸因子CcEF1a基因片段的克隆
通过比对已发表的EF1a基因cDNA序列,根据保守区域利用Primer5.0软件设计一对简并引物EF1aF和EF1aR,其中EF1aF,ATGGGYAARGARAAGWYYCAYATCAAC;EF1aR,GAAATCCACATCTGYTGGAA。 用 该对引物扩增香樟EF1a基因片段,PCR产物利用1.0%琼脂糖凝胶电泳分析,利用Axygen公司的DNA回收试剂盒回收目的条带,连接至TaKaRa公司的PMD19-T载体上,16 ℃连接过夜,转化大肠杆菌感受态TOP10,菌落PCR检测,挑选阳性克隆送往上海生工测序。
1.2.3 香樟延伸因子CcEF1a基因的生物信息学分析
将测序获得的香樟EF1a基因片段的核苷酸序 列, 在 http://www.ncbi.nlm.nih.gov网 站 上 使用BLAST工具进行基因序列相似性分析,利用DNAMAN和CLUSTALX等生物软件进行序列的比对和翻译,将推导的氨基酸与已公布的其他物种EF1a先利用生物分析软件CLUSTALX进行多序列比对,比对结果经Bioedit剪切对齐后用MEGA5.03进行系统进化树的建立。
1.2.4 香樟延伸因子CcEF1a基因低温胁迫下的荧光定量表达分析
取约200株长势一致的香樟组培苗置于人工气候箱进行4 ℃低温处理,分别于0、0.5、1、2、4、6、12、24、48和72 h取叶片作为实验材料,利用北京艾德莱EASYspin Plus植物RNA快速提取试剂盒提取香樟叶片总RNA,以TaKaRa公司的PrimeScriptII 1st strand cDNA Synthesis Kit进行cDNA第一链的反转录,起始RNA模板量为2 μg,反转录产物稀释10倍作为RT-qPCR反应模板。根据所获得香樟CcEF1a基因的核苷酸序列,按照荧光定量PCR引物的要求设计一对RT-qPCR引物EF1a-RTF和EF1a-RTR,其序列分别为:EF1a-RTF,TCCAAGGCACGGTATGAT;EF1a-RTR,CCTGAAGAGGGAGACGAA。扩增片段大小为232 bp,扩增产物经琼脂糖凝胶电泳检测条带单一、不含引物二聚体,并且测序确保扩增产物的特异性。RT-qPCR反应采用SYBR Green I法,利用罗氏(Roche)公司的FastStart Universal SYBR Green Master(ROX),在伯乐(BIO RAD)CFX96 实时定量PCR仪上进行。反应体系(10 μL):ddH2O 3.4 μL,2×Power Taq PCR MasterMix 5 μL,EF1a-RTF 和 EF1a-RTR(10 μmol) 各 0.3 μL,cDNA 1 μL。反应条件:94 ℃预变性5 min;94 ℃变性15 s,60 ℃退火30 s,72 ℃延伸1 min,共40次循环;溶解曲线在65~95℃之间,每0.05 s升高0.5 ℃。实验每一样品重复3次,每个实验设3个平行重复。基因表达分析软件为Bio-Rad CFX manager 2.0,依据各样品的CT值,利用Excell 2007和BestKeeper进行数据分析。BestKeeper程序依据各个样品的CT值用Excell软件计算标准偏差SD,SD值越小,基因的稳定性越好,若SD>1,则认为该基因不稳定[19-20]。
2 结果与分析
2.1 总RNA的提取及检测
以香樟叶片为材料提取叶片总RNA,琼脂糖凝胶电泳检测,结果(见图1A)显示,28 S和18 S条带清晰,未见拖带现象,表明实验提取的RNA完整性良好,可以用于反转录。
2.2 香樟延伸因子CcEF1a基因片段的克隆
2.2.1 PCR扩增产物的鉴定
根据TaKaRa公司PrimeScriptII 1st strand cDNA Synthesis Kit的操作说明,将2 μg总RNA反转录合成cDNA第一链,作为PCR反应模板,以EF1aF、EF1aR进行PCR扩增。反应产物用1.0%的琼脂糖凝胶电泳检测,在1 000 bp与2 000 bp之间有一条亮带(见图1B),与预期片段大小一致,可能是香樟EF1a基因片段,进一步测序鉴定。

图1 香樟叶片总RNA琼脂糖凝胶电泳检测(A)和香樟EF1a基因PCR扩增结果(B)Fig.1 Agarose gel electrophoresis of total RNA extracted from the C. camphora leaves and ampli fication results of CcEF1a by PCR
2.2.2 阳性克隆的鉴定
将回收纯化的目的片段连接到克隆载体pMD19-T上,转化大肠杆菌TOP10感受态细胞,随机挑选10株抗性菌进行菌落PCR检测,菌落PCR扩增结果显示阳性菌落PCR扩增产物比目的基因片段PCR产物稍大(见图2),挑取保存在平板上的相应阳性菌进行扩大培养,并测序。
2.3 香樟延伸因子CcEF1a基因的生物信息学分析
测序结果经分析并去除两端引物序列,获得了1个大小为1297 bp的cDNA序列片段,编码432个氨基酸(见图3)。将获得片段的核苷酸序列和推导氨基酸序列在NCBI中进行BLAST分析,结果显示该基因片段的核苷酸序列与BLAST结果的其它物种EF1a基因的核苷酸序列的相似度均在80%以上,其中与梅花EF1a相似性最高,相似度为87%;氨基酸序列的相似性均在96%以上,与大戟科木薯、蓖麻、小油桐以及棕榈科的油棕的EF1a氨基酸序列的相似度均达到98%。推测本实验所获得的基因片段是香樟EF1a基因片段,命名为CcEF1a,提交Genbank,登录号为KM086740。
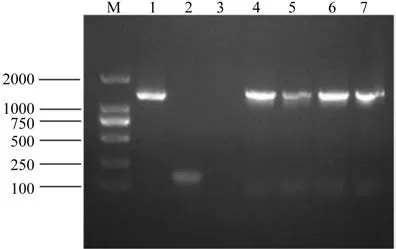
图2 部分菌落PCR筛选阳性重组子结果Fig.2 Positive recombinant screened from partial colony by PCR
将得到的香樟EF1a基因片段所编码蛋白质的氨基酸序列NCBI中已经登录的其它物种EF1a的氨基酸序列利用DNAMAN进行多序列比对,结果(见图4)显示,EF1a基因所编码蛋白质的氨基酸序列相当保守,进一步证明延伸因子EF1a是高度保守的蛋白质。
此外,利用MEGA5.03软件对香樟EF1a推导的氨基酸序列与橡胶树、毛果杨、油棕、烟草等14个物种的EF1a蛋白进行系统进化树分析,结果(见图5)表明,香樟与大戟科木薯、橡胶以及棕榈科油棕聚为一支,表明它们在氨基酸水平上亲缘关系较近;单子叶植物玉米与水稻聚为一支,表明它们起源相同;此外,单子叶植物油棕与双子叶植物橡胶树聚为一支,说明EF1a基因在单子叶植物和双子叶植物中的进化过程相互交叉进行,这也充分说明EF1a基因在不同物种行使相近的功能。通过一系列生物信息学分析,本研究所获得基因片段应为香樟延伸因子基因(CcEF1a)。
2.4 CcEF1a基因在低温胁迫下香樟叶片中的荧光定量表达分析
采用RT-qPCR技术进行CcEF1a基因在低温胁迫下香樟叶片中的表达分析,根据各个样品的CT值研究该基因的表达量及稳定性,其中基因的表达量与CT值呈反比例关系[21]。CT值是指反应体系累计足够的扩增产物,至可以产生可检测的荧光信号时的循环数,主要由扩增反应体系中模板的初始浓度决定,如果初始模板浓度低,需要较多的扩增循环才能产生足够的荧光信号;反之,只需要较少的扩增循环就可以累计足够的产物,从而产生高过背景的荧光信号。根据各个样品的CT平均值,利用Excell 2007作图,结果见图6。由图6可以看出,各个样品的CT平均值虽然有一定起伏,但差异并不明显;BestKeeper程序依据样品CT值进行计算,结果显示,各个样品的CT平均值为21.95~22.98,所得标准偏差SD为0.23,远小于1。综上可以得出,CcEF1a基因在低温胁迫下表达量基本恒定,可以作为实时定量PCR分析的内参基因。

图3 CcEF1a基因片段核苷酸序列与推导的对应氨基酸序列Fig.3 Nucleotide sequence and deduced amino acid sequenceof CcEF1a gene fragment from C. camphora
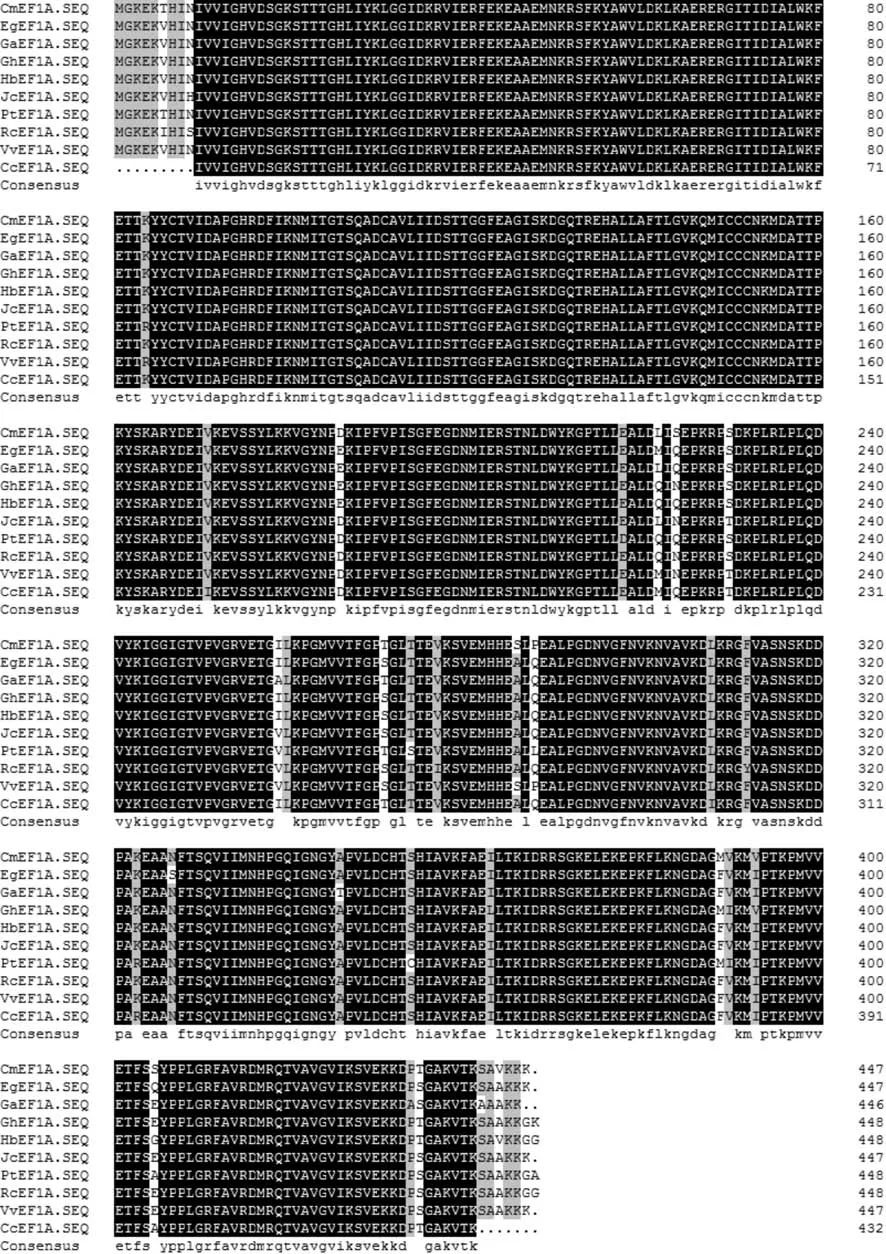
图4 香樟与其他植物EF1a氨基酸序列的多序列比对Fig.4 Multiple alignment of EF1a proteins from C. camphora and other plants
3 讨论与结论
真核生物延伸因子EF1a(elongation factor 1 alpha)与原核生物的EF-Tu 类似,是翻译延伸因子(elongation factor,EF)家族当中一个非常重要的成员,主要在蛋白质翻译过程中将氨酰tRNA 运输到核糖体A 位点,参与肽键的形成及多肽链的延伸反应,属于G 蛋白家族,并且它的基因序列及表达调控在真核生物中高度保守[22]。由于对樟科植物的相关研究十分有限,本实验在进行引物设计时,并未能找到樟科植物EF1a基因的核苷酸序列;而根据木本植物杨树、李树和蔷薇的EF1a基因的核苷酸序列的保守区域设计的1对兼并引物,以香樟cDNA为模板扩增即可获得约1 300 bp的特异条带,充分证明其核苷酸序列是高度保守的。将该基因片段推导的氨基酸序列在NCBI网站进行BLASTP分析,发现与大戟科植物的EF1a(木薯,O49169.1)氨基酸序列相似度最高,达98%。EF1a蛋白的高保守性表明EF1a基因对各个物种生存有着相当重要的意义并发挥相似的作用。EF1a基因由于的表达量高并且相对稳定,因而经常在基因的表达分析中被用作内参基因。RT-qPCR的准确性很大程度上取决于定量分析方法以及内参基因表达的稳定性[23],因此一个稳定表达的内参基因对于RT-qPCR必不可少。大量研究者报道,同一种内参基因,在不同实验条件下或不同物种中,甚至同一有机体的不同组织部位的表达模式也并非是一成不变的[24-25],因此,必须对内参基因在相应实验条件下的稳定性进行评估。

图5 香樟CcEF1a基因推导的氨基酸序列与其他植物EF1a氨基酸序列的系统进化树Fig.5 Phylogentic tree based on CcEF1a amino acid sequences from C. camphora and other plants

图6 CcEF1a基因在低温处理下的香樟叶片中的表达量Fig.6 Expression levels of CcEF1a gene in C. camphora under low temperature treaments
本实验通过同源克隆的方法结合RT-PCR技术从香樟叶片cDNA中克隆到了香樟EF1a基因的cDNA片段,利用生物信息学手段分析所推导氨基酸的序列特征,结果表明CcEF1a含有EF1a的典型结构域,属于Ras-like-GTPase 超家族。初步分析所获得片段与其他物种EF1a基因的同源性及进化关系,表明EF1a基因在分子进化水平上较为保守,也证明本实验获得的基因片段确实为香樟EF1a基因。本研究虽然并未克隆到香樟EF1a完整的cDNA全长,但是并不影响其作为内参基因的使用,通过设计RT-qPCR引物,利用RT-qPCR技术对其在低温处理下香樟叶片中的稳定性进行了分析,结果表明,CcEF1a基因在低温胁迫下表达量稳定,期望能成为香樟相关基因表达研究的内参基因。
[1]杜 丽, 庞振凌, 周 索, 等. 香樟胚性愈伤组织遗传转化体系建立[J].林业科学, 2008, 44(4): 54-59.
[2]廖建军, 李 晟, 杨喜生, 等. 城市园林树种规划探讨—以衡阳为例[J].中南林业科技大学学报, 2009, 29(3): 162-166.
[3]周 静, 张 炜, 李大明, 等. 蓝花楹JaCBF1转录因子片段的序列分析及耐寒性功能验证[J]. 林业科学研究, 2013, 26(4):414-419.
[4]陈 新, 王贵禧, 梁丽松, 等. 平榛冷适应相关基因CBF的克隆及时空表达特性分析[J]. 林业科学, 2012, 48(1): 167-172.
[5]王京京, 童再康, 黄程前, 等. 巨桉EgrCBF1和EgrCBF2基因的克隆和胁迫响应表达分析[J]. 林业科学, 2012, 48(10):41-48.
[6]Guo C, Zhang J Q, Peng T,et al.Structural and expression analyses of three PmCBFs fromPrunus mume[J]. Biologia Plantarum, 2014, 58 (2): 247-255.
[7]Selim M, Legay S, Berkelmann-Lo¨hnertz B,et al. Identi fication of suitable reference genes for real-time RT-PCR normalization in the grapevine-downy mildew pathosystem[J]. Plant Cell Rep.,2012, 31: 205-216.
[8]李 高, 杨 杞, 张 烨, 等. 柠条锦鸡儿肉桂酰辅酶A 还原酶基因克隆和分析[J]. 中国生物工程杂志, 2014, 34(1):50-56.
[9]Maksup S, Supaibulwatana K, Selvaraj G. High-quality reference genes for quantifying the transcriptional responses ofOryza sativa(ssp. indica and japonica) to abiotic stress conditions[J].Chin. Sci. Bull, 2013, 58: 1919-1930.
[10]王彦杰, 董 丽, 张 超, 等. 牡丹实时定量PCR分析中内参基因的选择[J]. 农业生物技术学报, 2012, 20(5): 521-528.
[11]Lopez-Pardo R, Ruiz de Galarreta J I, Ritter E. Selection of housekeeping genes for qRT-PCR analysis in potato tubers under cold stress[J]. Mol. Breeding, 2013,31: 39-45.
[12]蒋晓梅, 张新全, 严海东, 等.柳枝稷根组织实时定量PCR分析中内参基因的选择[J]. 农业生物技术学报, 2014, 22(1):55-63.
[13]Lin L Y, Han X J, Chen Y C,et al. Identi fication of appropriate reference genes for normalizing transcript expression by quantitative real-time PCR inLitsea cubeba[J]. Mol. Genet.Genomics, 2013, 288: 727-737.
[14]覃迎姿, 黄先益, 叶兴枝, 等. 植物延伸因子eEf1A研究进展[J].广西农业科学, 2009, 40 (5): 472-477.
[15]Wan H, Zhao Z, Qian C,et al. Selection of appropriate reference genes for gene expression studies by quan-titative real-time polymerase chain reaction in cucumber[J]. Anal. Biochem., 2010,399: 257-261.
[16]Mallona I, Lischewski S, Weiss J,et al. Validation of reference genes for quantitative real-time PCR during leaf and flower development inPetunia hybrida[J]. BMC Plant Biol., 2010, 10:4-14.
[17]Qi J, Yu S, Zhang F,et al. Reference gene selection for real-time quantitative polymerase chain reaction of mRNA transcript levels in Chinese cabbage (Brassica rapassp.pekinensis) [J]. Plant Mol. Biol. Rep., 2010, 28: 597-604.
[18]Kudo K, Oi T, Uno Y. Functional characterization and expression pro filing of a DREB2-type gene from lettuce (Lactuca sativa) [J].Plant Cell Tiss Organ Cult., 2014, 116(1): 97-109.
[19]Mehta R, Birerdinc A, Hossain N,et al.Validation of endogenous reference genes for qRTPCR analysis of human visceral adipose samples[J].BMC Mol. Biol., 2010, 11(39):1471-2199.
[20]苏晓娟,樊保国,袁丽钗,等. 实时荧光定量PCR分析中毛果杨内参基因的筛选和验证[J]. 植物学报,2013,48(5):507-518.
[21]肖 政, 李纪元, 范正琪, 等. 荔波连蕊茶肌动蛋白基因全长cDNA的克隆及表达分析[J]. 林业科学研究, 2014, 27(3):374-380.
[22]周 峰, 刘 燕, 马永贵, 等. 真核翻译延伸因子1A蛋白家族功能位点的进化踪迹分析[J].中国生物化学与分子生物学报,2013, 29( 8): 773-782.
[23]Kundu A, Patel A, Pal A. Defining reference genes for qPCR normalization to study biotic and abiotic stress responses in Vigna mungo[J]. Plant Cell Rep., 2013, 32:1647-1658.
[24]Li Q, Fan C M, Zhang X M,et al. Validation of reference genes for real-time quantitative PCR normalization in soybean developmental and germinating seeds [J]. Plant Cell Rep., 2012,31:1789-1798.
[25]Chaouachi M, Alaya A, Ali I H,et al. Development of real-time PCR method for the detection and the quantification of a new endogenous reference gene in sugar beet “Beta vulgarisL.”;GMO application[J]. Plant Cell Rep., 2013, 32:117-128.
Cloning and expression analysis ofEF1agene fragment of elongation factor fromCinnamomum camphora
ZHANG Li-wei, LI Yong-peng, YAO Yao, LIANG You, DU Li
(School of Life Science and Technology, Nangyang Normal University, Nanyang 473000, Henan, China)
A cDNA fragment encoding a putative protein of elongat ion factor 1 alpha (EF1a) was firstly isolated fromCinnamomum camphoraby using homology cloning, A pair of degenerate primers was designed based on the conserved sequences of otherEF1agenes from the different species from GenBank, and a fragment of 1 297 bp was obtained through Reverse Transcription Polymerase Chain Reaction (RT-PCR) techniques. The results of homologous alignment show thatCcEF1ashared over 80% nucleotide sequence identity and over 96% amino acid sequence identity withEF1agenes in other plants that had been submitted to GenBank. The cDNA fragment sequence was namedCcEF1aand registered in GenBank with accession number KM086740. According to the results of realtime quantitative PCR, the mRNA level of CcEF1a in leaves under chilling stress was identical. It could be used as an interrnal control in real-time quantitative PCR.
Cinnamomum camphora; elongation factor;EF1agene fragment; cloning; expression analysis
S792.23;Q786
A
1673-923X(2015)05-0122-07
10.14067/j.cnki.1673-923x.2015.05.021
2014-11-05
国家自然科学基金项目(31100511);河南省高校青年骨干教师计划项目(2010GGJS-161);南阳师范学院博士科研启动项目(nynu200746);2015年度研究生创新项目(2015CX008)
张力维,硕士研究生
杜 丽,副教授,博士;E-mail:dldldlucky@163.com
张力维,李勇鹏,姚 瑶,等. 香樟延伸因子EF1a基因片段的克隆及表达分析[J].中南林业科技大学学报,2015,35(5):122-128.
[本文编校:谢荣秀]
