利用CRISPR/Cas9n系统敲除人源SNF5基因
2015-11-29孙琰刘超毛赟赟王玺
孙琰,刘超,毛赟赟,王玺
1.天津医科大学 基础医学院,天津 300070;2.军事医学科学院 生物工程研究所,北京 100071
成簇的规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)是细菌的一种获得性免疫系统,用以保护细菌和古细菌不受病毒侵害。细菌被病毒侵染后,CRISPR关联蛋白(CRISPR-associated proteins,Cas)位点会编码多个核酸酶和解旋酶,形成Cas 蛋白复合物,对入侵的病毒DNA 进行切割,产生的新片段会整合到CRISPR重复序列中,从而对该病毒产生特异性免疫记忆[1-2]。当细菌再次遭到该病毒入侵时,便会转录出与入侵病毒DNA 序列相匹配的小分子RNA,Cas 蛋白复合物利用这些RNA 去切割外源病毒DNA,导致病毒不能在细菌内进行复制。CRISPR/Cas9 系统是基于细菌和古细菌的这种免疫防御机制而改造的一种基因组编辑技术[3],利用人工设计的单向导RNA(single-guide RNA,sgRNA)介导外源Cas9 蛋白与靶基因结合,切割带有5'-NGG 的间隔相邻基序(protospacer adjacent motifs,PAM),从而实现对靶基因DNA的特异性切割[4]。
表观遗传在癌症发生发展中有重要作用,染色质重塑是表观遗传的重要机制之一,染色质重塑复合物通过水解ATP 产生的能量来改变染色质结构,进而调节基因表达。染色质重塑复合物是较大的多亚基复合物,已知有SWI/SNF、ISWI、CHD 和IN080四大类。其中SWI/SNF是目前研究最多的,SNF5是SWI/SNF 染色质重塑复合物的核心组分之一,高度保守。研究发现SNF5是一个强抑癌基因,在许多恶性肿瘤中突变或缺失,如横纹肌样瘤、神经鞘瘤、淋巴瘤、乳腺癌、上皮样肉瘤及其他软组织的恶性肿瘤等[5-12]。目前有关SNF5的抗肿瘤机制研究不多。我们设计了针对SNF5基因的sgRNA,构建了SNF5基因的CRISPR/Cas9n 表达载体,旨在通过敲除SNF5基因为深入研究SNF5的抗肿瘤机制奠定基础。
1 材料与方法
1.1 材料
人胚肾293T 细胞、大肠杆菌DH5α、pX461 和pX462 质粒(本研究室保存);BbsⅠ、磷酸酯酶(Fermentas 公司);T4DNA 连接酶、T4多聚核苷酸激酶(NEB公司);胶回收试剂盒、无内毒素质粒提取试剂盒(天根生化科技有限公司);DMEM 高糖培养基(Gibco 公 司);胎牛血 清(PAN 公 司);LipofectAMINE 2000(Invitrogen 公 司);抗SNF5 抗 体(Abcam 公司);β-actin 抗体、辣根过氧化物酶偶联的羊抗鼠IgG(Santa Cruz公司)。
1.2 sgRNA靶点选择及其寡核苷酸链设计
应用CRISPR 在线设计工具(http://crispr.mit.edu/),在人源SNF5基因外显子1 处设计了一对sgRNA。设计过程如下:①从SNF5基因起始密码子处开始寻找长度约200 bp、富含NGG且位于同一外显子上的序列,将序列提交至该网站;②根据提交后网站显示的结果,选择分数较高的一对序列,自动生成的靶序列都是5'到3'共23 bp;③去掉序列3'端的NGG,并在5'端加上酶切位点CACC,其反向互补序列的5'端添加酶切位点AAAC,以便2条互补的寡核苷酸序列退火后,能够与经BbsⅠ酶切的质粒的粘性末端互补;如果靶序列5'端第一个碱基不是G,那么应先在5'端添加一个G,再加上酶切位点CACC,相应地其反向互补序列的3'端再增加一个C。
1.3 SNF5sgRNA表达载体的构建
首先用T4多聚核苷酸激酶对合成的2组引物分别进行磷酸化和退火,退火后的sgRNA 分别插入经BbsⅠ酶切的表达载体pSpCas9n(BB)-2A-GFP(简称pX461)和pSpCas9n(BB)-2A-Puro(简称pX462)中,然后将连接产物转化大肠杆菌DH5α感受态细胞,挑取单克隆菌落进行测序鉴定(由北京六合华大基因科技股份有限公司完成),测序引物为5'-ATACGATACAAGGCTGTTAGAGAGATA-3'。
1.4 细胞培养和转染
用含10%胎牛血清的DMEM 培养基培养293T细胞,将处于对数期生长的细胞接种于12 孔板,接种量以转染时细胞密度达到90%为宜,培养24 h后进行转染。转染过程按照LipofectAMINE 2000的说明书进行。
1.5 Westen印迹分析
转染293T 细胞后24~48 h 收集细胞,加入SDS上样缓冲液,煮沸10 min,离心后取上清液进行SDS-PAGE;电泳完毕转移至硝酸纤维素膜上,用5%脱脂奶粉室温封闭1 h,然后加入用5%脱脂奶粉稀释的SNF5 抗体,4℃过夜,TBST 洗膜3 次,每次7 min;加入用5%脱脂奶粉稀释的辣根过氧化物酶偶联的羊抗鼠IgG,室温轻摇1 h,TBST洗膜3次,每次7 min;显色后压片显影。
2 结果
2.1 sgRNA靶点及寡核苷酸序列设计
将选择的人源SNF5基因序列在线提交上述网站后,在外显子1 处设计了一对sgRNA(图1),网站生成的序列为5'-GACGGCGAGTTCTACATGATCG G-3'和5'-CTTCACGGGCTTCTGCCCGAAGG-3'。根据生成的靶序列,设计了4 条寡核苷酸序列(表1)。将合成的2 组寡核苷酸序列分别退火,退火后插入经BbsⅠ酶切的表达载体pX461和pX462,构建的表达载体分别为pX461-hSNF5sgRNA1、pX461-hSNF5sgRNA2、pX462-hSNF5sgRNA1 和pX462-hSNF5sgRNA2。
2.2 SNF5sgRNA表达载体构建的测序结果
将构建的4 个表达载体分别测序,结果显示sgRNA 靶序列均正确插入pX461 和pX462,证明SNF5sgRNA表达载体构建成功(图2)。
2.3 sgRNA对SNF5表达的影响
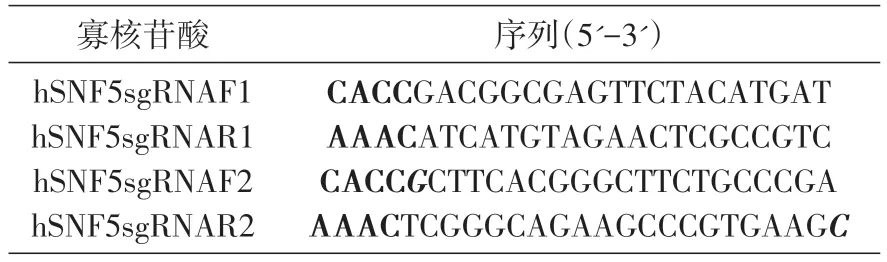
表1 合成的寡核苷酸序列

图1 一对靶向SNF5第1个外显子的sgRNA
将pX461、pX461-hSNF5sgRNA1 和pX461-hSNF5sgRNA2、pX462、pX462-hSNF5sgRNA1 和pX462-hSNF5sgRNA2 分别转染293T 细胞,转染后24、48 h 收集细胞裂解液进行Western 印迹。结果显示,与转染pX461 空载体组比,转染pX461-hSNF5sgRNA1 和pX461-hSNF5sgRNA2 后24 h,SNF5表达水平明显降低,转染后48 h,SNF5表达水平开始恢复;与转染pX462 空载体组比,转染pX462-hSNF5sgRNA1 和pX462-hSNF5sgRNA2 后24 h,SNF5 表达水平没有明显改变,转染后48 h,SNF5 表达水平显著降低(图3)。此现象表明,不同标签载体表达的sgRNA 起效时间并不一致,需要根据实验的具体情况进行摸索。实验结果表明,我们构建的针对人源SNF5基因的CRISPR/Cas9n系统能够敲除细胞中SNF5基因的表达。
3 讨论
Cas9 核酸内切酶有HNH 和RuvC 两个活性位点,切割后产生双链DNA断裂。基因组DNA通过启动同源重组和非同源末端连接机制进行修复,可能产生基因突变、插入或缺失[13]。CRISPR/Cas9的特异性与跟sgRNA 配对且靠近PAM 处的7~12 个碱基有关,因此CRISPR/Cas9 的靶向特异性非常低[14],会造成研究结果的不确定性及工作量的大量增加,这种脱靶效应会导致基因组中癌基因激活、其他序列突变等不良后果,给临床应用带来风险。张峰课题组将Cas9 的RuvC 催化位点进行突变,形成产生单链切口的核酸酶Cas9 nickase(Cas9n),通过一对sgRNA 引导来实现靶向双链切割,此方法能显著降低(约1/50~1/1500)CRISPR/Cas9系统在细胞系中的脱靶效应[15]。
本研究采用的即为上述CRISPR-Cas9n系统,本系统所用质粒是CRISPR 与Cas9n 的共质粒表达系统,可以提高基因敲除效率。在设计sgRNA 靶序列时应注意:靶序列应位于基因同一个外显子内,不能在外显子与内含子交界处,且靶序列应位于基因的CDS 区;如果外显子内CDS 序列少于60 bp,则没必要再进行设计,即使设计了效果也不佳;选择的靶序列离ATG 越近越好。在线提交所选基因序列后,得出的评分结果是针对脱靶效应的,分值越高脱靶效应越小。
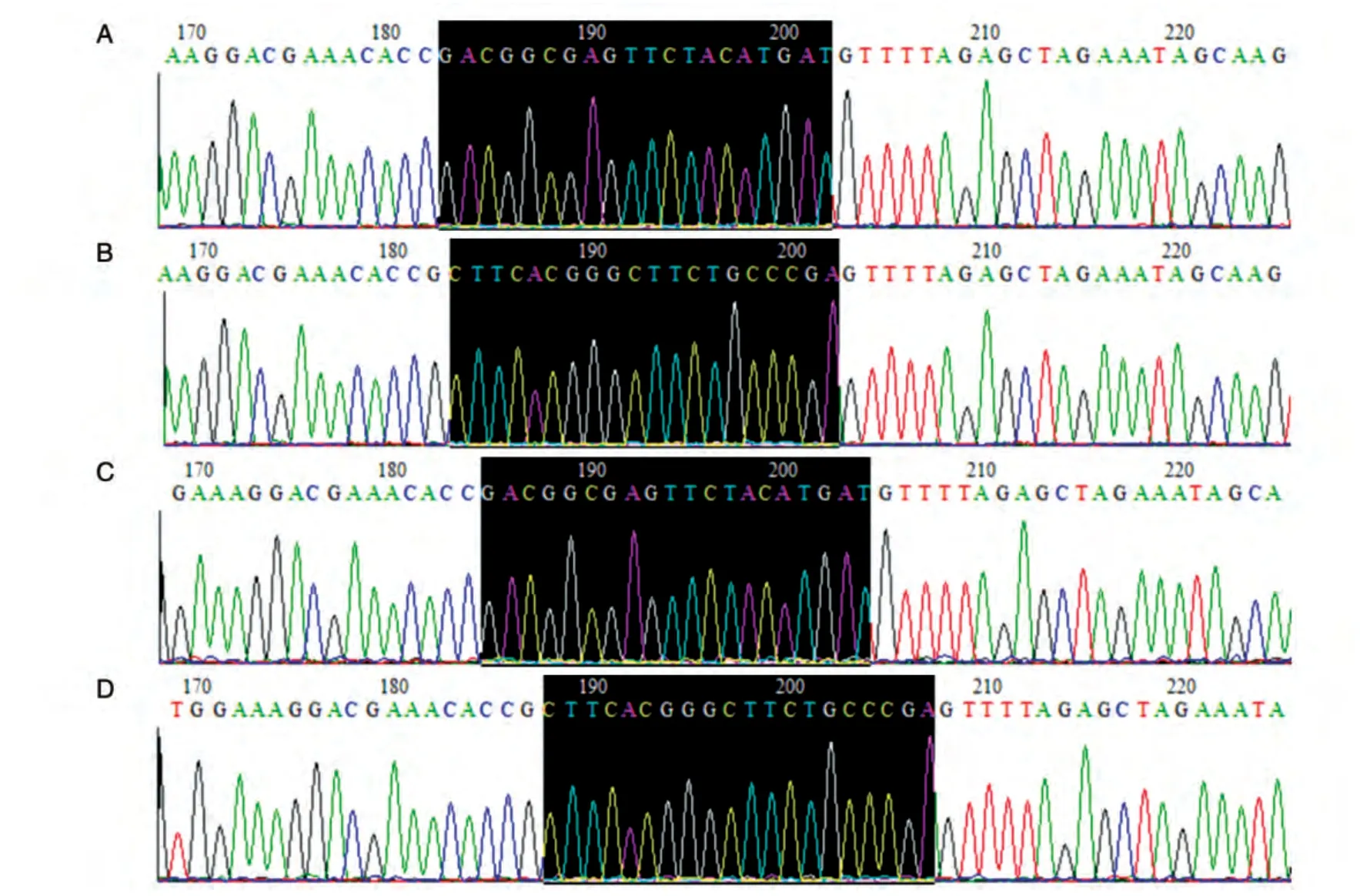
图2 构建的SNF5sgRNA表达载体测序峰图
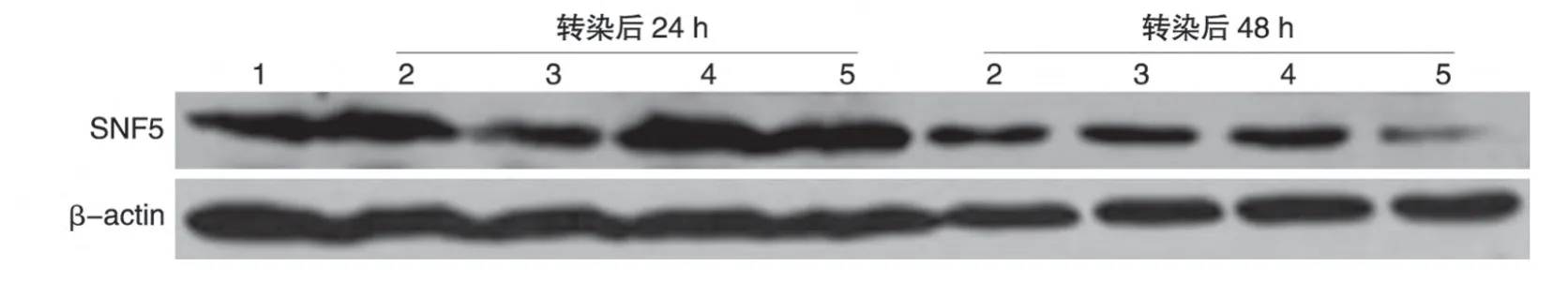
图3 Westren印迹检测SNF5蛋白表达水平
我们发现,293T 细胞内转入SNF5sgRNA 后,与转染空载体组比,SNF5 表达水平显著降低。这种SNF5表达水平改变反映的是SNF5在细胞群体水平敲除后的变化,CRISPR/Cas9 系统切割基因组DNA后,细胞会启动同源重组和非同源末端连接机制进行修复,这些修复是随机的,不同细胞个体突变修复后的基因型各不相同。本实验中我们选择了表达绿色荧光蛋白的pX461和表达嘌呤霉素的pX462两种载体,可以借助绿色荧光蛋白进行流式分选或借助嘌呤霉素进行抗性筛选,实现目的细胞的聚集,便于通过有限稀释法获取SNF5基因稳定敲除的单克隆细胞,通过测序即可得到基因型确定的敲除细胞株。
研究发现,SNF5基因的缺失是非典型畸胎瘤、横纹肌样瘤、黑色素瘤患者诊断的一个很好的标记物[16-18]。Roberts 等发现,SNF5条件性失活小鼠在平均11周内发生T细胞淋巴瘤,明显快于p53、RB等重要肿瘤抑制基因[19]。然而SNF5突变后如何导致染色质结构的改变,如何影响基因的转录,进而如何导致癌症的具体机制在很大程度上仍然不清楚。
综上,我们利用CRISPR/Cas9 技术建立了SNF5基因靶向敲除系统,通过敲除细胞株中SNF5基因,为研究其在疾病中的功能和具体作用机制提供了很好的工具。
[1]Chylinski K,Le Rhun A,Charpentier E.The tracrRNA and Cas9 families of type II CRISPRCas immunity systems[J].RNA Biol,2013,10(5):726-737.
[2]Jinek M,Chylinski K,Fonfara I,et al.A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J].Science,2012,337(6096):816-821.
[3]Sapranauskas R,Gasiunas G,Fremaux C,et al.The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli[J].Nucleic Acids Res,2011,39(21):9275-9282.
[4]Mali P,Esvelt K M,Church G M.Cas9 as a versatile tool for engineering biology[J].Nat Methods,2013,10(10):957-963.
[5]Brenca M,Rossi S,Lorenzetto E,et al.SMARCB1/INI1 genetic inactivation is responsible for tumorigenic properties of epithelioid sarcoma cell line VAESBJ[J].Mol Cancer Ther,2013,12(6):1060-1072.
[6]Carter J M,O'Hara C,Dundas G,et al.Epithelioid malig-nant peripheral nerve sheath tumor arising in a schwannoma,in a patient with“neuroblastoma-like”schwannomatosis and a novel germline SMARCB1 mutation[J].Am J Surg Pathol,2012,36(1):154-160.
[7]Hulsebos T J,Plomp A S,Wolterman R A,et al.Germline mutation of INI1/SMARCB1 in familial schwannomatosis[J].Am J Hum Genet,2007,80(4):805-810.
[8]Kohashi K,Oda Y,Yamamoto H,et al.SMARCB1/INI1 protein expression in round cell soft tissue sarcomas associated with chromosomal translocations involving EWS:a special reference to SMARCB1/INI1 negative variant extraskeletal myxoid chondrosarcoma[J].Am J Surg Pathol,2008,32(8):1168-1174.
[9]Mimori K,Inoue H,Shiraishi T,et al.A single-nucleotide polymorphism of SMARCB1 in human breast cancers[J].Genomics,2002,80(3):254-258.
[10]Rizzo D,Freneaux P,Brisse H,et al.SMARCB1 deficiency in tumors from the peripheral nervous system:a link between schwannomas and rhabdoid tumors[J]? Am J Surg Pathol,2012,36(7):964-972.
[11]Sullivan L M,Folpe A L,Pawel B R,et al.Epithelioid sarcoma is associated with a high percentage of SMARCB1 deletions[J].Mod Pathol,2013,26(3):385-392.
[12]Yuge M,Nagai H,Uchida T,et al.HSNF5/INI1 gene mutations in lymphoid malignancy[J].Cancer Genet Cytogenet,2000,122(1):37-42.
[13]Jinek M,Chylinski K,Fonfara I,et al.A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J].Science,2012,337(6096):816-821.
[14]Pattanayak V,Lin S,Guilinger J P,et al.High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity[J].Nat Biotechnol,2013,31(9):839-843.
[15]Ran F A,Hsu P D,Lin C Y,et al.Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J].Cell,2013,154(6):1380-1389.
[16]Vu-Han T L,Fruhwald M C,Hasselblatt M,et al.Identifying molecular markers for the sensitive detection of residual atypical teratoid rhabdoid tumor cells[J].Cancer Genet,2014,207(9):390-397.
[17]Kerl K,Oyen F,Leuschner I,et al.Detection of SMARCB1 loss in ascites cells in the diagnosis of an abdominal rhabdoid tumor[J].Pediatr Blood Cancer,2015,62(5):897-900.
[18]Stockman D L,Curry J L,Torres-Cabala C A,et al.Use of clinical next-generation sequencing to identified melanomas harboring SMARCB1 mutations[J].J Cutan Pathol,2015,42(5):308-317.
[19]Roberts C W,Leroux M M,Fleming M D,et al.Highly penetrant,rapid tumorigenesis through conditional inversion of the tumor suppressor gene Snf5[J].Cancer Cell,2002,2(5):415-425.
