PCR-DGGE研究青海农村户用沼气池微生物群落结构
2015-11-19陈来生刘德立华中师范大学生命科学学院遗传调控与整合生物学湖北省重点实验室湖北武汉40079青海省高原作物种质资源创新与利用国家重点实验室培育基地青海西宁810016青海省农林科学院青海省蔬菜遗传与生理重点实验室青海西宁810016
韩 睿,陈来生,李 莉,刘德立(1.华中师范大学生命科学学院,遗传调控与整合生物学湖北省重点实验室,湖北 武汉 40079;2.青海省高原作物种质资源创新与利用国家重点实验室培育基地,青海 西宁 810016;.青海省农林科学院,青海省蔬菜遗传与生理重点实验室,青海 西宁 810016)
PCR-DGGE研究青海农村户用沼气池微生物群落结构
韩 睿1,2,3,陈来生2,3,李 莉3,刘德立1*(1.华中师范大学生命科学学院,遗传调控与整合生物学湖北省重点实验室,湖北 武汉 430079;2.青海省高原作物种质资源创新与利用国家重点实验室培育基地,青海 西宁 810016;3.青海省农林科学院,青海省蔬菜遗传与生理重点实验室,青海 西宁 810016)
采用变性梯度凝胶电泳(DGGE)分析技术,对青海农村户用沼气池内细菌及古菌群落结构进行分析.结果表明,沼气池含有丰富的细菌及古菌类群,且泥样间的细菌和古菌的群落结构存在差异.4个户用沼气池发酵系统中,细菌分属于5个门,优势类群为拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria).在属分类水平上,属于24个类群,最优势类群为理研菌科佩特里单胞菌属(Petrimonas)、假单胞菌属(Pseudomona)、泰氏菌属(Tissierella)和梭菌属(Clostridium).同时,沼气池发酵系统中古菌主要包括热丝菌属(Thermofilum)、甲烷短杆菌属(Methanobrevibacter)、甲烷囊菌属(Methanoculleus)、产甲烷菌属(Methanogenium),其中,Methanobrevibacter 和Methanogenium是沼气池内最优势的产甲烷菌.说明沼气池的产甲烷途径主要是氢营养代谢类型,水解、酸化过程主要由来自动物消化系统内的细菌完成.
PCR-DGGE;青海;户用沼气池;微生物群落结构
沼气是一种极具应用前景的可再生清洁生物能源[1-2].发展沼气是解决我国农村能源危机、改善农村生态环境、实现农业可持续发展的有效途径[3].高效稳定的微生物生态系统是沼气产气率的保证,是沼气池稳定运行的关键.Zakrzewski等[4]对生物沼气池的样品进行研究,揭示了产甲烷菌群的代谢途径,认为在以后的生产中可以通过对微生物群落的监控优化生产;刘卫国等[5]对沼气生物脱硫系统中微生物群落构成进行分析,确定了系统中的优势菌种.研究并优化沼气发酵过程中的微生物群落结构,有利于充分发挥各种群的生态功能,使沼气发酵在粪污治理中得到科学合理应用[6].
青海省地处青藏高原东北部,平均海拔在4000m以上,具有气温低、昼夜温差大、太阳辐射强等特点.全省年平均气温在-4℃~8℃之间,冬季气温低(平均气温为-8℃~-18℃)且持续时间长.自2003年以来,青海省在农村沼气建设国债项目的支持下,在农村沼气建设和利用方面取得了一定成效.然而,由于青海省所处的地理位置及气候条件,导致其沼气技术开发落后,存在着诸多问题.进入冬季(当年11月~翌年4月),寒冷的气温导致户用沼气池内发酵微生物生理活性降低,产生的微量沼气不够应用,不能维持发酵系统自身的运转,甚至出现供气中断现象(用半年停半年).沼气池的闲置浪费,使农民建沼气池、用沼气的积极性降低,也在一定程度上影响了青海沼气的推广普及[9].据统计,到目前为止青海省绝大多数户用沼气池在冬季没有得到很好利用.由于农村户用沼气池发酵系统是一个复杂的微生物生态系统,微生物群落结构决定其生态功能.因此,深入解析青海沼气发酵生态系统的微生物群落结构特征,是指导青海沼气生产的关键.
绝大多数沼气发酵微生物难以培养,无法采用传统纯培养方法来研究微生物群落结构和代谢关系[6-9].变性梯度凝胶电泳技术(DGGE)以DNA分子为研究对象,能有效分析复杂微生物群落结构及其多样性,无需培养微生物,操作简单快速,已被广泛用于各种环境中微生物生态的研究[10-12].迄今为止,对寒区农村户用沼气发酵微生物群落结构与功能的研究相对较少,而青海地区的此类研究还未见报道.本研究利用PCR-DGGE技术,以青海省乐都县蒲台乡李家台村的沼气池为研究对象,探明青海农村户用沼气池发酵微生物的群落结构特征,为进一步揭示微生物群落结构与功能的关系,改进青海省沼气发酵工艺、增加沼气池产气量、实现农村户用沼气发酵的稳定运行提供有价值的参考.
1 材料与方法
1.1 户用沼气池
青海省乐都县蒲台乡李家台村(102°27′22″E,36°21′14″N,海拔2223.27m~2228.26m)有农户79 户.经初步调查分析,共建有沼气池65座,33.8%能够正常运行,运行效率不高,但相对青海省而言,该村沼气池的运行情况是比较好的.所建沼气池构造统一,为圆形,容积为8m3,采用“一池三改”的模式,将沼气池与改厕所、改暖棚猪舍与改厨房相结合,通过庭院的合理设计,粪便直接入池.发酵液pH值为7.0~7.5、主要投入原料为人畜的粪便.
1.2 样品采集
选取利用率相对较高,且能够在冬季供气不中断的4户沼气池底部取样.采样时间为2014年6月.样品采集后4℃封存,带回实验室后立即对样品进行处理.分别编号为LD1、LD2、LD3、LD4,其运行状况等基本情况见表1.
1.3 基因组DNA提取
将污泥样品置于2mL离心管中,用土壤基因组快速抽提试剂盒(上海生工)提取基因组DNA,经1%的琼脂糖凝胶电泳检测后保存于4℃备用.
1.4 PCR扩增
将污泥样品置于2mL离心管中,用土壤基因组快速抽提试剂盒(上海生工)提取基因组DNA,经1%的琼脂糖凝胶电泳检测后保存于4℃备用.
1.4.1 细菌PCR扩增 所用引物为细菌16S rDNA V3高变区357F-GC(5'-CGCCCGCCGCGCCCCGCGCCCGGCCCGCCGCCCCCGCCCCC CTACGGGAGGCAGCAG-3')和518R(5'-ATTACCGCGGCTGCTGG-3'),反应体系为50μL总体积,ddH2O 41.25μL,10×Buffer(含2.0mmol/L MgCl2)5μL,dNTP(10mmol/L)1μL,357F-GC(10μmol/L)1μL,518R(10μmol/L)1μL,Taq酶(5U/μL)0.25μL,模板DNA 0.5μL.反应程序:94℃预变性4min;30个循环(94℃ 30s;56℃ 1min;72℃ 30s);72℃延伸7min.
1.4.2 古菌PCR扩增 巢式PCR扩增,利用两轮引物.第一轮引物为ARCH46F(5'-YTAAGCCATGCRAGT-3')和ARCH1017R(5'-GGCCATGCACCWCCTCTC-3'),第二轮为PARCH344F(5'-CGCCCGCCGCGCGCGGCGGGCGGGGCG GGGGCACGGGGGGACGGGGHGCAGCAGGCGCGA-3')和UNIV 522R(5'-GWATTACCGCGGCKGCTG-3').反应体系为50μL总体积,ddH2O 41.25μL,10×Buffer(含2.0mmol/L MgCl2)5μL,dNTP(10mmol/L)1μL,上下游引物(10μmol/L)各1 μL,Taq酶(5U/μL)0.25μL,模板DNA 0.5μL.反应程序:94℃预变性4min;30个循环(94℃ 30s;56℃1min;72℃ 30s);72℃延伸7min.细菌和古菌PCR产物均用1.5%琼脂糖凝胶电泳检测.
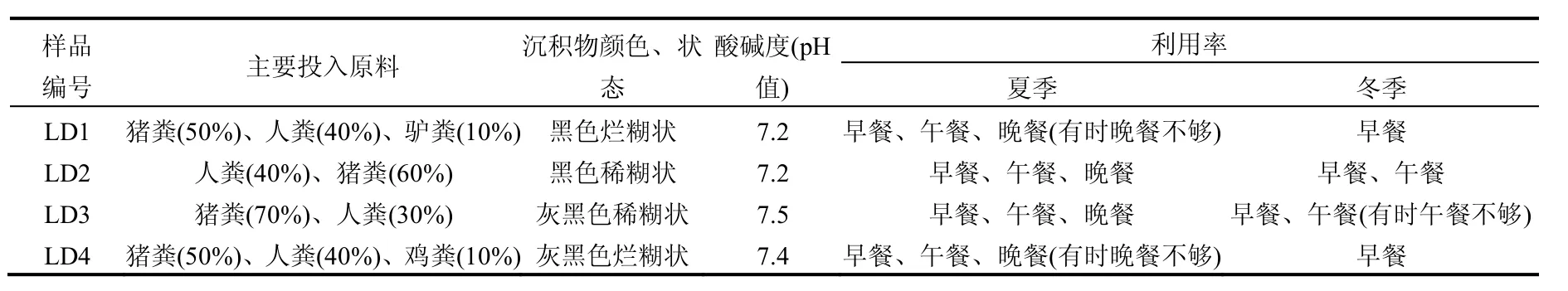
表1 户用沼气池基本情况调查表Table 1 Basic information of household biogas digesters
1.5 变性梯度凝胶电泳(DGGE)及数据分析
采用D-Code突变检测系统对样品进行DGGE分析.所用的聚丙烯酰胺凝胶浓度为8%(丙稀酰胺:双丙稀酰胺=37.5︰1),变性剂浓度从30%~60%(100%的变性剂为7mol/L尿素,40%甲酰胺).在60V电压下,60℃恒温,1×TAE中电泳16h.电泳完毕后,用超纯水冲洗胶,然后将胶放进含EB的染液中,置于摇床上染色30min后,凝胶成像系统进行拍照.
DGGE图谱用Quantity One 软件进行数字化处理,并进行聚类分析(UPGMA法).利用香农多样性指数(H′)、辛普森指数(D)和均匀度(E)等指标对各样品微生物的多样性进行评价.
1.6 DGGE条带的测序与系统发育树分析
选取较有代表性的条带,用洁净的手术刀片将目标DGGE条带完整的切下并装入1.5mL离心管中,用SanPrep柱式DNA胶回收试剂盒(上海生工)的方法进行回收.取适量做模板进行PCR扩增,除引物不带GC“夹板”外,扩增条件及步骤同1.4.将扩增DNA片段回收、纯化,分别与pMD18-T(TaKaRa)载体连接,热激转化E.coli DH5α感受态细胞.筛选阳性克隆子送上海生物工程技术服务有限公司测序.
序列测序后去除载体序列,与GenBank(http://www.ncbi.nl.nih.gov/BLAST/)及RDP(http://rdp.cme.msu.edu)数据库中的已有序列进行比对分析,确定其在原核生物界的分类地位,采用Clustal X 2.11和MEGA 5.2软件构建系统发育树.
2 结果与分析
2.1 青海沼气泥样DGGE图谱及多样性分析
以提取的4个泥样的基因组DNA为模板,对其进行16S rDNA片段的PCR扩增,将扩增产物进行变性梯度凝胶电泳,然后用凝胶成像分析系统和Quantity one分析软件对DGGE胶图进行条带识别和图谱分析.由图1和图2可见,细菌和古菌的DGGE图谱条带均清晰,显示了各户用沼气池泥样中微生物的DNA指纹.图谱中条带的数目、强度及迁移位置反映了泥样中微生物类群的多样性及丰度.不同位置的条带代表不同微生物类群,而同一水平位置的条带则代表同种微生物菌群,其条带的明暗程度则反映该微生物类群在不同样品中的相对丰度,得到的条带越多说明样品的多样性越高.由图1和表2可知,青海农村户用沼气池每个泥样DGGE图谱中的条带总数均在26条以上,D显示均在0.95以上,且LD4样品最高,这一规律同样可从H′中反映出来,说明泥样中细菌种类十分丰富.另外,4个泥样的E有一定差异,反映出泥样间细菌群落结构的差异.由图2和表2可知,每个泥样DGGE图谱中条带总数均在15条以上,H′与D的变化规律相同,且D显示均在0.91以上,说明也含有较为丰富的古菌类群.泥样样品中,LD1和LD4的S相近,但其他特征不同,说明泥样间古菌的多样性不同.同样,4个泥样的E差异较大,也说明泥样间的古菌群落结构的差异很大.
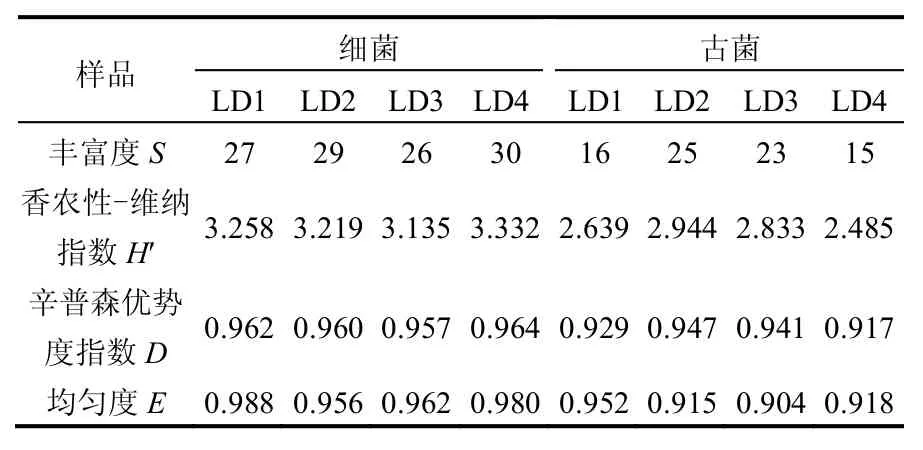
表2 不同样品中微生物特征指数计算结果Table 2 Microbial characteristics index of different samples from biogas slurry
2.2 不同泥样微生物群落的相似性分析
利用Quantity One提供的UPGMA法,在DGGE图谱的基础上,绘出微生物群落的聚类分析图(图3).从图3A可知,4个样品中细菌群落组成总体分为2大类,LD1、LD2、LD4的细菌聚为一类,LD3单独聚为另一类.对于前者,从DGGE图谱(图1)可以看出,LD1和LD2在带型、条带数目、条带亮度及共有条带数目上最近,与其余2个样有差别;从聚类分析图同样可以看出LD1和LD2相似性最高,因此又可以归为一亚类,而LD4则单独成为另一亚类,说明LD1和LD2泥样间的微生物最相似.从图3B可知,4个样品中古菌群落组成总体也可分为2大类,LD2、LD3、LD4的古菌聚为一类,LD1单独聚为一类.对于第一大类,LD2和LD3相似性最高,又可以归为一亚类,而LD4则单独成为另一亚类,这一结论同样可由DGGE图谱得出(图2).
2.3 沼气微生物菌群基因测序及系统发育分析
分别选取图1和图2中具代表性的条带,分别对编号为LDB-1~LDB-30的30个细菌条带和编号为LDA-1~LDA-25的25个古菌条带进行克隆测序,将得到的所有序列与GenBank及RDP数据库中的已有序列进行比对分析,获得各个序列的同源性信息(表3和表4),构建了系统发育树(图4和图5).由图4和表3可知,青海农村户用沼气池细菌的多样性十分丰富,其序列与近缘种的相似度从91.2%~100%,片段长度从169bp~196bp,分属于5个门,包括:放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)及疣微菌门(Verrucomicrobia),优势类群为Bacteroidetes、Firmicutes和Proteobacteria.在属分类水平属于24个微生物类群,包括:短小杆菌属(Curtobacterium)、紫单胞菌科帕鲁迪菌属(Paludibacter)、海滑菌科(Marinilabiaceae)未分类属、紫单胞菌科Proteiniphilum属(Proteiniphilum)、理研菌属(Rikenella)、理研菌科佩特里单胞菌属(Petrimonas)、理研菌科(Rikenellaceae)未分类属、噬胞菌属(Cytophaga)、噬几丁质杆菌科菌(Chitinophagaceae)、阿托波叶柄菌属(Atopostipes)、牛瘤胃菌属(Proteiniclasticum)、泰氏菌属(Tissierella)、粪球菌属(Coprococcus)、梭菌属(Clostridium)、梭菌科古根海姆艾拉菌属(Guggenheimella)、瘤胃菌科(Ruminococcaceae)未分类属、梭菌目未定科(Clostridiales_Incertae Sedis XI)未分类属、丹毒丝菌属(Erysipelothrix)、布鲁氏菌属(Brucella)、根瘤菌属(Rhizobium)、德沃斯氏菌属(Devosia)、苍白杆菌属(Ochrobactrum)、假单胞菌属(Pseudomonas)、疣微菌属(Verrucomicrobium).其中以Petrimonas、Pseudomonas、Tissierella和Clostridium为优势类群.
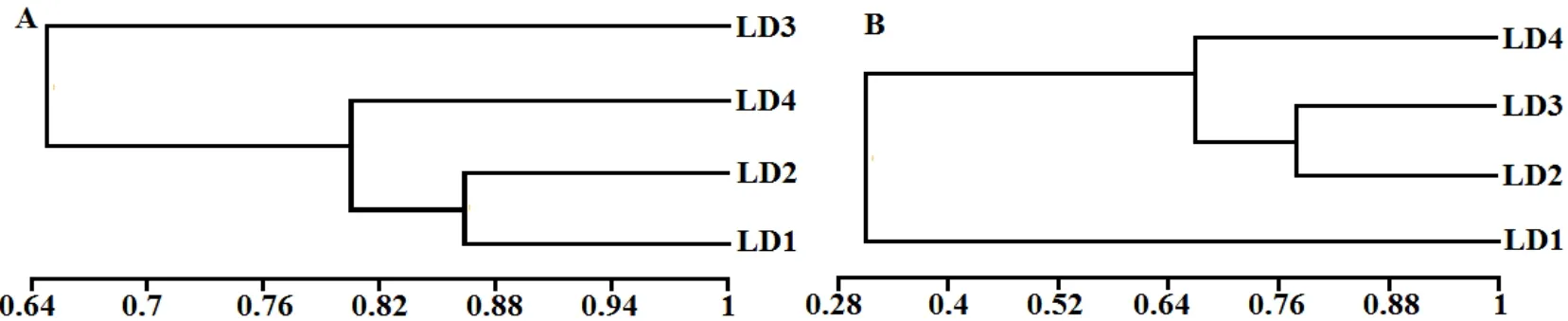
图3 沼气池泥样微生物群落的DGGE聚类分析Fig.3 The clustering dendrogram of the microbial community in biogas slurry by DGGE analysis
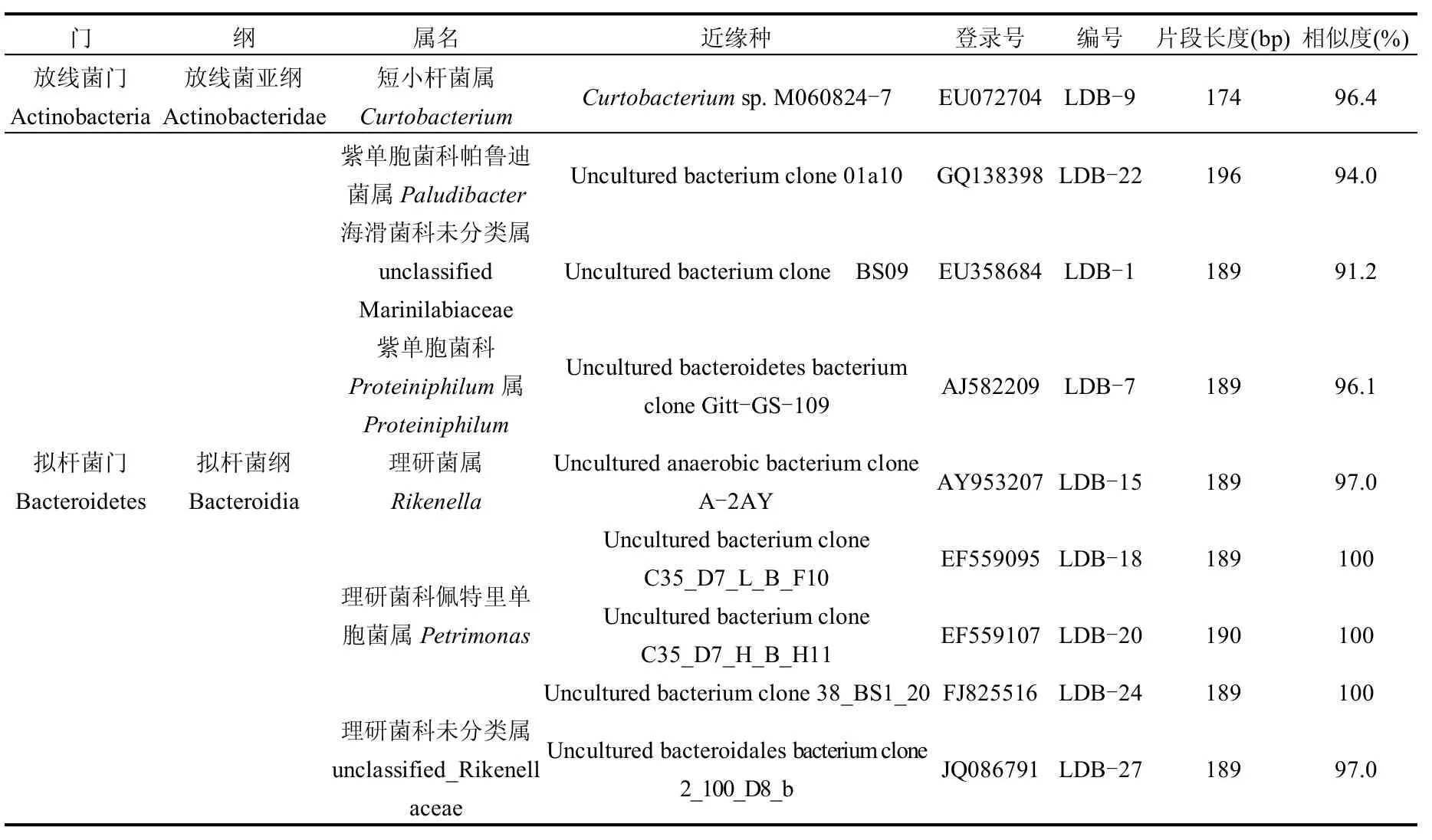
表3 细菌群落16S rDNA片断测序分析Table 3 Sequence analysis of 16S rDNA phrases in bacterial community

续表3
由图5和表4可知,古菌的片段长度为137~150bp,其序列与近缘种的相似度从80.1%~97.0%,分属于3个目,即热变形菌目(Thermoproteales)、甲烷杆菌目(Methanobacteriales)和甲烷微菌目(Methanomicrobiales),核酸序列比对结果显示,青海农村户用沼气池古菌包括热丝菌属(Thermofilum)、甲烷短杆菌属(Methanobrevibacter)、甲烷囊菌属(Methanoculleus)和产甲烷菌属(Methanogenium),其中,Methanobrevibacter和Methanogenium是沼气池内最优势的古菌类群.
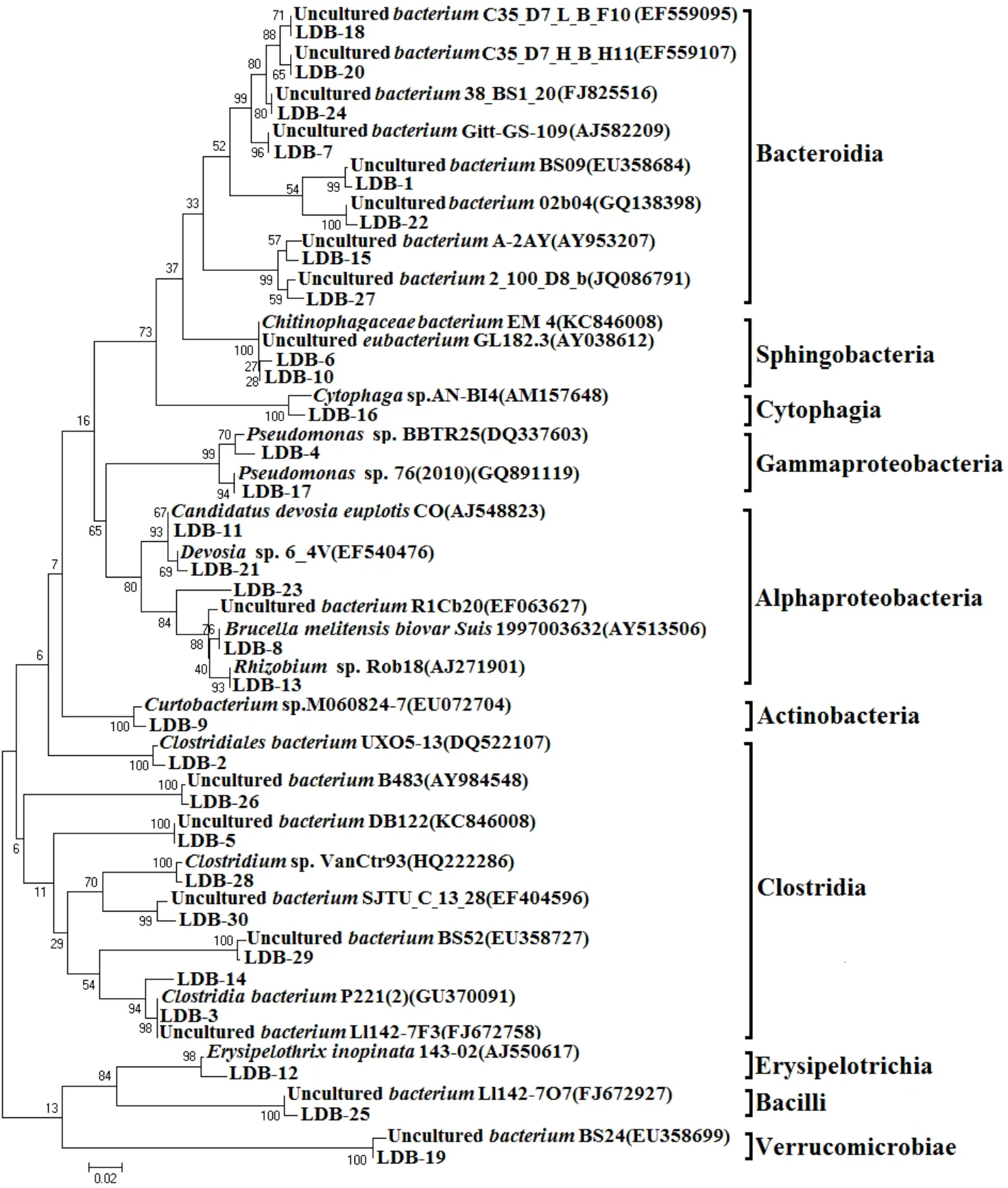
图4 沼气样品细菌系统发育分析Fig.4 Phylogenetic analysis of various bacteria in the samples from biogas slurry

表4 古菌群落16S rDNA片断测序分析Table 4 Sequence analysis of 16S rDNA phrases in archaeal community
续表4
3 讨论
理论上,DGGE图谱中每一条带代表一种微生物类群,带型、条带数目及亮度与微生物的种类及相对含量成正比[17-18].通过对比DGGE 图谱中不同泳道各条带的有无和明暗程度,即可判断各样品的微生物群落结构及种群丰富度状况.由图1和图2可知,细菌和古菌的DGGE图谱条带非常清晰,各样品的PCR产物获得了预期的分离效果,不同沼气池泥样细菌和古菌群落在带型、条带数目及亮度上均存在一定差异.青海农村户用沼气池每个泥样的DGGE图谱中,细菌的条带数在26条以上,古菌在14条以上,说明泥样中含有丰富的细菌及古菌菌群.不同泥样所对应的泳道有许多相同的条带,如LDB-3、LDB-4、LDB-5、LDB-16、LDB-18、LDB-19、LDB-20、LDB-27、LDB-28 和LDA-13、LDA-15、LDA-16、LDA-19,表明其所代表的微生物类群为青海4个农村户用沼气池所共有,但这些共有条带的亮度不尽相同,如LDB-3仅在LD4泳道非常亮,说明LDB-3条带对应的细菌群落在LD4的泥样中最为丰富.同样,LDA-19在LD2泳道非常亮,在LD3泳道比较亮,也说明了其对应的古菌群落的丰富度.从表2可以看出,每个样沼气池泥样中细菌和古菌特征指数不同,其中S、H′和D的特征变化规律相同,反映出其微生物的多样性和丰富性.细菌泥样间的D、S相差不大,而H′有一定差异.LD4样品的H′是泥样中最高的,为3.332,LD3则是最低的,为3.135.古菌泥样间的H′、D和S均有一定差异.LD2 和LD3的H′、D相近,LD1和LD4相近,且前两者指数要远高于后两者.其中,LD2最高,分别为2.944和0.947;而LD4分别仅有2.485和 0.917.LD1和LD4的S相近,但这2个泥样的S与其他2个相差很大.E是指微生物群落中各菌群的丰富度或重要类群的均匀程度,E不同即说明样品间微生物群落结构不同.细菌和古菌各泥样的E均不同,但细菌LD1和LD4的E相差不大,古菌LD2和LD4相差不大.
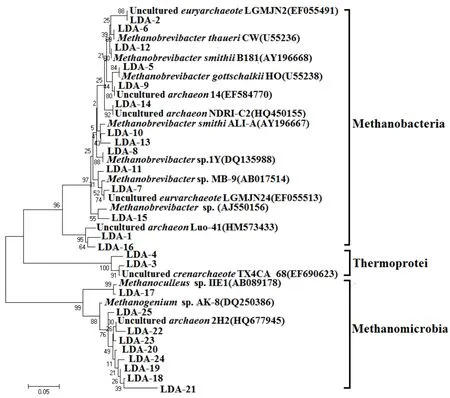
图5 沼气样品古菌系统发育分析Fig.5 Phylogenetic analysis of different archaea in the samples from biogas slurry
分别对30个细菌条带和25个古菌条带克隆测序分析,结果表明青海农村户用沼气池细菌多样性很高,优势类群主要为拟杆菌门、厚壁菌门和变形菌门.拟杆菌门和厚壁菌门主要分离自动物消化道、厌氧消化污泥、粪便、废水处理反应器等厌氧环境,前者有降解大分子碳水化合物产酸的功能,后者主要进行纤维素降解生成小分子物质[15-17].变形菌门主要分离自粪便、厌氧活性污泥及土壤等环境中,具有水解作用[18].在沼气发酵系统中曾多次发现这3门细菌是占主导地位的细菌类群,只是优势程度不同.Klocke等[19]发现青贮甜菜沼气系统中优势细菌主要归属于厚壁菌门、变形菌门和拟杆菌门.张蕾等[20]对秸秆沼气发酵反应器中微生物进行研究发现,细菌分属 9个门,大部分为水解与发酵性细菌,其中厚壁菌门、变形菌门和拟杆菌门为主要的优势种群.袁月祥等[21]研究发现,在以玉米秸秆厌氧发酵生产沼气的过程中,发酵系统含有20余个门的细菌,丰度最多的细菌为拟杆菌门、变形菌门和厚壁菌门.赵光等[22]对北方规模最大的海林农场沼气池内细菌及产甲烷古菌群落结构进行解析,发现细菌的优势菌群有厚壁菌门、拟杆菌门及变形菌门.在属分类水平上,青海农村户用沼气池主要优势类群为理研菌科佩特里单胞菌属、假单胞菌属、泰氏菌属和梭菌属,均属动物消化系统内常见的微生物类群,说明这些沼气池底物水解及产酸过程的主要功能微生物来源于动物消化系统内的细菌类群.同时,所含的古菌中,最优势的类群为甲烷短杆菌属和产甲烷菌属,该结果与前人的报道相符.郎会花等[23]利用分子生物学方法构建16S rDNA基因文库研究鸡粪沼气池发酵液中产甲烷菌的菌群结构,结果发现产甲烷菌属占总数的92%.Weiss等[24]利用DGGE技术分析市政沼气工程中古菌多样性后得知古菌包括甲烷短杆菌属、甲烷囊菌属、甲烷球形菌属、甲烷微菌属和未培养甲烷微菌目.甲烷短杆菌属常见于动物消化道、腐败木质、厌氧活性污泥及厌氧消化反应器等,产甲烷菌属则常见于海底沉积物、温泉、厌氧消化反应器、土壤、腐败木质、厌氧活性污泥等,两者均为氢营养型产甲烷古菌,可利用氢气或甲酸盐,不能利用乙酸和甲基胺[25-27].由此可知,青海农村户用沼气池的主要代谢方式为:来自动物消化系统内的细菌通过水解、酸化过程为产甲烷菌提供H2等营养物质,产甲烷菌则利用这些底物还原CO2生成甲烷,这一代谢途径维持着该发酵系统的稳定运行.
寒冷的气候条件是导致青海沼气池利用率低、产气少、供气中断等问题的重要原因之一.为解决沼气池越冬问题,青海省通常采用畜禽舍下建造沼气池、覆盖塑料薄膜、池体进出口加盖、太阳能加热、利用辅助燃烧式建造技术等办法来给沼气池增温[28-29].但冬季气温过低,以上措施不能克服温度对沼气池的影响,必需从发酵过程中的微生物入手.由于沼气发酵是一个涉及多种微生物的复杂的厌氧消化过程,对各类微生物在其中所起作用的研究非常重要,现已有通过研究微生物来解决低温对沼气产气不足问题的成功报道.姚利等[30]筛选出了具有耐低温和分解秸秆性能的菌种,并经复合成为沼气发酵菌剂.分别在采取不同保温升温措施的沼气池内添加该菌剂,对其冬季应用效果进行验证试验.结果表明,各组试验池产气量提高38.2%~45.5%,平均日产气量可达0.47~0.80m3.说明对沼气池采取保温升温措施并添加高效微生物菌剂,可显著提高沼气池的水温和产气量,确保沼气池在冬季正常使用.黄江丽等[31]选育出了低温沼气发酵功能菌群,沼气发酵实验结果表明,该菌群采用后最高日产气量均明显高于单独采用野生菌群和厌氧颗粒污泥菌群,具有改善低温沼气发酵性能.以此为基础研制的低温沼气发酵促进剂,在13℃下可使产气率平均提高46.6%,10℃下产气率平均提高41.1%.目前我们通过PCR-DGGE技术明确了青海农村户用沼气池6月份的微生物群落结构,正在开展温度变化对沼气池微生物群落结构变化的研究,旨在了解沼气发酵过程中微生物群落随温度变化的动态变化特征,进而优化群落结构、调整群落功能,为增加青海农村户用沼气池产气量,提高沼气池利用效率奠定基础.
4 结论
4.1 利用PCR-DGGE技术对青海省乐都县蒲台乡李家台村不同沼气池内细菌及古菌群落结构进行研究,结果显示,青海农村户用沼气池含有丰富的细菌及古菌类群,且泥样间的细菌和古菌的群落结构存在差异.4个户用沼气池发酵系统中,细菌分属于5个门,优势类群为拟杆菌门、厚壁菌门和变形菌门.在属分类水平上,属于24个类群,最优势类群为理研菌科佩特里单胞菌属、假单胞菌属、泰氏菌属和梭菌属,均为动物消化系统内常见的细菌类群.
4.2 4个户用沼气池发酵系统中,古菌主要包括热丝菌属、甲烷短杆菌属、甲烷囊菌属和产甲烷菌属,其中,甲烷短杆菌属和产甲烷菌属是最优势的产甲烷菌类群,均为氢营养型产甲烷古菌.说明沼气池的产甲烷途径主要是氢营养代谢类型,水解、酸化过程主要由来自动物消化系统内的细菌完成.
[1]Bachmaier H,Effenberger M,Gronauer A,et al.Changes in greenhouse gas balance and resource demand of biogas plants insouthern Germany after a period of three years[J].Waste Management and Research,2012,33(2):8-11.
[2]Feng Y Z,Guo Y,Yang G H,et al.Household biogas development in rural China: On policy support and othermacro sustainable condi-tions[J].Renewable and Sustainable Energy Reviews,2012,16:5617-5624.
[3]师晓爽,刘德立,郎志宏,等.PCR-DGGE 技术在农村户用沼气发酵微生物研究中的初步应用[J].山东师范大学学报,2007,22(2):120-122.
[4]Zakrzewski M,Goesmann A,Jaenicke S,et al.Profiling of the metabolically active community from a product ion scale biogas plant by means of high-through put metatran-scriptome sequencing[J].Biotechnol.,2012,158(4):248-258.
[5]刘卫国,梁存珍,马 梦,等.PCR-DGGE技术分析沼气生物脱硫系统中的优势菌种[J].中国沼气,2013,31(4):3-6.
[6]林代炎,叶美锋,吴飞龙,等.规模化养猪场粪污循环利用技术集成与模式构建研究[J].农业环境科学学报,2010,29(2):386-391.
[7]彭发基,田德宁.高寒地区一种新型辅助燃烧式沼气池简介[J].中国沼气,2011,29(2):41-42.
[8]Pathak H,Jain N,Bhatia A,et al.Global warming mitigation potential of biogas plants in India[J].Environmental Monitoring and Assessment,2009,157(1/4):407-418.
[9]Hofman-Bang J,Zheng D,Westermann P,et al.Molecular ecology of anaerobic reactor systems[J].Advances in Biochemical Engineering/Biotechnology,2003,81:151-203.
[10]满 鹏,齐鸿雁,呼 庆,等.利用PCR-DGGE分析未开发油气田地表微生物群落结构[J].环境科学,2012,33(1):305-313.
[11]Feligini M,Panelli S,Buffoni J N,et al.Identification of microbiota present on the surface of Taleggio cheese using PCR-DGGE and RAPD-PCR[J].Food Science,2012,77(11):609-615.
[12]Li S,Sun L,Wu H,et al.The intestinal microbial diversity in mud crab(Scylla paramamosain)as determined by PCR-DGGE and clone library analysis[J].Applied Microbiology and Biotechnology,2012,113(6):1341-1351.
[13]Konstantinov S R,Zhu W Y,Williams B A,et al.Effect of fermentable carbohydrates on piglet faecal bacterial communities as revealed by denaturing gradient gel electrophoresis analysis of 16S ribosomal DNA[J].FEMS Microbiology Ecology,2003,43(2):225-235.
[14]Muyzer G,Smalla K.Application of denaturing gradient gel electrophoresis(DGGE)and temperature gradient gel electrophoresis(TGGE)in microbial ecology[J].Antonie Van Leeuwenhoek International Journal of General and Molecular Microbiology,1998,73(1):127-141.
[15]Ye N F,Lü F,Shao L M,et al.Bacterial community dynamics and product distribution during pH-adjusted fermentation ofvegetablewastes[J].Journal of Applied Microbiology,2007,103(4):1055-1065.
[16]Narihiro T,Terada T,Kikuchi K,et al.Comparative analysis of bacterial and archaeal communities in methanogenic sludge granules from upflow anaerobic sludge blanket reactors treating various food-processing,high-strength organic wastewaters[J].Microbes and Environments,2008,24(2):88-96.
[17]Roest K,Heilig H G H J,Smidt H,et al.Community analysis of a fullscale anaerobic bioreactor treating paper mill wastewater[J].Systematic and Applied Microbiology,2005,28(2):175-185.
[18]Lai Q,Shao Z.Pseudomonas xiamenensis sp.nov.,a denitrifying bacterium isolated from activated sludge[J].International Journal of Systematic and Evolutionary Microbiology,2008,58(8):1911-1915.
[19]Klocke M,Mähnert P,Mundt K,et al.Microbial community analysis of a biogas-producing completely stirred tank reactor fed continuously with fodder beet silage as mono-substrate[J].Systematic and Applied Microbiology,2007,30(2):139-151.
[20]张 蕾,梁军锋,崔文文,等.规模化秸秆沼气发酵反应器中微生物群落特征[J].农业环境科学学报,2014,33(3):584-592.
[21]袁月祥,文昊深,黄显波,等.玉米秸秆产生物燃气及其微生物群落解析[J].化工学报,2014,65(5):1784-1791.
[22]赵 光,马 放,孙 婷,等.基于高通量测序的寒地沼气池微生物群落解析[J].哈尔滨工业大学学报,2014,46(4):36-42.
[23]郎会花,杨洪江,张永刚.鸡粪沼气池产甲烷菌多样性[J].微生物学通报,2010,37(4):508-512.
[24]Weiss A,Jérôme V,Freitag R.Diversity of the resident microbiota in a thermophilic municipal biogas plant[J].Applied Microbiology and Biotechnology,2008,81(1):163-173.
[25]Tajima K,Nagamine T,Matsui H,et al.Phylogenetic analysis of archaeal 16S rRNA libraries from the rumen suggests the existence of a novel group of archaea not associated with known methanogens[J].FEMS microbiology letters,2001,200(1):67-72.
[26]Savant D V,Shouche Y S,Prakash S,et al.Methanobrevibacter acididurans sp.nov.,a novel methanogen from a sour anaerobic digester[J].International Journal of Systematic and Evolutionary Microbiology,2002,52(4):1081-1087.
[27]Strapoc D,Picardal F W,Turich C,et al.Methane-producing microbial community in a coal bed of the Illinois Basin[J].Applied and Environmental Microbiology,2008,74(8):2424-2432.
[28]彭发基.高寒地区新型辅助燃烧式沼气池建造技术[J].现代农业科技,2009,13:260-261.
[29]崔卫芳,张婷婷,席建超,等.太阳能辅助沼气发酵装置设计与试验[J].资源科学,2012,34(11):2034-2038.
[30]姚 利,王艳芹,袁长波,等.高效沼气微生物菌剂的冬季产气试验[J].山东农业科学,2010,8:57-60.
[31]黄江丽,张国华,丁建南,等.低温沼气发酵促进剂的研究[J].江西科学,2012,30(1):39-43.
Microbial community of rural household biogas digesters in Qinghai by PCR-DGGE.
HAN Rui1,2,3,CHEN Lai-sheng2,3,LI Li3,LIU De-li1*(1.Hubei Key Laboratory of Genetic Regulation and Integrative Biology,School of Life Science,Central China Normal University,Wuhan 430079,China;2.State Key Laboratory Breeding Base,Key Laboratory of Qinghai Province for Plateau Crop Germplasm Innovation and Utilization,Xining 810016,China;3.Qinghai Key Laboratory of Vegetable Genetics and Physiology,Qinghai Academy of Agriculture and Forestry Science,Xining 810016,China).China Environmental Science,2015,35(6):1794~1804
The microbial community composition in rural household biogas digesters of Qinghai was investigated with polymerase chain reaction-denaturing gradient gel electrophoresis(PCR-DGGE)technology.The results showed that the diversity of bacteria and archaea were rich,and the shift of microbial community was obvious in digesters.The bacterial communities in four biogas digesters were classified into five phyla,Bacteroidetes,Firmicutes and Proteobacteria were identified to be dominant.At genome level,belonging to twenty-four communities,Petrimonas,Pseudomona,Tissierella and Clostridium were the abundant taxa.Meanwhile,the species of archaeal community were calssified as Methanobrevibacter,Methanogenium,Methanoculleus and Thermofilum,and the first two classes were dominant in the digesters.Notably,the methane produced by hydrogen nutrition methanogens,and the dominant fermentative bacteria during the hydrolysis and acidogenesis were detected from animal digestive system.
PCR-DGGE;Qinghai;household biogas digesters;microbial community structure
X172
A
1000-6923(2015)06-1794-11
韩 睿(1983-),女,河南许昌人,助理研究员,华中师范大学博士研究生,从事微生物分子生物学方面的研究.发表论文10余篇.
2014-11-20
青海省蔬菜遗传与生理重点实验室(2014-Z-Y15)
* 责任作者,教授,deliliu2013@163.com
